Atracamento molecular (Docking)
|
|
|
- Jonathan Ramires de Caminha
- 7 Há anos
- Visualizações:
Transcrição
1 Atracamento molecular (Docking)
2 O atracamento molecular O que é? Método para achar o melhor encaixe entre duas moléculas
3 O atracamento molecular O que é? Porquê? Método para achar o melhor encaixe entre duas moléculas Se assume que duas moléculas que encaixam bem devem ligar bem entre elas.
4 O atracamento molecular O que é? Porquê? Como? Método para achar o melhor encaixe entre dois moléculas Se assume que duas moléculas que encaixam bem devem ligar bem entre elas. Modificação das conformações/orientações delas para maximizar a interação. Minimização energética (a melhor modo de união deve ter a menor energia).
5 O atracamento molecular Objetivos 1. Determinar se o ligante pode-se unir com a proteína 2. Estimar a geometria do complexo 3. Estimar a energia de ligação Ferramenta s 1. Busca do modo de união ótima: atracamento rígido ou flexível 2. Uma função de avaliação (scoring function) para estimar a energia de união
6 O atracamento molecular Tipos 1. Atracamento Ligante-Proteína 2. Atracamento Proteína-Proteína 3. Atracamento Proteína-DNA
7 1. Identificação de sítios de ligação????
8 1. Identificação de sítios de ligação 1. Estrutura conhecida de complexos proteína/ligante 2. Conservação de resíduos entre espécies 3. Motivos estruturais típicos no sitio ativo de uma família de proteínas 4. Partes flexíveis das proteínas 5. Pontos quentes (Hot spots): A mutação deles por alanina causa um aumento significativo da energia de união 6. Dados de NMR como os desplazamentos químicos ou acoplamentos dipolares 7. Existência de grupos que podem formar enlaces de hidrogênio, de fissuras nas enzimas ou de grupos carregados na superfície da proteína 8. Ferramentas computacionais: predizem sitios de união baseados em distintas propriedades como áreas de superfície enterradas, desolvatação, energias de interação eletrostática, hidrofobicidade, conservação de resíduos
9 Prática 1 Vamos identificar cavidades e possíveis sitos de união de ligantes com o programa CASTp calculation Coloca um número de identificação do PDB ou sobe o PDB do HIV que enviei por . Olha os possíveis sitos de união achados.
10 Prática 1
11 Prática 1
12 2. Caracterização do sitio de união
13 2. Caracterização do sitio de união Duas formas principais de representação 1 Representação molecular: geométricos de forma que representam a. Descritores superfície molecular (+ propriedades físico-químicas) Superfície de van der Waals: é a superfície de união das superfícies atômicas esféricas definidas pelos raios de Van der Waals. Superfície acessível ao solvente e Connolly surface : Todos os pontos da superfície de Van der Waals que o solvente pode tocar.
14 2. Caracterização do sito de união Duas formas principais de representação 2. Representação em grades: armazena a informação acerca das contribuições energéticas das moléculas em pontos da grade que serão necessárias durante a avaliação do atracamento Os pontos da grade armazenam principalmente dois tipos de potenciais: eletrostático e de Lennard Jones (van der Waals + repulsão interatômica) +
15 Prática 2 No programa Chimera, abre o pdb da HIV_protease que enviei por Actions/Surface/Show Tools/Surface-Binding analysis/electros tacic potential Brinca com o resto de opções
16 Prática 2
17 3. Busca do modo de união Ligante rígido Receptor rígido (Atracamento rígido, geométrico ou por encaixe de superfícies) Ligante flexível Receptor rígido (Atracamento flexível) Ligante flexível Receptor flexível
18 3. Busca do modo de união Ligante rígido Receptor rígido (Atracamento rígido, geométrico Baseadoou na complementaridade entre ligante e receptor (chave / por encaixeestérica de superfícies) fechadura). Inclusão de propriedades físico-químicas. Objetivo: Achar a transformação (rotação + translação) que maximiza o número de pontos superficiais que encaixam entre ligante e proteína. Ligante Ligante Proteína Encaixe estérico Proteína Propriedades físico-químicas
19 3. Busca do modo de união Ligante fexível Receptor rígido (Atracamento flexível) Atracamento fragmentado Atracamento combinatorial
20 3. Busca do modo de união Ligante fexível Receptor rígido (Atracamento flexível) Atracamento fragmentado Ex. FlexX and Dock Dividir ligante em fragmentos Determinar o fragmento rígido central Adicionar fragmentos incrementamente Começar pelo fragmento rígido central
21 3. Busca do modo de união Ligante fexível Receptor flexível Atracamento combinatorial Atracamento Suave Métodos de amostragem
22 3. Busca do modo de união Ligante fexível Receptor flexível Atracamento combinatorial Atracamento Suave Métodos de amostragem
23 Métodos de amostragem Usados em: Busca conformacional de ligantes Busca conformacional de proteínas Busca conformacional de complexos São: Monte Carlo Dinâmica molecular Simmulated annealing Algoritmos genéticos Busca tabú Algoritmos genéticos Lamarkian
24 Métodos de amostragem Método de Monte Carlo (MC) Baseado em repetições de busca aleatória Passos: 1. Movimentos aleatórios no espaço conformacional (graus de liberdade rotacional, translacional e torsional) 2. Cálculo da energia da nova conformação 3. Aplicação do critério de aceitação. Critério Metrópolis: ΔE 0, Modo de união aceito ΔE 0, aplicação de um critério de probabilidade (P= ΔE/kT ) Combinado annealing com minimização e simmulated
25 Métodos de amostragem Dinâmica Molecular (MD) Baseado nas leis da mecânica molecular (Newton) No inicio é aplicada uma força para obrigar os átomos a se mexer, mas depois os átomos se mexem unicamente por as forças aplicadas neles por o resto do sistema As novas forças que moverão os átomos são calculadas a cada At. Vantagens É possível obter informação temporal dos movimentos Boa calculando mínimo local de energia Desvantagens Precisa um grande custo computacional Não é boa atravessando barreiras energéticas
26 Métodos de amostragem Simulated Annealing Baseado na aplicação rápida de altas temperaturas, e esfriamento lento do sistema Gera um movimento aleatório a partir de uma conformação inicial e calcula a nova energia ΔE 0, Pose aceita ΔE 0, aplicação de um critério de probabilidade (P= ΔE/kT ) Precisa aplicação de restrições Vantagens: Bom estudo do espaço conformacional Desvantagens: Não chega bem até o mínimo: minimização posterior
27 Métodos de amostragem Algoritmos genéticos Baseado no conceito de evolução Cromossoma = conformação, Gene = variável ou descritor (ângulo torção, distancia enlace,..), Fenótipo = energia Conformações de menor energia tem mais probabilidade de ser escolhidas para próximos cruzamentos
28 Métodos de amostragem Algoritmos genéticos Lamarkian Mesmo principio do algoritmo genético Diferencia com GA: as conformações (cromossomas) são minimizadas antes de fazer um novo cruzamento Mais rápida que SA e GA Busca tabú Busca aleatória. As novas soluções (conformações) são eliminadas se parecem com as anteriores, a menos que a energia delas seja menor que a melhor solução. Objetivo: Evitar ficar atracado no mínimo local.
29 3. Busca do modo de união Que método usar? Quando incluir flexibilidade??
30 4. A solvatação As moléculas na natureza estão presentes em meio aquoso
31 4. A solvatação Duas formas de tratar o problema da solvatação: Modelo explícito: A molécula que queremos simular é rodeada de uma capa de moléculas de água Modelo implícito: Representação do solvente como um meio contínuo, mediante a introdução de um término para a constante dielétrica na função de avaliação
32 4. A solvatação Exemplo (tratamento explícito): Molécula de água mediando a interação inibidor - HIV protease
33 5. Estimação da energia de união A energia de um sistema é calculada pela energia livre de Gibbs: Δ G = ΔH TΔ S G = Energia livre de Gibbs H = Entalpia S = Entropia Cálculo precisa alto custo computacional (Dinâmica Molecular) Inviável na busca do modo de união Funções de avaliação (estimação da energia de união)
34 5. Estimação da energia de união Funções de avaliação usadas com complexos liganteproteína: 1. Funções Baseadas em campo de forca (olhar tema 5, busca conformacional de ligantes) 2. Funções Baseadas em conhecimento (olhar tema 5, busca conformacional de ligantes) 3. Funções Empíricas Energias de ligação aproximadas pelo somatório de diferentes variáveis não correlacionadas (falso) Coeficientes obtidos por análises de regressão usando dados experimentais
35 6. Pós-processamento Inspeção visual Filtros Agrupamento (clusters) Cálculos energéticos mais precisos das soluções selecionadas
36 Programas
37 Programas LINUX FREE
38 Programas Free Linux and Window s
39 Programas linux, windows FREE
40 Programas LINUX FREE
41 Programas window s Require s pay license
42 Programas Windo ws, linux Require s pay license Não dá pra testar com seu complexo
43 Programas Windo ws, linux Require s pay license
44 Programas Windo ws, linux Require s pay license
45 Programas Windo ws, linux Require s pay license
46 Prática 3 Vamos fazer um docking com o programa SwissDock Entrar em SwissDock Clicar submit docking Subir o arquivo da proteína alvo HIVprotease.pdb que enviei por , esperar que o programa o aceite. Subir o arquivo do ligante
47 Prática 3
48 Prática 3
49 Prática 3
50 Atracamento proteínaproteína
51 Passos 1. Obtenção de informação sobre o sito ativo Superfícies, não cavidades!!!
52 Passos 1. Obtenção de informação sobre o sito ativo 2. Representação proteínas da superfície molecular das
53 Passos 1. Obtenção de informação sobre o sito ativo 2. Representação da superfície proteínas 3. Atracamento molecular rígido Porquê rígido??? molecular das
54 Passos 1. Obtenção de informação sobre o sito ativo 2. Representação da superfície proteínas 3. Atracamento molecular rígido molecular das Porquê rígido??? Atracamento de superfícies, se lembra?!!
55 Passos 1. Obtenção de informação sobre o sito ativo 2. Representação da superfície proteínas 3. Atracamento molecular rígido molecular das 4. Avaliação dos complexos formados e seleção do/s melhor/es (funções de avaliação e informação experimental)
56 Passos 1. Obtenção de informação sobre o sito ativo 2. Representação da superfície proteínas 3. Atracamento molecular rígido molecular das 4. Avaliação dos complexos formados e seleção do/s melhor/es (funções de avaliação e informação experimental) 5. Tratamento da flexibilidade
57 Passos 1. Obtenção de informação sobre o sito ativo 2. Representação da superfície proteínas 3. Atracamento molecular rígido molecular das 4. Avaliação dos complexos formados e seleção do/s melhor/es (funções de avaliação e informação experimental) 5. Tratamento da flexibilidade 6. Avaliação dos complexos formados e seleção do/s melhor/es (funções de avaliação e informação experimental)
58 Passos 1. Obtenção de informação sobre o sito ativo 2. Representação da superfície proteínas 3. Atracamento molecular rígido molecular das 4. Avaliação dos complexos formados e seleção do/s melhor/es (funções de avaliação e informação experimental) 5. Tratamento da flexibilidade 6. Avaliação dos complexos formados e seleção do/s melhor/es (funções de avaliação e informação experimental)
59 Tratamento da flexibilidade Atracamento suave (soft docking) Baseado na redução da alta penalidade de energia aplicada quando um átomo de uma proteína sobrepor um átomo da outra
60 Tratamento da flexibilidade Atracamento combinatorial Geração de diferentes conformações das proteínas para usar em atracamento rígido
61 Tratamento da flexibilidade Movimentos das cadeias laterais 1. Bibliotecas de rotâmeros Conformações das cadeias laterais são pegas de bibliotecas de rotâmeros derivadas de estruturas resolvidas de proteínas 2. Estudo do espaço conformacional: Monte Carlo (MC), Algoritmo genético (GA),... Todas as cadeias laterais são estudadas???
62 Tratamento da flexibilidade Movimentos das cadeias laterais Seleção das cadeias laterais a estudar: 1. Perto do sito de união 2. SOFTSPOTS: usa uma função baseada em conhecimento para determinar os resíduos que mais provavelmente vão sofrer mudança conformacional no atracamento. 3. Flexibilidade dos aminoácidos: lisina < arginina < metionina < glutamina < ácido glutâmico < isoleucin < leucina < asparagina < treonina < tirosina < serina < histidina < ácid aspártico < cisteína < triptófano < fenilalanina
63 Tratamento da flexibilidade Separação partes rígidas e flexíveis Todas as partes da proteína são estudadas??? Hinge-Find: analisa estruturas de dois proteínas homólogas em distintas conformações e identifica as partes rígidas.
64 Tratamento da flexibilidade Atracamento fragmentado Algoritmo FlexXDock: 1. Predição de regiões flexíveis e rígidas (HingeProt) 2. Partição da proteína por as dobradiças (loops) 3. Atracamento molecular simultânea e separadamente. de cada fragmento 4. Avaliação das distintas poses de cada fragmento. 5. Construção da proteína usando a pose melhor avaliada para cada fragmento
65 Tratamento da flexibilidade Análise de modos normais Perto do mínimo, a função de energia potencial pode ser aproximada como uma função harmônica (suma de modos normais) Função harmônica Função de Energia potencial Modo normal: oscilações harmônicas no redor do mínimo de energia Movimentos de grande amplitude correspondem a modos normais de baixa freqüência Grandes movimentos Baixa freqüência Cadeias laterais Proteín a Pequenos movimentos Alta freqüência
66 Tratamento da flexibilidade Análise de modos normais Podemos selecionar somente os modos de baixa frequência (grande amplitude) Diminuição de grados de liberdade Permitem representar a flexibilidade de toda a proteína sem grande custo computacional Possibilidade de estudar grandes movimentos
67 Tratamento da flexibilidade Dinâmica essencial Neste caso os modos normais de maior amplitude são obtidos a partir de um grupo de conformações da mesma molécula
68 Tratamento da flexibilidade Métodos de amostragem Monte Carlo, Dinâmica molecular, simulated annealing,... Maior custo computacional: Usados para gerar diferentes conformacoes Ou para um último estudo quando um/s poucos complexos finais foram selecionados.
69 Capri Critical Assesment Prediction of Interactions Experimento internacional desenhado para avaliar as aproximações computacionais disponíveis para o estudo do atracamento molecular proteína-proteína. Criada na conferencia de modelagem de interações de proteínas em genomas, em 2001 Processo: Quando os pesquisadores resolvem a estrutura de um complexo, enviam as estruturas das proteínas não ligadas para CAPRI. Eles tem umas poucas semanas para predizer a estrutura do complexo com as ferramentas computacionais disponíveis. Depois a estrutura experimental do complexo é publicada e comparada com os modelos de CAPRI.
70 Programas
71 Programas
72 Atracamento proteína-dna
73 Interfase Proteína DNA 1. Superfície de ligação rica em resíduos positivos e polares (R,K,T,S). 2. Abundancia de enlaces de hidrogênio, sobre todo com o esqueleto de DNA (inespecíficos) 3. Interações hidrofóbicas também importantes 4. Não existe preferência por um elemento de estrutura secundaria protéico 5. Grande quantidade de moléculas de água na interfase Padrão conservado de hidratação de DNA (regiões ricas em A-T) Estabilizacao DNA, e possível envolvimento na identificação da região de ligação 6. Preferências Arg-G, Asn-A e Lys-G 7. Flexibilidade intrínseca do DNA
74 Informação experimental 1. Mutação de resíduos na proteína: informação sobre envolvimento da ligação 2. Interferência por etilação do esqueleto de DNA: informação sobre envolvimento da ligação 3. Marcação das purinas com dimetilsulfato: informação sobre envolvimento da ligação 4. Mutação das proteínas a nucleasas com sito de reconhecimento especifico: informação sobre proximidade 5. Métodos DNA footprint (DNAase): informação sobre região de ligação no DNA 6. Espectrometria de massas ou NMR para o intercambio hidrogênio deutério: informação sobre acessibilidade de resíduos. El DNA no intercambia? 7. Informação de sitos de união e outros aspectos do reconhecimento DNA-proteína em bancos de dados (PFAM, PROSITE, SMART)
75 Passos 1. Preparação das estruturas de partida: proteína e DNA
76 Passos 1. Preparação das estruturas de partida: proteína e DNA Obtenção por métodos experimentais: Difícil Não ajuda muito a estrutura dela não-ligada Geração computacional: Geralmente usando blocos de nucleotídeos com parâmetros derivados de estudos de difração Exemplos de programas: NAMOT, NAB, 3DNA, MDDNA, DNAtools, CURVES/JUMNA. Grupos de conformações: Usando dados de dobradura obtidos por métodos bioquímicos: NAMOT e NAB. Usando estruturas de DNA em complexos de proteínas relacionadas. Usando simulações MD + informação sobre a dobratura.
77 Passos 1. Preparação das estruturas de partida: proteína e DNA 2. Estudo de atracamento rígido Igual que proteína-ligante, e proteínaproteína!!!
78 Passos 1. Preparação das estruturas de partida: proteína e DNA 2. Estudo de atracamento rígido 3. Introdução da flexibilidade
79 Passos 1. Preparação das estruturas de partida: proteína e DNA 2. Estudo de atracamento rígido 3. Introdução da flexibilidade Atracamento suave (Soft Docking) Atracamento de grupos de conformações Análise de modos normais Métodos de amostragem Bibliotecas de rotámeros (cadeias laterais)
80 Passos 1. Preparação das estruturas de partida: proteína e DNA 2. Estudo de atracamento rígido 3. Introdução da flexibilidade 4. Funções de avaliação
81 Passos 1. Preparação das estruturas de partida: proteína e DNA 2. Estudo de atracamento rígido 3. Introdução da flexibilidade 4. Funções de avaliação Conceitos específicos para a avaliação do atracamento proteína-dna Termo extra para favorecer soluções com maior número de enlaces de hidrogênio Usa complementaridade eletrostática para favorecer seleção de sitos polares na proteína Necessidade de uso de informação experimental para eliminar falsos positivos
82 Programas
Docking (ou atracamento) Molecular. Profa. Dra. Rafaela Ferreira Dept. de Bioquímica e Imunologia 9 de outubro de 2014
 Docking (ou atracamento) Molecular Profa. Dra. Rafaela Ferreira Dept. de Bioquímica e Imunologia rafaelasf@gmail.com 9 de outubro de 2014 O ATRACAMENTO MOLECULAR PERMITE ESTIMAR COMO DUAS MOLÉCULAS INTERAGEM
Docking (ou atracamento) Molecular Profa. Dra. Rafaela Ferreira Dept. de Bioquímica e Imunologia rafaelasf@gmail.com 9 de outubro de 2014 O ATRACAMENTO MOLECULAR PERMITE ESTIMAR COMO DUAS MOLÉCULAS INTERAGEM
Estrutura e Função de Proteínas O NMR tem a vantagem de puder apresentar mais do que uma estrura possivel
 Vamos trabalhar com a neurominidase. A m-glutinina liga o virs à celula. A neurominidasez o contrario e permite que o virus de despegue dessa celula e consegue ir infetar mais virus. O centro ativo mantem
Vamos trabalhar com a neurominidase. A m-glutinina liga o virs à celula. A neurominidasez o contrario e permite que o virus de despegue dessa celula e consegue ir infetar mais virus. O centro ativo mantem
Interações de van der Waals. Interações de van der Waals. Interações de van der Waals.
 Interações de van der Waals aandrico@if.sc.usp.br Também conhecidas como forças de dispersão de London, caracterizam-se pelo momento de dipolo induzido e ocorrem entre todos os átomos. Apesar de moléculas
Interações de van der Waals aandrico@if.sc.usp.br Também conhecidas como forças de dispersão de London, caracterizam-se pelo momento de dipolo induzido e ocorrem entre todos os átomos. Apesar de moléculas
04/10/2011 FORÇAS DE INTERAÇÃO MOLECULAR ENTRE LIGANTES E PROTEÍNAS. O que é um medicamento?
 FORÇAS DE INTERAÇÃO MOLECULAR ENTRE LIGANTES E PROTEÍNAS O que é um medicamento? 1 O que é um medicamento? Medicamento (produto final) Excipientes + Principio ativo ( fármaco, droga ) Como atua uma droga?
FORÇAS DE INTERAÇÃO MOLECULAR ENTRE LIGANTES E PROTEÍNAS O que é um medicamento? 1 O que é um medicamento? Medicamento (produto final) Excipientes + Principio ativo ( fármaco, droga ) Como atua uma droga?
Mecanismo de Inibição de Enzimas Preparação de Micro e Macromoléculas Docagem Molecular Exemplo de Aplicação
 bjetivos FFI0776 Modelagem e Engenharia de Proteínas Mecanismo de Inibição de Enzimas Preparação de Micro e Macromoléculas M l Docagem Molecular Exemplo de Aplicação Prof. Rafael V. C. Guido rvcguido@ifsc.usp.br
bjetivos FFI0776 Modelagem e Engenharia de Proteínas Mecanismo de Inibição de Enzimas Preparação de Micro e Macromoléculas M l Docagem Molecular Exemplo de Aplicação Prof. Rafael V. C. Guido rvcguido@ifsc.usp.br
Rafael Mesquita. Aminoácidos
 Aminoácidos As Proteínas são polímeros de Aminoácidos Os Aminoácidos apresentam pelo menos um grupo carboxílico e um grupo amino Aminoácidos têm como fórmula geral COOH + H 3 N - C - H R Aminoácidos constituintes
Aminoácidos As Proteínas são polímeros de Aminoácidos Os Aminoácidos apresentam pelo menos um grupo carboxílico e um grupo amino Aminoácidos têm como fórmula geral COOH + H 3 N - C - H R Aminoácidos constituintes
Aula 1. Referência. Bancos de Dados. Linguagem x Informação. Introdução a Bioquímica: Biomoléculas. Introdução ao Curso: Aminoácidos.
 Introdução a Bioquímica: Biomoléculas Aula 1 Introdução ao urso: Aminoácidos eferência Autores: Ignez aracelli e Julio Zukerman-Schpector Editora: EdUFSar Ignez aracelli BioMat DF UNESP/Bauru Julio Zukerman
Introdução a Bioquímica: Biomoléculas Aula 1 Introdução ao urso: Aminoácidos eferência Autores: Ignez aracelli e Julio Zukerman-Schpector Editora: EdUFSar Ignez aracelli BioMat DF UNESP/Bauru Julio Zukerman
Bioinformática Estrutural Aula 1
 Bioinformática Estrutural Aula 1 03 de Junho de 2013 Paula Kuser-Falcão Laboratório de Bioinformática Aplicada Embrapa Informática Agropecuária Paula.kuser-falcao@embrapa.br Pratique Atividade Física Paula
Bioinformática Estrutural Aula 1 03 de Junho de 2013 Paula Kuser-Falcão Laboratório de Bioinformática Aplicada Embrapa Informática Agropecuária Paula.kuser-falcao@embrapa.br Pratique Atividade Física Paula
QSAR 3D Planejamento de NCEs QSAR 3D. O processo de reconhecimento molecular é um processo tridimensional. Conformação bioativa.
 QSAR 3D Interação Fármaco-Receptor AULA 5 a maioria dos casos, a ação dos fármacos está ligada a interação fármaco-receptor, receptor, através de uma associação do tipo reversível e não- covalente Agonistas
QSAR 3D Interação Fármaco-Receptor AULA 5 a maioria dos casos, a ação dos fármacos está ligada a interação fármaco-receptor, receptor, através de uma associação do tipo reversível e não- covalente Agonistas
CURSO: ENFERMAGEM DISCIPLINA: BIOQUÍMICA HUMANA PROF. WILLAME BEZERRA. Aminoácidos. Prof. Willame Bezerra
 CURSO: ENFERMAGEM DISCIPLINA: BIOQUÍMICA HUMANA PROF. WILLAME BEZERRA Aminoácidos Prof. Willame Bezerra As proteínas são as biomoléculas mais abundantes nos seres vivos e exercem funções fundamentais em
CURSO: ENFERMAGEM DISCIPLINA: BIOQUÍMICA HUMANA PROF. WILLAME BEZERRA Aminoácidos Prof. Willame Bezerra As proteínas são as biomoléculas mais abundantes nos seres vivos e exercem funções fundamentais em
Aminoácidos. Prof. Dr. Walter F. de Azevedo Jr. Laboratório de Sistemas BioMoleculares. Departamento de Física. UNESP São José do Rio Preto. SP.
 Aminoácidos Prof. Dr. Walter F. de Azevedo Jr. Laboratório de Sistemas BioMoleculares. Departamento de Física. UNESP São José do Rio Preto. SP. Resumo Introdução Quiralidade Ligação peptídica Cadeia peptídica
Aminoácidos Prof. Dr. Walter F. de Azevedo Jr. Laboratório de Sistemas BioMoleculares. Departamento de Física. UNESP São José do Rio Preto. SP. Resumo Introdução Quiralidade Ligação peptídica Cadeia peptídica
2018 Dr. Walter F. de Azevedo Jr. Biofísica. Prof. Dr. Walter F. de Azevedo Jr.
 2018 Dr. Walter F. de Azevedo Jr. Biofísica Prof. Dr. Walter F. de Azevedo Jr. 1 Ao lado temos a estrutura de um peptídeo formado pelos aminoácidos Arginina - Prolina - Alanina - Tirosina - Serina, coloridos
2018 Dr. Walter F. de Azevedo Jr. Biofísica Prof. Dr. Walter F. de Azevedo Jr. 1 Ao lado temos a estrutura de um peptídeo formado pelos aminoácidos Arginina - Prolina - Alanina - Tirosina - Serina, coloridos
Proteínas São macromoléculas complexas, compostas de aminoácidos, e necessárias para os processos químicos que ocorrem nos organismos vivos
 Proteínas São macromoléculas complexas, compostas de aminoácidos, e necessárias para os processos químicos que ocorrem nos organismos vivos São os constituintes básicos da vida: tanto que seu nome deriva
Proteínas São macromoléculas complexas, compostas de aminoácidos, e necessárias para os processos químicos que ocorrem nos organismos vivos São os constituintes básicos da vida: tanto que seu nome deriva
Prof. Marcelo Langer. Curso de Biologia. Aula Genética
 Prof. Marcelo Langer Curso de Biologia Aula Genética CÓDIGO GENÉTICO Uma linguagem de códons e anticódons, sempre constituídos por 3 NUCLEOTÍDEOS. 64 CODONS = 4 tipos diferentes de nucleotídeos, combinação
Prof. Marcelo Langer Curso de Biologia Aula Genética CÓDIGO GENÉTICO Uma linguagem de códons e anticódons, sempre constituídos por 3 NUCLEOTÍDEOS. 64 CODONS = 4 tipos diferentes de nucleotídeos, combinação
ATRACAMENTO MOLECULAR
 UNIVERSIDADE FEDERAL DO ESPÍRITO SANTO CENTRO DE CIÊNCIAS DA SAÚDE PROGRAMA DE PÓS-GRADUAÇÃO EM BIOTECNOLOGIA BIOINFORMÁTICA ATRACAMENTO MOLECULAR VITÓRIA 2018 Ivana Alece Arantes Moreno Júlia de Assis
UNIVERSIDADE FEDERAL DO ESPÍRITO SANTO CENTRO DE CIÊNCIAS DA SAÚDE PROGRAMA DE PÓS-GRADUAÇÃO EM BIOTECNOLOGIA BIOINFORMÁTICA ATRACAMENTO MOLECULAR VITÓRIA 2018 Ivana Alece Arantes Moreno Júlia de Assis
Métodos de Química Computacional
 Universidade Federal de Ouro Preto Instituto de Ciências Exatas e Biológicas Departamento de Química Métodos de Química Computacional Professora: Melissa Soares Caetano Mecânica Molecular Mecânica Molecular
Universidade Federal de Ouro Preto Instituto de Ciências Exatas e Biológicas Departamento de Química Métodos de Química Computacional Professora: Melissa Soares Caetano Mecânica Molecular Mecânica Molecular
Aminoácidos não-essenciais: alanina, ácido aspártico, ácido glutâmico, cisteína, glicina, glutamina, hidroxiprolina, prolina, serina e tirosina.
 AMINOÁCIDOS Os aminoácidos são as unidades fundamentais das PROTEÍNAS. Existem cerca de 300 aminoácidos na natureza, mas nas proteínas podemos encontrar 20 aminoácidos principais Estruturalmente são formados
AMINOÁCIDOS Os aminoácidos são as unidades fundamentais das PROTEÍNAS. Existem cerca de 300 aminoácidos na natureza, mas nas proteínas podemos encontrar 20 aminoácidos principais Estruturalmente são formados
CÓDIGO GENÉTICO E SÍNTESE PROTEICA
 Terceirão Biologia 1 Professor João CÓDIGO GENÉTICO E SÍNTESE PROTEICA 1. Síntese de proteínas pelos ribossomos a partir do RNAm. a) RNAm: molécula de RNA que contem a informação genética necessária para
Terceirão Biologia 1 Professor João CÓDIGO GENÉTICO E SÍNTESE PROTEICA 1. Síntese de proteínas pelos ribossomos a partir do RNAm. a) RNAm: molécula de RNA que contem a informação genética necessária para
Métodos de Química Computacional
 Universidade Federal de Ouro Preto Instituto de Ciências Exatas e Biológicas Departamento de Química Métodos de Química Computacional Professora: Melissa Soares Caetano Dinâmica molecular Conhecendo posições
Universidade Federal de Ouro Preto Instituto de Ciências Exatas e Biológicas Departamento de Química Métodos de Química Computacional Professora: Melissa Soares Caetano Dinâmica molecular Conhecendo posições
Componentes Químicos das Células
 Componentes Químicos das Células Os seres vivos são um sistema Fundamentam-se em: químico! compostos de carbono, ou seja, na química orgânica. Dependem de reações químicas que ocorrem em soluções aquosas,
Componentes Químicos das Células Os seres vivos são um sistema Fundamentam-se em: químico! compostos de carbono, ou seja, na química orgânica. Dependem de reações químicas que ocorrem em soluções aquosas,
1164 BIOLOGIA ESTRUTURAL Aula 1 Prof. Dr. Valmir Fadel
 H C N O S Água Pontes de Hidrogênio -Interação fraca com grupos polares e carregados - orientada (Lehninger, cap. 4) AMINO-ÁCIDOS ISÔMERO L ISÔMERO D 20 amino-ácidos mais comuns Alanina CH3-CH(NH2)-COOH
H C N O S Água Pontes de Hidrogênio -Interação fraca com grupos polares e carregados - orientada (Lehninger, cap. 4) AMINO-ÁCIDOS ISÔMERO L ISÔMERO D 20 amino-ácidos mais comuns Alanina CH3-CH(NH2)-COOH
14/02/2017. Genética. Professora Catarina
 14/02/2017 Genética Professora Catarina 1 A espécie humana Ácidos nucleicos Tipos DNA ácido desoxirribonucleico RNA ácido ribonucleico São formados pela união de nucleotídeos. 2 Composição dos nucleotídeos
14/02/2017 Genética Professora Catarina 1 A espécie humana Ácidos nucleicos Tipos DNA ácido desoxirribonucleico RNA ácido ribonucleico São formados pela união de nucleotídeos. 2 Composição dos nucleotídeos
Biologia Molecular (Parte I)
 Biologia Molecular (Parte I) Introdução 1. Substâncias Inorgânicas 1.1. Água A água faz pontes de hidrogênio Polaridade Propriedades da água Solvente Universal Participa de reações químicas vitais Adesão
Biologia Molecular (Parte I) Introdução 1. Substâncias Inorgânicas 1.1. Água A água faz pontes de hidrogênio Polaridade Propriedades da água Solvente Universal Participa de reações químicas vitais Adesão
Funções Escores (MolDock Score) Prof. Dr. Walter F. de Azevedo Jr. ou
 205 Dr. Walter F. de zevedo Jr. Funções Escores (MolDock Score) Prof. Dr. Walter F. de zevedo Jr. walter.junior@pucrs.br ou walter.filgueira@hotmail.com Função MolDock Score função ajuste ou escore, usada
205 Dr. Walter F. de zevedo Jr. Funções Escores (MolDock Score) Prof. Dr. Walter F. de zevedo Jr. walter.junior@pucrs.br ou walter.filgueira@hotmail.com Função MolDock Score função ajuste ou escore, usada
Soluções de Conjunto de Problemas 1
 Soluções de 7.012 Conjunto de Problemas 1 Questão 1 a) Quais são os quatro tipos principais de moléculas biológicas discutidos na aula? Cite uma função importante de cada tipo de molécula biológica na
Soluções de 7.012 Conjunto de Problemas 1 Questão 1 a) Quais são os quatro tipos principais de moléculas biológicas discutidos na aula? Cite uma função importante de cada tipo de molécula biológica na
RMN em proteínas pequenas
 COSY COrrelated SpectroscopY Experimento 2D homonuclear ( 1 H- 1 H) mais simples Primeiro experimento 2D proposto (Jeener, 1971) Período de mistura: 1 único pulso de 90 Transferência da coerência entre
COSY COrrelated SpectroscopY Experimento 2D homonuclear ( 1 H- 1 H) mais simples Primeiro experimento 2D proposto (Jeener, 1971) Período de mistura: 1 único pulso de 90 Transferência da coerência entre
Rafael Mesquita. Proteínas. Estrutura
 Proteínas Estrutura Ligação peptídica Ligação peptídica - Rígida e plana - Ligação simples com caráter de dupla ligação Ângulos Φ e Ψ Ligação peptídica limitações estruturais Limitações estruturais impostas
Proteínas Estrutura Ligação peptídica Ligação peptídica - Rígida e plana - Ligação simples com caráter de dupla ligação Ângulos Φ e Ψ Ligação peptídica limitações estruturais Limitações estruturais impostas
REVISÃO: ENADE BIOQUÍMICA - 1
 FUNDAÇÃO CARMELITANA MÁRIO PALMÉRIO FACIHUS - FACULDADE DE CIÊNCIAS HUMANAS E SOCIAIS REVISÃO: ENADE BIOQUÍMICA - 1 Prof. Me. Cássio Resende de Morais Propriedades da Água Introdução Substância líquida,
FUNDAÇÃO CARMELITANA MÁRIO PALMÉRIO FACIHUS - FACULDADE DE CIÊNCIAS HUMANAS E SOCIAIS REVISÃO: ENADE BIOQUÍMICA - 1 Prof. Me. Cássio Resende de Morais Propriedades da Água Introdução Substância líquida,
Fundamentos da modelagem Molecular - 1
 Fundamentos da modelagem Molecular - 1 Departamento de Física UFPel ufpellogo Introdução Rotas para a pesquisa Conexão entre experimento, simulação e teoria Sistema real Fazer modelos do sistema Fazer
Fundamentos da modelagem Molecular - 1 Departamento de Física UFPel ufpellogo Introdução Rotas para a pesquisa Conexão entre experimento, simulação e teoria Sistema real Fazer modelos do sistema Fazer
Aminoácidos peptídeos e proteínas
 Pontifícia Universidade Católica de Goiás Departamento de Biologia Aminoácidos peptídeos e proteínas Prof. Macks Wendhell Gonçalves, Msc mackswendhell@gmail.com Algumas funções de proteínas A luz produzida
Pontifícia Universidade Católica de Goiás Departamento de Biologia Aminoácidos peptídeos e proteínas Prof. Macks Wendhell Gonçalves, Msc mackswendhell@gmail.com Algumas funções de proteínas A luz produzida
Aminoácidos e peptídeos. Prof.: Matheus de Souza Gomes Disciplina: Bioquímica I
 Aminoácidos e peptídeos Prof.: Matheus de Souza Gomes Disciplina: Bioquímica I Patos de Minas 2017 Conteúdo Aminoácidos e peptídeos Constituição das proteínas Aminoácidos Estrutura Classificação Ácido
Aminoácidos e peptídeos Prof.: Matheus de Souza Gomes Disciplina: Bioquímica I Patos de Minas 2017 Conteúdo Aminoácidos e peptídeos Constituição das proteínas Aminoácidos Estrutura Classificação Ácido
Bauru, 11 de agosto de 2008.
 Introdução a Bioquímica: Biomoléculas Aula 1 Introdução ao Curso: Aminoácidos Ignez Caracelli BioMat DF UNESP/Bauru Julio Zukerman Schpector LaCrEMM DQ UFSCar Bauru, 11 de agosto de 2008. 1 Avaliação 1seminário
Introdução a Bioquímica: Biomoléculas Aula 1 Introdução ao Curso: Aminoácidos Ignez Caracelli BioMat DF UNESP/Bauru Julio Zukerman Schpector LaCrEMM DQ UFSCar Bauru, 11 de agosto de 2008. 1 Avaliação 1seminário
Introdução. Estrutura dos Aminoácidos e Proteínas. Aminoácidos componentes de proteínas. Aminoácidos componentes de proteínas 10/02/2012.
 Introdução Estrutura dos Aminoácidos e Prof. Dr. Bruno Lazzari de Lima : Componentes celulares mais importantes. Diversidade de forma e função. Estruturais. Enzimáticas. Transportadoras. Ex.: Insulina,
Introdução Estrutura dos Aminoácidos e Prof. Dr. Bruno Lazzari de Lima : Componentes celulares mais importantes. Diversidade de forma e função. Estruturais. Enzimáticas. Transportadoras. Ex.: Insulina,
OBJETIVOS INTERAÇÕES INTERMOLECULARES INTERAÇÕES INTERMOLECULARES.
 OBJETIVOS aandrico@if.sc.usp.br Interações Intermoleculares Mecanismo de Ação Modo de Ligação Complexos Recetor-Ligante Exemplos e Exercícios INTERAÇÕES INTERMOLECULARES INTERAÇÕES INTERMOLECULARES O processo
OBJETIVOS aandrico@if.sc.usp.br Interações Intermoleculares Mecanismo de Ação Modo de Ligação Complexos Recetor-Ligante Exemplos e Exercícios INTERAÇÕES INTERMOLECULARES INTERAÇÕES INTERMOLECULARES O processo
Universidade Salgado de Oliveira Disciplina de Bioquímica Básica Proteínas
 Universidade Salgado de Oliveira Disciplina de Bioquímica Básica Proteínas Profª Larissa dos Santos Introdução As proteínas (ou também conhecidas como polipeptídeos) são as macromoléculas mais abundantes
Universidade Salgado de Oliveira Disciplina de Bioquímica Básica Proteínas Profª Larissa dos Santos Introdução As proteínas (ou também conhecidas como polipeptídeos) são as macromoléculas mais abundantes
Fundamentos da modelagem Molecular - 1
 Fundamentos da modelagem Molecular - 1 Departamento de Física UFPel Introdução Rotas para a pesquisa Conexão entre experimento, simulação e teoria Sistema real Fazer modelos do sistema Fazer experimentos
Fundamentos da modelagem Molecular - 1 Departamento de Física UFPel Introdução Rotas para a pesquisa Conexão entre experimento, simulação e teoria Sistema real Fazer modelos do sistema Fazer experimentos
Profº Lásaro Henrique
 Profº Lásaro Henrique Proteínas são macromoléculas complexas, compostas de aminoácidos. São os constituintes básicos da vida e necessárias para os processos químicos que ocorrem nos organismos vivos. Nos
Profº Lásaro Henrique Proteínas são macromoléculas complexas, compostas de aminoácidos. São os constituintes básicos da vida e necessárias para os processos químicos que ocorrem nos organismos vivos. Nos
Introdução à Modelagem Molecular
 Introdução à Modelagem Molecular Prof. Alex Fabiano C. Campos, Dr Introdução Métodos teóricos e técnicas computacionais para modelar ou imitar o comportamento de moléculas e sistemas atômicos. Empregados
Introdução à Modelagem Molecular Prof. Alex Fabiano C. Campos, Dr Introdução Métodos teóricos e técnicas computacionais para modelar ou imitar o comportamento de moléculas e sistemas atômicos. Empregados
Determinação da Estrutura de Proteínas
 Centro Brasileiro-Argentino de Biotecnologia Introdução à Biologia Computacional Determinação da Estrutura de Proteínas Paulo enrique C. Godoi Bioinformática objetivo principal é determinar a função de
Centro Brasileiro-Argentino de Biotecnologia Introdução à Biologia Computacional Determinação da Estrutura de Proteínas Paulo enrique C. Godoi Bioinformática objetivo principal é determinar a função de
PROTEÍNAS. A palavra proteína derivada de proteos, (grego = primeira ou a mais importante)
 PROTEÍNAS A palavra proteína derivada de proteos, (grego = primeira ou a mais importante) Macromolécula mais abundante nas células Produto final da informação genética (DNA-RNA-Proteína) Diversidade funcional
PROTEÍNAS A palavra proteína derivada de proteos, (grego = primeira ou a mais importante) Macromolécula mais abundante nas células Produto final da informação genética (DNA-RNA-Proteína) Diversidade funcional
Professor Antônio Ruas
 Universidade Estadual do Rio Grande do Sul Curso Superior de Tecnologia em Gestão Ambiental Componente curricular: BIOLOGIA GERAL Aula 4 Professor Antônio Ruas 1. Temas: Macromoléculas celulares Produção
Universidade Estadual do Rio Grande do Sul Curso Superior de Tecnologia em Gestão Ambiental Componente curricular: BIOLOGIA GERAL Aula 4 Professor Antônio Ruas 1. Temas: Macromoléculas celulares Produção
 Proteínas As proteínas são as macromoléculas mais abundantes nas células vivas. Elas são os instrumentos moleculares através dos quais a informação genética é expressa. O nome proteína vem do grego protos
Proteínas As proteínas são as macromoléculas mais abundantes nas células vivas. Elas são os instrumentos moleculares através dos quais a informação genética é expressa. O nome proteína vem do grego protos
ANDERSON ORZARI RIBEIRO
 Transformações Químicas ANDERSON ORZARI RIBEIRO Anderson.ribeiro@ufabc.edu.br Bloco B, 10º andar - Sala 1043 Transformações Químicas AULA 5 Compostos de carbono e interações intermoleculares CAPÍTULO 3
Transformações Químicas ANDERSON ORZARI RIBEIRO Anderson.ribeiro@ufabc.edu.br Bloco B, 10º andar - Sala 1043 Transformações Químicas AULA 5 Compostos de carbono e interações intermoleculares CAPÍTULO 3
BIOQUÍMICA PARA ODONTO
 BIOQUÍMICA PARA ODONTO Aula 3: Autoria: Ligação peptídica Proteínas globulares: estrutura primária, secundária e terciária Luiza Higa Programa de Biologia Estrutural Instituto de Bioquímica Médica Universidade
BIOQUÍMICA PARA ODONTO Aula 3: Autoria: Ligação peptídica Proteínas globulares: estrutura primária, secundária e terciária Luiza Higa Programa de Biologia Estrutural Instituto de Bioquímica Médica Universidade
BC 0307 Transformações Químicas
 BC 0307 Transformações Químicas ANDERSON ORZARI RIBEIRO Anderson.ribeiro@ufabc.edu.br Bloco B, 10º andar - Sala 1043 BC 0307 Transformações Químicas AULA 5 Compostos de carbono e interações intermoleculares
BC 0307 Transformações Químicas ANDERSON ORZARI RIBEIRO Anderson.ribeiro@ufabc.edu.br Bloco B, 10º andar - Sala 1043 BC 0307 Transformações Químicas AULA 5 Compostos de carbono e interações intermoleculares
Aminoácidos e Peptideos
 Aminoácidos e Peptideos O que são aminoácidos? Precursores de vários tipos de biomoléculas Compostos formados por : um grupo amina primário [ ] um grupo ácido carboxílico [ ] ambos ligados a um carbono
Aminoácidos e Peptideos O que são aminoácidos? Precursores de vários tipos de biomoléculas Compostos formados por : um grupo amina primário [ ] um grupo ácido carboxílico [ ] ambos ligados a um carbono
1. INTRODUÇÃO. Polímeros biológicos. Polissacarídeos Proteínas Ácidos Nucléicos. Reservas de energia. Armazenamento e Transmissão de informações
 AMINOÁIDOS 1. INTRODUÇÃO Polímeros biológicos Polissacarídeos Proteínas Ácidos Nucléicos Reservas de energia Materiais estruturais Marcadores bioquímicos Armazenamento e Transmissão de informações PROTEÍNAS
AMINOÁIDOS 1. INTRODUÇÃO Polímeros biológicos Polissacarídeos Proteínas Ácidos Nucléicos Reservas de energia Materiais estruturais Marcadores bioquímicos Armazenamento e Transmissão de informações PROTEÍNAS
Origem grego (protos) primeira, mais importante
 PROTEÍNAS Origem grego (protos) primeira, mais importante A palavra proteína que eu proponho vem derivada de proteos, porque ela parece ser a substância primitiva ou principal da nutrição animal, as plantas
PROTEÍNAS Origem grego (protos) primeira, mais importante A palavra proteína que eu proponho vem derivada de proteos, porque ela parece ser a substância primitiva ou principal da nutrição animal, as plantas
CÓDIGO GENÉTICO E SÍNTESE PROTEICA
 Terceirão Biologia 1 Professor João CÓDIGO GENÉTICO E SÍNTESE PROTEICA Dogma central da Biologia Descreve o fluxo unidirecional de informações, do DNA à síntese de proteínas. Duplicação/Replicação Síntese
Terceirão Biologia 1 Professor João CÓDIGO GENÉTICO E SÍNTESE PROTEICA Dogma central da Biologia Descreve o fluxo unidirecional de informações, do DNA à síntese de proteínas. Duplicação/Replicação Síntese
1. INTRODUÇÃO. 1.1 Motivação
 1. INTRODUÇÃO 1.1 Motivação As proteínas compõem uma das classes de moléculas biológicas mais estudadas atualmente. Isto é devido, entre várias outras razões, às descobertas das últimas décadas que mostram
1. INTRODUÇÃO 1.1 Motivação As proteínas compõem uma das classes de moléculas biológicas mais estudadas atualmente. Isto é devido, entre várias outras razões, às descobertas das últimas décadas que mostram
Professor Antônio Ruas
 Universidade Estadual do Rio Grande do Sul Curso Superior de Tecnologia em Gestão Ambiental Componente curricular: BIOLOGIA GERAL Aula 4 Professor Antônio Ruas 1. Temas: Macromoléculas celulares Produção
Universidade Estadual do Rio Grande do Sul Curso Superior de Tecnologia em Gestão Ambiental Componente curricular: BIOLOGIA GERAL Aula 4 Professor Antônio Ruas 1. Temas: Macromoléculas celulares Produção
Modelagem Comparativa de Proteínas
 Modelagem Comparativa de Proteínas Bioinformática Estrutural Aula 3 3 de Junho de 2013 Paula Kuser Falcão Laboratório de Bioinformática Aplicada Embrapa Informática Agropecuária Por que predizer a estrutura?
Modelagem Comparativa de Proteínas Bioinformática Estrutural Aula 3 3 de Junho de 2013 Paula Kuser Falcão Laboratório de Bioinformática Aplicada Embrapa Informática Agropecuária Por que predizer a estrutura?
Programa de Pós-Graduação em Química
 1/12 Programa de Pós-Graduação em Química PROVA DE CONHECIMENTOS ESPECÍFICOS Duração da Prova: 4 horas São José do Rio Preto, 24 de Janeiro de 2013. Departamento 1 2/12 Questão 1. Os haloalcanos, também
1/12 Programa de Pós-Graduação em Química PROVA DE CONHECIMENTOS ESPECÍFICOS Duração da Prova: 4 horas São José do Rio Preto, 24 de Janeiro de 2013. Departamento 1 2/12 Questão 1. Os haloalcanos, também
AULA 7. Cálculo das energias de interação estereoquímicas e eletrostáticas entre as moléculas do conjunto de
 Análise Comparativa dos Campos Moleculares AULA 7 Análise Comparativa dos Campos Moleculares A contribuição devido às forças de dispersão entre moléculas é descrita pelo potencial de Lennard-Jones e as
Análise Comparativa dos Campos Moleculares AULA 7 Análise Comparativa dos Campos Moleculares A contribuição devido às forças de dispersão entre moléculas é descrita pelo potencial de Lennard-Jones e as
Faculdade Anhanguera Curso de Graduação em Educação Física
 Faculdade Anhanguera Curso de Graduação em Educação Física Profa. Dra. Amabile Vessoni Arias E-mail: Amabile.arias@anhanguera.com 2016-2 Mês de agosto Conteúdo 9 Unidade 1 16 Unidade 1 23 Unidade 1 30
Faculdade Anhanguera Curso de Graduação em Educação Física Profa. Dra. Amabile Vessoni Arias E-mail: Amabile.arias@anhanguera.com 2016-2 Mês de agosto Conteúdo 9 Unidade 1 16 Unidade 1 23 Unidade 1 30
Interacções em moléculas biológicas
 Interacções em moléculas biológicas Interacções intramoleculares Ligações covalentes (manutenção da estrutura covalente das moléculas Interacções não-covalentes (conformções, folding, estrutura secundária)
Interacções em moléculas biológicas Interacções intramoleculares Ligações covalentes (manutenção da estrutura covalente das moléculas Interacções não-covalentes (conformções, folding, estrutura secundária)
COLÉGIO PEDRO II CAMPUS TIJUCA II. DEPARTAMENTO DE BIOLOGIA E CIÊNCIAS COORD.: PROFa. CRISTIANA LIMONGI
 COLÉGIO PEDRO II CAMPUS TIJUCA II DEPARTAMENTO DE BIOLOGIA E CIÊNCIAS COORD.: PROFa. CRISTIANA LIMONGI 1º & 2º TURNOS 3ª SÉRIE / ENSINO MÉDIO REGULAR & INTEGRADO ANO LETIVO 2015 PROFESSORES: FRED & PEDRO
COLÉGIO PEDRO II CAMPUS TIJUCA II DEPARTAMENTO DE BIOLOGIA E CIÊNCIAS COORD.: PROFa. CRISTIANA LIMONGI 1º & 2º TURNOS 3ª SÉRIE / ENSINO MÉDIO REGULAR & INTEGRADO ANO LETIVO 2015 PROFESSORES: FRED & PEDRO
Princípios básicos da modelagem molecular e sua aplicação no desenvolvimento de novos fármacos
 II JRADA DE IVER DA QUÍMICA Princípios básicos da modelagem molecular e sua aplicação no desenvolvimento de novos fármacos Ana Carolina de liveira Introdução MDELAGEM MLECULAR DEFIIÇÃ: Trata da investigação
II JRADA DE IVER DA QUÍMICA Princípios básicos da modelagem molecular e sua aplicação no desenvolvimento de novos fármacos Ana Carolina de liveira Introdução MDELAGEM MLECULAR DEFIIÇÃ: Trata da investigação
BIOQUÍMICA I 1º ano de Medicina Ensino teórico 2010/2011
 BIOQUÍMICA I 1º ano de Medicina Ensino teórico 2010/2011 7ª aula teórica 11 Outubro 2010 Proteínas estruturais e funcionais Organização estrutural das proteínas Estrutura e diferentes funções de proteínas
BIOQUÍMICA I 1º ano de Medicina Ensino teórico 2010/2011 7ª aula teórica 11 Outubro 2010 Proteínas estruturais e funcionais Organização estrutural das proteínas Estrutura e diferentes funções de proteínas
OBJETIVOS AULA 10 DESENVOLVIMENTO DESCOBERTA. ¾ Otimização de Moléculas Bioativas. ¾ Integração com o processo de HTS
 AULA 10 2 DESCOBERTA DESENVOLVIMENTO Pesquisa Básica (Pré-clínica) (Fases Clínicas I IV) Identificação do alvo molecular Triagens clínicas conduzidas em humanos: avaliação da segurança, impacto no estado
AULA 10 2 DESCOBERTA DESENVOLVIMENTO Pesquisa Básica (Pré-clínica) (Fases Clínicas I IV) Identificação do alvo molecular Triagens clínicas conduzidas em humanos: avaliação da segurança, impacto no estado
- Apresentam uma fórmula básica: um átomo central de carbono onde se ligam:
 1 4 Aminoácidos e proteínas a) Aminoácidos - São encontrados polimerizados formando proteínas ou livres - São degradados, originando moléculas intermediárias da síntese de glicose e lipídeos - Alguns são
1 4 Aminoácidos e proteínas a) Aminoácidos - São encontrados polimerizados formando proteínas ou livres - São degradados, originando moléculas intermediárias da síntese de glicose e lipídeos - Alguns são
Aminoácidos. Bioquímica Prof. Dr. Marcelo Soares
 Aminoácidos Aminoácidos Nutrição Século XIX: produtos contendo Nitrogênio eram essenciais para a sobrevivência dos animais G. J. Mulder (1839): termo Proteínas Gr Proteios (Primário) Teoria: Proteínas
Aminoácidos Aminoácidos Nutrição Século XIX: produtos contendo Nitrogênio eram essenciais para a sobrevivência dos animais G. J. Mulder (1839): termo Proteínas Gr Proteios (Primário) Teoria: Proteínas
Aminoácido: um composto que contém tanto um grupo amino como um grupo carboxila
 Aminoácidos e Peptídios 1 Aminoácidos Aminoácido: um composto que contém tanto um grupo amino como um grupo carboxila aaminoácido: têm um grupo carboxila e um grupo amino ligados ao mesmo átomo de carbono
Aminoácidos e Peptídios 1 Aminoácidos Aminoácido: um composto que contém tanto um grupo amino como um grupo carboxila aaminoácido: têm um grupo carboxila e um grupo amino ligados ao mesmo átomo de carbono
Disciplina de Proteômica. Caroline Rizzi Doutoranda em Biotecnologia -UFPel
 Disciplina de Proteômica Caroline Rizzi Doutoranda em Biotecnologia -UFPel Bibliografia REVISÃO QUÍMICA Principais grupos funcionais em proteômica O que é uma ligação de hidrogênio? átomo eletronegativo
Disciplina de Proteômica Caroline Rizzi Doutoranda em Biotecnologia -UFPel Bibliografia REVISÃO QUÍMICA Principais grupos funcionais em proteômica O que é uma ligação de hidrogênio? átomo eletronegativo
Macromolécula mais abundante nas células
 PROTEÍNAS Origem grego (protos) primeira, mais importante A palavra proteína que eu proponho vem derivada de proteos, porque ela parece ser a substância primitiva ou principal da nutrição animal, as plantas
PROTEÍNAS Origem grego (protos) primeira, mais importante A palavra proteína que eu proponho vem derivada de proteos, porque ela parece ser a substância primitiva ou principal da nutrição animal, as plantas
BIOLOGIA EXERCÍCIOS. Anabolismo Nuclear
 Anabolismo Nuclear EXERCÍCIOS 1. mesmo responsável pela decodificação do genoma humano em 2001, o presidente dos EUA, Barack Obama, pediu a seus conselheiros especializados em biotecnologia para analisarem
Anabolismo Nuclear EXERCÍCIOS 1. mesmo responsável pela decodificação do genoma humano em 2001, o presidente dos EUA, Barack Obama, pediu a seus conselheiros especializados em biotecnologia para analisarem
AMINOÁCIDOS. - Os aminoácidos são os elementos constituintes de peptídeos, proteínas; - Precursores de hormônios, neurotransmissores, nucleotídeos;
 1 AMINOÁCIDOS - Os aminoácidos são os elementos constituintes de peptídeos, proteínas; - Precursores de hormônios, neurotransmissores, nucleotídeos; - Ocorrem em grande variedade, sendo descritos cerca
1 AMINOÁCIDOS - Os aminoácidos são os elementos constituintes de peptídeos, proteínas; - Precursores de hormônios, neurotransmissores, nucleotídeos; - Ocorrem em grande variedade, sendo descritos cerca
MACRONUTRIENTES III PROTEÍNAS
 MACRONUTRIENTES III PROTEÍNAS 1 PROTEÍNAS As proteínas são compostos orgânicos de estrutura complexa e massa molecular elevada, elas são sintetizadas pelos organismos vivos através da condensação de um
MACRONUTRIENTES III PROTEÍNAS 1 PROTEÍNAS As proteínas são compostos orgânicos de estrutura complexa e massa molecular elevada, elas são sintetizadas pelos organismos vivos através da condensação de um
INTRODUÇÃO Á BIOQUÍMICA
 INTRODUÇÃO Á BIOQUÍMICA BIOQUÍMICA ENFERMAGEM FIO Faculdades Integradas de Ourinhos. Prof. Esp. Roberto Venerando Fundação Educacional Miguel Mofarrej. FIO robertovenerando@fio.edu.br 1 - Introdução à
INTRODUÇÃO Á BIOQUÍMICA BIOQUÍMICA ENFERMAGEM FIO Faculdades Integradas de Ourinhos. Prof. Esp. Roberto Venerando Fundação Educacional Miguel Mofarrej. FIO robertovenerando@fio.edu.br 1 - Introdução à
BÁSICA EM IMAGENS. Aminoácidos, peptídeos e proteínas
 Universidade Federal de Pelotas Instituto de Química e Geociências Departamento de Bioquímica 04 BÁSICA EM IMAGENS - um guia para a sala de aula Aminoácidos, peptídeos e proteínas Generalidades AAs x Proteínas
Universidade Federal de Pelotas Instituto de Química e Geociências Departamento de Bioquímica 04 BÁSICA EM IMAGENS - um guia para a sala de aula Aminoácidos, peptídeos e proteínas Generalidades AAs x Proteínas
AMINOÁCIDOS.! São biomoléculas que apresentam na sua constituição as funções amina primária e ácido carboxílico NH2 I R - C - C = 0O I I H OH
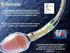 Aminoácidos AMNOÁCDOS! São biomoléculas que apresentam na sua constituição as funções amina primária e ácido carboxílico radical R C-alfa N2 R - C - C = 0O O amina primária ácido carboxílico Aminoácidos
Aminoácidos AMNOÁCDOS! São biomoléculas que apresentam na sua constituição as funções amina primária e ácido carboxílico radical R C-alfa N2 R - C - C = 0O O amina primária ácido carboxílico Aminoácidos
Composição química celular
 Natália Paludetto Composição química celular Proteínas Enzimas Ácidos nucléicos Proteínas Substâncias sólidas; Componente orgânico mais abundante da célula. Podem fornecer energia quando oxidadas, mas
Natália Paludetto Composição química celular Proteínas Enzimas Ácidos nucléicos Proteínas Substâncias sólidas; Componente orgânico mais abundante da célula. Podem fornecer energia quando oxidadas, mas
Mecânica Molecular. Dinâmica Molecular. Exemplos de aplicação. é um método relativamente. energia e conformação de micro ou macromoléculas
 Objetivos FFI0776 Modelagem e Engenharia de Proteínas Prof. Rafael V. C. Guido rvcguido@ifsc.usp.br Aula 04 Mecânica Molecular Campo de força Exemplos de Campo de Força Dinâmica Molecular Etapas envolvidas
Objetivos FFI0776 Modelagem e Engenharia de Proteínas Prof. Rafael V. C. Guido rvcguido@ifsc.usp.br Aula 04 Mecânica Molecular Campo de força Exemplos de Campo de Força Dinâmica Molecular Etapas envolvidas
Aminoácidos FUNDAÇÃO CARMELITANA MÁRIO PALMÉRIO FACIHUS - FACULDADE DE CIÊNCIAS HUMANAS E SOCIAIS
 FUNDAÇÃO CARMELITANA MÁRIO PALMÉRIO FACIHUS - FACULDADE DE CIÊNCIAS HUMANAS E SOCIAIS Aminoácidos Disciplina: Bioquímica I Prof. Me. Cássio Resende de Morais Introdução Proteínas são as macromoléculas
FUNDAÇÃO CARMELITANA MÁRIO PALMÉRIO FACIHUS - FACULDADE DE CIÊNCIAS HUMANAS E SOCIAIS Aminoácidos Disciplina: Bioquímica I Prof. Me. Cássio Resende de Morais Introdução Proteínas são as macromoléculas
ATIVIDADE - 3 ARTIGO: Proteínas: Hidrólise, Precipitação e um Tema para o Ensino de Química RESUMO: Questão 1:
 ATIVIDADE - 3 ARTIGO: FRANCISCO JUNIOR, W. E.; FRANCISCO, W. Proteínas: Hidrólise, Precipitação e um Tema para o Ensino de Química. 2006. Disponível em: .
ATIVIDADE - 3 ARTIGO: FRANCISCO JUNIOR, W. E.; FRANCISCO, W. Proteínas: Hidrólise, Precipitação e um Tema para o Ensino de Química. 2006. Disponível em: .
Alinhamento de seqüências
 Alinhamento de seqüências Qual a importância do alinhamento de seqüências Permite estabelecer identidades entre sequências Permite a dedução de função de proteínas baseado em similaridade Permite a definição
Alinhamento de seqüências Qual a importância do alinhamento de seqüências Permite estabelecer identidades entre sequências Permite a dedução de função de proteínas baseado em similaridade Permite a definição
Triagem Virtual em Larga Escala. Profa. Dra. Rafaela Ferreira Dept. de Bioquímica e Imunologia 10 de outubro de 2014
 Triagem Virtual em Larga Escala Profa. Dra. Rafaela Ferreira Dept. de Bioquímica e Imunologia rafaelasf@gmail.com 10 de outubro de 2014 1 O que é triagem virtual? Grande conjunto de compostos Estratégia
Triagem Virtual em Larga Escala Profa. Dra. Rafaela Ferreira Dept. de Bioquímica e Imunologia rafaelasf@gmail.com 10 de outubro de 2014 1 O que é triagem virtual? Grande conjunto de compostos Estratégia
13/08/2018. Escala de ph. Escala de ph. Crescimento básico. Crescimento ácido. Neutro. básico
 Escala de ph Crescimento básico Neutro Crescimento ácido Escala de ph básico 1 Sistema tampão Um par conjugado ácido-base tende a resistir a alteração de ph, quando pequenas quantidades de ácido ou base
Escala de ph Crescimento básico Neutro Crescimento ácido Escala de ph básico 1 Sistema tampão Um par conjugado ácido-base tende a resistir a alteração de ph, quando pequenas quantidades de ácido ou base
Tipos de Dados Biológicos e Multimídia
 Tipos de Dados Biológicos e Multimídia Arthur Emanuel de O. Carosia Felipe Alves da Louza Luana Peixoto Annibal 1 Dados Biológicos São dados ou medidas coletadas a partir de fontes biológicas São geralmente
Tipos de Dados Biológicos e Multimídia Arthur Emanuel de O. Carosia Felipe Alves da Louza Luana Peixoto Annibal 1 Dados Biológicos São dados ou medidas coletadas a partir de fontes biológicas São geralmente
Gabriela Sehnem Heck 1,2, Prof. Dr. Walter F. de Azevedo Jr 2. (orientador)
 Pontifícia Universidade Católica do Rio Grande do Sul Gabriela Sehnem Heck 1,2, Prof. Dr. Walter F. de Azevedo Jr 2. (orientador) 1 Acadêmica de Curso de Ciêncas Biológicas da PUCRS, 2 Laboratório de Bioquímica
Pontifícia Universidade Católica do Rio Grande do Sul Gabriela Sehnem Heck 1,2, Prof. Dr. Walter F. de Azevedo Jr 2. (orientador) 1 Acadêmica de Curso de Ciêncas Biológicas da PUCRS, 2 Laboratório de Bioquímica
BIOQUÍMICA. Profº André Montillo
 BIOQUÍMICA Profº André Montillo www.montillo.com.br Definição: É uma Molécula Orgânica que contém simultaneamente grupo funcionais amina (NH2) e carboxílico (COOH) É formado pelos seguintes Átomos: o Carbono
BIOQUÍMICA Profº André Montillo www.montillo.com.br Definição: É uma Molécula Orgânica que contém simultaneamente grupo funcionais amina (NH2) e carboxílico (COOH) É formado pelos seguintes Átomos: o Carbono
Aminoácidos, Péptidos e Proteínas
 Aminoácidos, Péptidos e Proteínas Proteínas: -São as macromoléculas biológicas mais abundantes, presentes em todas as células. - Ocorrem numa variedade enorme numa mesma célula. - Exibem uma enorme diversidade
Aminoácidos, Péptidos e Proteínas Proteínas: -São as macromoléculas biológicas mais abundantes, presentes em todas as células. - Ocorrem numa variedade enorme numa mesma célula. - Exibem uma enorme diversidade
Aminoácidos e Peptídeos. Profa. Alana Cecília
 Aminoácidos e Peptídeos Profa. Alana Cecília O que são aminoácidos? A estrutura geral dos aminoácidos inclui um grupo amina e um grupo carboxila, ambos ligados ao carbono α (aquele próximo ao grupo carboxila);
Aminoácidos e Peptídeos Profa. Alana Cecília O que são aminoácidos? A estrutura geral dos aminoácidos inclui um grupo amina e um grupo carboxila, ambos ligados ao carbono α (aquele próximo ao grupo carboxila);
Soluções para Conjunto de Problemas 1
 Soluções para 7.012 Conjunto de Problemas 1 Pergunta 1 a) Quais são os quatro principais tipos de moléculas biológicas discutidas na palestra? Cite uma função importante para cada tipo de molécula biológica
Soluções para 7.012 Conjunto de Problemas 1 Pergunta 1 a) Quais são os quatro principais tipos de moléculas biológicas discutidas na palestra? Cite uma função importante para cada tipo de molécula biológica
AMINOÁCIDOS. Dra. Flávia Cristina Goulart. Bioquímica
 AMINOÁCIDOS Dra. Flávia Cristina Goulart Bioquímica Os aminoácidos (aa). O que são? AMINOÁCIDOS São as unidades fundamentais das PROTEÍNAS. São ácidos orgânicos formados por átomos de carbono, hidrogênio,
AMINOÁCIDOS Dra. Flávia Cristina Goulart Bioquímica Os aminoácidos (aa). O que são? AMINOÁCIDOS São as unidades fundamentais das PROTEÍNAS. São ácidos orgânicos formados por átomos de carbono, hidrogênio,
UNIVERSO TERRA SERES VIVOS ORIGEM
 UNIVERSO TERRA SERES VIVOS ORIGEM BIOLOGIA Surgiu da observação, da curiosidade de se compreender a vida e da utilização da natureza em benefício humano Grande salto com Aristóteles Baseada na observação
UNIVERSO TERRA SERES VIVOS ORIGEM BIOLOGIA Surgiu da observação, da curiosidade de se compreender a vida e da utilização da natureza em benefício humano Grande salto com Aristóteles Baseada na observação
FFI0750 Biologia Molecular Estrutural
 Objetivos FFI0750 Biologia Molecular Estrutural Visualização de estruturas de macromoléculas Representação de estruturas de macromoléculas Prof. Rafael V. C. Guido rvcguido@ifsc.usp.br Aula 02 Bacharelado
Objetivos FFI0750 Biologia Molecular Estrutural Visualização de estruturas de macromoléculas Representação de estruturas de macromoléculas Prof. Rafael V. C. Guido rvcguido@ifsc.usp.br Aula 02 Bacharelado
4. Dinâmica Molecular
 54 4. Dinâmica Molecular Neste capitulo será descrita a metodologia de dinâmica molecular utilizada neste trabalho, com inclusão de solvente explícito e controle de temperatura e pressão. 4.1 Simulação
54 4. Dinâmica Molecular Neste capitulo será descrita a metodologia de dinâmica molecular utilizada neste trabalho, com inclusão de solvente explícito e controle de temperatura e pressão. 4.1 Simulação
AMINOÁCIDOS E PROTEÍNAS: ESTRUTURA E FUNÇÕES
 Universidade Estadual Paulista Júlio de Mesquita Filho Faculdade de Odontologia de Araçatuba Departamento de Ciências Básicas AMINOÁCIDOS E PROTEÍNAS: ESTRUTURA E FUNÇÕES Professora Marcelle Danelon Tópicos
Universidade Estadual Paulista Júlio de Mesquita Filho Faculdade de Odontologia de Araçatuba Departamento de Ciências Básicas AMINOÁCIDOS E PROTEÍNAS: ESTRUTURA E FUNÇÕES Professora Marcelle Danelon Tópicos
Aminoácidos (aas) Prof.ª: Suziane Antes Jacobs
 Aminoácidos (aas) Prof.ª: Suziane Antes Jacobs Introdução Pequenas moléculas propriedades únicas Unidades estruturais (UB) das proteínas N- essencial para a manutenção da vida; 20 aminoácidos-padrão -
Aminoácidos (aas) Prof.ª: Suziane Antes Jacobs Introdução Pequenas moléculas propriedades únicas Unidades estruturais (UB) das proteínas N- essencial para a manutenção da vida; 20 aminoácidos-padrão -
MÓDULO 3 BIOLOGIA MOLECULAR
 MÓDULO 3 BIOLOGIA MOLECULAR Aula 1 - Estrutura e Propriedades dos Ácidos Nucleicos Evidências de que o DNA constitui o material genético Experimento de Frederick Griffith (1928) Pneumococcus pneumoniae
MÓDULO 3 BIOLOGIA MOLECULAR Aula 1 - Estrutura e Propriedades dos Ácidos Nucleicos Evidências de que o DNA constitui o material genético Experimento de Frederick Griffith (1928) Pneumococcus pneumoniae
Entendem-se por compostos heterocíclicos, aqueles compostos orgânicos cíclicos estáveis, que contem no seu anel um ou mais átomos diferentes do
 1 2 3 Entendem-se por compostos heterocíclicos, aqueles compostos orgânicos cíclicos estáveis, que contem no seu anel um ou mais átomos diferentes do carbono. 4 5 O prefixo ribo também é aceitável para
1 2 3 Entendem-se por compostos heterocíclicos, aqueles compostos orgânicos cíclicos estáveis, que contem no seu anel um ou mais átomos diferentes do carbono. 4 5 O prefixo ribo também é aceitável para
Aula de Bioquímica I. Tema: Água. Prof. Dr. Júlio César Borges
 Aula de Bioquímica I Tema: Água Prof. Dr. Júlio César Borges Depto. de Química e Física Molecular DQFM Instituto de Química de São Carlos IQSC Universidade de São Paulo USP E-mail: borgesjc@iqsc.usp.br
Aula de Bioquímica I Tema: Água Prof. Dr. Júlio César Borges Depto. de Química e Física Molecular DQFM Instituto de Química de São Carlos IQSC Universidade de São Paulo USP E-mail: borgesjc@iqsc.usp.br
TRADUÇÃO SÍNTESE PROTEICA
 TRADUÇÃO SÍNTESE PROTEICA Formação do Aminoacil-tRNA Durante a formação do aminoacil-trna, o aminoácido é primeiramente ativado, reagindo com o ATP. Após, é transferido do aminoacil-amp para a extremidade
TRADUÇÃO SÍNTESE PROTEICA Formação do Aminoacil-tRNA Durante a formação do aminoacil-trna, o aminoácido é primeiramente ativado, reagindo com o ATP. Após, é transferido do aminoacil-amp para a extremidade
Pontifícia Universidade Católica do Rio Grande do Sul Faculdade de Informática Programa de Pós-Graduação em Ciência da Computação.
 Pontifícia Universidade Católica do Rio Grande do Sul Faculdade de Informática Programa de Pós-Graduação em Ciência da Computação Uma proposta para a predição computacional da estrutura 3D aproximada de
Pontifícia Universidade Católica do Rio Grande do Sul Faculdade de Informática Programa de Pós-Graduação em Ciência da Computação Uma proposta para a predição computacional da estrutura 3D aproximada de
2 Contexto Biológico Genômica
 15 2 Contexto Biológico Neste capítulo abordaremos o contexto biológico para o entendimento deste trabalho. Serão abordados os aspectos gerais da genômica, expostos os processos do sequenciamento genético
15 2 Contexto Biológico Neste capítulo abordaremos o contexto biológico para o entendimento deste trabalho. Serão abordados os aspectos gerais da genômica, expostos os processos do sequenciamento genético
BIOQUÍMICA CELULAR E MOLECULAR DA DEPRESSÃO PROF.DR.PAULO CESAR NAOUM ACADEMIA DE CIÊNCIA E TECNOLOGIA DE SÃO JOSÉ DO RIO PRETO, SP 2014/15
 BIOQUÍMICA CELULAR E MOLECULAR DA DEPRESSÃO PROF.DR.PAULO CESAR NAOUM ACADEMIA DE CIÊNCIA E TECNOLOGIA DE SÃO JOSÉ DO RIO PRETO, SP 2014/15 PARA ENTENDER A BIOQUÍMICA DA DEPRESSÃO É PRECISO SABER QUE ENTRE
BIOQUÍMICA CELULAR E MOLECULAR DA DEPRESSÃO PROF.DR.PAULO CESAR NAOUM ACADEMIA DE CIÊNCIA E TECNOLOGIA DE SÃO JOSÉ DO RIO PRETO, SP 2014/15 PARA ENTENDER A BIOQUÍMICA DA DEPRESSÃO É PRECISO SABER QUE ENTRE
Características AULA 11. Características e Propriedades. drug-like (fármaco-similar) Bioativo-similar
 AULA 11 rganização e gerenciamento de bases de dados padrões Emprego de pré-filtros: otimização das bases de dados (e.g., seleção por tipo de átomo: C,,,, S, F, Cl, Br, P) Características e Propriedades
AULA 11 rganização e gerenciamento de bases de dados padrões Emprego de pré-filtros: otimização das bases de dados (e.g., seleção por tipo de átomo: C,,,, S, F, Cl, Br, P) Características e Propriedades
