Funções Escores (MolDock Score) Prof. Dr. Walter F. de Azevedo Jr. ou
|
|
|
- Luiz Eduardo Salvado Ferretti
- 6 Há anos
- Visualizações:
Transcrição
1 205 Dr. Walter F. de zevedo Jr. Funções Escores (MolDock Score) Prof. Dr. Walter F. de zevedo Jr. ou
2 Função MolDock Score função ajuste ou escore, usada pelo programa Molegro Virtual Docker (Thomsen & Christensen, 2006) é chamada MolDock Score. Esta função é uma aproximação da energia de interação entre o ligante e a proteína alvo. Usamos as coordenadas atômicas do ligante e da proteína, bem como, eventualmente, co-fatores e moléculas de água para calcular a função escore. ssim, a entrada da função do MolDock Score é o sistema biológico em estudo, ou seja, proteína, ligante, co-fatores e água. saída é o valor da função calculada para o sistema biológico. No caso do docking, temos o cálculo da função MolDock Score para cada posição possível do ligante (pose). É a função que é usada como critério de seleção das poses. Sistema biológico MolDock Scoring Function Score Thomsen R., Christensen M.H. MolDock: a new technique for highaccuracy molecular docking. J. Med. Chem., 2006, 49,
3 Função MolDock Score (MVD) baixo temos a seleção da função MolDock Score do programa MVD. Na opção mostrada, temos a seleção de termos de energia do ligante (Internal ES, Internal HB, Sp2-Sp2 torsions e Displaceable Water com Entropy Reward de 0,50). 3
4 Função MolDock Score (equação) função MolDock Score é definida da seguinte forma: E score = E inter + E intra Onde E score é a função MolDock Score, E inter é a energia de interação intermolecular (proteína e ligante) e E intra é a energia intramolecular (ligante). baixo temos a equação da E inter, E inter N M i j E PLP (r ij ) 332 q q i r ij j Onde E PLP é o termo de energia PLP (piecewise linear potential) e o segundo termo leva em conta as interações eletrostáticas intermoleculares. N e M são os números de átomos do ligante e da proteína, respectivamente. Os termos q i e q j são as cargas dos átomo i e j, respectivamente. O termo r ij indica a distância interatômica entre os átomos i e j. São considerados átomos diferentes de hidrogênio na somatória, incluindo co-fatores, se presentes. 4
5 Função MolDock Score (um olhar matemático) função que vimos no slide anterior, usa o símbolo para representar somatória, é uma forma condensada de representarmos somas. Por exemplo, podemos colocar a soma dos 0 primeiros números naturais da seguinte forma: Soma = Ou podemos representar de forma condensada: Soma 0 i i Veja, se expandirmos a somatória acima, temos a soma dos números, como indicado abaixo: Soma 0 i i
6 Função MolDock Score (um olhar matemático) representação na forma de somatória facilita a notação, por exemplo, se queremos representar a soma dos quadrados dos 0 primeiros número naturais, Soma dos quadrados = Na forma condensada temos: Soma dos quadrados 0 i i 2 Expandindo-se a soma temos: 0 Soma dos quadrados i i
7 Função MolDock Score (termo eletrostático) Voltando para a energia intermolecular, mostrada pela equação a seguir: E inter i j E O termo eletrostático, dentro da circunferência vermelha, usa um esquema de atribuição de cargas completas e parciais aos átomos (q i e q j ), conforme mostrado na tabela abaixo. Carga Átomos dos ligantes Átomos da proteína 0,5 N no grupo C(NH 2 ) 2 His (ND e NE2) rg (NH e NH2),0 N nos grupos N(CH 3 ) 2 e (NH 3 ) -0,5 O nos grupos COO, SO 4, PO 2 e PO 2-0,66 O no grupo PO 3 N M PLP (r ij ) qiq 332 r ij j Lys (NZ) sp (OD e OD2) Glu (OE e OE2) -0,33 O no grupo SO 3 -,0 O no grupo SO 2 NH 7
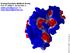
8 Função MolDock Score (termo eletrostático) s figuras abaixo ilustram os aminoácidos que apresentam carga na cadeia lateral. CE NE2 ND CD2 CG CB C C O N Histidina (carga = 0,5) NH2 CD NE CZ NH N CB O CG C C rginina (carga = 0,5) N CD NZ CE CG CB Lisina (carga =,0) C N C O OE2 OE CD CG CB N O OD C CG C CB OD2 spartato (carga = -0,5) C C O Glutamato (carga = -0,5) 8
9 Função MolDock Score (PLP) O termo E PLP usa a mesma função para interações estéricas (van der Waals) e de ligações de hidrogênio. variação está nos parâmetros da função, que está dividida em pedaços, para cada faixa de distância interatômica, indicada por r ij. E PLP = 0 0 R r 2 ij R 0 R R r R ij r 4 ij R R 3 3 para rij R para R r para R r para R r ij R 2 2 ij R3 3 ij R4 para r ij R 4 9
10 Função MolDock Score (PLP) Temos valores de R, R 2, R 3, R 4, e 0 distintos para as interações de van der Waals e ligações de hidrogênio, como mostrado na figura abaixo. R R 2 R 3 R 4 0 E PLP R R 2 R 3 R 4 0
11 Função MolDock Score (PLP) Um par de átomos é considerado como participante de ligação de hidrogênio, se há um átomo doador de hidrogênio, como oxigênio, nitrogênio e enxofre e um outro aceitador. Para o cálculo da função escore PLP, são usados os critérios indicados na tabela abaixo. implementação da PLP para o cálculo da energia da interação de ligação de hidrogênio é dependente da distância interatômica. Tal situação leva a uma cálculo rápido da interação, mas não é realista do ponto de vista físico, visto que a ligação de hidrogênio depende da orientação relativa dos átomos. Tipo de participante da ligação de H ceitador Doador mbos polar Átomo N e O sem H ligados N e S com um ou mais Hs ligados O com um H ligado ou O em moléculas de água Todos os outros átomos
12 Função MolDock Score (PLP) Para levarmos em conta a direção na ligação de hidrogênio, um fator de correção (H factor ) é multiplicado pela energia calculada para a ligação de hidrogênio. s figuras abaixo ilustram a geometria da ligação de hidrogênio. D H D H D H DH H D DH H D D: átomo doador de hidrogênio : átomo aceitador de hidrogênio : átomo antecessor do aceitador de ligação de hidrogênio : ângulo entre D-H- : ângulo entre H-- : ângulo entre D-- 2
13 Função MolDock Score (PLP) equação abaixo ilustra o fator H factor que é uma função do tipo rampa, factor o o o o o o 90 ;50. ;90 ;00. ;90 ; H 00 DH ; H D Os ângulos indicados entre parênteses são os valores mínimos ( min ) e máximo ( max ), respectivamente. O H factor varia entre 0 e, assim em situações angulares desfavoráveis à formação da ligação de hidrogênio, o H factor será zero, em situações favoráveis o valor final é, ou seja, o termo do E PLP referente à ligação de hidrogênio fica integralmente considerado. DH H D D: átomo doador de hidrogênio : átomo aceitador de hidrogênio : átomo antecessor do aceitador de ligação de hidrogênio : ângulo entre D-H- : ângulo entre H-- : ângulo entre D-- 3
14 Função MolDock Score (PLP) Os gráficos das funções rampas, para cada um dos ângulos das ligações de hidrogênio, são mostrados abaixo. o o o o DH ;90 ; 50 H ;90 ; 00 0 DH o o o D;90 ; 00 0 DH H D H o D: átomo doador de hidrogênio : átomo aceitador de hidrogênio : átomo antecessor do aceitador de ligação de H : ângulo entre D-H- : ângulo entre H-- : ângulo entre D-- 0 D o
15 Função MolDock Score (termo intramolecular) O último termo da função MolDock Score, é referente à energia interna do ligante (E intra ), que tem a seguinte forma. E intra EPLP rij ) iligante jligante ( cos m. 0 Eclash ligações flexíveis somatória dupla indica que a soma é realizada para todo par de átomos do ligante, que esteja a mais de duas ligações de distância. O segundo termo da equação refere-se à energia de torção, com os parâmetros mostrados na tabela abaixo. Tipo de ligação Θ 0 ( o ) m sp 2 -sp 3 0 6,5 sp 3 -sp ,0 sp 2 -sp ,0 O termo E clash é uma penalização para átomos próximos, abaixo de 2,0 Å, o valor assumido neste caso é
16 Função MolDock Score (uso) Durante uma simulação de docking, o MVD calcula a função escore e armazena os valores no arquivo DockingResults.mvdresults. lém do MoDock Score, o MVD calcula outros termos de energia, como interação de van der Waals e interações eletrostáticas de longa distância, entre outros. Esses termos são usados para calcular a função Rerank Score. Cada termo energético usado no Rerank Score tem um peso que está armazenado no arquivo RerankingCoefficients.txt. Podemos modificar esses pesos para obtermos funções escores adequadas para o sistema biológico que estamos estudando. Para avaliarmos os resultados da simulação de docking, podemos salvar os resultados num arquivo no formato CSV editando o arquivo DockingResults.mvdresults. 6
17 Função MolDock Score (uso) De uma forma geral, podemos selecionar os seguintes termos: Moldock Score, Rerank Score, Total Interaction Energy Between the Pose and the Target, Internal Energy of the Pose, HBond Score, Ligand Efficiency (MolDock Score Divided by Number of Heavy toms), Ligand Efficiency 3 (Rerank Score Divided by Number of Heavy toms), Docking Score (Evaluated Before Post-processing), Energy Contribution from Non-displaced and Displaced Water Interactions o lado temos a tabela parcial com os termos energéticos selecionados. 7
18 Virtual Screening in silico in vitro in vivo Moléculas puras Base de dados moleculares Seleção de alvo molecular Simulações de docking Na descoberta de fármacos podemos usar as abordagens in silico. Para isso, temos que ter a estrutura 3D da proteína, que foi selecionada a partir do conhecimento do sistema biológico. Usamos uma metodologia chamada docking molecular, para procurarmos chaves em potencial que se encaixem do sítio ativo da enzima. O teste de várias moléculas é chamado Virtual Screening. s melhores moléculas selecionadas são submetidas aos testes posteriores. Cálculo da afinidade proteína-ligante teórico Testes in vivo Testes in vitro Seleção dos melhores resultados 8
19 Material dicional (rtigos Indicados) Bio-inspired algorithms applied to molecular docking simulations. Heberlé G, de zevedo WF Jr. Curr Med Chem. 20;8(9): MolDock applied to structure-based virtual screening. De zevedo WF Jr. Curr Drug Targets. 200 Mar;(3): Revista científica Current Drug Targets Revista científica Current Medicinal Chemistry Imagem disponível em: < Fonte da imagem: < > cesso em : 0 de julho de 205. cesso em : 0 de julho de
Scoring Functions (MolDock Score) Prof. Dr. Walter F. de Azevedo Jr. walter.junior@pucrs.br ou walter.filgueira@hotmail.com
 2015 Dr. Walter F. de Azevedo Jr. Scoring Functions (MolDock Score) Prof. Dr. Walter F. de Azevedo Jr. walter.junior@pucrs.br ou walter.filgueira@hotmail.com 1 O que é desenho de fármacos baseado em computador?
2015 Dr. Walter F. de Azevedo Jr. Scoring Functions (MolDock Score) Prof. Dr. Walter F. de Azevedo Jr. walter.junior@pucrs.br ou walter.filgueira@hotmail.com 1 O que é desenho de fármacos baseado em computador?
2016 Dr. Walter F. de Azevedo Jr.
 2016 Dr. Walter F. de Azevedo Jr. 000000000000000000000000000000000000000 000000000000000000000000000000000000000 000000000000111111111110001100000000000 000000000001111111111111111111000000001 000000000111111111111111111111111000000
2016 Dr. Walter F. de Azevedo Jr. 000000000000000000000000000000000000000 000000000000000000000000000000000000000 000000000000111111111110001100000000000 000000000001111111111111111111000000001 000000000111111111111111111111111000000
Visualização de Hélices
 Grupo: Curso: Turma: Data: Visualização de Hélices Objetivos Visualizar a estrutura tridimensional de hélices presentes na estrutura de proteínas e peptídeos, usandose recursos computacionais. Analisar
Grupo: Curso: Turma: Data: Visualização de Hélices Objetivos Visualizar a estrutura tridimensional de hélices presentes na estrutura de proteínas e peptídeos, usandose recursos computacionais. Analisar
2018 Dr. Walter F. de Azevedo Jr. Biofísica. Prof. Dr. Walter F. de Azevedo Jr.
 2018 Dr. Walter F. de Azevedo Jr. Biofísica Prof. Dr. Walter F. de Azevedo Jr. 1 Ao lado temos a estrutura de um peptídeo formado pelos aminoácidos Arginina - Prolina - Alanina - Tirosina - Serina, coloridos
2018 Dr. Walter F. de Azevedo Jr. Biofísica Prof. Dr. Walter F. de Azevedo Jr. 1 Ao lado temos a estrutura de um peptídeo formado pelos aminoácidos Arginina - Prolina - Alanina - Tirosina - Serina, coloridos
Interações de van der Waals. Interações de van der Waals. Interações de van der Waals.
 Interações de van der Waals aandrico@if.sc.usp.br Também conhecidas como forças de dispersão de London, caracterizam-se pelo momento de dipolo induzido e ocorrem entre todos os átomos. Apesar de moléculas
Interações de van der Waals aandrico@if.sc.usp.br Também conhecidas como forças de dispersão de London, caracterizam-se pelo momento de dipolo induzido e ocorrem entre todos os átomos. Apesar de moléculas
Mecanismo de Inibição de Enzimas Preparação de Micro e Macromoléculas Docagem Molecular Exemplo de Aplicação
 bjetivos FFI0776 Modelagem e Engenharia de Proteínas Mecanismo de Inibição de Enzimas Preparação de Micro e Macromoléculas M l Docagem Molecular Exemplo de Aplicação Prof. Rafael V. C. Guido rvcguido@ifsc.usp.br
bjetivos FFI0776 Modelagem e Engenharia de Proteínas Mecanismo de Inibição de Enzimas Preparação de Micro e Macromoléculas M l Docagem Molecular Exemplo de Aplicação Prof. Rafael V. C. Guido rvcguido@ifsc.usp.br
OBJETIVOS INTERAÇÕES INTERMOLECULARES INTERAÇÕES INTERMOLECULARES.
 OBJETIVOS aandrico@if.sc.usp.br Interações Intermoleculares Mecanismo de Ação Modo de Ligação Complexos Recetor-Ligante Exemplos e Exercícios INTERAÇÕES INTERMOLECULARES INTERAÇÕES INTERMOLECULARES O processo
OBJETIVOS aandrico@if.sc.usp.br Interações Intermoleculares Mecanismo de Ação Modo de Ligação Complexos Recetor-Ligante Exemplos e Exercícios INTERAÇÕES INTERMOLECULARES INTERAÇÕES INTERMOLECULARES O processo
OBJETIVOS AULA 10 DESENVOLVIMENTO DESCOBERTA. ¾ Otimização de Moléculas Bioativas. ¾ Integração com o processo de HTS
 AULA 10 2 DESCOBERTA DESENVOLVIMENTO Pesquisa Básica (Pré-clínica) (Fases Clínicas I IV) Identificação do alvo molecular Triagens clínicas conduzidas em humanos: avaliação da segurança, impacto no estado
AULA 10 2 DESCOBERTA DESENVOLVIMENTO Pesquisa Básica (Pré-clínica) (Fases Clínicas I IV) Identificação do alvo molecular Triagens clínicas conduzidas em humanos: avaliação da segurança, impacto no estado
Gabriela Sehnem Heck 1,2, Prof. Dr. Walter F. de Azevedo Jr 2. (orientador)
 Pontifícia Universidade Católica do Rio Grande do Sul Gabriela Sehnem Heck 1,2, Prof. Dr. Walter F. de Azevedo Jr 2. (orientador) 1 Acadêmica de Curso de Ciêncas Biológicas da PUCRS, 2 Laboratório de Bioquímica
Pontifícia Universidade Católica do Rio Grande do Sul Gabriela Sehnem Heck 1,2, Prof. Dr. Walter F. de Azevedo Jr 2. (orientador) 1 Acadêmica de Curso de Ciêncas Biológicas da PUCRS, 2 Laboratório de Bioquímica
Docking (ou atracamento) Molecular. Profa. Dra. Rafaela Ferreira Dept. de Bioquímica e Imunologia 9 de outubro de 2014
 Docking (ou atracamento) Molecular Profa. Dra. Rafaela Ferreira Dept. de Bioquímica e Imunologia rafaelasf@gmail.com 9 de outubro de 2014 O ATRACAMENTO MOLECULAR PERMITE ESTIMAR COMO DUAS MOLÉCULAS INTERAGEM
Docking (ou atracamento) Molecular Profa. Dra. Rafaela Ferreira Dept. de Bioquímica e Imunologia rafaelasf@gmail.com 9 de outubro de 2014 O ATRACAMENTO MOLECULAR PERMITE ESTIMAR COMO DUAS MOLÉCULAS INTERAGEM
AULA 7. Cálculo das energias de interação estereoquímicas e eletrostáticas entre as moléculas do conjunto de
 Análise Comparativa dos Campos Moleculares AULA 7 Análise Comparativa dos Campos Moleculares A contribuição devido às forças de dispersão entre moléculas é descrita pelo potencial de Lennard-Jones e as
Análise Comparativa dos Campos Moleculares AULA 7 Análise Comparativa dos Campos Moleculares A contribuição devido às forças de dispersão entre moléculas é descrita pelo potencial de Lennard-Jones e as
Descritores moleculares para aprendizagem automática ( Machine learning ) João Aires de Sousa
 Descritores moleculares para aprendizagem automática ( Machine learning ) 1 Pode um computador aprender Química? 2 Aprender o quê? Por ex., aprender a prever propriedades a partir da estrutura molecular
Descritores moleculares para aprendizagem automática ( Machine learning ) 1 Pode um computador aprender Química? 2 Aprender o quê? Por ex., aprender a prever propriedades a partir da estrutura molecular
QUI109 QUÍMICA GERAL (Ciências Biológicas) 8ª aula /
 QUI109 QUÍMICA GERAL (Ciências Biológicas) 8ª aula / 2016-2 Prof. Mauricio X. Coutrim (disponível em: http://professor.ufop.br/mcoutrim) A LIGAÇÃO COVALENTE É BASEADA NO COMPARTILHAMENTO DE ELÉTRONS Ex.
QUI109 QUÍMICA GERAL (Ciências Biológicas) 8ª aula / 2016-2 Prof. Mauricio X. Coutrim (disponível em: http://professor.ufop.br/mcoutrim) A LIGAÇÃO COVALENTE É BASEADA NO COMPARTILHAMENTO DE ELÉTRONS Ex.
Polaridade, Geometria Molecular e Forças Intermoleculares
 Polaridade, Geometria Molecular e Forças Intermoleculares Polaridade das Ligações x Polaridade das Moléculas Polaridade das Ligações Ligações covalentes Apolares: elétrons são compartilhados igualmente
Polaridade, Geometria Molecular e Forças Intermoleculares Polaridade das Ligações x Polaridade das Moléculas Polaridade das Ligações Ligações covalentes Apolares: elétrons são compartilhados igualmente
Bioquímica. Prof. Dr. Walter F. de Azevedo Jr Dr. Walter F. de Azevedo Jr.
 2018 Dr. Walter F. de Azevedo Jr. Bioquímica Prof. Dr. Walter F. de Azevedo Jr. E-mail: walter@azevedolab.net 1 As proteínas que tiveram sua estrutura tridimensional determinadas estão disponíveis no site
2018 Dr. Walter F. de Azevedo Jr. Bioquímica Prof. Dr. Walter F. de Azevedo Jr. E-mail: walter@azevedolab.net 1 As proteínas que tiveram sua estrutura tridimensional determinadas estão disponíveis no site
Mecânica Molecular. Dinâmica Molecular. Exemplos de aplicação. é um método relativamente. energia e conformação de micro ou macromoléculas
 Objetivos FFI0776 Modelagem e Engenharia de Proteínas Prof. Rafael V. C. Guido rvcguido@ifsc.usp.br Aula 04 Mecânica Molecular Campo de força Exemplos de Campo de Força Dinâmica Molecular Etapas envolvidas
Objetivos FFI0776 Modelagem e Engenharia de Proteínas Prof. Rafael V. C. Guido rvcguido@ifsc.usp.br Aula 04 Mecânica Molecular Campo de força Exemplos de Campo de Força Dinâmica Molecular Etapas envolvidas
Enzimologia Aula 2. Leitura sugerida. Biochemistry Voet & Voet 3ª edição Capítulo 10 parte 1Bb (página 323), parte 4A, 4B e 4C (páginas )
 Enzimologia Aula 2 Leitura sugerida Biochemistry Voet & Voet 3ª edição Capítulo 10 parte 1Bb (página 323), parte 4A, 4B e 4C (páginas 346-350) Structure and Mechanism in Protein Science Alan Fersht 1999
Enzimologia Aula 2 Leitura sugerida Biochemistry Voet & Voet 3ª edição Capítulo 10 parte 1Bb (página 323), parte 4A, 4B e 4C (páginas 346-350) Structure and Mechanism in Protein Science Alan Fersht 1999
SOLUÇÃO PRATIQUE EM CASA - QUÍMICA
 SOLUÇÃO PC. Percebe-se pela geometria apresentada, que: 3 2 () A hibridização de é sp d. 2 2 6 2 6 2 0 6 2 0 6 Xe (Z 54) : s 2s 2p 3s 3p 4s 3d 4p 5s 4d 5p Xe (camada de valência): SOLUÇÃO PRATIQUE EM CASA
SOLUÇÃO PC. Percebe-se pela geometria apresentada, que: 3 2 () A hibridização de é sp d. 2 2 6 2 6 2 0 6 2 0 6 Xe (Z 54) : s 2s 2p 3s 3p 4s 3d 4p 5s 4d 5p Xe (camada de valência): SOLUÇÃO PRATIQUE EM CASA
ATRACAMENTO MOLECULAR
 UNIVERSIDADE FEDERAL DO ESPÍRITO SANTO CENTRO DE CIÊNCIAS DA SAÚDE PROGRAMA DE PÓS-GRADUAÇÃO EM BIOTECNOLOGIA BIOINFORMÁTICA ATRACAMENTO MOLECULAR VITÓRIA 2018 Ivana Alece Arantes Moreno Júlia de Assis
UNIVERSIDADE FEDERAL DO ESPÍRITO SANTO CENTRO DE CIÊNCIAS DA SAÚDE PROGRAMA DE PÓS-GRADUAÇÃO EM BIOTECNOLOGIA BIOINFORMÁTICA ATRACAMENTO MOLECULAR VITÓRIA 2018 Ivana Alece Arantes Moreno Júlia de Assis
Química A Extensivo V. 5
 Química A Extensivo V. 5 Exercícios 01) Respostas: a) f) k) b) g) l) c) h) m) d) i) n) e) j) o) 02) E 03) A 04) 12 05) 60 * A representação dos ângulos de ligação é apenas aproximada. 01. Errada. A molécula
Química A Extensivo V. 5 Exercícios 01) Respostas: a) f) k) b) g) l) c) h) m) d) i) n) e) j) o) 02) E 03) A 04) 12 05) 60 * A representação dos ângulos de ligação é apenas aproximada. 01. Errada. A molécula
Space, the final frontier... Como explorar o espaço de funções escores para o desenho de fármacos. Prof. Dr. Walter F. de Azevedo Jr. azevedolab.
 Space, the final frontier... Como explorar o espaço de funções escores para o desenho de fármacos. Prof. Dr. Walter F. de Azevedo Jr. azevedolab.net Doutor em Ciências Física Aplicada Universidade de São
Space, the final frontier... Como explorar o espaço de funções escores para o desenho de fármacos. Prof. Dr. Walter F. de Azevedo Jr. azevedolab.net Doutor em Ciências Física Aplicada Universidade de São
Interacções em moléculas biológicas
 Interacções em moléculas biológicas Interacções intramoleculares Ligações covalentes (manutenção da estrutura covalente das moléculas Interacções não-covalentes (conformções, folding, estrutura secundária)
Interacções em moléculas biológicas Interacções intramoleculares Ligações covalentes (manutenção da estrutura covalente das moléculas Interacções não-covalentes (conformções, folding, estrutura secundária)
ESCOLA SECUNDÁRIA PINHAL DO REI FICHA FORMATIVA 2ºTESTE
 Ano Letivo 2016/2017 ESCOLA SECUNDÁRIA PINHAL DO REI FICHA FORMATIVA 2ºTESTE Física e Química A 10ºAno 1. A energia dos eletrões nos átomos inclui: (A) apenas o efeito das atrações entre os eletrões e
Ano Letivo 2016/2017 ESCOLA SECUNDÁRIA PINHAL DO REI FICHA FORMATIVA 2ºTESTE Física e Química A 10ºAno 1. A energia dos eletrões nos átomos inclui: (A) apenas o efeito das atrações entre os eletrões e
Ajuda a descrever o compartilhamento dos elétrons entre os átomos
 Polaridade, Geometria Molecular e Forças Intermoleculares Polaridade das Ligações Ajuda a descrever o compartilhamento dos elétrons entre os átomos Ligações covalentes Apolares: elétrons são compartilhados
Polaridade, Geometria Molecular e Forças Intermoleculares Polaridade das Ligações Ajuda a descrever o compartilhamento dos elétrons entre os átomos Ligações covalentes Apolares: elétrons são compartilhados
Aula 1. Referência. Bancos de Dados. Linguagem x Informação. Introdução a Bioquímica: Biomoléculas. Introdução ao Curso: Aminoácidos.
 Introdução a Bioquímica: Biomoléculas Aula 1 Introdução ao urso: Aminoácidos eferência Autores: Ignez aracelli e Julio Zukerman-Schpector Editora: EdUFSar Ignez aracelli BioMat DF UNESP/Bauru Julio Zukerman
Introdução a Bioquímica: Biomoléculas Aula 1 Introdução ao urso: Aminoácidos eferência Autores: Ignez aracelli e Julio Zukerman-Schpector Editora: EdUFSar Ignez aracelli BioMat DF UNESP/Bauru Julio Zukerman
2015 Dr. Walter F. de Azevedo Jr.
 2015 Dr. Walter F. de Azevedo Jr. 110100111010 101101011100 100110011000 1111111100111 101101011010 100111100111 111110011000 110100111100 Evolução Diferencial e Sua Aplicação em Docking do Sistema Proteína-Ligante
2015 Dr. Walter F. de Azevedo Jr. 110100111010 101101011100 100110011000 1111111100111 101101011010 100111100111 111110011000 110100111100 Evolução Diferencial e Sua Aplicação em Docking do Sistema Proteína-Ligante
QSAR 3D Planejamento de NCEs QSAR 3D. O processo de reconhecimento molecular é um processo tridimensional. Conformação bioativa.
 QSAR 3D Interação Fármaco-Receptor AULA 5 a maioria dos casos, a ação dos fármacos está ligada a interação fármaco-receptor, receptor, através de uma associação do tipo reversível e não- covalente Agonistas
QSAR 3D Interação Fármaco-Receptor AULA 5 a maioria dos casos, a ação dos fármacos está ligada a interação fármaco-receptor, receptor, através de uma associação do tipo reversível e não- covalente Agonistas
Fundamentos da modelagem Molecular - 1
 Fundamentos da modelagem Molecular - 1 Departamento de Física UFPel Introdução Rotas para a pesquisa Conexão entre experimento, simulação e teoria Sistema real Fazer modelos do sistema Fazer experimentos
Fundamentos da modelagem Molecular - 1 Departamento de Física UFPel Introdução Rotas para a pesquisa Conexão entre experimento, simulação e teoria Sistema real Fazer modelos do sistema Fazer experimentos
Métodos de Química Computacional
 Universidade Federal de Ouro Preto Instituto de Ciências Exatas e Biológicas Departamento de Química Métodos de Química Computacional Professora: Melissa Soares Caetano Mecânica Molecular Mecânica Molecular
Universidade Federal de Ouro Preto Instituto de Ciências Exatas e Biológicas Departamento de Química Métodos de Química Computacional Professora: Melissa Soares Caetano Mecânica Molecular Mecânica Molecular
Evolução Diferencial e Sua Aplicação em Docking do Sistema Proteína-Ligante Prof. Dr. Walter F. de Azevedo Jr.
 2017 Dr. Walter F. de Azevedo Jr. 110100111010 101101011100 100110011000 1111111100111 101101011010 100111100111 111110011000 110100111100 Evolução Diferencial e Sua Aplicação em Docking do Sistema Proteína-Ligante
2017 Dr. Walter F. de Azevedo Jr. 110100111010 101101011100 100110011000 1111111100111 101101011010 100111100111 111110011000 110100111100 Evolução Diferencial e Sua Aplicação em Docking do Sistema Proteína-Ligante
PMT-3131 Química dos Materiais Aplicada à Engenharia Elétrica. TURMA 3 AULA 1 Augusto Neiva
 PMT-3131 Química dos Materiais Aplicada à Engenharia Elétrica TURMA 3 AULA 1 Augusto Neiva APOSTILA 2017 PARTE 1 - LIGAÇÕES QUÍMICAS PMT-3131 Química dos Materiais Aplicada à Engenharia Elétrica PQI-3110
PMT-3131 Química dos Materiais Aplicada à Engenharia Elétrica TURMA 3 AULA 1 Augusto Neiva APOSTILA 2017 PARTE 1 - LIGAÇÕES QUÍMICAS PMT-3131 Química dos Materiais Aplicada à Engenharia Elétrica PQI-3110
2.1.2 Ligação covalente
 2.1.2 Ligação covalente Adaptado pelo Prof. Luís Perna Ligações covalentes Quando a partilha de eletrões é significativa e localizada entre átomos denomina-se de ligação covalente. Nem todos os átomos
2.1.2 Ligação covalente Adaptado pelo Prof. Luís Perna Ligações covalentes Quando a partilha de eletrões é significativa e localizada entre átomos denomina-se de ligação covalente. Nem todos os átomos
Bioinformática. Uma Viagem Pelas Moléculas da Vida
 Bioinformática. Uma Viagem Pelas Moléculas da Vida Prof. Dr. Walter F. de Azevedo Jr. azevedolab.net Representação CPK Diversos programas gráficos permitem a visualização molecular, como a representada
Bioinformática. Uma Viagem Pelas Moléculas da Vida Prof. Dr. Walter F. de Azevedo Jr. azevedolab.net Representação CPK Diversos programas gráficos permitem a visualização molecular, como a representada
Fundamentos da modelagem Molecular - 1
 Fundamentos da modelagem Molecular - 1 Departamento de Física UFPel ufpellogo Introdução Rotas para a pesquisa Conexão entre experimento, simulação e teoria Sistema real Fazer modelos do sistema Fazer
Fundamentos da modelagem Molecular - 1 Departamento de Física UFPel ufpellogo Introdução Rotas para a pesquisa Conexão entre experimento, simulação e teoria Sistema real Fazer modelos do sistema Fazer
2018 Dr. Walter F. de Azevedo Jr. Problema da Fase
 2018 Dr. Walter F. de Azevedo Jr. 000000000000000000000000000000000000000 000000000000000000000000000000000000000 000000000000111111111110001100000000000 000000000001111111111111111111000000001 000000000111111111111111111111111000000
2018 Dr. Walter F. de Azevedo Jr. 000000000000000000000000000000000000000 000000000000000000000000000000000000000 000000000000111111111110001100000000000 000000000001111111111111111111000000001 000000000111111111111111111111111000000
Forças intermoleculares. Formação de fases condensadas Forças íon-dipolo Forças dipolo-dipolo Forças de London Ligação de hidrogênio
 Forças intermoleculares Formação de fases condensadas Forças íon-dipolo Forças dipolo-dipolo Forças de London Ligação de hidrogênio Estado gasoso O estado da matéria mais simples é o gás. Descrevemos esse
Forças intermoleculares Formação de fases condensadas Forças íon-dipolo Forças dipolo-dipolo Forças de London Ligação de hidrogênio Estado gasoso O estado da matéria mais simples é o gás. Descrevemos esse
ESCOLA SECUNDÁRIA PINHAL DO REI FICHA FORMATIVA 2ºTESTE
 Ano Letivo 2016/2017 ESOLA SEUNDÁRIA PINAL DO REI FIA FORMATIVA 2ºTESTE Física e Química A 10ºAno 1. A energia dos eletrões nos átomos inclui: (A) apenas o efeito das atrações entre os eletrões e o núcleo.
Ano Letivo 2016/2017 ESOLA SEUNDÁRIA PINAL DO REI FIA FORMATIVA 2ºTESTE Física e Química A 10ºAno 1. A energia dos eletrões nos átomos inclui: (A) apenas o efeito das atrações entre os eletrões e o núcleo.
2019 Dr. Walter F. de Azevedo Jr.
 2019 Dr. Walter F. de Azevedo Jr. 000000000000000000000000000000000000000 000000000000000000000000000000000000000 000000000000111111111110001100000000000 000000000001111111111111111111000000001 000000000111111111111111111111111000000
2019 Dr. Walter F. de Azevedo Jr. 000000000000000000000000000000000000000 000000000000000000000000000000000000000 000000000000111111111110001100000000000 000000000001111111111111111111000000001 000000000111111111111111111111111000000
2.1 Princípios gerais da ligação química. Ligações químicas
 2.1 Princípios gerais da ligação química Ligações químicas A associação de átomos formando moléculas, ou em agregados de maiores dimensões como, por exemplo, nos metais, é possível pelo estabelecimento
2.1 Princípios gerais da ligação química Ligações químicas A associação de átomos formando moléculas, ou em agregados de maiores dimensões como, por exemplo, nos metais, é possível pelo estabelecimento
BC 0307 Transformações Químicas
 BC 0307 Transformações Químicas ANDERSON ORZARI RIBEIRO Anderson.ribeiro@ufabc.edu.br Bloco B, 10º andar - Sala 1043 BC 0307 Transformações Químicas AULA 5 Compostos de carbono e interações intermoleculares
BC 0307 Transformações Químicas ANDERSON ORZARI RIBEIRO Anderson.ribeiro@ufabc.edu.br Bloco B, 10º andar - Sala 1043 BC 0307 Transformações Químicas AULA 5 Compostos de carbono e interações intermoleculares
"Biologia de Sistemas Computacional Aplicada ao Desenvolvimento de Fármacos. Um Estudo de Inibidores da Quinase Dependente de Ciclina (CDK)"
 "Biologia de Sistemas Computacional Aplicada ao Desenvolvimento de Fármacos. Um Estudo de Inibidores da Quinase Dependente de Ciclina (CDK)" print(cdk) Prof. Dr. Walter F. de Azevedo Jr. Laboratório de
"Biologia de Sistemas Computacional Aplicada ao Desenvolvimento de Fármacos. Um Estudo de Inibidores da Quinase Dependente de Ciclina (CDK)" print(cdk) Prof. Dr. Walter F. de Azevedo Jr. Laboratório de
ANDERSON ORZARI RIBEIRO
 Transformações Químicas ANDERSON ORZARI RIBEIRO Anderson.ribeiro@ufabc.edu.br Bloco B, 10º andar - Sala 1043 Transformações Químicas AULA 5 Compostos de carbono e interações intermoleculares CAPÍTULO 3
Transformações Químicas ANDERSON ORZARI RIBEIRO Anderson.ribeiro@ufabc.edu.br Bloco B, 10º andar - Sala 1043 Transformações Químicas AULA 5 Compostos de carbono e interações intermoleculares CAPÍTULO 3
Polaridade das moléculas
 Polaridade das moléculas Determinar a polaridade de uma molécula é identificar se ela apresenta polos negativos e positivos (molécula polar) ou não (molécula apolar). Para isso, é necessário conhecer o
Polaridade das moléculas Determinar a polaridade de uma molécula é identificar se ela apresenta polos negativos e positivos (molécula polar) ou não (molécula apolar). Para isso, é necessário conhecer o
RESOLUÇÃO DE EXERCÍCIOS PROPOSTOS AULA 02 TURMA FMJ
 RESOLUÇÃO DE EXERCÍCIOS PROPOSTOS AULA 02 TURMA FMJ 03. Item C O equador da figura mostrada pode ser representado como abaixo. 01. Item B I Correto. A energia para quebrar a ligação H F (568 kj/mol) é
RESOLUÇÃO DE EXERCÍCIOS PROPOSTOS AULA 02 TURMA FMJ 03. Item C O equador da figura mostrada pode ser representado como abaixo. 01. Item B I Correto. A energia para quebrar a ligação H F (568 kj/mol) é
Módulo 1. CONTEÚDOS E OBJETIVOS
 Módulo 1. CONTEÚDOS E OBJETIVOS CONTEÚDOS OBJETIVOS 1. Elementos químicos e sua organização: - Massa e tamanho dos átomos: - Massa isotópica e massa atómica relativa média Quantidade de matéria e massa
Módulo 1. CONTEÚDOS E OBJETIVOS CONTEÚDOS OBJETIVOS 1. Elementos químicos e sua organização: - Massa e tamanho dos átomos: - Massa isotópica e massa atómica relativa média Quantidade de matéria e massa
Física Moderna II - FNC376
 Universidade de São Paulo Instituto de Física Física Moderna II - FNC376 Profa. Márcia de Almeida Rizzutto 1o. Semestre de 2008 1 MQ átomos > < Moléculas moléculas e sólidos núcleos e partículas Moléculas
Universidade de São Paulo Instituto de Física Física Moderna II - FNC376 Profa. Márcia de Almeida Rizzutto 1o. Semestre de 2008 1 MQ átomos > < Moléculas moléculas e sólidos núcleos e partículas Moléculas
Ligações Interatômicas: IÔNICA = metal + não-metal COVALENTE = não-metais METÁLICA = metais
 Ligações Químicas Ligações Interatômicas: IÔNICA = metal + não-metal COVALENTE = não-metais METÁLICA = metais Ligação iônica Transferência de elétrons de um átomo para outro Íons de cargas opostas Forças
Ligações Químicas Ligações Interatômicas: IÔNICA = metal + não-metal COVALENTE = não-metais METÁLICA = metais Ligação iônica Transferência de elétrons de um átomo para outro Íons de cargas opostas Forças
Química Bio-inorgânica - roteiro de aulas
 Química Bio-inorgânica - roteiro de aulas 2014 - Prof. André Ferraz Pense: Como esta um metal em um sistema vivo? Para entender a questão dos "metais em sistemas biológicos" precisamos entender: 1. Ligação
Química Bio-inorgânica - roteiro de aulas 2014 - Prof. André Ferraz Pense: Como esta um metal em um sistema vivo? Para entender a questão dos "metais em sistemas biológicos" precisamos entender: 1. Ligação
04/10/2011 FORÇAS DE INTERAÇÃO MOLECULAR ENTRE LIGANTES E PROTEÍNAS. O que é um medicamento?
 FORÇAS DE INTERAÇÃO MOLECULAR ENTRE LIGANTES E PROTEÍNAS O que é um medicamento? 1 O que é um medicamento? Medicamento (produto final) Excipientes + Principio ativo ( fármaco, droga ) Como atua uma droga?
FORÇAS DE INTERAÇÃO MOLECULAR ENTRE LIGANTES E PROTEÍNAS O que é um medicamento? 1 O que é um medicamento? Medicamento (produto final) Excipientes + Principio ativo ( fármaco, droga ) Como atua uma droga?
Ficha de preparação para o 2º teste- Física e Química 10º CPM e CPSI
 Ficha de preparação para o 2º teste- Física e Química 10º CPM e CPSI 1. A substância composta fluoreto de hidrogénio, HF, está representada na figura segundo notação de Lewis. 1.1 O que representam os
Ficha de preparação para o 2º teste- Física e Química 10º CPM e CPSI 1. A substância composta fluoreto de hidrogénio, HF, está representada na figura segundo notação de Lewis. 1.1 O que representam os
Química Orgânica Ambiental
 Química Orgânica Ambiental Aula 2 Formas de representação e interações moleculares Prof. Dr. Leandro Vinícius Alves Gurgel 1. Introdução: Estrutura de Lewis 1) Estruturas de Lewis mostram as conexões entre
Química Orgânica Ambiental Aula 2 Formas de representação e interações moleculares Prof. Dr. Leandro Vinícius Alves Gurgel 1. Introdução: Estrutura de Lewis 1) Estruturas de Lewis mostram as conexões entre
Ligações Interatômicas: IÔNICA = metal + não-metal COVALENTE = não-metais METÁLICA = metais
 Ligações Químicas Ligações Interatômicas: IÔNICA = metal + não-metal COVALENTE = não-metais METÁLICA = metais Ligação iônica Transferência de elétrons de um átomo para outro Íons de cargas opostas Forças
Ligações Químicas Ligações Interatômicas: IÔNICA = metal + não-metal COVALENTE = não-metais METÁLICA = metais Ligação iônica Transferência de elétrons de um átomo para outro Íons de cargas opostas Forças
Métodos de Química Computacional
 Universidade Federal de Ouro Preto Instituto de Ciências Exatas e Biológicas Departamento de Química Métodos de Química Computacional Professora: Melissa Soares Caetano Química Medicinal Matemática Química
Universidade Federal de Ouro Preto Instituto de Ciências Exatas e Biológicas Departamento de Química Métodos de Química Computacional Professora: Melissa Soares Caetano Química Medicinal Matemática Química
História antiga da química de proteínas é a da Hb Hb cristalina atlas fotográfico com cristais de 109 espécies. Urease 1926.
 História antiga da química de proteínas é a da Hb. 1849 Hb cristalina. 1909 atlas fotográfico com cristais de 109 espécies. Urease 1926. Uma das primeiras proteínas a ter a massa molecular determinada,
História antiga da química de proteínas é a da Hb. 1849 Hb cristalina. 1909 atlas fotográfico com cristais de 109 espécies. Urease 1926. Uma das primeiras proteínas a ter a massa molecular determinada,
Algumas EXCEÇÕES à TEORIA DO OCTETO. H Be H. H Be H. O berílio ficou estável com 4 elétrons na camada de valência
 Algumas EXCEÇÕES à TEORIA DO OCTETO H Be H H Be H O berílio ficou estável com 4 elétrons na camada de valência Algumas EXCEÇÕES à TEORIA DO OCTETO B B O boro ficou estável com 6 elétrons na camada de valência
Algumas EXCEÇÕES à TEORIA DO OCTETO H Be H H Be H O berílio ficou estável com 4 elétrons na camada de valência Algumas EXCEÇÕES à TEORIA DO OCTETO B B O boro ficou estável com 6 elétrons na camada de valência
Física Molecular Estrutura das Ligações Químicas
 Física Molecular Estrutura das Ligações Químicas 100 É razoável focalizar o estudo de moléculas nas interações entre elétrons e núcleos já reagrupados em caroço atômico (núcleo + camadas fechadas internas),
Física Molecular Estrutura das Ligações Químicas 100 É razoável focalizar o estudo de moléculas nas interações entre elétrons e núcleos já reagrupados em caroço atômico (núcleo + camadas fechadas internas),
CURSO: ENFERMAGEM DISCIPLINA: BIOQUÍMICA HUMANA PROF. WILLAME BEZERRA. Aminoácidos. Prof. Willame Bezerra
 CURSO: ENFERMAGEM DISCIPLINA: BIOQUÍMICA HUMANA PROF. WILLAME BEZERRA Aminoácidos Prof. Willame Bezerra As proteínas são as biomoléculas mais abundantes nos seres vivos e exercem funções fundamentais em
CURSO: ENFERMAGEM DISCIPLINA: BIOQUÍMICA HUMANA PROF. WILLAME BEZERRA Aminoácidos Prof. Willame Bezerra As proteínas são as biomoléculas mais abundantes nos seres vivos e exercem funções fundamentais em
Métodos de Química Computacional
 Universidade Federal de Ouro Preto Instituto de Ciências Exatas e Biológicas Departamento de Química Métodos de Química Computacional Professora: Melissa Soares Caetano Dinâmica molecular Conhecendo posições
Universidade Federal de Ouro Preto Instituto de Ciências Exatas e Biológicas Departamento de Química Métodos de Química Computacional Professora: Melissa Soares Caetano Dinâmica molecular Conhecendo posições
TEORIA DA LIGAÇÃO QUÍMICA
 TERIA DA LIGAÇÃ QUÍMICA s exercícios seguintes estão directamente relacionados com a matéria exposta no capítulo do manual da disciplina de Química Geral. São apresentados vários tipos de exercícios resposta
TERIA DA LIGAÇÃ QUÍMICA s exercícios seguintes estão directamente relacionados com a matéria exposta no capítulo do manual da disciplina de Química Geral. São apresentados vários tipos de exercícios resposta
LIGAÇÕES QUÍMICAS INTERMOLECULARES
 LIGAÇÕES QUÍMICAS INTERMOLECULARES Ligações Químicas INTRAmoleculares INTERmoleculares Tipo de Ligação Química INTERMOLECULAR INTRAMOLECULAR Magnitude Comparativamente FRACA FORTE Polaridade em Ligações
LIGAÇÕES QUÍMICAS INTERMOLECULARES Ligações Químicas INTRAmoleculares INTERmoleculares Tipo de Ligação Química INTERMOLECULAR INTRAMOLECULAR Magnitude Comparativamente FRACA FORTE Polaridade em Ligações
Universidade do Estado de Santa Catarina UDESC - CCT Química Geral Profª Fabíola Corrêa viel
 Ligações químicas Universidade do Estado de Santa Catarina UDESC - CCT Química Geral Profª Fabíola Corrêa viel Ligações Químicas Sacarina Vitamina E Amônia Fulereno LIGAÇÕES IÔNICAS Acontece por atração
Ligações químicas Universidade do Estado de Santa Catarina UDESC - CCT Química Geral Profª Fabíola Corrêa viel Ligações Químicas Sacarina Vitamina E Amônia Fulereno LIGAÇÕES IÔNICAS Acontece por atração
Docking de Ligantes em Proteínas
 110100111010 101101011100 100110011000 1111111100111 101101011010 100111100111 111110011000 110100111100 Docking de Ligantes em Proteínas Prof. Dr. Walter F. de Azevedo Jr. walter.junior@pucrs.br ou walter@azevedolab.net
110100111010 101101011100 100110011000 1111111100111 101101011010 100111100111 111110011000 110100111100 Docking de Ligantes em Proteínas Prof. Dr. Walter F. de Azevedo Jr. walter.junior@pucrs.br ou walter@azevedolab.net
Características AULA 11. Características e Propriedades. drug-like (fármaco-similar) Bioativo-similar
 AULA 11 rganização e gerenciamento de bases de dados padrões Emprego de pré-filtros: otimização das bases de dados (e.g., seleção por tipo de átomo: C,,,, S, F, Cl, Br, P) Características e Propriedades
AULA 11 rganização e gerenciamento de bases de dados padrões Emprego de pré-filtros: otimização das bases de dados (e.g., seleção por tipo de átomo: C,,,, S, F, Cl, Br, P) Características e Propriedades
UNIVERSIDADE FEDERAL DO CEARÁ CENTRO DE CIÊNCIAS DEPARTAMENTO DE QUÍMICA ORGÂNICA E INORGÂNICA PROGRAMA DE PÓS-GRADUAÇÃO EM QUÍMICA
 UNIVERSIDADE FEDERAL DO CEARÁ CENTRO DE CIÊNCIAS DEPARTAMENTO DE QUÍMICA ORGÂNICA E INORGÂNICA PROGRAMA DE PÓS-GRADUAÇÃO EM QUÍMICA SÉRGIO XAVIER BARBOSA ARAÚJO VIRTUAL SCREENING DE POSSÍVEIS INIBIDORES
UNIVERSIDADE FEDERAL DO CEARÁ CENTRO DE CIÊNCIAS DEPARTAMENTO DE QUÍMICA ORGÂNICA E INORGÂNICA PROGRAMA DE PÓS-GRADUAÇÃO EM QUÍMICA SÉRGIO XAVIER BARBOSA ARAÚJO VIRTUAL SCREENING DE POSSÍVEIS INIBIDORES
8/30/2010. Aula 4 Interações fracas. Introdução a Bioquímica: Biomoléculas. Intermolecular Intramolecular
 Introdução a Bioquímica: Biomoléculas Intermolecular Intramolecular Aula 4 Interações fracas Forças Intermoleculares são forças de atração entre moléculas. 41 kj para vaporizar 1 mol of água (inter) Forças
Introdução a Bioquímica: Biomoléculas Intermolecular Intramolecular Aula 4 Interações fracas Forças Intermoleculares são forças de atração entre moléculas. 41 kj para vaporizar 1 mol of água (inter) Forças
Você deve desenhar o aminoácido com o grupo amino protonado (pka > 7) e o grupo carboxílico desprotonado (pka <7).
 QBQ4010 Introdução à Bioquímica Exercícios sobre sistema tampão, aminoácidos e estrutura de proteínas 1. Você estagiário de um laboratório bioquímica, recebeu a função de preparar uma solução tamponada
QBQ4010 Introdução à Bioquímica Exercícios sobre sistema tampão, aminoácidos e estrutura de proteínas 1. Você estagiário de um laboratório bioquímica, recebeu a função de preparar uma solução tamponada
QB70C:// Química (Turmas S71/S72) Ligação Química
 QB70C:// Química (Turmas S71/S72) Ligação Química Prof. Dr. Eduard Westphal (http://paginapessoal.utfpr.edu.br/eduardw) Formação das Ligações O modelo RPECV, baseado principalmente nas estruturas de Lewis,
QB70C:// Química (Turmas S71/S72) Ligação Química Prof. Dr. Eduard Westphal (http://paginapessoal.utfpr.edu.br/eduardw) Formação das Ligações O modelo RPECV, baseado principalmente nas estruturas de Lewis,
Qui. Semana. Allan Rodrigues Xandão (Gabriel Pereira)
 Semana 6 Allan Rodrigues Xandão (Gabriel Pereira) Este conteúdo pertence ao Descomplica. Está vedada a cópia ou a reprodução não autorizada previamente e por escrito. Todos os direitos reservados. CRONOGRAMA
Semana 6 Allan Rodrigues Xandão (Gabriel Pereira) Este conteúdo pertence ao Descomplica. Está vedada a cópia ou a reprodução não autorizada previamente e por escrito. Todos os direitos reservados. CRONOGRAMA
Cinética Química. Prof. Alex Fabiano C. Campos. Rapidez Média das Reações
 Cinética Química Prof. Alex Fabiano C. Campos Rapidez Média das Reações A cinética é o estudo da rapidez com a qual as reações químicas ocorrem. A rapidez de uma reação pode ser determinada pela variação
Cinética Química Prof. Alex Fabiano C. Campos Rapidez Média das Reações A cinética é o estudo da rapidez com a qual as reações químicas ocorrem. A rapidez de uma reação pode ser determinada pela variação
Ligações Químicas Parte - 3
 Ligações Químicas Parte - 3 Lewis: modelo para combinação dos átomos e formação das moléculas Ligação covalente : Baseia-se no compartilhamento de elétrons pelos átomos 1H : 1s 1 1H : 1s 1 1H : 1s 2 1H
Ligações Químicas Parte - 3 Lewis: modelo para combinação dos átomos e formação das moléculas Ligação covalente : Baseia-se no compartilhamento de elétrons pelos átomos 1H : 1s 1 1H : 1s 1 1H : 1s 2 1H
2018 Dr. Walter F. de Azevedo Jr.
 2018 Dr. Walter F. de Azevedo Jr. 000000000000000000000000000000000000000 000000000000000000000000000000000000000 000000000000111111111110001100000000000 000000000001111111111111111111000000001 000000000111111111111111111111111000000
2018 Dr. Walter F. de Azevedo Jr. 000000000000000000000000000000000000000 000000000000000000000000000000000000000 000000000000111111111110001100000000000 000000000001111111111111111111000000001 000000000111111111111111111111111000000
Quiminformática e o planejamento de fármacos
 Quiminformática e o planejamento de fármacos Banco de Dados Otimização do Matriz Análise e Modelagem Estatística Quiminformática Carlos Montanari NEQUIMED/DQFM/IQSC/USP montana@iqsc.usp.br Evolução do
Quiminformática e o planejamento de fármacos Banco de Dados Otimização do Matriz Análise e Modelagem Estatística Quiminformática Carlos Montanari NEQUIMED/DQFM/IQSC/USP montana@iqsc.usp.br Evolução do
LIGAÇÕES INTERATÔMICAS X LIGAÇÕES INTERMOLECULARES
 Prof. Edson Cruz LIGAÇÕES INTERATÔMICAS X LIGAÇÕES INTERMOLECULARES Já estudamos que qualquer tipo de matéria é formado por átomos. Vimos também que cada agrupamento de átomos, combinados em uma determinada
Prof. Edson Cruz LIGAÇÕES INTERATÔMICAS X LIGAÇÕES INTERMOLECULARES Já estudamos que qualquer tipo de matéria é formado por átomos. Vimos também que cada agrupamento de átomos, combinados em uma determinada
Click to edit Master title style
 Moléculas poliatómicas Considere-se a molécula H 2 O As ligações nesta molécula seriam explicadas com base na coalescência de duas orbitais p do átomo de oxigénio com orbitais 1s dos átomos de hidrogénio.
Moléculas poliatómicas Considere-se a molécula H 2 O As ligações nesta molécula seriam explicadas com base na coalescência de duas orbitais p do átomo de oxigénio com orbitais 1s dos átomos de hidrogénio.
A Dualidade Onda-Partícula
 A Dualidade Onda-Partícula O fato de que as ondas têm propriedades de partículas e viceversa se chama Dualidade Onda-Partícula. Todos os objetos (macroscópicos também!) são onda e partícula ao mesmo tempo.
A Dualidade Onda-Partícula O fato de que as ondas têm propriedades de partículas e viceversa se chama Dualidade Onda-Partícula. Todos os objetos (macroscópicos também!) são onda e partícula ao mesmo tempo.
Ligações Químicas. Profª. Ms. Loraine Cristina do Valle Jacobs DAQBI.
 Ligações Químicas Profª. Ms. Loraine Cristina do Valle Jacobs lorainejacobs@utfpr.edu.br http://paginapessoal.utfpr.edu.br/lorainejacobs DAQBI Propriedades das Ligações Químicas TEORIA DA REPULSÃO DOS
Ligações Químicas Profª. Ms. Loraine Cristina do Valle Jacobs lorainejacobs@utfpr.edu.br http://paginapessoal.utfpr.edu.br/lorainejacobs DAQBI Propriedades das Ligações Químicas TEORIA DA REPULSÃO DOS
Propriedades Físicas e Químicas dos Compostos Orgânicos
 Aumenta Propriedades das Moléculas Orgânicas Propriedades Físicas e Químicas dos Compostos Orgânicos A partir das ligações químicas que unem os átomos de suas moléculas, e das forças que agem entre elas
Aumenta Propriedades das Moléculas Orgânicas Propriedades Físicas e Químicas dos Compostos Orgânicos A partir das ligações químicas que unem os átomos de suas moléculas, e das forças que agem entre elas
Teoria da Ligação de Valência. Prof. Jean Marcel R. Gallo
 Teoria da Ligação de Valência Prof. Jean Marcel R. Gallo Os orbitais Os orbitais Teoria do Orbital de Valência É uma das teorias usada para explicar a ligação química. Primeira teoria de ligação a usar
Teoria da Ligação de Valência Prof. Jean Marcel R. Gallo Os orbitais Os orbitais Teoria do Orbital de Valência É uma das teorias usada para explicar a ligação química. Primeira teoria de ligação a usar
A Troposfera. A Troposfera é a camada da atmosfera que está em contacto com a superfície terrestre que contém o ar que respiramos.
 1 A Troposfera A Troposfera é a camada da atmosfera que está em contacto com a superfície terrestre que contém o ar que respiramos. A atmosfera é constituída, maioritariamente, por moléculas. Espécies
1 A Troposfera A Troposfera é a camada da atmosfera que está em contacto com a superfície terrestre que contém o ar que respiramos. A atmosfera é constituída, maioritariamente, por moléculas. Espécies
Interações intermoleculares
 Comparações entre ligações e interações químicas Ligação covalente : Interação intramolecular Ligação de hidrogênio: Interação intermolecular Forças íon-dipolo Existem entre um íon e a carga parcial em
Comparações entre ligações e interações químicas Ligação covalente : Interação intramolecular Ligação de hidrogênio: Interação intermolecular Forças íon-dipolo Existem entre um íon e a carga parcial em
Estrutura de Lewis, Formas de representação e Interações intermoleculares. Aula 2
 Universidade Federal de Ouro Preto Estrutura de Lewis, Formas de representação e Interações intermoleculares Aula 2 Flaviane Francisco ilário 1 1 Estrutura de Lewis Um símbolo de Lewis é um símbolo no
Universidade Federal de Ouro Preto Estrutura de Lewis, Formas de representação e Interações intermoleculares Aula 2 Flaviane Francisco ilário 1 1 Estrutura de Lewis Um símbolo de Lewis é um símbolo no
Química I Exame de 2ª Época, 7/2/2012 Resolução 1. a)
 Química I Exame de 2ª Época, 7/2/2012 Resolução 1. a) b) A intensidade de corrente que passa no circuito (dada pelo nº de electrões que passa por unidade de tempo) depende da intensidade da radiação incidente.
Química I Exame de 2ª Época, 7/2/2012 Resolução 1. a) b) A intensidade de corrente que passa no circuito (dada pelo nº de electrões que passa por unidade de tempo) depende da intensidade da radiação incidente.
Geometria Molecular. A forma geométrica de uma molécula é determinada pelo arranjo dos pares eletrônicos em torno dos átomos.
 Geometria Molecular A forma geométrica de uma molécula é determinada pelo arranjo dos pares eletrônicos em torno dos átomos. O método pelo qual se estabelece a orientação mais estável dos pares eletrônicos
Geometria Molecular A forma geométrica de uma molécula é determinada pelo arranjo dos pares eletrônicos em torno dos átomos. O método pelo qual se estabelece a orientação mais estável dos pares eletrônicos
RESOLUÇÃO DE EXERCÍCIOS PROPOSTOS AULA 02 TURMA INTENSIVA
 RESOLUÇÃO DE EXERCÍCIOS PROPOSTOS AULA 02 TURMA INTENSIVA 01. Item B I Correto. A energia para quebrar a ligação H (568 kj/mol) é a maior da tabela. Isto torna mais difícil a sua quebra, portanto ionizando
RESOLUÇÃO DE EXERCÍCIOS PROPOSTOS AULA 02 TURMA INTENSIVA 01. Item B I Correto. A energia para quebrar a ligação H (568 kj/mol) é a maior da tabela. Isto torna mais difícil a sua quebra, portanto ionizando
Na Atmosfera da Terra: Radiação, Matéria e Estrutura Unidade temática 2
 Sumário Na : Radiação, Matéria e Estrutura Unidade temática 2 Moléculas na Troposfera espécies maioritárias e espécies vestigiais. Ligação covalente. Notação de Lewis. Regra do octeto. Como se estabelece
Sumário Na : Radiação, Matéria e Estrutura Unidade temática 2 Moléculas na Troposfera espécies maioritárias e espécies vestigiais. Ligação covalente. Notação de Lewis. Regra do octeto. Como se estabelece
Biologia Estrutural. Cálculo da Densidade Eletrônica. Prof. Dr. Walter Filgueira de Azevedo Jr. wfdaj.sites.uol.com.br
 Biologia Estrutural Cálculo da Densidade Eletrônica Prof. Dr. Walter Filgueira de Azevedo Jr. Resumo Introdução Cálculo da densidade eletrônica Densidade eletrônica de um cristal unidimensional Densidade
Biologia Estrutural Cálculo da Densidade Eletrônica Prof. Dr. Walter Filgueira de Azevedo Jr. Resumo Introdução Cálculo da densidade eletrônica Densidade eletrônica de um cristal unidimensional Densidade
Biologia Estrutural. Fatores de Estrutura. Prof. Dr. Walter Filgueira de Azevedo Jr. wfdaj.sites.uol.com.br Dr. Walter F. de Azevedo Jr.
 Biologia Estrutural Fatores de Estrutura Prof. Dr. Walter Filgueira de Azevedo Jr. Resumo Fator de Espalhamento Atômico Fator de Estrutura Cálculo Computacional do Fator de Estrutura Arquivos PDB Fator
Biologia Estrutural Fatores de Estrutura Prof. Dr. Walter Filgueira de Azevedo Jr. Resumo Fator de Espalhamento Atômico Fator de Estrutura Cálculo Computacional do Fator de Estrutura Arquivos PDB Fator
nome do arquivo com informações das moléculas outname = ch4_nvt nome dos arquivos de saída init = yes
 SIMULAÇÃO DE METANO (CH 4 ) NO ENSEMBLE NVT 100 molécula, a temperatura de 25 o C e densidade inicial de 0.8 g/cm 3. ch4.txt (contem informações [1] sobre a molécula a ser simulada) * 1 5 na X Y Z OPLS
SIMULAÇÃO DE METANO (CH 4 ) NO ENSEMBLE NVT 100 molécula, a temperatura de 25 o C e densidade inicial de 0.8 g/cm 3. ch4.txt (contem informações [1] sobre a molécula a ser simulada) * 1 5 na X Y Z OPLS
LIGAÇÃO QUÍMICA. Forças que mantêm os átomos unidos e os fazem funcionar como um todo.
 Porque é que os átomos se ligam para formar moléculas? Como é que os átomos se ligam para formar moléculas? Como é que uma molécula ou um ião poliatómico se mantém unido? LIGAÇÃO QUÍMICA LIGAÇÃO QUÍMICA
Porque é que os átomos se ligam para formar moléculas? Como é que os átomos se ligam para formar moléculas? Como é que uma molécula ou um ião poliatómico se mantém unido? LIGAÇÃO QUÍMICA LIGAÇÃO QUÍMICA
QUÍMICA ORGÂNICA I. INTRODUÇÃO À QUÍMICA ORGÂNICA (Parte II)
 QUÍMICA RGÂNICA I INTRDUÇÃ À QUÍMICA RGÂNICA (Parte II) CURS FARMÁCIA SEMESTRE 2016/2 Professoras: Alceni Augusta Werle Tânia Márcia Sacramento Melo 1 1 Ressonância ESTRUTURA DE LEWIS Explica de maneira
QUÍMICA RGÂNICA I INTRDUÇÃ À QUÍMICA RGÂNICA (Parte II) CURS FARMÁCIA SEMESTRE 2016/2 Professoras: Alceni Augusta Werle Tânia Márcia Sacramento Melo 1 1 Ressonância ESTRUTURA DE LEWIS Explica de maneira
Origem grego (protos) primeira, mais importante
 PROTEÍNAS Origem grego (protos) primeira, mais importante A palavra proteína que eu proponho vem derivada de proteos, porque ela parece ser a substância primitiva ou principal da nutrição animal, as plantas
PROTEÍNAS Origem grego (protos) primeira, mais importante A palavra proteína que eu proponho vem derivada de proteos, porque ela parece ser a substância primitiva ou principal da nutrição animal, as plantas
FUNDAÇÃO CARMELITANA MÁRIO PALMÉRIO FACIHUS - FACULDADE DE CIÊNCIAS HUMANAS E SOCIAIS. Enzimas. Prof. Me. Cássio Resende de Morais
 FUNDAÇÃO CARMELITANA MÁRIO PALMÉRIO FACIHUS - FACULDADE DE CIÊNCIAS HUMANAS E SOCIAIS Enzimas Prof. Me. Cássio Resende de Morais Enzimas Catalisadores biológicos: substâncias de origem biológica que aceleram
FUNDAÇÃO CARMELITANA MÁRIO PALMÉRIO FACIHUS - FACULDADE DE CIÊNCIAS HUMANAS E SOCIAIS Enzimas Prof. Me. Cássio Resende de Morais Enzimas Catalisadores biológicos: substâncias de origem biológica que aceleram
LIPOFILIA LIPOFILIA COEFICIENTE DE PARTIÇÃO.
 LIPOFILIA A lipofilia de um composto é comumente estimada usando Log P a partir da partição de um sistema octanol/água aandrico@ifsc.usp.br A lipofilia é uma das propriedades mais importantes relacionadas
LIPOFILIA A lipofilia de um composto é comumente estimada usando Log P a partir da partição de um sistema octanol/água aandrico@ifsc.usp.br A lipofilia é uma das propriedades mais importantes relacionadas
O QUE É UMA PROTEÍNA?
 PROTEÍNAS O QUE É UMA PROTEÍNA? Macromoléculas que agem em processos biológicos como: Mediação por catálise proteica, transporte de outras moléculas, controle de condições intra/extra celulares e transporte
PROTEÍNAS O QUE É UMA PROTEÍNA? Macromoléculas que agem em processos biológicos como: Mediação por catálise proteica, transporte de outras moléculas, controle de condições intra/extra celulares e transporte
Química A MESTRADO INTEGRADO EM ENGENHARIA DO AMBIENTE. 1º Semestre /2014. Doutor João Paulo Noronha.
 MESTRAD INTEGRAD EM ENGENARIA D AMBIENTE 1º Semestre - 2013/2014 Doutor João Paulo Noronha jpnoronha@fct.unl.pt (VII a VIII) UNIVERSIDADE NVA DE LISBA Faculdade de Ciências e Tecnologia Propriedades físicas
MESTRAD INTEGRAD EM ENGENARIA D AMBIENTE 1º Semestre - 2013/2014 Doutor João Paulo Noronha jpnoronha@fct.unl.pt (VII a VIII) UNIVERSIDADE NVA DE LISBA Faculdade de Ciências e Tecnologia Propriedades físicas
Bioinformática Estrutural Aula 1
 Bioinformática Estrutural Aula 1 03 de Junho de 2013 Paula Kuser-Falcão Laboratório de Bioinformática Aplicada Embrapa Informática Agropecuária Paula.kuser-falcao@embrapa.br Pratique Atividade Física Paula
Bioinformática Estrutural Aula 1 03 de Junho de 2013 Paula Kuser-Falcão Laboratório de Bioinformática Aplicada Embrapa Informática Agropecuária Paula.kuser-falcao@embrapa.br Pratique Atividade Física Paula
QUÍMICA. Interações Atômicas: Geometria Molecular, Polaridade da Ligação e da Molécula, Forças Intermoleculares e Número de Oxidação Parte 2
 QUÍMICA Interações Atômicas: Geometria Molecular, Polaridade da Ligação e da Molécula, Forças Intermoleculares e Número de Oxidação Parte 2 Prof a. Giselle Blois Forças ou Ligações Intermoleculares Em
QUÍMICA Interações Atômicas: Geometria Molecular, Polaridade da Ligação e da Molécula, Forças Intermoleculares e Número de Oxidação Parte 2 Prof a. Giselle Blois Forças ou Ligações Intermoleculares Em
Interações Atômicas e Moleculares
 Interações Atômicas e Moleculares 7. Interações Intermoleculares Prof. Pieter Westera pieter.westera@ufabc.edu.br http://professor.ufabc.edu.br/~pieter.westera/iam.html São de natureza eletromagnética.
Interações Atômicas e Moleculares 7. Interações Intermoleculares Prof. Pieter Westera pieter.westera@ufabc.edu.br http://professor.ufabc.edu.br/~pieter.westera/iam.html São de natureza eletromagnética.
