2019 Dr. Walter F. de Azevedo Jr.
|
|
|
- Edite Beltrão
- 5 Há anos
- Visualizações:
Transcrição
1 2019 Dr. Walter F. de Azevedo Jr
2 Interação Proteína-Ligante Consideremos um sistema biológico formado por uma proteína (P) e um ligante (L) em solução. No sistema o ligante é uma pequena molécula que apresenta afinidade pela proteína, por exemplo, o fármaco indinavir, um inibidor a protease do HIV. Tal sistema possibilita a formação de um complexo binário proteína-ligante (PL), com constante de reação indicada por k 1 e constante reversa de reação dada por k -1, ambos descritos na equação reversível abaixo, Ligante Proteína P + L k 1 k -1 PL A reação pode ser caracterizada pela constante de equilíbrio (K eq ). Na prática são usadas as constantes de dissociação (K d ) e inibição (K i ). Formação do complexo proteína (P) com ligante (L), complexo proteína-ligante (PL). A estrutura em destaque é o complexo protease do HIV (P) e o fármaco indinavir (L), usado no tratamento contra a AIDS. 2
3 Interação Proteína-Ligante As interações entre o inibidor de CDK2 (roscovitine mostrado ao lado) e a proteína alvo (CDK2) são não covalentes. Tais contatos intermoleculares apresentam uma interação energética favorável, o que leva o equilíbrio da reação química indicada abaixo para o lado da formação do complexo binário (PL). P + L k 1 k -1 PL A análise da energia da interação proteína-ligante indica a formação de um composto de transição, onde a energia do sistema é elevada e o composto formado instável. Este estado é chamado estado de transição, e a reação caminha para uma menor energia, com a formação do complexo binário PL. Estrutura molecular do inibidor de CDK2 roscovitine. A figura foi gerada com o programa VMD e a opção CPK. As coordenadas foram extraídas do arquivo PDB: 2a4l. 3
4 Energia Interação Proteína-Ligante + Proteína + Ligante Estado de transição Complexo proteína-ligante E a = Energia de ativação para associação E d = Energia de ativação para dissociação E a E = Variação total da energia E d E Coordenada da interação 4
5 Interação Proteína-Ligante Podemos representar a formação do complexo proteína-ligante (PL) a partir dos componentes proteína (P) e ligante(l), como esquematizado abaixo. Temos a proteína (P) e o ligante (L) em solução, no caso do ligante apresentar afinidade pela proteína, o equilíbrio desta reação será favorável à formação do complexo proteína-ligante (PL). P + L PL k 1 k -1 5
6 Interação Proteína-Ligante No caso do complexo binário proteínaligante (PL) apresentar uma energia menor que as moléculas livres (proteína e ligante sem interação), a formação do complexo proteína-ligante ocorrerá de forma espontânea, ou seja, sem a necessidade de fornecimento de energia para que ocorra a formação. Uma forma de estudar a formação do complexo binário é a partir da avaliação da variação de energia livre de Gibbs ( G), definida pela seguinte equação, P PL G = H -T S Onde H é a entalpia do sistema proteína-ligante, T é a temperatura e S é a variação da entropia do sistema. Na figura acima temos a formação espontânea de um complexo proteína-ligante, pois este apresenta energia menor que das moléculas livres (proteína e ligante 6sem interação) L
7 Interação Proteína-Ligante O termo H indica a entalpia do sistema, que representa as forças moleculares envolvidas nas interações proteínaligante. O termo S é a variação da entropia do sistema, que pode ser entendida como a quantidade de energia que não pode ser convertida em trabalho. Na equação anterior, o produto da temperatura absoluta (T) pela variação da entropia ( S), indica que um aumento da entropia favorece a formação do complexo binário. P PL G < 0 Um valor negativo de G indica formação espontânea do complexo. G = H -T S G > 0 Com G > 0 a reação não é favorável à formação do complexo, como na figura inferior. Com G < 0 temos uma reação favorável à formação do complexo, como 7 na figura superior. L
8 Interação Proteína-Ligante O termo entálpico ( H) contribui para variação da energia livre de Gibbs, por meio de interações não-covalentes, resultantes da quebra ou formação destas interações, são elas: 1) Ligações de hidrogênio. Esta ligação envolve um átomo doador (D) e um átomo aceitador (A). 2) Interações eletrostáticas (iônicas e polares). 3) Interações aromáticas. 4) Interações dispersivas (van der Waals). 8
9 Interação Proteína-Ligante O termo entrópico ( S) está relacionado com a variação de graus de liberdades das moléculas presentes no sistema proteína-ligante. Na parte de cima da figura ao lado temos o sítio ativo da enzima (esquerda) e o ligante livre (direita). Temos que lembrar que a interação proteína-ligante não ocorre no vácuo, temos no sistema moléculas de água, que têm participação decisiva na interação intermolecular. As cadeias laterais dos resíduos de aminoácido do sítio ativo estão livres para girarem, o ligante também está livre para girar, se apresentar ligações simples. A proteína e ligante interagem com água. Sítio de ligação solvatado Complexo proteína-ligante solvatado Ligações de hidrogênio Contatos hidrofóbicos Fonte: Caceres RA, Pauli I, Timmers LFS, de Azevedo Jr. WF. Curr. Drug Targets 2008; 9(12):
10 Interação Proteína-Ligante A figura ao lado ilustra a formação do complexo proteína-ligante, a liberação das moléculas de água tende a aumentar a entropia do sistema, o que compensa a perda de entropia devida à formação do complexo. O ligante e a proteína perdem liberdade na estrutura do complexo binário. Sítio de ligação solvatado Complexo proteína-ligante solvatado Ligações de hidrogênio Contatos hidrofóbicos Fonte: Caceres RA, Pauli I, Timmers LFS, de Azevedo Jr. WF. Curr. Drug Targets 2008; 9(12):
11 Interação Proteína-Ligante Quando o sistema proteína-ligante está em equilíbrio, a formação do complexo binário e a dissociação ocorrem, assim a constante de equilíbrio (K eq ) da reação é dada por, K eq k k 1 1 PL P L onde os termos [P], [L], [PL] indicam as concentrações molares da proteína, ligante e complexo proteína-ligante, respectivamente. A partir da análise dimensional da constante de equilíbrio vemos claramente que sua unidade é M -1. Estrutura do complexo entre a proteína quinase dependente de ciclina 2 (Cyclin-Dependent Kinase 2, CDK2) e o inibidor roscovitine. 11
12 Inibidores da Protease do HIV A AIDS surgiu como pandemia há algumas décadas atrás e causou grande temor, pois inicialmente não havia conhecimento da sua causa. O estudo da AIDS registra um dos maiores sucessos da moderna abordagem do desenho de fármacos, usando-se recursos computacionais. A AIDS é causada pelo HIV. Os vírus são formados por uma capa de proteína que envolve seu material genético, no caso do HIV é o RNA. Aqui descreveremos o uso da protease do HIV, como alvo para o desenvolvimento de fármacos contra a AIDS. Concepção artística da pesquisa de novos fármacos contra a AIDS. Imagem disponível em: < Acesso em: 30 de março de
13 Inibidores da Protease do HIV As proteases são enzimas que catalisam a clivagem de outras proteínas, a protease do HIV catalisa tal clivagem. Esta protease realiza uma importante etapa no ciclo da infecção viral. Como em outros vírus, o HIV leva a célula infectada a produzir muitas cópias de suas proteínas. Tais proteínas apresentam inicialmente como um única cadeia polipeptídica (poliproteína), que apresenta várias proteínas coladas numa cadeia. A função da protease do HIV é catalisar a clivagem da poliproteína em unidades menores funcionais. A correta execução de tal clivagem é crítica para o processo de infecção viral. Célula (linfócito T)(em verde) infectada com o HIV (em vermelho). As esferas vermelhas são partículas repletas de HIV, que saem da linfócito T para infectar outras células. Imagem disponível em: Acesso em: 30 de março de
14 Inibidores da Protease do HIV A poliproteína intacta é necessária no início do processo de infecção, quando monta a forma imatura do vírus. Em seguida a poliproteína tem que ser clivada, para formar o vírus maduro, que pode então infectar uma nova célula. As reações de clivagem da poliproteína têm que ser coordenadas perfeitamente, o que permite a montagem do vírus. Devido a tal sensibilidade e seu papel essencial para infecção viral, a protease do HIV é um alvo importante para o desenho de fármacos contra a AIDS. A inibição da protease do HIV impede a maturação do vírus, cessando a progressão da infecção. A estrutura da protease do HIV é dimérica, com duas unidades idênticas. Os inibidores ligam-se na cavidade entre as duas unidades. O inibidor está indicado pela esferas verde s, no meio da estrutura. Imagem disponível em: ftp://resources.rcsb.org/motm/tiff/6- HIV-1Protease-7hvp_activesite.tif Acesso em: 30 de março de
15 Inibidores da Protease do HIV A variação da energia livre de Gibbs da interação intermolecular entre proteína e ligante é dada pela seguinte equação, G G o [PL] RT ln( ) [P][L] onde G o é a variação da energia livre de Gibbs padrão, ou seja, a variação em G que acompanha a formação do complexo no estado padrão de equilíbrio (temperatura de 25 o C, pressão de 100 kpa). Estrutura cristalográfica da protease do HIV em complexo com inibidor. A estrutura destaca as interações intermoleculares entre um fármaco (ligante) e sua proteína alvo. 15
16 Inibidores da Protease do HIV No equilíbrio termodinâmico temos G = 0 (estado estacionário), assim temos, 0 G G o o [PL] RT ln( ) [P][L] [PL] RT ln( ) [P][L] O termo G o é chamado variação na energia livre de Gibbs da ligação ( G binding ), assim temos, [PL] G binding RT ln( ) [P][L] 16
17 Inibidores da Protease do HIV De uma forma geral podemos expressar a afinidade proteína-ligante pela variação da energia livre de Gibbs ( G binding ), quanto menor a energia, maior a afinidade do ligante pela proteína. Esta grandeza física depende das concentrações do complexo proteínaligante [PL], das concentrações da proteína [P] e do ligante [L], bem como da temperatura. O R (R=1,99 cal/mol.k = 8,31 J/mol.K) na equação indica a constante dos gases. G G G binding binding binding [ PL] RT ln( ) [ P][ L] 1 RT ln( K RT ln( K A constante de dissociação (K d ) é dada por: d d ) ) RT ln( K d ) K d [ P][ L] [ PL] Assim temos como calcular a energia livre de uma interação proteína-ligante, a partir da constante de dissociação determinada experimentalmente. 17
18 Inibidores da Protease do HIV Vamos considerar o estudo do desenvolvimento de drogas contra o HIV. Vimos que o principal alvo para o desenho de fármacos contra HIV é a protease do HIV. Os dados sobre a constante de dissociação (K d ) de 4 inibidores da protease estão indicados na tabela abaixo, todos feitos na temperatura de 25 º C. As estruturas são mostradas ao lado. A partir dessa informação calcularemos a variação na energia livre de Gibbs da ligação ( G binding ). Inibidor K d (nm) G binding (kj/mol) Indinavir 1,07 Saquinavir 0,31 Ritonavir 0,6 Nelfinavir 0,0122 Estruturas de alguns inibidores da protease do HIV. A) Indinavir. B) Saquinavir. C) Ritonavir. D) Nelfinavir. 18
19 Inibidores da Protease do HIV Estudo do indinavir. As constantes de dissociação foram determinadas a uma temperatura de 25 º C, para converter para Kelvin é só somar 273, assim temos T= 298 K G R.T. ln(k ) 8, ln 1, ,8236. ln 1,07.10 G G binding binding binding d 2478,8236.(-20,6556) ,6 J/mol - 51,2 kj/mol Inibidor K d (nm) G binding (kj/mol) Indinavir 1,07-51,2 Saquinavir 0,31 Ritonavir 0,6 Nelfinavir 0,0122 Estrutura molecular do fármaco indinavir. A figura foi gerada com o programa VMD e a opção CPK. As coordenadas foram extraídas do arquivo PDB: 1hsg. 19
20 Inibidores da Protease do HIV A partir da análise da tabela abaixo vemos que o inibidor que apresenta menor energia livre de Gibbs de ligação é o nelfinavir, cujo a estrutura do complexo proteína-ligante está mostrada abaixo. Inibidor K d (nm) G binding (kj/mol) Indinavir 1,07-51,2 Saquinavir 0,31-54,3 Ritonavir 0,6-52,6 Nelfinavir 0, ,3 20
21 Fonte da imagem: Consultado em 30/03/2019 Desenho de Fármacos Muitos fármacos são desenvolvidos a partir do uso de simulações computacionais, inclusive baseados nas ideias da evolução darwiniana. Para entender tais estudos, temos que ver a interação do fármaco com uma proteína alvo. Podemos usar a analogia da interação da chave com a fechadura, onde o fármaco é a chave e a proteína alvo é a fechadura. No caso de enzimas a fechadura será o sítio ativo, e o inibidor a chave. Temos que destacar que tal analogia é só para captarmos a essencial da interação, o fenômeno da interação proteína-fármaco é mais complexo, mas para nossos propósitos será suficiente tal comparação. Interação da chave (fármaco) com a fechadura (sítio ativo da enzima) 21
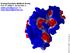
22 Desenho de Fármacos Na figura ao lado temos a representação gráfica da interação de um inibidor (chave) com o sítio ativo de uma enzima (fechadura). A estrutura representada é da CDK2 (cyclin-dependent kinase 2), uma enzima envolvida no controle da progressão do ciclo celular. A inibição dessa enzima leva a célula a parar a progressão do ciclo celular. Tal parada pode levar a célula à apoptose. Este efeito mostrou atividade anticancerígena. Por isso grande esforço tem sido feito no desenvolvimento de inibidores de CDK2. Na figura ao lado temos a superfície molecular da CDK2 interagindo com inibidor (em verde). As cores da superfície molecular da CDK2 representam a carga elétrica parcial, com vermelho indicando concentração 22 de carga negativa, azul positiva e ciano neutro.
23 Docking Molecular A interação de um possível inibidor de uma enzima pode ser obtida por meio de algoritmos evolucionários, num método chamado docking molecular (docagem molecular). Nesta simulação computacional, são tentadas várias posições possíveis para a chave (possível inibidor), mudando-se sua orientação relativa dentro do sítio ativo da enzima. Ao final da simulação temos várias posições possíveis para a chave (possível inibidor) no sítio ativo da enzima (fechadura). O melhor resultado (a melhor posição da chave na fechadura) é escolhido a partir de uma função de ajuste (também chamada de escore). Zoom do sítio ativo da CDK2. Vemos o inibidor (chave) em verde bem acomodado no sítio ativo (fechadura). As cores da superfície molecular da CDK2 representam a carga elétrica parcial, com vermelho indicando concentração de carga negativa, azul positiva e ciano neutro. 23
24 Docking Molecular Toda simulação de docking molecular tem duas etapas. Na primeira etapa são geradas posições para a molécula no sítio ativo da enzima. Numa segunda etapa selecionamos, dentro de um critério (função ajuste), a melhor posição. Na simulação de docking molecular, as posições do inibidor (chave) são geradas aleatoriamente. Depois selecionamos as melhores posições. No passo seguinte operamos o cruzamento entre as melhores posições, gerando as posições filhas. Aplicamos de forma aleatória mutações às posições filhas. Selecionamos os melhores, entre pais e filhas e iniciamos novo ciclo (iteração). Cinco posições possíveis de um inibidor no sítio ativo da CDK2. A melhor posição será selecionada a partir da função ajuste (também chamada função escore). A superfície da CDK2 está colorida para indicar potencial elétrico, com vermelho indicando potencial negativo, azul positivo e branco neutro. 24
25 Docking Molecular A simulação computacional finaliza ao atingirmos um critério de parada, que pode ser o número de repetições da iteração, ou um critério de convergência. As novas posições em um novo ciclo não mostram grande variação com relações às posições do ciclo anterior. A simulação de docking está disponível em diversos programas de computador. E são usadas de forma intensa por laboratórios farmacêuticos para o desenvolvimento de novos fármacos. Um dos maiores sucessos de tal abordagem, foi a descoberta de inibidores da protease do HIV, mostrada ao lado. Tais inibidores são usados no coquetel contra AIDS. Inibidor no sítio ativo da HIV protease. 25
26 Docking Molecular Como toda metodologia, a simulação de docking tem uma etapa de validação e refinamento do protocolo. Tal etapa é que chamamos de redocking, onde achamos o melhor protocolo capaz de recuperar a posição cristalográfica de um ligante acoplado ao sítio ativo de uma enzima. A condição para que possamos validar nosso protocolo de docking é que haja informações sobre pelo menos uma estrutura cristalográfica de um complexo proteína-ligante. Chamamos da posição obtida computacionalmente de pose (em vermelho) e a comparamos com a cristalográfica (em cinza) a partir do RMSD (Root-Mean Squared Deviation) que pode ser pensado como uma régua que mede a distância da posição cristalográfica da pose. Bio-Inspired Algorithms Applied to Molecular Docking Simulations. Heberlé G, De Azevedo WF Jr. Curr Med Chem 2011; 18 (9):
27 Introdução Uma situação comumente encontrada na análise de sistemas biológicos é a pergunta se duas variáveis, ou conjuntos de medidas, estão relacionadas de alguma forma. Por exemplo, podemos dizer que a massa corporal dos seres humanos tem relação com a altura, ou seja, pessoas mais altas apresentam maior massa corporal. Obviamente tal observação não é uma verdade absoluta, mas uma tendência. Esta situação pode ser avaliada a partir do coeficiente de correlação. Há dois tipos principais de coeficientes usados na análise estatística de dados biológicos, os coeficientes de Pearson e Spearman. O coeficiente de Pearson pode ser usado para avaliarmos se há uma relação linear entre duas variáveis. Gráfico de duas variáveis que apresentam correlação, conforme x aumenta a variável y também aumenta. A relação entre as duas variáveis não é linear. 27
28 Introdução O coeficiente de Spearman é usado para verificar se há uma relação monotônica entre duas variáveis. Mas afinal o que é uma relação monotônica? Consideremos o exemplo citado da relação da altura e a massa corporal dos seres humanos. Pessoas mais altas tendem a ter uma massa corporal maior, ou seja, conforme aumentamos a variável altura, esperamos que a variável massa corporal aumente. Todas vez que temos uma relação entre duas variáveis, por exemplo x e y, em que ambas aumentam juntas, temos uma relação monotônica (monotônica crescente), mostrada na figura 1. Podemos ter também uma relação monotônica para duas variáveis, onde quando uma aumenta a outra diminui decrescente. (monotônica decrescente), figura Figura 1. Duas variáveis x e y com relação monotônica crescente. Figura 2. Duas variáveis x e y com relação monotônica
29 Coeficiente de Correlação de Spearman O coeficiente de correlação de Spearman (ρ) apresenta valores que variam no intervalo entre -1 e 1. Valores de correlação próximos a -1 ou a 1, indicam que há correlação entre as duas variáveis, resultados próximos de zero indicam que não há correlação entre as duas variáveis. A principal aplicação da análise do coeficiente de correlação entre duas variáveis, é o teste de hipóteses, ou seja, eu realizo um experimento, ou coleto dados de um sistema biológico, levanto hipóteses e testo a partir da análise estatística. Para nossos propósitos, vamos considerar uma aplicação para o desenvolvimento de fármacos. Figura 1. Duas variáveis x e y com relação monotônica crescente. Figura 2. Duas variáveis x e y com relação monotônica decrescente. 29
30 Previsão de Afinidade de CDKs Vamos considerar a avaliação computacional da afinidade para a proteína quinase dependente de ciclina (CDK). Neste estudo foram realizadas avaliação da afinidade para 20 estruturas cristalográficas da CDK, para os quais havia informação sobre o IC 50. Tal parâmetro indica a constante de inibição à 50 %, ou seja, qual a concentração do inibidor para que a atividade da enzima caia pela metade. Quanto menor o IC 50, mais efetivo é o inibidor. Superfície eletrostática da CDK2 30
31 Previsão de Afinidade de CDKs A tabela abaixo mostra os potenciais de Lennard-Jones (v_lj_12_6), ligação de hidrogênio (v_hb_12_10), eletrostático (v_elec) e de solvatação (v_sol) para 20 moléculas inibidoras de CDK. A última coluna traz o valor experimental de log(ic 50 ). Como o valor de IC 50 pode variar muitas ordens de grandeza, usamos a escala logarítmica. A primeira coluna indica o código PDB (Protein Data Bank) seguida da informação do ligante (Lig, Chain, Number). PDB Lig Chain Number v_lj_12_6 v_hb_12_10 v_elec v_sol Torsions Experimental 1DI8 DTQ A DM2 HMD A E9H INR A FVT 106 A FVV 107 A GII 1PU A H00 FAP A H07 MFP A H0W 207 A H1Q 2A6 A H1R 6CP A e JVP LIG P KE5 LS1 A KE6 LS2 A KE7 LS3 A KE8 LS4 A KE9 LS5 A OGU ST8 A OI9 N20 A OIQ HDU A
32 Previsão de Afinidade de CDKs O coeficiente de Spearman é calculado sobre a posição de cada valor, ou seja, classificamos do menor para o maior valor e substituímos o valor numérico pela sua posição. O coeficiente de Spearman é calculado a partir da seguinte equação: i=1 N 2 d i onde N é número de medidas, d i2 a diferença ao ρ = 1 6 quadrado das posições dos valores das variáveis. N N 2 1 PDB Lig Chain Number v_lj_12_6 v_hb_12_10 v_elec v_sol Torsions Experimental 1DI8 DTQ A DM2 HMD A E9H INR A FVT 106 A FVV 107 A GII 1PU A H00 FAP A H07 MFP A H0W 207 A H1Q 2A6 A H1R 6CP A e JVP LIG P KE5 LS1 A KE6 LS2 A KE7 LS3 A KE8 LS4 A KE9 LS5 A OGU ST8 A OI9 N20 A OIQ HDU A
33 Previsão de Afinidade de CDKs Nossa hipótese a ser testada pelo coeficiente de correlação é que as variáveis dos potenciais e log(ic 50 ) estão relacionados, ou seja, quanto menor o valor do potencial, menor o log(ic 50 ). O tamanho da nossa amostra é N = 20. Usaremos o programa Molegro Data Modeller (MDM) para análise da planilha de dados. PDB Lig Chain Number v_lj_12_6 v_hb_12_10 v_elec v_sol Torsions Experimental 1DI8 DTQ A DM2 HMD A E9H INR A FVT 106 A FVV 107 A GII 1PU A H00 FAP A H07 MFP A H0W 207 A H1Q 2A6 A H1R 6CP A e JVP LIG P KE5 LS1 A KE6 LS2 A KE7 LS3 A KE8 LS4 A KE9 LS5 A OGU ST8 A OI9 N20 A OIQ HDU A
34 Previsão de Afinidade de CDKs A planilha com os dados inseridos no programa MDM estão mostradas abaixo. 34
35 Previsão de Afinidade de CDKs Para calcularmos o coeficiente de correlação de Spearman, clicamos em Modelling- >Bivariate Statatistics... Como mostrado abaixo. 35
36 Previsão de Afinidade de CDKs Temos o novo menu mostrado abaixo. Nesta janela iremos selecionar os eixos para calcularmos a correlação. O eixo x (First column) será a coluna experimental. O eixo y (Second column) será a coluna do potencial calculado (Lennard-Jones e outros) 36
37 Previsão de Afinidade de CDKs Escolhemos os dados experimentais (Experimental). 37
38 Previsão de Afinidade de CDKs Depois escolhemos o potencial de Lennard-Jones (v_lj_12_6) para o eixo y. 38
39 Previsão de Afinidade de CDKs O programa MDM gera o gráfico de dispersão e calcula os coeficientes de correlação, como mostrado abaixo. Podemos ver que o valor do coeficiente de Spearman (ρ) está baixo, por volta de 0,36. Esperamos valores próximos de 1. 39
40 Previsão de Afinidade de CDKs Repetimos o processo para os outros potenciais. Para o potencial de ligação de hidrogênio (v_hb_12_10), houve um aumento da correlação, pouco acima de 0,50. 40
41 Previsão de Afinidade de CDKs O potencial eletrostático tem baixa correlação, ρ = 0,
42 Previsão de Afinidade de CDKs O potencial de solvatação apresenta correlação de 0,39. 42
43 Previsão de Afinidade de CDKs O número de ângulos de torção não é um potencial, mas podemos verificar a correlação com o log(ic 50 ), neste caso é baixa, por volta de 0,18. 43
44 Previsão de Afinidade de CDKs Uma forma de melhorarmos o poder de previsão do modelo computacional é considerarmos uma equação que combina vários potenciais num único modelo. Chamamos este modelo de função escore que pode ser expresso por uma equação. Nesta equação cada potencial tem um peso que aproxima o valor previsto da afinidade (predicted binding affinity) (PBA) do valor experimental (log(ic 50 )) o que aumenta a correlação do PBA com o valor experimental da afinidade. A equação do PBA tem a forma mostrada abaixo. PBA = α 0 α 1 V_LJ_ α 2 V_HB_12=10 + α 3 V_Elec + α 4 V_Sol + α 5 Torsions Os valores de α s são determinados de forma a aproximar o valor previsto (PBA) do valor experimental (log(ic 50 )). São usados métodos de aprendizado de máquina para a determinação dos pesos. 44
45 Previsão de Afinidade de CDKs Podemos usar o programa MDM para gerarmos o modelo (função escore). Para isto clique em Modelling->Multiple Linear Regression... 45
46 Previsão de Afinidade de CDKs Teremos um novo menu onde temos que escolher a variável alvo (Target variable) também chamada de variável dependente, é a variável Experimental. Depois de escolhermos a variável Experimental clicamos no botão Next. 46
47 Previsão de Afinidade de CDKs Agora escolhemos os descritores (descriptors) que são as variáveis independentes que receberão o peso. No caso escolheremos: v_lj_12_6, v_hb_12_10, v_elec, V_Sol e Torsions, como mostrado abaixo. Depois clicamos no botão Next. 47
48 Previsão de Afinidade de CDKs Deixamos o algoritmo como Multiple Linear Regression e depois clicamos no botão Next. 48
49 Previsão de Afinidade de CDKs Deixamos a opção Using CDK_IC50_20_structures (subset: All) as traning set clicada e depois clicamos no botão Start. 49
50 Previsão de Afinidade de CDKs Depois clicamos OK. 50
51 Previsão de Afinidade de CDKs Veja que temos uma nova coluna na planilha, destacada em verde. Esta coluna traz os valores previstos pelo novo modelo. Podemos calcular a correção. 51
52 Previsão de Afinidade de CDKs Clique em Modelling->Bivariate Statistics... 52
53 Previsão de Afinidade de CDKs Escolhemos para First column a coluna Experimenal e no Second column MLR-Train (5D). Como mostrado abaixo. 53
54 Previsão de Afinidade de CDKs Veja que a correlação de Spearman (ρ) subiu significativamente, temos um ρ de 0, Para termos o modelo, clicamos no botão Close. 54
55 Previsão de Afinidade de CDKs Agora no Workspace Explorer temos acesso ao modelo. 55
56 Previsão de Afinidade de CDKs Colocando-se o cursor sobre o modelo, clicamos com o botão da direita do mouse (no Windows) sobre o MLR (50)->Show details..., como mostrado abaixo. 56
57 Previsão de Afinidade de CDKs Na nova janela, clicamos no botão Model. 57
58 Previsão de Afinidade de CDKs Agora temos a equação do novo modelo, veja que cada potencial tem um peso. Os pesos foram determinados de forma a aproximar ao máximo o valor previsto do experimental. 58
59 Virtual Screening in silico in vitro in vivo Moléculas puras Base de dados moleculares Seleção de alvo molecular Simulações de docking Neste fluxograma, a seleção dos resultados do virtual screening é realizada a partir da análise da função score, no caso da CDK citada. Cálculo da afinidade proteína-ligante teórico Testes in vivo Testes in vitro Seleção dos melhores resultados 59
Funções Escores (MolDock Score) Prof. Dr. Walter F. de Azevedo Jr. ou
 205 Dr. Walter F. de zevedo Jr. Funções Escores (MolDock Score) Prof. Dr. Walter F. de zevedo Jr. walter.junior@pucrs.br ou walter.filgueira@hotmail.com Função MolDock Score função ajuste ou escore, usada
205 Dr. Walter F. de zevedo Jr. Funções Escores (MolDock Score) Prof. Dr. Walter F. de zevedo Jr. walter.junior@pucrs.br ou walter.filgueira@hotmail.com Função MolDock Score função ajuste ou escore, usada
2018 Dr. Walter F. de Azevedo Jr. Biofísica. Prof. Dr. Walter F. de Azevedo Jr.
 2018 Dr. Walter F. de Azevedo Jr. Biofísica Prof. Dr. Walter F. de Azevedo Jr. 1 Ao lado temos a estrutura de um peptídeo formado pelos aminoácidos Arginina - Prolina - Alanina - Tirosina - Serina, coloridos
2018 Dr. Walter F. de Azevedo Jr. Biofísica Prof. Dr. Walter F. de Azevedo Jr. 1 Ao lado temos a estrutura de um peptídeo formado pelos aminoácidos Arginina - Prolina - Alanina - Tirosina - Serina, coloridos
Bioquímica. Prof. Dr. Walter F. de Azevedo Jr Dr. Walter F. de Azevedo Jr.
 2018 Dr. Walter F. de Azevedo Jr. Bioquímica Prof. Dr. Walter F. de Azevedo Jr. E-mail: walter@azevedolab.net 1 As proteínas que tiveram sua estrutura tridimensional determinadas estão disponíveis no site
2018 Dr. Walter F. de Azevedo Jr. Bioquímica Prof. Dr. Walter F. de Azevedo Jr. E-mail: walter@azevedolab.net 1 As proteínas que tiveram sua estrutura tridimensional determinadas estão disponíveis no site
Gabriela Sehnem Heck 1,2, Prof. Dr. Walter F. de Azevedo Jr 2. (orientador)
 Pontifícia Universidade Católica do Rio Grande do Sul Gabriela Sehnem Heck 1,2, Prof. Dr. Walter F. de Azevedo Jr 2. (orientador) 1 Acadêmica de Curso de Ciêncas Biológicas da PUCRS, 2 Laboratório de Bioquímica
Pontifícia Universidade Católica do Rio Grande do Sul Gabriela Sehnem Heck 1,2, Prof. Dr. Walter F. de Azevedo Jr 2. (orientador) 1 Acadêmica de Curso de Ciêncas Biológicas da PUCRS, 2 Laboratório de Bioquímica
Aplicação de Docking Molecular para o Estudo de Inibidores da Protease do HIV-1
 2015 Dr. Walter F. de Azevedo Jr. Aplicação de Docking Molecular para o Estudo de Inibidores da Protease do HIV-1 Prof. Dr. Walter F. de Azevedo Jr. walter.junior@pucrs.br ou walter.filgueira@hotmail.com
2015 Dr. Walter F. de Azevedo Jr. Aplicação de Docking Molecular para o Estudo de Inibidores da Protease do HIV-1 Prof. Dr. Walter F. de Azevedo Jr. walter.junior@pucrs.br ou walter.filgueira@hotmail.com
Biofísica. Métodos Teóricos em Biofísica Algoritmos Evolucionários. Prof. Dr. Walter F. de Azevedo Jr Dr. Walter F. de Azevedo Jr.
 2015 Dr. Walter F. de Azevedo Jr. Biofísica Métodos Teóricos em Biofísica Algoritmos Evolucionários Prof. Dr. Walter F. de Azevedo Jr. 110100111010 101101011100 100110011000 1111111100111 101101011010
2015 Dr. Walter F. de Azevedo Jr. Biofísica Métodos Teóricos em Biofísica Algoritmos Evolucionários Prof. Dr. Walter F. de Azevedo Jr. 110100111010 101101011100 100110011000 1111111100111 101101011010
2018 Dr. Walter F. de Azevedo Jr.
 2018 Dr. Walter F. de Azevedo Jr. 000000000000000000000000000000000000000 000000000000000000000000000000000000000 000000000000111111111110001100000000000 000000000001111111111111111111000000001 000000000111111111111111111111111000000
2018 Dr. Walter F. de Azevedo Jr. 000000000000000000000000000000000000000 000000000000000000000000000000000000000 000000000000111111111110001100000000000 000000000001111111111111111111000000001 000000000111111111111111111111111000000
Docking (ou atracamento) Molecular. Profa. Dra. Rafaela Ferreira Dept. de Bioquímica e Imunologia 9 de outubro de 2014
 Docking (ou atracamento) Molecular Profa. Dra. Rafaela Ferreira Dept. de Bioquímica e Imunologia rafaelasf@gmail.com 9 de outubro de 2014 O ATRACAMENTO MOLECULAR PERMITE ESTIMAR COMO DUAS MOLÉCULAS INTERAGEM
Docking (ou atracamento) Molecular Profa. Dra. Rafaela Ferreira Dept. de Bioquímica e Imunologia rafaelasf@gmail.com 9 de outubro de 2014 O ATRACAMENTO MOLECULAR PERMITE ESTIMAR COMO DUAS MOLÉCULAS INTERAGEM
Interações de van der Waals. Interações de van der Waals. Interações de van der Waals.
 Interações de van der Waals aandrico@if.sc.usp.br Também conhecidas como forças de dispersão de London, caracterizam-se pelo momento de dipolo induzido e ocorrem entre todos os átomos. Apesar de moléculas
Interações de van der Waals aandrico@if.sc.usp.br Também conhecidas como forças de dispersão de London, caracterizam-se pelo momento de dipolo induzido e ocorrem entre todos os átomos. Apesar de moléculas
Visualização de Hélices
 Grupo: Curso: Turma: Data: Visualização de Hélices Objetivos Visualizar a estrutura tridimensional de hélices presentes na estrutura de proteínas e peptídeos, usandose recursos computacionais. Analisar
Grupo: Curso: Turma: Data: Visualização de Hélices Objetivos Visualizar a estrutura tridimensional de hélices presentes na estrutura de proteínas e peptídeos, usandose recursos computacionais. Analisar
Mecanismo de Inibição de Enzimas Preparação de Micro e Macromoléculas Docagem Molecular Exemplo de Aplicação
 bjetivos FFI0776 Modelagem e Engenharia de Proteínas Mecanismo de Inibição de Enzimas Preparação de Micro e Macromoléculas M l Docagem Molecular Exemplo de Aplicação Prof. Rafael V. C. Guido rvcguido@ifsc.usp.br
bjetivos FFI0776 Modelagem e Engenharia de Proteínas Mecanismo de Inibição de Enzimas Preparação de Micro e Macromoléculas M l Docagem Molecular Exemplo de Aplicação Prof. Rafael V. C. Guido rvcguido@ifsc.usp.br
Scoring Functions (MolDock Score) Prof. Dr. Walter F. de Azevedo Jr. walter.junior@pucrs.br ou walter.filgueira@hotmail.com
 2015 Dr. Walter F. de Azevedo Jr. Scoring Functions (MolDock Score) Prof. Dr. Walter F. de Azevedo Jr. walter.junior@pucrs.br ou walter.filgueira@hotmail.com 1 O que é desenho de fármacos baseado em computador?
2015 Dr. Walter F. de Azevedo Jr. Scoring Functions (MolDock Score) Prof. Dr. Walter F. de Azevedo Jr. walter.junior@pucrs.br ou walter.filgueira@hotmail.com 1 O que é desenho de fármacos baseado em computador?
FUNDAÇÃO CARMELITANA MÁRIO PALMÉRIO FACIHUS - FACULDADE DE CIÊNCIAS HUMANAS E SOCIAIS. Enzimas. Prof. Me. Cássio Resende de Morais
 FUNDAÇÃO CARMELITANA MÁRIO PALMÉRIO FACIHUS - FACULDADE DE CIÊNCIAS HUMANAS E SOCIAIS Enzimas Prof. Me. Cássio Resende de Morais Enzimas Catalisadores biológicos: substâncias de origem biológica que aceleram
FUNDAÇÃO CARMELITANA MÁRIO PALMÉRIO FACIHUS - FACULDADE DE CIÊNCIAS HUMANAS E SOCIAIS Enzimas Prof. Me. Cássio Resende de Morais Enzimas Catalisadores biológicos: substâncias de origem biológica que aceleram
2016 Dr. Walter F. de Azevedo Jr.
 2016 Dr. Walter F. de Azevedo Jr. 000000000000000000000000000000000000000 000000000000000000000000000000000000000 000000000000111111111110001100000000000 000000000001111111111111111111000000001 000000000111111111111111111111111000000
2016 Dr. Walter F. de Azevedo Jr. 000000000000000000000000000000000000000 000000000000000000000000000000000000000 000000000000111111111110001100000000000 000000000001111111111111111111000000001 000000000111111111111111111111111000000
Estrutura e Função de Proteínas O NMR tem a vantagem de puder apresentar mais do que uma estrura possivel
 Vamos trabalhar com a neurominidase. A m-glutinina liga o virs à celula. A neurominidasez o contrario e permite que o virus de despegue dessa celula e consegue ir infetar mais virus. O centro ativo mantem
Vamos trabalhar com a neurominidase. A m-glutinina liga o virs à celula. A neurominidasez o contrario e permite que o virus de despegue dessa celula e consegue ir infetar mais virus. O centro ativo mantem
Aula: 28 Temática: Efeito da Temperatura na Velocidade de Reação
 Aula: 28 Temática: Efeito da Temperatura na Velocidade de Reação Em grande parte das reações, as constantes de velocidade aumentam com o aumento da temperatura. Vamos analisar esta dependência. A teoria
Aula: 28 Temática: Efeito da Temperatura na Velocidade de Reação Em grande parte das reações, as constantes de velocidade aumentam com o aumento da temperatura. Vamos analisar esta dependência. A teoria
AULA 7. Cálculo das energias de interação estereoquímicas e eletrostáticas entre as moléculas do conjunto de
 Análise Comparativa dos Campos Moleculares AULA 7 Análise Comparativa dos Campos Moleculares A contribuição devido às forças de dispersão entre moléculas é descrita pelo potencial de Lennard-Jones e as
Análise Comparativa dos Campos Moleculares AULA 7 Análise Comparativa dos Campos Moleculares A contribuição devido às forças de dispersão entre moléculas é descrita pelo potencial de Lennard-Jones e as
OBJETIVOS BIBLIOGRAFIA ENZIMAS E INIBIDORES ENZIMÁTICOS
 OBJETIVOS Enzimas: Funções, Nomenclatura e Propriedades Fundamentos da Cinética Enzimática Cinética Enzimática: Michaelis-Menten Ensaios Cinéticos: Padronização e Validação Parâmetros Cinéticos: vo, KM,
OBJETIVOS Enzimas: Funções, Nomenclatura e Propriedades Fundamentos da Cinética Enzimática Cinética Enzimática: Michaelis-Menten Ensaios Cinéticos: Padronização e Validação Parâmetros Cinéticos: vo, KM,
Fundamentos da modelagem Molecular - 1
 Fundamentos da modelagem Molecular - 1 Departamento de Física UFPel ufpellogo Introdução Rotas para a pesquisa Conexão entre experimento, simulação e teoria Sistema real Fazer modelos do sistema Fazer
Fundamentos da modelagem Molecular - 1 Departamento de Física UFPel ufpellogo Introdução Rotas para a pesquisa Conexão entre experimento, simulação e teoria Sistema real Fazer modelos do sistema Fazer
04/10/2011 FORÇAS DE INTERAÇÃO MOLECULAR ENTRE LIGANTES E PROTEÍNAS. O que é um medicamento?
 FORÇAS DE INTERAÇÃO MOLECULAR ENTRE LIGANTES E PROTEÍNAS O que é um medicamento? 1 O que é um medicamento? Medicamento (produto final) Excipientes + Principio ativo ( fármaco, droga ) Como atua uma droga?
FORÇAS DE INTERAÇÃO MOLECULAR ENTRE LIGANTES E PROTEÍNAS O que é um medicamento? 1 O que é um medicamento? Medicamento (produto final) Excipientes + Principio ativo ( fármaco, droga ) Como atua uma droga?
Aula de Bioquímica I. Tema: Água. Prof. Dr. Júlio César Borges
 Aula de Bioquímica I Tema: Água Prof. Dr. Júlio César Borges Depto. de Química e Física Molecular DQFM Instituto de Química de São Carlos IQSC Universidade de São Paulo USP E-mail: borgesjc@iqsc.usp.br
Aula de Bioquímica I Tema: Água Prof. Dr. Júlio César Borges Depto. de Química e Física Molecular DQFM Instituto de Química de São Carlos IQSC Universidade de São Paulo USP E-mail: borgesjc@iqsc.usp.br
Cinética Enzimática. Prof Karine P. Naidek Novembro/2016
 UNIVERSIDADE DO ESTADO DE SANTA CATARINA CENTRO DE CIÊNCIAS TECNOLÓGICAS DEPARTAMENTO DE QUÍMICA DQMC BIOQUÍMICA BIO0001 Cinética Enzimática Prof Karine P. Naidek Novembro/2016 Cinética das Reações Bioquímicas
UNIVERSIDADE DO ESTADO DE SANTA CATARINA CENTRO DE CIÊNCIAS TECNOLÓGICAS DEPARTAMENTO DE QUÍMICA DQMC BIOQUÍMICA BIO0001 Cinética Enzimática Prof Karine P. Naidek Novembro/2016 Cinética das Reações Bioquímicas
Aula de Bioquímica I. Tema: Enzimas. Prof. Dr. Júlio César Borges
 Aula de Bioquímica I Tema: Enzimas Prof. Dr. Júlio César Borges Depto. de Química e Física Molecular DQFM Instituto de Química de São Carlos IQSC Universidade de São Paulo USP E-mail: borgesjc@iqsc.usp.br
Aula de Bioquímica I Tema: Enzimas Prof. Dr. Júlio César Borges Depto. de Química e Física Molecular DQFM Instituto de Química de São Carlos IQSC Universidade de São Paulo USP E-mail: borgesjc@iqsc.usp.br
TERMODINÂMICA QUÍMICA
 TERMODINÂMICA QUÍMICA Processos Espontâneos 1ª Lei da termodinâmica: Energia de um sistema é conservada ΔE = variação da energia interna q = calor absorvido pelo sistema w = trabalho realizado pela vizinhança
TERMODINÂMICA QUÍMICA Processos Espontâneos 1ª Lei da termodinâmica: Energia de um sistema é conservada ΔE = variação da energia interna q = calor absorvido pelo sistema w = trabalho realizado pela vizinhança
Métodos de Química Computacional
 Universidade Federal de Ouro Preto Instituto de Ciências Exatas e Biológicas Departamento de Química Métodos de Química Computacional Professora: Melissa Soares Caetano Dinâmica molecular Conhecendo posições
Universidade Federal de Ouro Preto Instituto de Ciências Exatas e Biológicas Departamento de Química Métodos de Química Computacional Professora: Melissa Soares Caetano Dinâmica molecular Conhecendo posições
Docking de Ligantes em Proteínas
 110100111010 101101011100 100110011000 1111111100111 101101011010 100111100111 111110011000 110100111100 Docking de Ligantes em Proteínas Prof. Dr. Walter F. de Azevedo Jr. walter.junior@pucrs.br ou walter@azevedolab.net
110100111010 101101011100 100110011000 1111111100111 101101011010 100111100111 111110011000 110100111100 Docking de Ligantes em Proteínas Prof. Dr. Walter F. de Azevedo Jr. walter.junior@pucrs.br ou walter@azevedolab.net
Métodos de Química Computacional
 Universidade Federal de Ouro Preto Instituto de Ciências Exatas e Biológicas Departamento de Química Métodos de Química Computacional Professora: Melissa Soares Caetano Química Medicinal Matemática Química
Universidade Federal de Ouro Preto Instituto de Ciências Exatas e Biológicas Departamento de Química Métodos de Química Computacional Professora: Melissa Soares Caetano Química Medicinal Matemática Química
Forças intermoleculares. Formação de fases condensadas Forças íon-dipolo Forças dipolo-dipolo Forças de London Ligação de hidrogênio
 Forças intermoleculares Formação de fases condensadas Forças íon-dipolo Forças dipolo-dipolo Forças de London Ligação de hidrogênio Estado gasoso O estado da matéria mais simples é o gás. Descrevemos esse
Forças intermoleculares Formação de fases condensadas Forças íon-dipolo Forças dipolo-dipolo Forças de London Ligação de hidrogênio Estado gasoso O estado da matéria mais simples é o gás. Descrevemos esse
CINÉTICA QUÍMICA. Profa. Loraine Jacobs DAQBI.
 CINÉTICA QUÍMICA Profa. Loraine Jacobs DAQBI lorainejacobs@utfpr.edu.br http://paginapessoal.utfpr.edu.br/lorainejacobs Reações de Segunda Ordem Exercício 05: Entre os possíveis destinos do NO 2 na química
CINÉTICA QUÍMICA Profa. Loraine Jacobs DAQBI lorainejacobs@utfpr.edu.br http://paginapessoal.utfpr.edu.br/lorainejacobs Reações de Segunda Ordem Exercício 05: Entre os possíveis destinos do NO 2 na química
Fisica do Corpo Humano ( ) Prof. Adriano Mesquita Alencar Dep. Física Geral Instituto de Física da USP B01. Temperatura Aula 5 e 1/2 da 6
 Fisica do Corpo Humano (4300325) Prof. Adriano Mesquita Alencar Dep. Física Geral Instituto de Física da USP B01 Temperatura Aula 5 e 1/2 da 6 1. Existem em torno de uma centena de átomos 2. Cada átomo
Fisica do Corpo Humano (4300325) Prof. Adriano Mesquita Alencar Dep. Física Geral Instituto de Física da USP B01 Temperatura Aula 5 e 1/2 da 6 1. Existem em torno de uma centena de átomos 2. Cada átomo
Função proteica envolve a ligação reversível com outras moléculas O ligante se liga na proteína e um sítio específico, chamado SÍTIO DE LIGAÇÃO.
 Estrutura Função 1. Função proteica envolve a ligação reversível com outras moléculas. A molécula que se liga reversivelmente à proteina LIGANTE 2. O ligante se liga na proteína e um sítio específico,
Estrutura Função 1. Função proteica envolve a ligação reversível com outras moléculas. A molécula que se liga reversivelmente à proteina LIGANTE 2. O ligante se liga na proteína e um sítio específico,
2018 Dr. Walter F. de Azevedo Jr.
 2018 Dr. Walter F. de Azevedo Jr. 000000000000111111111110001100000000000 000000000001111111111111111111000000001 000000000111111111111111111111111000000 000000000111111111111111111111111000000 000000000011111111111111111111100000000
2018 Dr. Walter F. de Azevedo Jr. 000000000000111111111110001100000000000 000000000001111111111111111111000000001 000000000111111111111111111111111000000 000000000111111111111111111111111000000 000000000011111111111111111111100000000
Fundamentos da modelagem Molecular - 1
 Fundamentos da modelagem Molecular - 1 Departamento de Física UFPel Introdução Rotas para a pesquisa Conexão entre experimento, simulação e teoria Sistema real Fazer modelos do sistema Fazer experimentos
Fundamentos da modelagem Molecular - 1 Departamento de Física UFPel Introdução Rotas para a pesquisa Conexão entre experimento, simulação e teoria Sistema real Fazer modelos do sistema Fazer experimentos
Evolução Diferencial e Sua Aplicação em Docking do Sistema Proteína-Ligante Prof. Dr. Walter F. de Azevedo Jr.
 2017 Dr. Walter F. de Azevedo Jr. 110100111010 101101011100 100110011000 1111111100111 101101011010 100111100111 111110011000 110100111100 Evolução Diferencial e Sua Aplicação em Docking do Sistema Proteína-Ligante
2017 Dr. Walter F. de Azevedo Jr. 110100111010 101101011100 100110011000 1111111100111 101101011010 100111100111 111110011000 110100111100 Evolução Diferencial e Sua Aplicação em Docking do Sistema Proteína-Ligante
OBJETIVOS INTERAÇÕES INTERMOLECULARES INTERAÇÕES INTERMOLECULARES.
 OBJETIVOS aandrico@if.sc.usp.br Interações Intermoleculares Mecanismo de Ação Modo de Ligação Complexos Recetor-Ligante Exemplos e Exercícios INTERAÇÕES INTERMOLECULARES INTERAÇÕES INTERMOLECULARES O processo
OBJETIVOS aandrico@if.sc.usp.br Interações Intermoleculares Mecanismo de Ação Modo de Ligação Complexos Recetor-Ligante Exemplos e Exercícios INTERAÇÕES INTERMOLECULARES INTERAÇÕES INTERMOLECULARES O processo
2.1 Princípios gerais da ligação química. Ligações químicas
 2.1 Princípios gerais da ligação química Ligações químicas A associação de átomos formando moléculas, ou em agregados de maiores dimensões como, por exemplo, nos metais, é possível pelo estabelecimento
2.1 Princípios gerais da ligação química Ligações químicas A associação de átomos formando moléculas, ou em agregados de maiores dimensões como, por exemplo, nos metais, é possível pelo estabelecimento
Universidade Federal do Ceará Centro de Ciências Departamento de Bioquímica e Biologia Molecular Disciplina de Biofísica.
 Universidade Federal do Ceará Centro de Ciências Departamento de Bioquímica e Biologia Molecular Disciplina de Biofísica Termodinâmica 1. Introdução 2. Conceitos Importantes em Termodinâmica 3. Leis da
Universidade Federal do Ceará Centro de Ciências Departamento de Bioquímica e Biologia Molecular Disciplina de Biofísica Termodinâmica 1. Introdução 2. Conceitos Importantes em Termodinâmica 3. Leis da
Aula 14 Equilíbrio de Fases: Substâncias Puras
 Aula 14 Equilíbrio de Fases: Substâncias Puras 1. A condição de estabilidade Inicialmente precisamos estabelecer a importância da energia de Gibbs molar na discussão das transições de fase. A energia de
Aula 14 Equilíbrio de Fases: Substâncias Puras 1. A condição de estabilidade Inicialmente precisamos estabelecer a importância da energia de Gibbs molar na discussão das transições de fase. A energia de
A Dualidade Onda-Partícula
 A Dualidade Onda-Partícula O fato de que as ondas têm propriedades de partículas e viceversa se chama Dualidade Onda-Partícula. Todos os objetos (macroscópicos também!) são onda e partícula ao mesmo tempo.
A Dualidade Onda-Partícula O fato de que as ondas têm propriedades de partículas e viceversa se chama Dualidade Onda-Partícula. Todos os objetos (macroscópicos também!) são onda e partícula ao mesmo tempo.
Aula de Bioquímica Avançada. Tema: Enzimas. Prof. Dr. Júlio César Borges
 Aula de Bioquímica Avançada Tema: Enzimas Prof. Dr. Júlio César Borges Depto. de Química e Física Molecular DQFM Instituto de Química de São Carlos IQSC Universidade de São Paulo USP E-mail: borgesjc@iqsc.usp.br
Aula de Bioquímica Avançada Tema: Enzimas Prof. Dr. Júlio César Borges Depto. de Química e Física Molecular DQFM Instituto de Química de São Carlos IQSC Universidade de São Paulo USP E-mail: borgesjc@iqsc.usp.br
OBJETIVOS AULA 10 DESENVOLVIMENTO DESCOBERTA. ¾ Otimização de Moléculas Bioativas. ¾ Integração com o processo de HTS
 AULA 10 2 DESCOBERTA DESENVOLVIMENTO Pesquisa Básica (Pré-clínica) (Fases Clínicas I IV) Identificação do alvo molecular Triagens clínicas conduzidas em humanos: avaliação da segurança, impacto no estado
AULA 10 2 DESCOBERTA DESENVOLVIMENTO Pesquisa Básica (Pré-clínica) (Fases Clínicas I IV) Identificação do alvo molecular Triagens clínicas conduzidas em humanos: avaliação da segurança, impacto no estado
Termodinâmica de Sistemas Biológicos
 Aula de Bioquímica I Tema: Termodinâmica de Sistemas Biológicos Prof. Dr. Júlio César Borges Depto. de Química e Física Molecular DQFM Instituto de Química de São Carlos IQSC Universidade de São Paulo
Aula de Bioquímica I Tema: Termodinâmica de Sistemas Biológicos Prof. Dr. Júlio César Borges Depto. de Química e Física Molecular DQFM Instituto de Química de São Carlos IQSC Universidade de São Paulo
2015 Dr. Walter F. de Azevedo Jr.
 2015 Dr. Walter F. de Azevedo Jr. 110100111010 101101011100 100110011000 1111111100111 101101011010 100111100111 111110011000 110100111100 Evolução Diferencial e Sua Aplicação em Docking do Sistema Proteína-Ligante
2015 Dr. Walter F. de Azevedo Jr. 110100111010 101101011100 100110011000 1111111100111 101101011010 100111100111 111110011000 110100111100 Evolução Diferencial e Sua Aplicação em Docking do Sistema Proteína-Ligante
Triagem Virtual em Larga Escala. Profa. Dra. Rafaela Ferreira Dept. de Bioquímica e Imunologia 10 de outubro de 2014
 Triagem Virtual em Larga Escala Profa. Dra. Rafaela Ferreira Dept. de Bioquímica e Imunologia rafaelasf@gmail.com 10 de outubro de 2014 1 O que é triagem virtual? Grande conjunto de compostos Estratégia
Triagem Virtual em Larga Escala Profa. Dra. Rafaela Ferreira Dept. de Bioquímica e Imunologia rafaelasf@gmail.com 10 de outubro de 2014 1 O que é triagem virtual? Grande conjunto de compostos Estratégia
Tutorial Visualização Gráfica com Pymol
 Sobre o Pymol Tutorial Visualização Gráfica com Pymol Aula Prática 21/06/2018 PyMOL é um software de visualização molecular criado por Warren Lyford DeLano. É um programa open source, lançado sob a licença
Sobre o Pymol Tutorial Visualização Gráfica com Pymol Aula Prática 21/06/2018 PyMOL é um software de visualização molecular criado por Warren Lyford DeLano. É um programa open source, lançado sob a licença
Termodinâmica em Bioquímica
 Instituto de Química de São Carlos IQSC Universidade de São Paulo Termodinâmica em Bioquímica Disciplina: Bioquímica I Docente: Profa. Dra. Fernanda Canduri Sugestão de leitura: Cap. 1 do Lehninger A vida
Instituto de Química de São Carlos IQSC Universidade de São Paulo Termodinâmica em Bioquímica Disciplina: Bioquímica I Docente: Profa. Dra. Fernanda Canduri Sugestão de leitura: Cap. 1 do Lehninger A vida
Físico-Química I. Profa. Dra. Carla Dalmolin. Misturas Simples. Termodinâmica das Misturas Propriedades das Soluções Atividade
 Físico-Química I Profa. Dra. Carla Dalmolin Misturas Simples Termodinâmica das Misturas Propriedades das Soluções Atividade Misturas Simples Misturas de substâncias que não reagem Modelo simples para posteriormente
Físico-Química I Profa. Dra. Carla Dalmolin Misturas Simples Termodinâmica das Misturas Propriedades das Soluções Atividade Misturas Simples Misturas de substâncias que não reagem Modelo simples para posteriormente
CINÉTICA QUÍMICA. Profa. Loraine Jacobs DAQBI.
 CINÉTICA QUÍMICA Profa. Loraine Jacobs DAQBI lorainejacobs@utfpr.edu.br http://paginapessoal.utfpr.edu.br/lorainejacobs Cinética Química Estudo da velocidade das reações químicas. REAGENTES PRODUTOS Termodinâmica
CINÉTICA QUÍMICA Profa. Loraine Jacobs DAQBI lorainejacobs@utfpr.edu.br http://paginapessoal.utfpr.edu.br/lorainejacobs Cinética Química Estudo da velocidade das reações químicas. REAGENTES PRODUTOS Termodinâmica
Macromoléculas Biológicas
 Macromoléculas Biológicas Objetivos Visualizar a estrutura tridimensional de peptídeos e proteínas, usando-se recursos computacionais. Montar modelos estruturais de proteínas, usando-se kit de plástico.
Macromoléculas Biológicas Objetivos Visualizar a estrutura tridimensional de peptídeos e proteínas, usando-se recursos computacionais. Montar modelos estruturais de proteínas, usando-se kit de plástico.
L. G. Oliveira, S. P. Nascimento, T. D. Romão, J. A. Cavalcante, N. A. Costa
 APLICATIVO EXCEL/VBA PARA ESTIMATIVAS DE COEFICIENTES DE ATIVIDADES A DILUIÇÃO INFINITA ATRAVÉS DO MODELO MOSCED E VERIFICAÇÃO DE AZEOTROPIA DE MISTURAS BINÁRIAS L. G. Oliveira, S. P. Nascimento, T. D.
APLICATIVO EXCEL/VBA PARA ESTIMATIVAS DE COEFICIENTES DE ATIVIDADES A DILUIÇÃO INFINITA ATRAVÉS DO MODELO MOSCED E VERIFICAÇÃO DE AZEOTROPIA DE MISTURAS BINÁRIAS L. G. Oliveira, S. P. Nascimento, T. D.
As letras ASHLC correspondem a Actions, Show, Hide, Label e Color, respectivamente.
 Tutorial Visualização Gráfica com Pymol Profa. Rafaela Salgado Ferreira Departamento de Bioquímica e Imunologia - Universidade Federal de Minas Gerais Objetivos do tutorial: Preparação de uma figura, análise
Tutorial Visualização Gráfica com Pymol Profa. Rafaela Salgado Ferreira Departamento de Bioquímica e Imunologia - Universidade Federal de Minas Gerais Objetivos do tutorial: Preparação de uma figura, análise
Métodos de Química Computacional
 Universidade Federal de Ouro Preto Instituto de Ciências Exatas e Biológicas Departamento de Química Métodos de Química Computacional Professora: Melissa Soares Caetano Mecânica Molecular Mecânica Molecular
Universidade Federal de Ouro Preto Instituto de Ciências Exatas e Biológicas Departamento de Química Métodos de Química Computacional Professora: Melissa Soares Caetano Mecânica Molecular Mecânica Molecular
QUI109 QUÍMICA GERAL (Ciências Biológicas) 8ª aula /
 QUI109 QUÍMICA GERAL (Ciências Biológicas) 8ª aula / 2016-2 Prof. Mauricio X. Coutrim (disponível em: http://professor.ufop.br/mcoutrim) A LIGAÇÃO COVALENTE É BASEADA NO COMPARTILHAMENTO DE ELÉTRONS Ex.
QUI109 QUÍMICA GERAL (Ciências Biológicas) 8ª aula / 2016-2 Prof. Mauricio X. Coutrim (disponível em: http://professor.ufop.br/mcoutrim) A LIGAÇÃO COVALENTE É BASEADA NO COMPARTILHAMENTO DE ELÉTRONS Ex.
Capítulo by Pearson Education
 QUÍMICA A Ciência Central 9ª Edição Termodinâmica química David P. White Processos espontâneos A termodinâmica está relacionada com a pergunta: uma reação pode ocorrer? A primeira lei de termodinâmica:
QUÍMICA A Ciência Central 9ª Edição Termodinâmica química David P. White Processos espontâneos A termodinâmica está relacionada com a pergunta: uma reação pode ocorrer? A primeira lei de termodinâmica:
Entropia. Energia de Gibbs e Equilíbrio
 Entropia Energia de Gibbs e Equilíbrio Mestrado integrado em Engenharia Biológica Disciplina Química II, 2º semestre 2009/10 Professsora Ana Margarida Martins TRANSFORMAÇÕES ESPONTÂNEAS Uma reacção diz-se
Entropia Energia de Gibbs e Equilíbrio Mestrado integrado em Engenharia Biológica Disciplina Química II, 2º semestre 2009/10 Professsora Ana Margarida Martins TRANSFORMAÇÕES ESPONTÂNEAS Uma reacção diz-se
Aula 2 - Introdução à Bioquímica
 Aula 2 - Introdução à Bioquímica -Interações intermoleculares: interação eletrostática, interações de van der Waals -Solubilidade de compostos polares em água -Solubilidade de compostos apolares em água
Aula 2 - Introdução à Bioquímica -Interações intermoleculares: interação eletrostática, interações de van der Waals -Solubilidade de compostos polares em água -Solubilidade de compostos apolares em água
Profª Eleonora Slide de aula. Revisão: Termodinâmica e Bioenergética
 Revisão: Termodinâmica e Bioenergética Revisão:Termodinâmica As leis da termodinâmica governam o comportamento de s bioquímicos As leis da termodinâmica são princípios gerais aplicados a todos os processos
Revisão: Termodinâmica e Bioenergética Revisão:Termodinâmica As leis da termodinâmica governam o comportamento de s bioquímicos As leis da termodinâmica são princípios gerais aplicados a todos os processos
Cinética Química. a temperatura na qual a reação ocorre e a presença de um catalisador.
 Cinética Química Cinética é o estudo da velocidade com a qual as reações químicas ocorrem. Existem quatro fatores importantes que afetam as velocidades das reações: o estado físico do reagente, as concentrações
Cinética Química Cinética é o estudo da velocidade com a qual as reações químicas ocorrem. Existem quatro fatores importantes que afetam as velocidades das reações: o estado físico do reagente, as concentrações
CINÉTICA QUÍMICA. Profa. Loraine Jacobs DAQBI.
 CINÉTICA QUÍMICA Profa. Loraine Jacobs DAQBI lorainejacobs@utfpr.edu.br http://paginapessoal.utfpr.edu.br/lorainejacobs Cinética Química Lei de Velocidade Integrada Mostra a variação das concentrações
CINÉTICA QUÍMICA Profa. Loraine Jacobs DAQBI lorainejacobs@utfpr.edu.br http://paginapessoal.utfpr.edu.br/lorainejacobs Cinética Química Lei de Velocidade Integrada Mostra a variação das concentrações
CQ110 : Princípios de FQ
 CQ 110 Princípios de Físico Química Curso: Farmácia 1º semestre de 2011 Quartas / Quintas: 9:30 11:30 Prof. Dr. Marcio Vidotti www.quimica.ufpr.br/mvidotti mvidotti@ufpr.br criação de modelos CQ110 : Princípios
CQ 110 Princípios de Físico Química Curso: Farmácia 1º semestre de 2011 Quartas / Quintas: 9:30 11:30 Prof. Dr. Marcio Vidotti www.quimica.ufpr.br/mvidotti mvidotti@ufpr.br criação de modelos CQ110 : Princípios
Opte então pela opção Suplementos disponível do lado esquerdo da tela que se abriu.
 2.4 - Solução computacional utilizando ferramenta solver do MS Excel O problema envolvendo a produção de CPUs é um problema de maximização: queremos o máximo lucro. Vamos aproveitá-lo mais uma vez como
2.4 - Solução computacional utilizando ferramenta solver do MS Excel O problema envolvendo a produção de CPUs é um problema de maximização: queremos o máximo lucro. Vamos aproveitá-lo mais uma vez como
Universidade Tecnológica Federal do Paraná Departamento Acadêmico de Química e Biologia. Aula Interlúdio molecular
 Universidade Tecnológica Federal do Paraná Departamento Acadêmico de Química e Biologia Aula Interlúdio molecular Energia Interna somatório de energias (cinética + potencial) Energia cinética relacionada
Universidade Tecnológica Federal do Paraná Departamento Acadêmico de Química e Biologia Aula Interlúdio molecular Energia Interna somatório de energias (cinética + potencial) Energia cinética relacionada
QSAR 3D Planejamento de NCEs QSAR 3D. O processo de reconhecimento molecular é um processo tridimensional. Conformação bioativa.
 QSAR 3D Interação Fármaco-Receptor AULA 5 a maioria dos casos, a ação dos fármacos está ligada a interação fármaco-receptor, receptor, através de uma associação do tipo reversível e não- covalente Agonistas
QSAR 3D Interação Fármaco-Receptor AULA 5 a maioria dos casos, a ação dos fármacos está ligada a interação fármaco-receptor, receptor, através de uma associação do tipo reversível e não- covalente Agonistas
Soluções para Conjunto de Problemas 1
 Soluções para 7.012 Conjunto de Problemas 1 Pergunta 1 a) Quais são os quatro principais tipos de moléculas biológicas discutidas na palestra? Cite uma função importante para cada tipo de molécula biológica
Soluções para 7.012 Conjunto de Problemas 1 Pergunta 1 a) Quais são os quatro principais tipos de moléculas biológicas discutidas na palestra? Cite uma função importante para cada tipo de molécula biológica
REAÇÕES ESPECÍFICAS PARA aa
 REAÇÕES ESPECÍFICAS PARA aa Organismo Desaminação não sintetiza Transaminação Descarboxilação TRANSAMINAÇÃO Ác.glutâmico Ceto-ácido ALANINA-piruvato ÁC. ASPÁRTICO- oxalacetato OBJETIVO : CENTRALIZAR TODOS
REAÇÕES ESPECÍFICAS PARA aa Organismo Desaminação não sintetiza Transaminação Descarboxilação TRANSAMINAÇÃO Ác.glutâmico Ceto-ácido ALANINA-piruvato ÁC. ASPÁRTICO- oxalacetato OBJETIVO : CENTRALIZAR TODOS
Aula 05 INTRODUÇÃO. Prezado(a) Aluno(a),
 Aula 05 Computação para Engenharia Civil INTRODUÇÃO Prezado(a) Aluno(a), Na aula de hoje, iremos tratar de uma questão muito importante e que poderá ser muito útil ao longo de sua vida profissional: encontrar
Aula 05 Computação para Engenharia Civil INTRODUÇÃO Prezado(a) Aluno(a), Na aula de hoje, iremos tratar de uma questão muito importante e que poderá ser muito útil ao longo de sua vida profissional: encontrar
Segunda Lei da Termodinâmica
 Físico-Química I Profa. Dra. Carla Dalmolin Segunda Lei da Termodinâmica Espontaneidade das reações químicas Entropia Terceira Lei da Termodinâmica Primeira Lei da Termodinâmica Estabelece que as transformações
Físico-Química I Profa. Dra. Carla Dalmolin Segunda Lei da Termodinâmica Espontaneidade das reações químicas Entropia Terceira Lei da Termodinâmica Primeira Lei da Termodinâmica Estabelece que as transformações
Estatística Aplicada ao Serviço Social
 Estatística Aplicada ao Serviço Social Módulo 7: Correlação e Regressão Linear Simples Introdução Coeficientes de Correlação entre duas Variáveis Coeficiente de Correlação Linear Introdução. Regressão
Estatística Aplicada ao Serviço Social Módulo 7: Correlação e Regressão Linear Simples Introdução Coeficientes de Correlação entre duas Variáveis Coeficiente de Correlação Linear Introdução. Regressão
Notas de aula - Profa. Zélia Aulas 07,08,09 e 10. Capítulo 3 Relações formais e sistemas amostrais (exemplares)
 Notas de aula - Profa. Zélia Aulas 07,08,09 e 10 Livro texto: Thermodynamics and an Introduction to Thermostatistics (2nd edition) H. B. Callen. Capítulo 3 Relações formais e sistemas amostrais (exemplares)
Notas de aula - Profa. Zélia Aulas 07,08,09 e 10 Livro texto: Thermodynamics and an Introduction to Thermostatistics (2nd edition) H. B. Callen. Capítulo 3 Relações formais e sistemas amostrais (exemplares)
ATRACAMENTO MOLECULAR
 UNIVERSIDADE FEDERAL DO ESPÍRITO SANTO CENTRO DE CIÊNCIAS DA SAÚDE PROGRAMA DE PÓS-GRADUAÇÃO EM BIOTECNOLOGIA BIOINFORMÁTICA ATRACAMENTO MOLECULAR VITÓRIA 2018 Ivana Alece Arantes Moreno Júlia de Assis
UNIVERSIDADE FEDERAL DO ESPÍRITO SANTO CENTRO DE CIÊNCIAS DA SAÚDE PROGRAMA DE PÓS-GRADUAÇÃO EM BIOTECNOLOGIA BIOINFORMÁTICA ATRACAMENTO MOLECULAR VITÓRIA 2018 Ivana Alece Arantes Moreno Júlia de Assis
ADSORÇÃO. Adsorção é a fixação de uma substância em uma interface, seja ela sólido-gás, sólido-líquido, líquido-gás (vapor).
 ADSORÇÃO Adsorção é a fixação de uma substância em uma interface, seja ela sólido-gás, sólido-líquido, líquido-gás (vapor). Adsorção física não ocorre ligação química entre substrato e adsorbato. O adsorbato
ADSORÇÃO Adsorção é a fixação de uma substância em uma interface, seja ela sólido-gás, sólido-líquido, líquido-gás (vapor). Adsorção física não ocorre ligação química entre substrato e adsorbato. O adsorbato
Universidade de São Paulo Instituto de Física Energia em Sistemas Biológicos Edi Carlos Sousa
 Universidade de São Paulo Instituto de Física Energia em Sistemas Biológicos Edi Carlos Sousa edisousa@if.usp.br Metabolismo Celular Cada reação que ocorre em um organismo vivo requer o uso de energia
Universidade de São Paulo Instituto de Física Energia em Sistemas Biológicos Edi Carlos Sousa edisousa@if.usp.br Metabolismo Celular Cada reação que ocorre em um organismo vivo requer o uso de energia
AULA 5 Adsorção, isotermas e filmes monomoleculares. Prof a Elenice Schons
 AULA 5 Adsorção, isotermas e filmes monomoleculares Prof a Elenice Schons ADSORÇÃO É um processo de acumulação e concentração seletiva de um ou mais constituintes contidos num gás ou líquido sobre superfícies
AULA 5 Adsorção, isotermas e filmes monomoleculares Prof a Elenice Schons ADSORÇÃO É um processo de acumulação e concentração seletiva de um ou mais constituintes contidos num gás ou líquido sobre superfícies
INTRODUÇÃO Á BIOQUÍMICA
 INTRODUÇÃO Á BIOQUÍMICA BIOQUÍMICA ENFERMAGEM FIO Faculdades Integradas de Ourinhos. Prof. Esp. Roberto Venerando Fundação Educacional Miguel Mofarrej. FIO robertovenerando@fio.edu.br 1 - Introdução à
INTRODUÇÃO Á BIOQUÍMICA BIOQUÍMICA ENFERMAGEM FIO Faculdades Integradas de Ourinhos. Prof. Esp. Roberto Venerando Fundação Educacional Miguel Mofarrej. FIO robertovenerando@fio.edu.br 1 - Introdução à
CEDERJ - CENTRO DE EDUCAÇÃO SUPERIOR A DISTÂNCIA DO ESTADO DO RIO DE JANEIRO
 CEDERJ - CENTRO DE EDUCAÇÃO SUPERIOR A DISTÂNCIA DO ESTADO DO RIO DE JANEIRO MATERIAL DIDÁTICO IMPRESSO CURSO: Física DISCIPLINA: Informática para o Ensino de Física CONTEUDISTA: Carlos Eduardo Aguiar
CEDERJ - CENTRO DE EDUCAÇÃO SUPERIOR A DISTÂNCIA DO ESTADO DO RIO DE JANEIRO MATERIAL DIDÁTICO IMPRESSO CURSO: Física DISCIPLINA: Informática para o Ensino de Física CONTEUDISTA: Carlos Eduardo Aguiar
QUI109 QUÍMICA GERAL (Ciências Biológicas) 9ª aula /
 QUI109 QUÍMICA GERAL (Ciências Biológicas) 9ª aula / 2016-2 Prof. Mauricio X. Coutrim (disponível em: http://professor.ufop.br/mcoutrim) LIGAÇÃO COVALENTE HIBRIDIZAÇÃO DE ORBITAIS sp 2 Orientação dos orbitais
QUI109 QUÍMICA GERAL (Ciências Biológicas) 9ª aula / 2016-2 Prof. Mauricio X. Coutrim (disponível em: http://professor.ufop.br/mcoutrim) LIGAÇÃO COVALENTE HIBRIDIZAÇÃO DE ORBITAIS sp 2 Orientação dos orbitais
Inibidores Enzimáticos
 Inibidores Enzimáticos BIOQUÍMICA - INIBIÇÃO ENZIMÁTICA NOÇÕES BÁSICAS SOBRE ENZIMAS CONCEITO As enzimas são proteínas especializadas na catálise de reações biológicas MECANISMO DE AÇÃO As enzimas aceleram
Inibidores Enzimáticos BIOQUÍMICA - INIBIÇÃO ENZIMÁTICA NOÇÕES BÁSICAS SOBRE ENZIMAS CONCEITO As enzimas são proteínas especializadas na catálise de reações biológicas MECANISMO DE AÇÃO As enzimas aceleram
Primeira Lista de Exercícios de Biofísica II. c n
 Primeira Lista de Exercícios de Biofísica II 1. Considere uma célula composta por um corpo celular ao qual está preso um longo e fino processo tubular (por exemplo, o axônio de um neurônio ou o flagelo
Primeira Lista de Exercícios de Biofísica II 1. Considere uma célula composta por um corpo celular ao qual está preso um longo e fino processo tubular (por exemplo, o axônio de um neurônio ou o flagelo
CATÁLISE ENZIMÁTICA. CINÉTICA Controle da velocidade de reações. CINÉTICA Equilíbrio e Estado Estacionário
 CATÁLISE ENZIMÁTICA Equilíbrio e Estado Estacionário P U T F M A Velocidade: período inicial Tempo As medidas de velocidade inicial (v 0 ) são obtidas com a variação da concentração de S btenção de várias
CATÁLISE ENZIMÁTICA Equilíbrio e Estado Estacionário P U T F M A Velocidade: período inicial Tempo As medidas de velocidade inicial (v 0 ) são obtidas com a variação da concentração de S btenção de várias
Dois exercícios desta lista entrarão na primeira prova teórica
 Dois exercícios desta lista entrarão na primeira prova teórica 01. Quais são os três estados físicos da matéria? Quais as diferenças entre eles do ponto de vista de movimentação de moléculas e interações
Dois exercícios desta lista entrarão na primeira prova teórica 01. Quais são os três estados físicos da matéria? Quais as diferenças entre eles do ponto de vista de movimentação de moléculas e interações
QUÍMICA GERAL EXPERIMENTAL 2º SEMESTRE DE 2016 EXPERIMENTO VIRTUAL DE CINÉTICA QUÍMICA
 QUÍMICA GERAL EXPERIMENTAL 2º SEMESTRE DE 2016 EXPERIMENTO VIRTUAL DE CINÉTICA QUÍMICA Acesse a página da Internet http://salvadorhurtado.wikispaces.com/file/view/cinequim.swf e siga as orientações para
QUÍMICA GERAL EXPERIMENTAL 2º SEMESTRE DE 2016 EXPERIMENTO VIRTUAL DE CINÉTICA QUÍMICA Acesse a página da Internet http://salvadorhurtado.wikispaces.com/file/view/cinequim.swf e siga as orientações para
NOTAS DE AULAS DE ESTRUTURA DA MATÉRIA
 NOTAS DE AULAS DE ESTRUTURA DA MATÉRIA Prof. Carlos R. A. Lima CAPÍTULO 11 MOLÉCULAS Primeira Edição junho de 2005 CAPÍTULO 11 MOLÉCULAS ÍNDICE 11-1- Introdução 11.2- Ligação por Tunelamento e a Molécula
NOTAS DE AULAS DE ESTRUTURA DA MATÉRIA Prof. Carlos R. A. Lima CAPÍTULO 11 MOLÉCULAS Primeira Edição junho de 2005 CAPÍTULO 11 MOLÉCULAS ÍNDICE 11-1- Introdução 11.2- Ligação por Tunelamento e a Molécula
CQ049 FQ Eletroquímica
 CQ049 FQ Eletroquímica prof. Dr. Marcio Vidotti LEAP Laboratório de Eletroquímica e Polímeros mvidotti@ufpr.br www.quimica.ufpr.br/mvidotti criação de modelos Modelos são criados para explicar os fenômenos
CQ049 FQ Eletroquímica prof. Dr. Marcio Vidotti LEAP Laboratório de Eletroquímica e Polímeros mvidotti@ufpr.br www.quimica.ufpr.br/mvidotti criação de modelos Modelos são criados para explicar os fenômenos
Bioinformática. Uma Viagem Pelas Moléculas da Vida
 Bioinformática. Uma Viagem Pelas Moléculas da Vida Prof. Dr. Walter F. de Azevedo Jr. azevedolab.net Representação CPK Diversos programas gráficos permitem a visualização molecular, como a representada
Bioinformática. Uma Viagem Pelas Moléculas da Vida Prof. Dr. Walter F. de Azevedo Jr. azevedolab.net Representação CPK Diversos programas gráficos permitem a visualização molecular, como a representada
FORÇAS INTERMOLECULARES QUÍMICA GERAL
 FORÇAS INTERMOLECULARES QUÍMICA GERAL FORÇAS INTERMOLECULARES As forças intermoleculares, forças que existem entre as moléculas, contribuem em grande parte com as propriedades físicas apresentadas pelos
FORÇAS INTERMOLECULARES QUÍMICA GERAL FORÇAS INTERMOLECULARES As forças intermoleculares, forças que existem entre as moléculas, contribuem em grande parte com as propriedades físicas apresentadas pelos
Algumas considerações da aula anterior e Interações moleculares
 Algumas considerações da aula anterior e Interações moleculares From: lbe et al, at Rev Drug Discovery 2:132, 2003 Effect of route of administration on plasma-concentration-response relationships based
Algumas considerações da aula anterior e Interações moleculares From: lbe et al, at Rev Drug Discovery 2:132, 2003 Effect of route of administration on plasma-concentration-response relationships based
Estudo da velocidade da reação enzimática e como ela se altera em função de diferentes parâmetros
 Estudo da velocidade da reação enzimática e como ela se altera em função de diferentes parâmetros Importante abordagem para o entendimento do mecanismo de ação de uma enzima. Vários fatores afetam a atividade
Estudo da velocidade da reação enzimática e como ela se altera em função de diferentes parâmetros Importante abordagem para o entendimento do mecanismo de ação de uma enzima. Vários fatores afetam a atividade
TEORIA DO ESTADO ESTACIONÁRIO E SIMULAÇÃO COMPUTACIONAL.
 EXERCÍCIOS SOBRE: TEORIA DA COLISÃO, TEORIA DO ESTADO ESTACIONÁRIO E SIMULAÇÃO COMPUTACIONAL. Glauber Silva Godoi META Desenvolver no aluno a capacidade de extrair os parâmetros cinéticos de uma reação
EXERCÍCIOS SOBRE: TEORIA DA COLISÃO, TEORIA DO ESTADO ESTACIONÁRIO E SIMULAÇÃO COMPUTACIONAL. Glauber Silva Godoi META Desenvolver no aluno a capacidade de extrair os parâmetros cinéticos de uma reação
Guilherme Menegon Arantes. Instituto de Química Universidade de São Paulo
 Termodinâmica metabólica Guilherme Menegon Arantes Instituto de Química Universidade de São Paulo garantes@iq.usp.br http://gaznevada.iq.usp.br Universidade de São Paulo Termodinâmica ultra -básica Condições
Termodinâmica metabólica Guilherme Menegon Arantes Instituto de Química Universidade de São Paulo garantes@iq.usp.br http://gaznevada.iq.usp.br Universidade de São Paulo Termodinâmica ultra -básica Condições
2 Metodologia. Simulação de defeitos pontuais em um sólido de Lennard-Jones
 Simulação de defeitos pontuais em um sólido de Lennard-Jones SILVA JÚNIOR, Domingos Lopes da 1 ; COSTA, Lucas Modesto da 1 ; RABELO, José Nicodemos Teixeira 2 ; CÂNDIDO, Ladir 3 1 Introdução Apesar de
Simulação de defeitos pontuais em um sólido de Lennard-Jones SILVA JÚNIOR, Domingos Lopes da 1 ; COSTA, Lucas Modesto da 1 ; RABELO, José Nicodemos Teixeira 2 ; CÂNDIDO, Ladir 3 1 Introdução Apesar de
1 semestre de 2014 Gabarito Lista de exercícios 3 - Estatística Descritiva III C A S A
 Exercício 1. (1,0 ponto). A tabela a seguir mostra o aproveitamento conjunto em Física e Matemática para os alunos do ensino médio de uma escola. Notas Notas Notas Física/Matemática Altas Regulares Baixas
Exercício 1. (1,0 ponto). A tabela a seguir mostra o aproveitamento conjunto em Física e Matemática para os alunos do ensino médio de uma escola. Notas Notas Notas Física/Matemática Altas Regulares Baixas
Físico-Química I. Profa. Dra. Carla Dalmolin. Misturas Simples. Físico-Química, cap. 5: Transformações Físicas de Substâncias Puras
 Físico-Química I Profa. Dra. Carla Dalmolin Misturas Simples Físico-Química, cap. 5: Transformações Físicas de Substâncias Puras Misturas Simples Misturas de substâncias que não reagem Modelo simples para
Físico-Química I Profa. Dra. Carla Dalmolin Misturas Simples Físico-Química, cap. 5: Transformações Físicas de Substâncias Puras Misturas Simples Misturas de substâncias que não reagem Modelo simples para
Idade da mãe, em anos completos, no dia do parto. Numérico * A ausência de preenchimento identifica a não obtenção do valor no momento da coleta
 Plano de Trabalho 1)Tabelas, variáveis e significados 2) Transformar peso em gramas em 5 faixas. Método: Função SE 3) Montar a Tabela Univariada do peso em 5 faixas. Método Subtotais 4) Montar a tabela
Plano de Trabalho 1)Tabelas, variáveis e significados 2) Transformar peso em gramas em 5 faixas. Método: Função SE 3) Montar a Tabela Univariada do peso em 5 faixas. Método Subtotais 4) Montar a tabela
Aula 2 Lei de Coulomb
 Aula Lei de Coulomb Introdução Vimos na aula anterior que corpos carregados com carga sofrem interação mutua podendo ser atraídos ou repelidos entre si. Nessa aula e na próxima trataremos esses corpos
Aula Lei de Coulomb Introdução Vimos na aula anterior que corpos carregados com carga sofrem interação mutua podendo ser atraídos ou repelidos entre si. Nessa aula e na próxima trataremos esses corpos
FÍSICA E QUÍMICA A 10º ANO
 DEPARTAMENTO DE MATEMÁTICA E CIÊNCIAS EXPERIMENTAIS Planificação Anual FÍSICA E QUÍMICA A 10º ANO 2017 / 2018 1º Período - QUÍMICA Apresentação e divulgação de critérios 2 1. ELEMENTOS QUÍMICOS E SUA ORGANIZAÇÃO
DEPARTAMENTO DE MATEMÁTICA E CIÊNCIAS EXPERIMENTAIS Planificação Anual FÍSICA E QUÍMICA A 10º ANO 2017 / 2018 1º Período - QUÍMICA Apresentação e divulgação de critérios 2 1. ELEMENTOS QUÍMICOS E SUA ORGANIZAÇÃO
