Universidade de Brasília
|
|
|
- Benedicta Lombardi Bardini
- 8 Há anos
- Visualizações:
Transcrição
1 Universidade de Brasília Instituto de Ciências Exatas Departamento de Ciência da Computação SOM-PORTRAIT: um método para identificar RNA não codificador utilizando Mapas Auto Organizáveis Tulio Conrado Campos da Silva Monografia apresentada como requisito parcial para conclusão do Bacharelado em Ciência da Computação Orientador Prof. Dr. Pedro de Azevedo Berger Coorientadora Prof.ª Dr.ª Maria Emília M. Telles Walter Brasília 2009
2 Universidade de Brasília UnB Instituto de Ciências Exatas Departamento de Ciência da Computação Bacharelado em Ciência da Computação Coordenadora: Prof.ª Dr.ª Carla Maria Chagas e Cavalcante Koike Banca examinadora composta por: Prof. Dr. Pedro de Azevedo Berger (Orientador) CIC/UnB Prof.ª Dr.ª Maria Emília M. Telles Walter (Coorientadora) CIC/UnB Prof.ª Dr.ª Aletéia P. Favacho de Araújo CIC/UnB Prof. Dr. Roberto Coiti Togawa Embrapa Rec. Genéticos e Biotecnologia CIP Catalogação Internacional na Publicação da Silva, Tulio Conrado Campos. SOM-PORTRAIT: um método para identificar RNA não codificador utilizando Mapas Auto Organizáveis / Tulio Conrado Campos da Silva. Brasília : UnB, p. : il. ; 29,5 cm. Monografia (Graduação) Universidade de Brasília, Brasília, ncrna, 2. Bioinformática, 3. SOM, 4. redes de Kohonen, 5. Mapas Auto Organizáveis, 6. Computação Distribuída, 7. Paracoccidioides brasiliensis, 8. EELA-2 CDU Endereço: Universidade de Brasília Campus Universitário Darcy Ribeiro Asa Norte CEP Brasília DF Brasil
3 Universidade de Brasília Instituto de Ciências Exatas Departamento de Ciência da Computação SOM-PORTRAIT: um método para identificar RNA não codificador utilizando Mapas Auto Organizáveis Tulio Conrado Campos da Silva Monografia apresentada como requisito parcial para conclusão do Bacharelado em Ciência da Computação Prof. Dr. Pedro de Azevedo Berger (Orientador) CIC/UnB Prof.ª Dr.ª Maria Emília M. Telles Walter CIC/UnB Prof.ª Dr.ª Aletéia P. Favacho de Araújo CIC/UnB Prof. Dr. Roberto Coiti Togawa Embrapa Rec. Genéticos e Biotecnologia Prof.ª Dr.ª Carla Maria Chagas e Cavalcante Koike Coordenadora do Bacharelado em Ciência da Computação Brasília, 03 de julho de 2009
4 Agradecimentos Agradecimentos especiais aos professores Pedro Berger e Maria Emília, pela atenção, paciência e muita disposição em ensinar. De forma geral, a todos os professores que me auxiliaram durante estes nove semestres de graduação, exemplos valiosos de pessoas trabalhadoras e esforçadas. Agradeço aos meus amigos próximos e aos meus amigos de outras cidades, de outros países, que fiz na UnB. Muito mais do que colegas: amigos para toda hora. Um agradecimento especial à equipe do projeto EELA-2 pela disponibilidade e dedicação em auxiliar-nos e assessorar-nos. Agradeço também à minha família, pela paciência, pelos inúmeros bom-dia, durma bem, fique com Deus : esse amor incondicional me fez chegar onde estou, tenho absoluta certeza. Finalmente, agradeço a Deus, pela possibilidade de, através do meu trabalho, louvar a sua criação. iv
5 Resumo Os recentes esforços para descoberta e inferência da função dos RNAs não codificadores nos organismos concentram-se na seleção de critérios relevantes para a identificação e posterior classificação de tais sequências. A solução deste novo problema da biologia molecular por mapas auto organizáveis de Kohonen é uma alternativa inovadora, ao adicionar a possibilidade de categorização de sequências em mais do que duas classes. Também é uma alternativa viável e eficiente do ponto de vista computacional, por suas características de aprendizado não supervisionado de conjuntos de critérios variáveis para classificação de sequências, o que reduz os riscos associados ao treinamento do classificador. Neste contexto, o método SOM-PORTRAIT foi proposto, implementado e utilizado em um conjunto de assembled ESTs do transcriptoma do fungo P. brasiliensis, e os resultados analisados indicaram uma boa acurácia do método. Em conjunto com o uso da infraestrutura do projeto EELA-2, uma implementação distribuída do método também foi proposta, e alcançou melhorias expressivas no tempo de execução para grande volume de dados de entrada. Palavras-chave: ncrna, Bioinformática, SOM, redes de Kohonen, Mapas Auto Organizáveis, Computação Distribuída, Paracoccidioides brasiliensis, EELA-2 v
6 Abstract The recent efforts in non coding RNAs discovery and inference of its function in the organism concentrates in the selection of the relevant criteria for the classification of such sequences. The solution of this new molecular biology problem by Kohonen self-organized maps is seen as an innovative alternative, for its possibility of multiple classes categorization of sequences. Also, it is a viable and efficient alternative, for its characteristic of unsupervised learning of sets of variable criteria for sequence classification, which in turn reduces the classificator training associated risks. In this context, the SOM-PORTRAIT method was proposed, implemented and applied to a set containing assembled ESTs of the P. brasiliensis fungus transcriptome, and the analized results indicated a good accuracy for the method. Together with the use of EELA-2 s project infrastructure, a distributed implementation of the method was also proposed, and obtained expressive improvement in execution time for large input data. Keywords: ncrna, Bioinformatics, SOM, Kohonen networks, Self Organized Maps, Grid Computing, Paracoccidioides brasiliensis, EELA-2 vi
7 Dedicatória Em memória do meu avô Arthur Osvaldo de Campos e seu infindável gênio criativo. vii
8 Sumário 1 Introdução 1 2 Biologia Molecular, Bioinformática e o Projeto Genoma Pb Biologia Molecular Proteínas Ácidos nucléicos Dogma Central da Biologia Molecular Técnicas de cópia e replicação de sequências em Biologia Molecular Bioinformática Algoritmos em Bioinformática Programas e bancos de dados utilizados na Biologia Molecular Projeto do Genoma Funcional do fungo Paracoccidioides brasiliensis Morfologia e Genética Paracoccidiodomicose O projeto ncrnas no P. brasiliensis RNAs não codificadores Definição Classificação de ncrnas Abordagens experimentais para identificação de ncrnas Avaliação termodinâmica Avaliação composicional Avaliação utilizando aprendizado de máquina Avaliação comparativa viii
9 4 Mapas Auto Organizáveis Redes Neurais Artificiais Inteligência Representação do Conhecimento Redes Neurais Artificiais e Inteligência Artificial Arquitetura de Redes Neurais Processos de Aprendizado Aprendizado Competitivo Perceptrons Auto-Organização Mapas Auto Organizáveis Processamento Distribuído e Projeto EELA Processamento Distribuído Classificação de Flynn Desafios do processamento distribuído Arquiteturas Distribuídas Middleware glite Elementos do glite Fluxo de uma tarefa Projeto EELA Materiais e métodos O método SOM-PORTRAIT Ambiente de trabalho utilizado Construção do conjunto de treinamento Descrição do método O método Dist-SOM-PORTRAIT Descrição do método Ambiente operacional do grid Estratégia de distribuição de processamento Resultados e Discussões Estudo de caso com o fungo Paracoccidioides brasiliensis ix
10 7.1.1 Construção do conjunto de testes Configuração do PORTRAIT Testes comparativos: SOM-PORTRAIT PORTRAIT Configuração do MDC Testes comparativos: PORTRAIT MDC Testes comparativos: SOM-PORTRAIT MDC Dist-SOM-PORTRAIT: gridificação do método Dados de teste Teste comparativo de tempo de execução Conclusões Perspectivas Futuras A Exemplo de um arquivo JDL 80 I Artigo de descrição do método SOM-PORTRAIT (BSB 2009) 81 Referências Bibliográficas 95 x
11 Lista de Figuras 1.1 Um esquema para o Dogma Central da Biologia Grupos formadores de aminoácidos Ligação peptídica e orientações φ e ψ do carbono Cα Pentose principal do nucleotídeo formador do DNA: a desoxirribose Bases nitrogenadas que compõem um nucleotídeo de molécula DNA Estrutura do DNA e suas partes integrantes Pentose principal do nucleotídeo formador do RNA: a ribose Uracila - base pirimidina que compõe um nucleotídeo de molécula RNA Fases da síntese de proteínas em organismo eucarioto (esquerda) e procarioto (direita) Imagem da estrutura secundária do trna Exemplo de funções do ncrna nas atividades de transcrição, tradução e excisão em eucariotos. As interrogações representam funcionalidades e processos identificados, mas ainda desconhecidos aos biólogos Representação de um neurônio artificial A relação entre os três componentes chave de sistemas inteligentes: Representação, Aprendizado e Conhecimento (Haykin, 1999) Arquitetura de uma rede neural acíclica de camada simples Arquitetura de um perceptron de camada simples Exemplos de classes Arquitetura simplificada de uma rede neural baseada em mapas auto organizáveis de Kohonen e seus principais componentes Tipos de organização básica de computadores (Wu, 1999) Camadas de serviços de software e hardware em sistemas distribuídos xi
12 5.3 Diagrama representativo da arquitetura cliente-servidor. Os quadrados cinza-escuros são os elementos de processamento, e os círculos claros são os processos Diagrama representativo da arquitetura de um serviço distribuído em múltiplos servidores Diagrama representativo da arquitetura de servidores de proxy Diagrama representativo da arquitetura de peers Camadas de abstração de um grid baseado no middleware glite (Projeto EEGE, 2007) Fluxo de uma tarefa através dos componentes integrantes do grid e seus correspondentes estados (Burke et al., 2008) Passos para confecção do conjunto de treinamento (Arrial, 2008) O workflow do método SOM-PORTRAIT U-matriz para o mapa do modelo proteína dependente U-matriz para o mapa do modelo proteína independente O workflow do método Dist-SOM-PORTRAIT Comparações entre resultados do SOM-PORTRAIT (faixas) e resultados do PORTRAIT (círculos) para os vários arquivos de teste U-matrizes calculadas para os dois modelos do MDC Comparações entre resultados do SOM-PORTRAIT (faixas) e resultados do MDC (círculos) para as assembled ESTs do Pb Comparações entre resultados do MDC (faixas) e resultados do POR- TRAIT (círculos) para os vários arquivos de teste Comparação de tempos de execução entre Dist-SOM-PORTRAIT e SOM- PORTRAIT para o conjunto de arquivos de teste. A curva tracejada corresponde aos valores encontrados para o método SOM-PORTRAIT, enquanto que a curva contínua (em cinza) corresponde aos valores encontrados para o método Dist-SOM-PORTRAIT xii
13 Lista de Tabelas 2.1 Lista dos 22 aminoácidos encontrados na natureza Mapeamento de códons para aminoácidos e sequências de controle Matriz de programação dinâmica para o algoritmo de comparação global de duas sequências Alguns tipos de RNAs não codificadores e suas funções conhecidas (Eddy, 2001; Lakshmi and Agrawal, 2007) Passos do algoritmo de aprendizado por erro-correção de um perceptron (Kasabov, 1998; Mendes and Oliveira, 2009) Passos do algoritmo de aprendizado de um mapa auto organizável de Kohonen (Kasabov, 1998) Composição do conjunto de treinamento (Arrial, 2008) Tabela com o atributo, o programa usado para extraí-lo, seu número total de variáveis de cada atributo e a qual conjunto (dependente de ORF predita ou independente de ORF predita) de operações de extração de parâmetro pertence Nomes dos arquivos de treinamento, seu propósito e a quantidade de sequências que o compõe Arquivos criados durante a etapa de treinamento e funcionalidade Nomes dos arquivos de treinamento, seu propósito e a quantidade de sequências que o compõe Nome, filtro utilizado e quantidade de sequências dos arquivos de teste Comparação entre o método SOM-PORTRAIT e o método PORTRAIT. A porcentagem é relativa ao total de assembled ESTs no arquivo Pb ests.fas Comparação entre o método SOM-PORTRAIT e o método PORTRAIT. A porcentagem é relativa ao total de assembled ESTs no arquivo Pb ests.fas Comparação entre o método SOM-PORTRAIT e o método PORTRAIT. A porcentagem é relativa ao total de assembled ESTs no arquivo Pb ests.fas xiii
14 7.5 Comparação entre o método SOM-PORTRAIT e o método PORTRAIT. A porcentagem é relativa ao total de assembled ESTs no arquivo Pb ests.fas Comparação entre o MDC e o método PORTRAIT. A porcentagem é relativa ao total de assembled ESTs no arquivo Pb ests.fas Comparação entre o MDC e o método PORTRAIT. A porcentagem é relativa ao total de assembled ESTs no arquivo Pb ests.fas Comparação entre o MDC e o método PORTRAIT. A porcentagem é relativa ao total de assembled ESTs no arquivo Pb ests.fas Comparação entre o MDC e o método PORTRAIT. A porcentagem é relativa ao total de assembled ESTs no arquivo Pb ests.fas Comparação entre o método SOM-PORTRAIT e o modelo MDC. A comparação foi realizada sobre as sequências do transcriptoma do Pb Arquivos de teste criados para a comparação de tempos de execução dos métodos SOM-PORTRAIT e Dist-SOM-PORTRAIT Tempos de execução encontrados para cada arquivo de testes. O número de sequências do arquivo denota a coluna, e seu correspondente tempo de execução no método SOM-PORTRAIT e Dist-SOM-PORTRAIT xiv
15 Capítulo 1 Introdução Desde a definição funcional e estrutural do DNA, na década de 50, por Watson e Crick (Watson and Crick, 1953), o trabalho de mapeamento genético vem possibilitando um conhecimento cada vez mais amplo das funções e mecanismos genéticos, fisiológicos e metabólicos, entre outros, dos mais diversos organismos. Desses estudos pioneiros, definiuse o chamado dogma central da biologia molecular, a codificação indireta do DNA para proteínas (Eddy, 2001). Figura 1.1: Um esquema para o Dogma Central da Biologia. Recentemente, a Biologia Molecular fez mais uma importante descoberta. Boa parcela do material genético dos organismos estudados - inclusive organismos humanos - não codi- 1
16 fica proteínas, isto é, o fluxo apresentado na figura 1.1 pode não explicar completamente a função e ação de todo o material genético que compõe os organismos. Nos humanos, a proporção de RNA codificador está avaliada por volta de somente 2% do total do genoma (Szymanski et al., 2003). Este material genético que não está envolvido de forma direta na codificação de proteínas, chamado de RNA não codificador de proteínas ou ncrna, é o alvo dos estudos mais recentes de cientistas por todo o mundo. Experimentalmente é conhecido o vínculo de tais produtos genéticos com a regulação e com funções estruturais, algumas relacionadas à transcrição e tradução de mrnas. As predições de Crick e estudos posteriores sobre os RNAs de transferência (Hoagland et al., 1958), e RNAs ribossomais (Eddy, 2001) confirmam indubitavelmente tais vínculos. Mais um desafio computacional se desvela através dessa constatação experimental das inúmeras possibilidades de funções desse material genético não codificador, pois a análise de tais sequências seguindo os atuais processos de comparação e classificação de sequências não têm obtido bons resultados (Arrial, 2008). O processo de classificação dessas novas classes de RNAs deve considerar novos critérios, levantados experimentalmente sobre sua função no organismo. Esta análise é facilitada pela proposta de novos algoritmos capazes de identificar sequências potencialmente não codificadoras. Várias abordagens computacionais foram propostas, obtendo bons resultados para problemas específicos de identificação de uma determinada classe de ncrna ou para identificação de ncrnas em determinado organismo. Recentemente, o método PORTRAIT abriu novas perpectivas ao possibilitar a avaliação de forma independente de transcritos de diversos organismos, levando em conta aspectos e carências reais de projetos de análise de transcriptoma e de investigação de ncrnas, obtendo bons resultados experimentais (Arrial et al., 2007). Uma nova abordagem é proposta no presente trabalho, utilizando uma rede de Kohonen, uma rede neural artificial não supervisionada com capacidade de auto organização (Haykin, 1999; Kohonen, 2001). Partindo, de forma semelhante ao método PORTRAIT, da capacidade de generalização do problema de identificação de ncrnas para diversos organismos, e levando em conta as mesmas restrições de projetos biológicos nesta incipiente área de pesquisa, um novo método, batizado de SOM-PORTRAIT, é proposto. O método SOM-PORTRAIT possibilitará a identificação de sequências em três classes distintas: a classe de RNAs potencialmente codificadores (Coding), a classe de RNAs potencialmente não codificadores (Noncoding) e uma terceira classe hipotética, treinada com o propósito de confirmar a capacidade de categorização em classes de ncrna do método baseado em mapas auto organizáveis. Esta classe é nomeada Undefined, e indícios experimentais serão obtidos para confirmar sua boa delimitação pela rede neural treinada. A característica inédita da inclusão de mais de duas classes para identificação de ncrnas ampliará as possibilidades de classificação e categorização dos ncrnas em suas diversas subclasses, acelerando o processo de pesquisa e reconhecimento por parte dos biólogos. O artigo de descrição do método SOM-PORTRAIT (Silva et al., 2009) foi submetido ao Simpósio Brasileiro de Bioinformática BSB 2009 e aceito para compor a publicação do simpósio, feita pela Springer na revista Lecture Notes in Bioinformatics (LNBI ). 2
17 Estabelecido o método, sequências do transcriptoma do organismo Paracoccidioides brasiliensis, mapeado pelo Laboratório de Biologia Molecular da Universidade de Brasília, serão utilizadas para a validação do algoritmo através da confrontação direta com o método PORTRAIT (Arrial, 2008). Um método especialmente fabricado para esta etapa de avaliação, constituído de um mapa auto organizável com somente duas possibilidades de classificação também será confeccionado, para testes comparativos e avaliação da composição da classe Undefined do método SOM-PORTRAIT. Com a finalidade de maximizar o desempenho do algoritmo para grandes volumes de dados, uma versão distribuída do algoritmo, batizada Dist-SOM-PORTRAIT será especificamente implementada para rodar sobre o ambiente computacional distribuído do projeto EELA-2. O projeto é um consórcio de universidades e centros de pesquisa entre América Latina e Europa com a finalidade de difundir a infraestrutura distribuída de grids entre centros de pesquisa e conhecimento (Gavillet, 2008). A confrontação de tempos de execução do SOM-PORTRAIT e do Dist-SOM-PORTRAIT será avaliada, bem como as vantagens e desvantagens da implementação distribuída do método em relação à sua implementação não distribuída. Os objetivos do presente trabalho, resumidamente, pautam-se em: ˆ Propor o método SOM-PORTRAIT, um identificador de ncrnas baseado em mapas auto organizáveis, e implementá-lo; ˆ Realizar um estudo de caso com o organismo P. brasiliensis; ˆ Implementar uma versão distribuída do método SOM-PORTRAIT no âmbito do projeto EELA-2. O presente trabalho se divide, para estas finalidades, em: ˆ Capítulo 2: detalhamento de conceitos fundamentais relativos a Biologia Molecular, Bioinformática e explanação do fungo P. brasiliensis e seu Projeto do Genoma Funcional; ˆ Capítulo 3: detalhamento de conceitos fundamentais relativos a RNAs não codificadores; ˆ Capítulo 4: detalhamento de teoria e conceitos relativos a redes neurais artificias, mapas auto organizáveis (SOMs) e redes de Kohonen; ˆ Capítulo 5: detalhamento de conceitos relacionados a processamento distribuído e do middleware de acesso ao grid, seus serviços e ferramentas, bem como uma rápida explanação do projeto EELA-2 e seu contexto operacional; ˆ Capítulo 6: explicação sobre materiais, ferramentas e ambiente de trabalho utilizados no trabalho; ˆ Capítulo 7: exposição de resultados e explicações relativas ao trabalho desenvolvido; ˆ Capítulo 8: algumas considerações sobre os resultados obtidos e perpectivas futuras para o projeto; 3
18 ˆ Apêndice A: exemplo de arquivo JDL utilizado; ˆ Anexo I: artigo de descrição do método SOM-PORTRAIT submetido à conferência BSB 2009 e aprovado para publicação. 4
19 Capítulo 2 Biologia Molecular, Bioinformática e o Projeto Genoma Pb O presente capítulo dá as bases teóricas dos estudos e técnicas em Biologia Molecular e Bioinformática, com exemplificações práticas de problemas da área, bem como uma breve ilustração de ferramentas e bancos de dados utilizados na pesquisa biológica. Também é apresentado o organismo Paracoccidioides brasiliensis (Pb), suas principais características biológicas conhecidas e o projeto do seqüenciamento de seu genoma funcional, resultados obtidos e as expectativas com relação à presença de RNAs não codificadores em seu genoma. 2.1 Biologia Molecular Biologia Molecular é o ramo da Biologia responsável, basicamente, pelo estudo da estrutura de proteínas e ácidos nucléicos, processos relacionados e outros atores envolvidos, como organelas celulares e enzimas (Setubal and Meidanis, 2000). As primeiras formas de vida reportadas na Terra foram datadas, através de estudos geológicos, em aproximadamente 3,5 bilhões de anos atrás (Setubal and Meidanis, 2000). Avançando para as atuais formas de vida conhecidas, estas formas primordiais sofreram um processo de evolução que culminou no aparecimento de organismos complexos, pluricelulares, convivendo com organismos mais simples, unicelulares, como os procariotos. Partindo do mesmo ponto inicial - estas formas de vidas primordiais - é fácil compreender que todos os organismos, unicelulares ou pluricelulares, dividem uma composição química muito semelhante. A composição química de células de organismos vivos é predominantemente formada por carbono C, oxigênio O, nitrogênio N e hidrogênio H, onde o hidrogênio é responsável por 99% da massa atômica de tais compostos (Clote and Backofen, 2000). Nos seres vivos, os compostos aparecem, muitas vezes, como cadeias de outros pequenos compostos interligados. A estes pequenos compostos damos o nome de monômeros. Às cadeias formadas pela junção repetida de monômeros, dá-se o nome de polímeros. Os 5
20 polímeros mais importantes para os organismos são as proteínas e os ácidos nucléicos, formados respectivamente por cadeias de aminoácidos e cadeias da bases nitrogenadas Proteínas Proteínas participam direta ou indiretamente de quase todas as atividades celulares de um organismo vivo (Setubal and Meidanis, 2000). Dividem-se em três categorias, de acordo com sua função: globular, fibrilar e membranar. Proteínas globulares têm função principalmente enzimática e de ligação (antígenos). Proteínas fibrilares formam tecidos elásticos, e proteínas membranares participam da composição de membranas celulares (Clote and Backofen, 2000). Composição química Proteínas, como dito anteriormente, são formadas por longas cadeias de aminoácidos. A estrutura do monômero aminoácido é formada, basicamente, por um carbono central: um grupo amina: um grupo carboxila: C α NH 2 COOH e uma cadeia complementar (radical), que é a responsável pela unicidade dos vários aminoácidos existentes na natureza (Setubal and Meidanis, 2000). A figura 2.1 exibe os grupos formadores. Somente aminoácidos entram na composição de proteínas (Lesk, 2002). Esta composição é feita unindo uma sequência de aminoácidos através de ligações peptídicas. Ligações peptídicas ocorrem pela retirada de uma molécula de água entre um grupo carboxila de um aminoácido e o grupo amina de outro aminoácido. A figura 2.2 ilustra esta ligação. Os radicais R e Rŕepresentam as cadeias complementares dos dois aminoácidos originais que compõem a ligação. Desta forma, proteínas não são exatamente compostas de aminoácidos, mas sim do resíduo desta ligação, os peptídeos (Clote and Backofen, 2000). Essencialmente cadeias desses peptídeos, proteínas costumam ser chamadas também de polipeptídeos. Por convenção, as extremidades desta longa cadeia são caracterizadas por um grupo amina (N-terminal) e um grupo carboxila (C-terminal), e a proteína orienta-se do N-terminal (início) para o C-terminal (fim). O grupo entitulado backbone é um resultado importante desta ligação para a estrutura final da proteína. É uma estrutura coplanar - isto é, cada átomo compreendido na área escurecida está num mesmo plano. Na natureza, são catalogados 22 aminoácidos conhecidos (Lesk, 2002), sendo 20 nãopolares, comumente achados em proteínas, e 2 polares, mais raramente encontrados em polipeptídeos. 6
21 Figura 2.1: Grupos formadores de aminoácidos. Figura 2.2: Ligação peptídica e orientações φ e ψ do carbono Cα. A tabela 2.1 mostra o nome, abreviação e o código de uma letra usado para identificar o aminoácido. Em asterisco (*), os aminoácidos menos comumente encontrados em proteínas. Estrutura da proteína A função de uma proteína é determinada pela sua estrutura espacial (Clote and Backofen, 2000). Os peptídeos que a compõem combinam-se por meio de ligações de hidrogênio 7
22 Tabela 2.1: Lista dos 22 aminoácidos encontrados na natureza. Nome Abreviação Código 1 Alanina Ala A 2 Arginina Arg R 3 Asparagina Asn N 4 Ácido Aspártico Asp D 5 Asparagina ou Ácido Aspártico * Asx B 6 Cisteína Cys C 7 Glutamina Gln Q 8 Ácido Glutâmico Glu E 9 Glutamina ou Ácido Glutâmico * Glx Z 10 Glicina Gly G 11 Histidina His H 12 Isoleucina Ile I 13 Leucina Leu L 14 Lisina Lys K 15 Metionina Met M 16 Fenilalanina Phe F 17 Prolina Pro P 18 Serina Ser S 19 Treonina Thr T 20 Triptofano Trp W 21 Tirosina Tyr Y 22 Valina Val V (chamadas também de pontes de hidrogênio), ligações iônicas e ligações dissulfídicas (entre átomos de enxofre dos resíduos de aminoácidos Cisteína (Cys)). Outros determinantes da conformação espacial de proteínas são a hidrofobicidade de regiões do polipeptídeo - isto é, o grau de afinidade com moléculas de água - e a rotação dos eixos φ e ψ (figura 2.2). A sequência de resíduos que forma a proteína é dita estrutura primária da proteína. Tal estrutura linear é importante para leitura dos aminoácidos que compõem a proteína, mas não caracteriza sua função (Setubal and Meidanis, 2000). A estrutura secundária de uma proteína é formada pelo alinhamento e dobramento da sequência de resíduos, principalmente pelo dobramento nos eixos φ e ψ, e pelo alinhamento de backbones (figura 2.2), que formam estruturas cilíndricas chamadas α-hélice. Também surgem nesta configuração os alinhamentos do tipo β-folha, que são alinhamentos de regiões entre diferentes cadeias de resíduos. Nessa configuração espacial, repetições de padrões de alinhamento e dobramento da sequência de resíduos, chamadas de motivos, são evidenciadas. Motivos são especialmente importantes para inferência de funções e grau de similaridade entre diferentes proteínas (Clote and Backofen, 2000). Estruturas terciárias evidenciam o formato tridimensional de proteínas no organismo, dita estrutura nativa. Se a proteína for na verdade um agregado de várias subproteínas 8
23 (chamadas proteínas diméricas), a estrutura terciária considera cada subproteína isoladamente. Finalmente, a estrutura quaternária de uma proteína considera sua totalidade, em forma tridimensional nativa - isto é, sua forma naturalmente encontrada no organismo -, plenamente funcional, portanto Ácidos nucléicos Ácidos nucléicos, segundo a biologia moderna, têm a função principal de armazenar informação necessária para criação de proteínas e possibilitar a transferência desta informação para outros organismos, através de processos de reprodução celular (Setubal and Meidanis, 2000). DNA - ácido desoxirribonucléico - e RNA - ácido ribonucléico - são ambos compostos por cadeias de elementos menores, assim como as proteínas. No caso de DNAs e RNAs, tem-se um grupo fosfato P O 3 4 um açúcar central e uma base nitrogenada, formando o monômero chamado nucleotídeo (Clote and Backofen, 2000; Setubal and Meidanis, 2000). A composição em cadeias de nucleotídeos forma uma sequência RNA ou DNA, dependendo da composição deste nucleotídeo. DNA No ácido desoxirribonucléico - DNA -, o nucleotídeo é formado por um açúcar central - a pentose (açúcar com cinco átomos de carbono) desoxirribose (figura 2.3), e um de quatro tipos diferentes de bases nitrogenadas - moléculas com ciclos de carbono e nitrogênio (figura 2.4). Figura 2.3: Pentose principal do nucleotídeo formador do DNA: a desoxirribose. Na figura 2.3, os carbonos são numerados de 1 a 5, por uma convenção em relação à estrutura química do composto. Ao carbono 1 está associada uma base nitrogenada, ao 9
24 carbono 5 o fosfato. O carbono 3 desempenha importante papel, pois é nele que ocorre a reação de ligação entre o fosfato de um nucleotídeo com o grupo hidroxila do carbono 3 de outro nucleotídeo, compondo o polímero. Por causa desta ligação, a molécula do DNA é orientada do carbono 5 ao carbono 3, naturalmente (Clote and Backofen, 2000). A figura 2.4 mostra também as ligações de hidrogênio entre duas bases nitrogenadas, chamadas complementares. O modelo de Watson-Crick do DNA já definia tal afinidade entre bases complementares, por causa de sua disposição espacial e afinidade eletrônica da molécula. Bases purinas (Adenina e Guanina) somente se ligam a bases pirimidinas (Timina e Citosina) (Watson and Crick, 1953). Figura 2.4: Bases nitrogenadas que compõem um nucleotídeo de molécula DNA. Pela característica das bases complementares, é possível extrair o complemento de uma faixa de DNA aplicando a seguinte regra: Adenina T imina Guanina Citosina (2.1) Da disposição espacial de uma fita DNA, indo do carbono 5 ao 3, também se conclui que seu complemento é o exato oposto, indo do 3 ao 5. Portanto, uma faixa é o exato complemento reverso da outra, possibilitando a duplicação de trechos do código DNA. A figura 2.5 ilustra a famosa estrutura de dupla hélice do DNA. O DNA pode ser encontrado na forma de cromossomos (aglomerado com proteínas para reduzir espaço), em forma circular (principalmente em organismos menos complexos, como bactérias) e em sua forma linear, assim como apresentado na figura
25 Figura 2.5: Estrutura do DNA e suas partes integrantes. Grande parte do material genético encontrado em DNA não codifica para proteínas, em organismos eucariotos (Szymanski et al., 2003). Dá-se o nome de genes para as regiões delimitadas do DNA que codificam para proteínas ou RNAs (Setubal and Meidanis, 2000), isto é, no ato de transcrição, o DNA é transcrito para um RNA funcional válido ou para um RNA mensageiro válido (veja a seção 2.1.3). RNA RNAs - ácidos ribonucléicos - agem em conjunto com DNAs na produção de proteínas e herança celular (Setubal and Meidanis, 2000). De forma semelhante ao DNA, o nucleotídeo é formado por um açúcar central - a pentose ribose (figura 2.6), e um de quatro tipos diferentes de bases nitrogenadas. Em RNAs, entretanto, a base nitrogenada Timina (T) é substituída pela base nitrogenada Uracila (U) (figura 2.7), também uma base pirimidina. Identicamente ao DNA, a orientação do RNA se dá do carbono 5 ao carbono 3. Podese reescrever as regras de complementaridade das bases nitrogenadas em 2.1 simplesmente trocando-se a base Timina pela base Uracila. 11
26 Figura 2.6: Pentose principal do nucleotídeo formador do RNA: a ribose. A estrutura de um filamento do RNA o torna mais vulnerável a danos e erros, e portanto menos apto a transportar informação genética (Clote and Backofen, 2000). Por essa característica, além da estrutura química mais simplificada tanto da base Uracila, quando confrontada com a base Timina, como da estrutura do RNA, existem várias teorias de que o RNA teria sido o primeiro ácido nucléico a ser usado como transportador de material genético (Eddy, 2001). Figura 2.7: Uracila - base pirimidina que compõe um nucleotídeo de molécula RNA Dogma Central da Biologia Molecular O dogma central da Biologia Molecular, determinado pelos estudos de Watson e Crick relacionados a ácidos nucléicos (Watson and Crick, 1953) relaciona os principais agentes da Biologia Molecular, ácidos nucléicos e proteínas, com atividades celulares muito importantes, o processo de replicação de trechos de DNA genômico, de transcrição, e de tradução. 12
27 Transcrição Transcrição envolve mecanismos e proteínas celulares com o objetivo de transformar genes do DNA em RNA. Já a tradução utiliza cadeias de RNA chamadas mensageiro - o mrna - para traduzir sua sequência de bases nitrogenadas em aminoácidos, ligando-os com o auxílio de organelas celulares e outros tipos de RNA para formar proteínas (Setubal and Meidanis, 2000). A figura 2.8 ilustra as fases de transcrição e de tradução em um organismo eucarioto - com núcleo - e em um organismo procarioto, que são diferentes. Figura 2.8: Fases da síntese de proteínas em organismo eucarioto (esquerda) e procarioto (direita). A transcrição em organismos procariotos é feita de forma direta. Uma região de codificação é identificada no DNA através de seu promotor, uma região específica do DNA que identifica o início de um novo gene. Usualmente, este promotor é uma sequência das bases Timina e Adenina, formando uma TATA-box (Clote and Backofen, 2000). Uma enzima de RNA - a RNA polimerase é capaz de identificar este promotor, e segmentar o DNA no ponto exato do início do gene. Esta enzima age como uma ponte entre as bases nitrogenadas na faixa de DNA sendo percorrida - sempre do carbono 5 ao 3 - e as bases nitrogenadas complementares - levando-se em conta a substituição da Timina pela Uracila para formar o RNA - que vão sendo ligadas pela enzima. Quando o processo chega ao fim do gene, identificado por outra área de finalização, o RNA recém criado - dito RNA transcrito - é liberado e pode ser imediatamente usado para tradução. Portanto, o RNA transcrito em células procariotas é um RNA mensageiro (Setubal and Meidanis, 2000). Em organismos eucariotos, o processo é similar. Regiões de codificação - identificadas da mesma forma - são reconhecidas pela enzima RNA polimerase. Porém, o gene, em 13
28 organismos eucariotos, não é representado por uma sequência contínua de bases nitrogenadas no DNA. Esta sequência é interrompida muitas vezes por trechos que não integram o RNA mensageiro final: os íntrons. Logo após a transcrição do RNA, estes trechos são descartados do RNA transcrito para formar o mrna com os trechos restantes - chamados exons - no processo de excisão (Clote and Backofen, 2000). O processo reverso de síntese de DNA a partir do mrna, através da enzima RNA-transcriptase reversa, resulta no DNA complementar de um gene, ou cdna, uma cadeia sem íntrons do gene, mais facilmente manipulável e muito importante para a biologia molecular (Setubal and Meidanis, 2000). Trechos destes genes que codificam em proteínas recebem o nome de ESTs - Expressed Sequence Tags. O mrna, ao final do processo, atravessa a parede nuclear e se deposita no citoplasma da célula, pronto para a etapa de tradução em proteínas. Tradução O processo de tradução ocorre de forma semelhante em ambos os organismos. Tanto em procariotos como em eucariotos, o mrna é articulado a um ribossomo e a uma série de RNAs de transferência - trnas. Ribossomos são organelas celulares formadas por duas extremidades formadas por RNA ribossomal - rrna - envolvido em proteínas, que provêm o meio necessário para o correto acoplamento de trnas ao mrna para formar proteínas. Figura 2.9: Imagem da estrutura secundária do trna. O laço do anticódon na figura 2.9 é um códon - isto é, uma sequência de três bases nitrogenadas - complementar do códon correspondente no mrna. Um códon de inicia- 14
29 lização demarca o início do mrna e a primeira ligação de trnas ao mrna. Após esta primeira ligação, a estrutura do ribossomo permite o deslizamento dos trnas pelo ribossomo, e o avanço da fita mrna. Cada trna, quando transporta em seu sítio ACC (sítio de ligação) um aminoácido, é chamado de aminoacil-trna (Clote and Backofen, 2000), e denotado pelo aminoácido que carrega. Após a ligação pelo anticódon ao códon correspondente e realização da ligação peptídica com o restante da cadeia de resíduos no ribossomo, o trna é retirado do ribossomo. A tabela 2.2 representa o mapeamento dos 20 aminoácidos comumente encontrados em organismos com os correspondentes códons em genes. Nota-se também a presença de sequências de parada (STOP). A sequência de início de uma proteína é sempre o aminoácido Metionina (Met), representado pelo códon AUG (Clote and Backofen, 2000). Tabela 2.2: Mapeamento de códons para aminoácidos e sequências de controle. U C A G Phe Ser Tyr Cys U Phe Ser Tyr Cys C U Leu Ser STOP STOP A Leu Ser STOP Trp G Leu Pro His Arg U Leu Pro His Arg C C Leu Pro Gln Arg A Leu Pro Gln Arg G Ile Thr Asn Ser U Ile Thr Asn Ser C G Ile Thr Lys Arg A Met Thr Lys Arg G Val Ala Asp Gly U Val Ala Asp Gly C A Val Ala Glu Gly A Val Ala Glu Gly G Técnicas de cópia e replicação de sequências em Biologia Molecular As técnicas de cópia e replicação de sequências biológicas em laboratório permitem aos cientistas estudar de forma eficiente e rápida os mais variados organismos. Alguns conceitos são explanados abaixo para compreender tais técnicas. 15
30 Fases de Leitura As sequências de bases nitrogenadas que formam cadeias de DNA e RNA podem ser agrupadas em códons (tripla de bases nitrogenadas) de diferentes maneiras, sempre obedecendo à ordem de leitura do carbono 5 ao 3. Uma fase de leitura, em inglês reading frame, é um possível agrupamento em triplas da sequência, ao se adotar um determinado ponto da cadeia para começar o agrupamento (Clote and Backofen, 2000), por exemplo, seja a cadeia DNA: 5 T ACT GCGT T ACGACG 3 Uma primeira possível fase de leitura é: 5 T ACT GCGT T ACGACG 3 O que resulta, referindo-se à tabela 2.2, ao seguinte polipeptídeo: Met T hr Gln Cys Cys Mas, deslocado de uma base, a segunda fase de leitura será: O que resulta no seguinte polipeptídeo: 5 T ACT GCGT T ACGACG 3 ST OP Arg Asn Ala Note que as bases T inicial e CG finais foram descartadas. Esta sequência, bem como qualquer outra sequência, admite três fases de leitura diferentes. A partir da quarta, pode-se considerá-la uma subsequência da primeira fase de leitura. No caso de cadeias DNA, deve-se levar em conta ainda a possibilidade de transcrição da fita complementar. Por isso, mais três fases de leitura são possíveis. Uma fase aberta de leitura, ou open reading frame - ORF - é uma configuração em que a escolha de triplas resulta numa sequência contínua (excluídos introns) de triplas que representam exclusivamente aminoácidos, sem sequências STOP, e que é múltipla de três, ou seja, não deixa bases residuais ao ser agrupado (Setubal and Meidanis, 2000). Técnicas utilizadas As técnicas utilizadas para coleta de sequências RNA e DNA de um organismo são variadas e avançadas, sendo agrupadas em operações de cópia e quebra de cadeias, leitura dos fragmentos e remontagem da cadeia original a partir de seus fragmentos. O objetivo principal é o seqüenciamento completo dos genes, ou do DNA genômico - mapeamento do genoma do organismo. O DNA complementar - recuperado pelo processo inverso da transcrição aplicado nas cadeias de mrna, também é alvo do seqüenciamento em laboratórios, resultando no transcriptoma do organismo. A precisa localização dos genes no cromossomo e sua função também são objetivos do seqüenciamento destas cadeias. 16
31 A cópia de trechos de DNA é imprescindível para as atividades laboratoriais de seqüenciamento. A clonagem de trechos de DNA pode ser feita através da enzima catalizadora DNA-polimerase ou através de enxerto de material genético em organismos hospedeiros (Setubal and Meidanis, 2000). A enzima DNA-polimerase atua de forma similar à RNA-polimerase (veja seção 2.1.3). Através de processos de pirólise e adição de promotores, o DNA inicial e suas sucessivas cópias são replicados várias outras vezes. Organismos hospedeiros, chamados de vetores, são muito utilizados para replicação de DNA. Os vetores mais utilizados são procariotos, como a levedura (Saccharomyces Cerevisiae), que contém DNA circular (o plasmídeo), sem presença de íntrons, facilmente adaptável. Vírus também são muito utilizados, como a classe de vírus bacteriófagos. O método é desenvolvido da seguinte forma: 1. O organismo tem seu plasmídeo quebrado por uma enzima catalítica, e recomposto com o enxerto do DNA a ser replicado, por uma enzima DNA-ligase. 2. Naturalmente, o organismo reproduz-se, cultivado em laboratório, até que a cultura atinja a quantidade desejada. 3. Cada organismo novo é coletado, seu DNA (o DNA do vetor) é removido através de enzimas catalíticas do DNA hospedado. O processo de remoção do DNA do vetor não é completo. Podem haver resíduos restantes no DNA original. Excertos de DNA com resíduos são ditos contaminados, e sua detecção é muito difícil (Setubal and Meidanis, 2000). A quebra de cadeias de ácidos nucléicos é um processo muito importante na Biologia Molecular, pois a análise de cadeias muito longas em laboratório é inviável através das técnicas atuais (Setubal and Meidanis, 2000). Usam-se enzimas catalíticas ou processos mecânicos para a quebra de cadeias. As enzimas utilizadas são ditas enzimas de restrição. Enzimas de restrição agem sobre sítios da cadeia DNA, trechos da sequência com mesma codificação da parte ativa da enzima. Estes trechos têm a característica marcante de serem palíndromos, isto é, um trecho é idêntico ao seu complementar. As enzimas conjugam e extraem a sequência combinante, e o restante do DNA pode sofrer uma recombinação ou manter-se separado. Outro processo envolve a quebra mecânica de sequências. Este procedimento, chamado método shotgun (escopeta, em inglês) é feito imergindo-se várias amostras do mesmo DNA replicado em solução. Através de centrífuga ou outro processo mecânico, esta solução é misturada violentamente, causando cisões e recombinações aleatórias na cadeia. A leitura de seqüencias se dá por uso de uma técnica especial de eletroforese em gel, ou seja, de sedimentação de material submetido a um campo elétrico. Aproveitando-se das diferenças de massa molecular e afinidade eletrônica, as diferentes cadeias são imersas em uma placa gel e submetidas a um campo elétrico forte constante. Cadeias com bases nitrogenadas de maior massa se movimentarão vagarosamente na placa, enquanto que cadeias com bases nitrogenadas leves sedimentarão mais à frente na placa. Por interpolação 17
32 aritmética, sabendo o tamanho da cadeia, é possível calcular sua composição (Setubal and Meidanis, 2000). 1 Após a replicação e quebra em fragmentos, os trechos de ácido nucléico podem ser finalmente analisados e seqüenciados, isto é, sua sequência de bases nitrogenadas descoberta. Fragmentos de cdna ou mrna, ou DNA genômico, podem ser analisados com relação ao seu produto final, a proteína que codifica, geralmente resultando uma sequência de aminoácidos (Mount, 2003). A comparação de sequências tem por objetivo descobrir similaridades entre duas ou mais sequências e também o alinhamento dos trechos semelhantes entre estas várias sequências. O problema de comparação pode ser generalizado para o problema de reconhecimento k-aproximado de padrões (Baase, 1978; Setubal and Meidanis, 2000). Conceitos de semelhança e distância são usados para a modelagem do problema de alinhamento de sequências. Após corretamente seqüenciados, os fragmentos de sequências devem ser remontados para formar a cadeia inicial. Dados vários fragmentos, procura-se chegar à sequência de consenso que os gerou. São vários os problemas envolvidos na remontagem de fragmentos. Entre vários, cita-se a contaminação da sequência por DNA do vetor, trocas, deleções ou inserções acidentais nas bases da cadeia, além de quimeras, falta de cobertura, orientação desconhecida da cadeia e fragmentos de regiões repetidas (Setubal and Meidanis, 2000). Durante todo o procedimento de cópia, fragmentação e leitura, a cadeia pode sofrer alterações acidentais na sua sequência de bases: uma ou mais bases, ou trechos de bases, podem ser adicionados ao fragmento. Estes erros são detectados por uma análise dos fragmentos. Quimeras acontecem quando fragmentos não contínuos na cadeia original são, todavia, passíveis de serem remontados como fragmentos contínuos, gerando um consenso errado da cadeia. Erros de fragmentos quiméricos são evitados também pela análise dos fragmentos (Setubal and Meidanis, 2000). Falta de cobertura, como o nome já indica, acontece quando, para remontar certa região do DNA, não são coletados nenhum ou um número insuficiente de fragmentos para se obter o consenso. Para algumas sequências, não se conhece a orientação, se é a cadeia 5 3 ou se é sua complementar 3 5. Fragmentos de regiões repetidas podem ser confundidos por erros de troca de bases na sequência (Setubal and Meidanis, 2000). 2.2 Bioinformática Bioinformática é o ramo sinérgico entre Computação, Matemática e Biologia Molecular que contribui com modelos, análises estatísticas, algoritmos e sistemas de computação, entre outras contribuições teóricas e práticas à área de Biologia Molecular (Clote and Backofen, 2000). Assim, a Bioinformática é especialmente dedicada aos vários e complexos problemas que a Biologia Molecular oferece. 1 Máquinas atuais de eletroforese gel permitem a contagem de cadeias de aproximadamente 700 a 2000 pares de bases - bp, automaticamente. 18
33 2.2.1 Algoritmos em Bioinformática Os principais algoritmos que solucionam problemas relacionados a comparação e remontagem de sequências representam abordagens para solucionar problemas mais específicos envolvendo estes dois problemas centrais. Para o problema de comparação, busca-se na verdade o melhor alinhamento entre duas sequências. Já para o problema de remontagem, as heurísticas conhecidas se apóiam no problema de alinhamento múltiplo de sequências (neste caso, os fragmentos são as sequências a serem alinhadas para obter a cadeia de consenso) (Setubal and Meidanis, 2000). Na seguinte subseção, o exemplo do algoritmo básico para comparação global entre duas sequências é apresentado. Algoritmo para Comparação Global de Duas Sequências Sejam duas sequências conforme abaixo: T ACGGC T AAGC (2.2) Observa-se que a sequência T AAGC não coincide em tamanho com a sequência T ACGGC. Para alinhar suas bases de forma otimizada, insere-se um espaço (GAP) entre a segunda base Adenina e a base Guanina, da seguinte forma: T ACGGC T AA GC (2.3) Dado este alinhamento inicial, pode-se definir um escore associado a este alinhamento, através de uma função de cálculo da similaridade entre duas sequências p e q: α, se p = q sim(p, q) = β, se p q γ, se p = GAP ou q = GAP (2.4) Os valores α, β e γ são definidos de acordo com a análise do problema em questão. De forma geral, α > β, para avaliar positivamente alinhamentos idênticos de bases. Espaços costumam ter avaliação γ < β, para desencorajar a alteração das sequências (Setubal and Meidanis, 2000). Através de táticas de programação dinâmica, constrói-se uma matriz com os valores de escores obtidos para os alinhamentos de subsequências das duas sequências a serem 19
34 alinhadas. A matriz 2.3 ilustra a matriz de programação dinâmica para o problema de comparação global de duas sequências. Tabela 2.3: Matriz de programação dinâmica para o algoritmo de comparação global de duas sequências. 0 q[1]... q[n] 0 sim(0, 0) sim(0, q[1])... sim(0, q[1..n]) p[1] sim(p[1], 0) sim(p[1], q[1])... sim(p[1], q[1..n]) p[1..2] sim(p[1..2], 0) sim(p[1..2], q[1])... sim(p[1..2], q[1..n])... p[1..m] sim(p[1..m], 0) sim(p[1..m], q[1])... sim(p[1..m], q[1..n]) A obtenção desta matriz (n+1)x(m+1) é feita calculando-se, para cada entrada (i, j), i = 0..n, j = 0..m, o valor do alinhamento entre p[1..i] - a subsequência da cadeia p até a base na posição i - e q[1..j] - a subsequência da cadeia q. A função de similaridade sim(p,q) pode ser então definida da seguinte forma: sim(p[1..i], q[1..j 1]) γ sim(p[1..i], q[1..j]) = max sim(p[1..i 1], q[1..j 1]) + p(i, j) sim(p[1..i 1], q[1..j]) γ (2.5) Onde a função p(i, j) aplicada sobre os caracteres i e j das respectivas sequências p e q, é da seguinte forma: p(i, j) = { α, se i = j β, se i j (2.6) O algoritmo é capaz de realizar a construção da matriz utilizando estas duas funções iterativamente sobre cada elemento da matriz. Importante notar que a primeira linha e primeira coluna - que correspondem, respectivamente, ao alinhamento da sequência p com espaços vazios e o alinhamento da sequência q com espaços vazios - serão calculadas diretamente. O algoritmo tem complexidade espaço-temporal, de O(m) + O(n) + O(mn), respectivamente, para percorrer o tamanho de p e q na etapa de inicialização e para preencher cada entrada da tabela (Setubal and Meidanis, 2000). Para extrair o alinhamento ótimo para as duas sequências, o algoritmo vale-se da rotina Align(p[1..i], q[1..j]), que recebe em cada iteração de sim(p[i], q[j]) a matriz preenchida até o ponto (i, j), e percorre o caminho completo até o início da matriz. Dada a entrada 20
35 de p[1..i], q[1..j] a rotina Align(p[1..i], q[1..j]), conforme descrita em Setubal and Meidanis (2000), realiza o seguinte cálculo recursivo para escolha do caminho: Align(p[1..i], q[1..j 1]) e alinha um espaço com q[j] Align(p[1..i], q[1..j]) = Align(p[1..i 1], q[1..j 1]) e alinha p[i] com q[j] Align(p[1..i 1], q[1..j]) e alinha um espaço com p[i] (2.7) Podemos interpretar este caminho criado pela rotina Align(p[1..i], q[1..j]) como as setas na matriz 2.3, da seguinte forma: Align(p[1..i], q[1..j 1]) e caminha para cima: Align(p[1..i], q[1..j]) = Align(p[1..i 1], q[1..j 1]) e caminha na diagonal: Align(p[1..i 1], q[1..j]) e caminha para a esquerda: (2.8) A rotina de procura pelo alinhamento ótimo tem complexidade O(m+n), o custo para percorrer as duas sequências p e q (Setubal and Meidanis, 2000). Outros problemas de Biologia Molecular Além dos problemas mostrados nas seções e acima, vários outros problemas desafiam, como dito por Donald E. Knuth, as áreas da Computação e da Matemática. A modelagem de tais problemas podem ser reduzidos a certas classes de problemas. Por exemplo, o problema de comparação de sequências pode ser reduzido ao problema de reconhecimento k-aproximado de padrões. Assim, o limite de tempo superior para solução de problemas desta classe é da ordem de O(3 n ) (Baase, 1978). Uma ordem exponencial deste tipo exige capacidade de processamento muito elevada dos computadores para que o problema seja resolvido em tempo hábil. O desafio na confecção de peças hardware e de soluções em computação distribuída (veja capítulo 5) é enorme também Programas e bancos de dados utilizados na Biologia Molecular Os programas e bancos de dados utilizados em Biologia Molecular são muitos e dos mais variados tipos. Abaixo, uma pequena descrição dos programas e bancos de dados que foram utilizados neste trabalho. BLAST O programa BLAST, Basic Local Alignment Search Tool, é bastante utilizado na comparação entre sequências. É um algoritmo de alinhamento local, que utiliza comparações estatísticas entre regiões de similaridade da seqüencia com várias outras sequências de 21
36 comparação em um banco de dados de sequências. O BLAST pode ser utilizado para identificar uma relação evolucionária, inferir funções, ou identificar uma família de genes entre sequências (Altschul et al., 1990). CD-HIT e BLASTCLUST O programa CD-HIT é utilizado para agrupar fragmentos de DNA (cdnas) em grandes bancos de dados de proteínas, removendo redundâncias e criando um banco de dados com somente contigs representativos (Li and Godzik, 2006; Arrial, 2008). Mais especificamente, o programa CD-HIT-EST pode ser efetivamente utilizado para eliminar redundâncias em um conjunto de sequências genéticas. Já o programa BLASTCLUST agrupa proteínas utilizando o BLAST para a identificação de regiões de similaridade, e agrupa sequências de DNA utilizando o MegaBLAST para a identificação de regiões de similaridade (Altschul et al., 1990). EMBL O banco de dados EMBL (EMBL Nucleotide Database) (Cochrane et al., 2008) é constituído de diversos índices e integrações de teor colaborativo entre os mais diversos bancos de dados, com elevado volume de dados e número de anotações, tornando-o uma referência muito confiável para coleções de sequências de nucleotídeos. SwissProt O banco SwissProt (Boeckmann et al., 2002) é especializado em sequências protéicas, concentrando-se na anotação de entradas do projeto de sequenciamento do genoma humano e de outros projetos de organismos modelo. Mantém, desta forma, anotações de boa qualidade. RNAdb O RNAdb (Pang et al., 2005) é composto por mais de 800 sequências de ncrnas estudadas experimentalmente e especialmente selecionadas por sua associação com doenças e processos de crescimento/desenvolvimento em organismos. O banco não contém RNAs de transmissão ou RNAs ribossômicos, e também engloba várias sequências do genoma humano. Rfam Incluindo RNAs estruturais não codificadores e regulatórios, o Rfam (Gardner et al., 2009) reúne diversas famílias de RNAs amplamente estudadas e anotadas, tais como RNAs de transmissão e RNAs ribossômicos, como outras famílias de ncrna com número mais limitado de anotações. 22
37 NONCODE NONCODE (Liu et al., 2005) é um banco de dados integrado dedicado exclusivamente a catalogar e armazenar informações relativas a ncrnas. Suas entradas são oriundas de dados obtidos de outros bancos de dados, notadamente o GenBank, e também de literaturas científicas relacionadas. Suas características composicionais incluem a ausência de RNAs de trasmissão e RNAs ribossômicos e a corroboração por meio de confronto com produções científicas relacionadas de mais de 80% de suas entradas. Em sua primeira versão, o banco conta com 5339 sequências não redundantes dos mais variados organismos unicelulares. 2.3 Projeto do Genoma Funcional do fungo Paracoccidioides brasiliensis O Paracoccidioides brasiliensis é um fungo dimórfico encontrado na forma de micélio ou esporos à temperatura de 24 a 26, e na forma de levedura à temperatura de 37. Pouco é sabido sobre os hospedeiros naturais do P. brasiliensis, mas presume-se que, além do homem, animais como o tatu ou morcegos possam ser hospedeiros silvestres do fungo (Andrade, 2006). O P. brasiliensis, conforme estudos e observações, vive como um fungo saprófita em solos e vegetais, preferencialmente em regiões de florestas tropicais úmidas. A forma de transmissão do organismo se dá, presumidamente, pelo solo, principalmente pela inalação de seus esporos ou por pequenas escoriações na pele (Andrade, 2006) Morfologia e Genética O fungo P. brasiliensis, em forma de micélio, é pluricelular, com células alongadas agrupadas em longos filamentos (hifas). Já na sua forma de levedura, é constituído de células arredondadas multinucleadas que apresentam um formato característico inconfundível ( roda de leme ) (Andrade, 2006). Estima-se que a variabilidade genética do P. brasiliensis é elevada. Vários agrupamentos destes isolados foram propostos, baseados em semelhança genética dos organismos, por distribuição geográfica, entre outros (Andrade, 2006) Paracoccidiodomicose O P. brasiliensis é o agente etiológico da Paracoccidiodomicose (PCM ), também chamada de Blastomicose sul americana ou Blastomicose brasileira. A PCM é uma micose sistêmica com expressivo número de infectados (10 milhões em toda a América Latina), sendo endêmica em regiões não contínuas da América Latina (México e América do Sul, exceto Guianas e Chile) (Andrade, 2006). 23
38 A PCM se manifesta em aproximadamente 2% dos indivíduos infectados (Felipe et al., 2005), ficando em estado latente na grande maioria dos casos. Os dados epidemiológicos coletados relatam incidência da doença principalmente na população rural, em membros do sexo masculino com idade de 30 a 60 anos, e em indivíduos imuno-deprimidos (Andrade, 2006). O organismo, em sua forma infectante, instala-se inicialmente nos pulmões do hospedeiro, causando reações alérgicas e o surgimento de granulomas característicos. Após este estado inicial, ocorre a diferenciação do fungo para seu estado de levedura, reação presumidamente desencadeada pela temperatura corpórea e por uma combinação de fatores fisiológicos do hospedeiro (Andrade, 2006). A PCM pode se manifestar de forma aguda ou de forma crônica. Em sua forma aguda, a PCM é fatal, acometendo de forma grave o sistema respiratório do indivíduo e progredindo rapidamente para seu sistema linfático, baço, fígado e medulas ósseas. Já em sua forma crônica, geralmente um órgão ou sistema é afetado pela PCM, podendo ser afetado qualquer órgão do indivíduo hospedeiro. Em acometimentos ao sistema nervoso central, por exemplo, a PCM causa granulomas e lesões irreversíveis aos órgãos (Felipe et al., 2005; Andrade, 2006). Os dados científicos a respeito da área de incidência e as estatísticas levantadas a respeito dos infectados pela doença são de difícil coleta pelo caráter restrito de sua distribuição geográfica e pelo difícil diagnóstico causado pelo longo período de latência (aproximadamente 15 anos) do fungo no organismo (Felipe et al., 2003; Andrade, 2006) O projeto O Projeto do Genoma Funcional do P. brasiliensis (Felipe and Brígido, 2009) envolve diversos laboratórios da região central do Brasil com o objetivo de coletar informações sobre o transcriptoma do fungo referente à sua forma miceliana como em sua forma de levedura. O projeto conseguiu gerar ESTs de alta qualidade através de dados de cdna do micélio e da levedura do fungo. As ESTs passaram por uma etapa de remontagem (assembly) através do CAP3 (Huang and Madan, 1999), resultando em contigs e singlets, que juntos constituem assembled ESTs (Felipe et al., 2005). Essas assembled ESTs correspondem a aproximadamente 80% do genoma do fungo (Andrade, 2006) ncrnas no P. brasiliensis Os conceitos teóricos relativos a RNAs não codificadores são abordados em detalhes no capítulo 3. A análise funcional do transcriptoma do P. brasiliensis é fundamental para a compreensão de seu mecanismo de diferenciação e de sua patogenicidade e/ou virulência, mas somente se torna possível através de abordagens utilizando sirnas e supressão de genes (Felipe et al., 2005). Desta forma, o escrutínio do transcriptoma do fungo para possíveis identificações de RNAs não codificadores abre possibilidades para a descoberta do meca- 24
39 nismo dimórfico do organismo e o combate eficiente à PCM através de vacinas (Andrade, 2006). O método PORTRAIT foi aplicado às assembled ESTs do fungo e identificou 970 assembled ESTs como potenciais RNAs não codificadores, com boa margem de certeza para a maioria das predições. Comparativamente, o método CPC identificou assembled ESTs como potenciais RNAs não codificadores (Arrial, 2008). 25
40 Capítulo 3 RNAs não codificadores Neste capítulo, as bases biológicas de RNAs não codificadores é explanada, bem como métodos experimentais e computacionais utilizados para identificação de ncrnas em organismos. 3.1 Definição RNAs não codificadores (non coding RNAs ou ncrnas) são transcritos de genes que não expressam mrnas codificadores de proteínas. Pelo contrário, agem diretamente na célula em funções estruturais, catalíticos ou regulatórios (Eddy, 2001). As linhas de pesquisa atuais apontam relações mais extensas entre ncrnas e os mais diversos processos de um organismo. Ainda muito pouco é conhecido, porém, principalmente pela grande dificuldade em verificar experimentalmente qual é exatamente a funcionalidade do determinado gene não codificador no organismo (Machado-Lima et al., 2007). ncrnas têm fundamental papel no controle da expressão de genes em proteínas (Jossinet et al., 2007). Um modelo detalhado da etapa de transcrição, excisão e tradução (abordado na figura 2.8 do capítulo 2), incluindo alguns transcritos de ncrnas atuantes, ajuda a visualizar a amplitude de funções de ncrnas em um organismo eucarioto (figura 3.1). Historicamente, a identificação de trechos de DNA que, transcritos, resultavam em RNAs não codificadores ocorreu na identificação de regiões inter-gênicas sem função aparente, presentes em grandes quantidades em organismos eucariotos complexos (Setubal and Meidanis, 2000). Estudos posteriores sobre o processo de transcrição e tradução realizados por Watson e Crick já postulavam, controversamente, a hipótese de um gene, um ribossomo e uma proteína, dando caráter exclusivo para o RNA de mero sintetizador de proteínas a nível citoplasmático, mas também a provável existência de estruturas mais complexas de RNA como intermediadores de atividades de tradução, como os mais tarde identificados RNAs de transporte (trnas) vieram confirmar (Watson and Crick, 1953; Eddy, 2001). Mais notadamente, hoje é sabido que a porção de RNA não codificador estimada para seres humanos é de 98% (Szymanski et al., 2003). Antes chamados de DNAs lixo - 26
41 Figura 3.1: Exemplo de funções do ncrna nas atividades de transcrição, tradução e excisão em eucariotos. As interrogações representam funcionalidades e processos identificados, mas ainda desconhecidos aos biólogos. junk DNAs - hoje as funcionalidades conhecidas para ncrnas abrangem silenciamento de genes, replicação, regulação da expressão de genes, transcrição, estabilidade de cromossomos, estabilidade de proteínas, translocação, localização e modificação, processamento e estabilidade de RNA (Machado-Lima et al., 2007). Estudos nesta área têm por objetivo principal estabelecer os critérios para distinguir precisamente ncrnas de mrnas e possibilitar uma maior compreensão do mecanismo genético e seus produtos (Frith et al., 2006). 3.2 Classificação de ncrnas As classificações de ncrnas variam conforme sua aparente funcionalidade visualizada no organismo. Ainda há muita discussão sobre a quantidade de ncrnas e sobre como estes são divididos (Eddy, 2001). A tabela 3.1 lista alguns tipos de ncrnas e suas funções mais conhecidas no organismo. 27
42 Tabela 3.1: Alguns tipos de RNAs não codificadores e suas funções conhecidas (Eddy, 2001; Lakshmi and Agrawal, 2007). Sigla Nome Função frna RNA funcional essencialmente idêntico ao RNA não codificador mirna micro RNA família putativa de genes reguladores da tradução. Pertence à classe dos ncrnas estruturais rrna RNA ribossômico RNA constituinte do ribossomo sirna RNA pequeno de interferência moléculas ativas na interferência de RNA. Junto com o mirna, constitui a classe dos ncrnas estruturais snrna Pequeno RNA nuclear incluem RNAs relacionados ao processo de excisão snmrna Pequeno não-mrna essencialmente pequenos RNAs não codificadores snorna Pequeno RNA nucleolar envolvidos na modificação do rrna strna Pequeno RNA temporal interrompem a tradução de mrnas trna RNA de transferência envolvidos na tradução de mrnas pirna RNA Piwi-interativo regulação de tradução e estabilidade de mrna, entre outras funções rasirna Repeat-associated small interfering RNA Silenciamento da transcrição de genes via remodelagem da cromatina Antes de prosseguir aos métodos e abordagens para identificação de ncrnas, é importante frisar a diferença entre classificar e identificar ncrnas. Métodos identificadores, atualmente, têm por objetivo separar seqüências potencialmente não codificadoras de seqüências codificadoras, de forma inequívoca e única. Já métodos classificatórios, mais avançados e com escassa literatura encontrada à respeito, são mais apurados em sua operação, discernindo seqüências potenciais entre diversas classes de ncrna. 3.3 Abordagens experimentais para identificação de ncrnas Não existe um consenso em métodos ou atributos para distinção precisa entre ncrna e mrna na Biologia Molecular contemporânea (Eddy, 2001; Frith et al., 2006). Este fato gerou, por sua vez, a criação de diversos métodos computacionais para resolver o problema de identificação de ncrnas. Estratégias consagradas para identificação e comparação de genes codificadores de proteínas falham ao serem aplicadas em transcritos não 28
43 codificadores (Machado-Lima et al., 2007). Mesmo as seqüências anotadas manualmente têm uma relevante porcentagem de erro: aproximadamente 10% das seqüências manualmente traduzidas no Swiss-Prot são na verdade ncrnas (Frith et al., 2006). A dificuldade em discriminar seqüências genéticas como ncrnas ou mrnas é ainda maior aplicada a longos transcritos, com tamanho superior a 200 nucleotídeos (Dinger et al., 2008). As estratégias computacionais para discriminar transcritos atualmente baseiam-se na identificação de atributos e características específicas de certas classes de ncrnas ou na identificação menos restrita de atributos genéricos de famílias de ncrnas em transcritos. Os atributos utilizados para discriminação podem ser extraídos diretamente da seqüência, por processos ab initio, ou podem ser inferidos por comparação com um banco de dados estabelecido, realizando uma avaliação comparativa (Machado-Lima et al., 2007; Dinger et al., 2008). Apesar do êxito experimental de vários métodos, a discriminação entre seqüências de RNA capazes de atuar tanto funcionalmente na célula quanto gerar produtor protéicos - fenômeno pouco observado mas possivelmente muito comum - não é realizada por nenhum dos métodos atuais (Dinger et al., 2008). Abaixo, as abordagens consideradas dão as linhas gerais de funcionamento de vários métodos baseados nestas estratégias Avaliação termodinâmica A composição e ordenação de nucleotídeos em uma molécula de RNA é responsável por sua conformação no espaço tridimensional. Uma investigação desta conformação, por sua vez, resulta num conhecimento aproximado sobre a organização da molécula e suas propriedades fisiológicas. A avaliação termodinâmica de moléculas de RNA pode ser utilizada em conjunto com várias regras estruturais e topológicas para inferir a estrutura secundária ativa da molécula de RNA (Zuker and Stiegler, 1981). RNAs com uma estrutura secundária bem definida têm energia livre associada menor do que seqüências com mesma freqüência de nucleotídeos, porém sem estrutura secundária definida (Machado- Lima et al., 2007). A partir da análise da mínima energia livre de uma molécula de RNA, é possível, portanto, inferir se a molécula tem uma conformação estável de sua estrutura secundária e se é possível sua atuação a nível funcional na célula Avaliação composicional Há um forte indício em estudos e experimentos de que a ocorrência dos nucleotídeos G ou C é significativamente maior em transcritos de ncrnas (Machado-Lima et al., 2007). Ocorrências do dinucleotídeo CG indicam, em estudos realizados com discriminadores utilizando máquinas de aprendizado supervisionado (Arrial, 2008), que sua conformação quimicamente mais estável do que a dupla AT e T A possibilitam a formação de uma estrutura secundária funcional. Além de avaliações porcentuais de ocorrência, a avaliação por comprimento de fases de leitura ou do próprio transcrito são bastante utilizadas. Experimentalmente, várias classes e tipos de ncrna contém de 15 a 300 nucleotídeos (nt). Aplicado a cadeias de proteínas putativas, a divisão orbita em torno de 100 peptídeos. Estes tipos de divisões, 29
44 quando aplicadas a algoritmos discriminatórios, têm obtido bons resultados experimentais (Arrial, 2008; Liu et al., 2006) Avaliação utilizando aprendizado de máquina Métodos que utilizam aprendizado de máquina mostraram um grau de acurácia elevado (Machado-Lima et al., 2007). CONC (Kong et al., 2007), CPC (Liu et al., 2006) e PORTRAIT (Arrial, 2008) utilizaram máquinas de vetor de suporte treinadas com dois conjuntos: o positivo sendo constituído de características extraídas de trancritos de mr- NAs e o negativo constituído de características extraídas de transcritos de ncrnas. Os atributos extraídos foram, por exemplo, tamanho da fase aberta de leitura, composição de nucleotídeos, estrutura secundária, entre outros (Dinger et al., 2008) Avaliação comparativa Através de comparação de genomas entre duas ou mais espécies, as regiões de similaridade comuns a todas as espécies comparadas é submetida a outras formas de avaliação, como a termodinâmica, para inferir regiões comuns de ncrna. Como dependem previamente de boas bases de genomas para efetuar as comparações, estes métodos são pouco viáveis na prática, e portanto, são pouco utilizados atualmente (Machado-Lima et al., 2007). 30
45 Capítulo 4 Mapas Auto Organizáveis Este capítulo objetiva uma breve explanação dos métodos teórico-matemáticos base para o entendimento do funcionamento de redes neurais artificiais e de mapas auto organizáveis de Kohonen, bem como a explicação dos conceitos importantes utilizados pelo método proposto neste trabalho. 4.1 Redes Neurais Artificiais Uma rede neural artificial é um modelo computacional inspirado no funcionamento do cérebro (Kasabov, 1998). O modelo neuronal humano pode ser visto como um sistema em três estágios: a recepção da informação, sua identificação e a decisão apropriada (Haykin, 1999). Assim como um cérebro é constituído pela associação de vários neurônios, uma rede neural artificial é formada pela associação de vários elementos de processamento: os neurônios artificiais (figura 4.1). Uma unidade neuronal é constituída de: ˆ conexões de entrada (inputs) x 1..x m ; ˆ pesos das conexões de entrada w 1..w m ; ˆ conexão de entrada fixa w 0 : uma conexão de entrada especial, com valor constante C (Kasabov, 1998); ˆ função de entrada : calcula o valor agregado de entrada u = f(x i, w i ), onde x i são as entradas e w i seus respectivos pesos. A função basicamente efetua o somatório u = n i=1 x i.w i ; ˆ um sinal (função) de ativação s: calcula o nível de ativação do neurônio a = s(u). Esta função de ativação pode ser do tipo limiar, linear, sigmóide, hiperbólica e gaussiana (Kasabov, 1998; Mendes and Oliveira, 2009); ˆ uma função de saída f(): calcula o sinal de saída emitido pelo neurônio no seu terminal de saída y k = f(a). O sinal de saída é comumente definido pelo nível de ativação do neurônio, isto é, y k = a. 31
46 Figura 4.1: Representação de um neurônio artificial. A arquitetura neuronal acima é baseada no primeiro neurônio artificial proposto por McCulloch e Pitts (Mendes and Oliveira, 2009). As conexões de entrada e a conexão de saída de um neurônio podem capturar e transmitir qualquer valor pré-estabelecido para outros neurônios ou para si mesmo, através de ligações sinápticas entre sua conexão de saída e uma ou mais conexões de entrada vizinhas. Os valores mais comumente utilizados para o valor transmitido são em escala binária (0, 1), bivalente ( 1, 1), contínua (0, 1) ou através de valores discretos entre dois limitantes, no formato a 0, a 1,.., a n (Kasabov, 1998). Além das unidades neuronais, uma rede neural artificial é caracterizada por sua topologia (as conexões entre os neurônios), por um algoritmo de treinamento, responsável pelo aprendizado da rede, e por um algoritmo de processamento, responsável pela avaliação de novos dados tendo por base o conhecimento adquirido durante o treinamento (Kasabov, 1998) Inteligência Redes neurais associam seus neurônios artificiais refletindo a associação neuronal biológica. Alcançam, a seu modo, os critérios de inteligência identificados para uma rede neural biológica (Kasabov, 1998): ˆ Aprendizado e adaptação: uma rede neural é capaz de reter nova informação, moldando seu circuito através de supressões e estímulos a determinadas entradas do seu conjunto x i de entradas. Também é capaz de adaptar-se a novas informações, alterando estes valores indefinidamente; 32
47 ˆ Generalização: redes neurais generalizam dados recebidos para formalizar uma série de características que os identificam. Essas características, mais ou menos generalizadas a critério da rede, são utilizadas para identificar novos dados; ˆ Paralelismo massivo: Assim como o cérebro, as redes neurais são constituídas de milhares de ligações entre neurônios, e funcionalidades redundantes para neurônios. Assim sendo, o mesmo processamento pode ser realizado concomitantemente por várias unidades neuronais ligadas em paralelo; ˆ Robustez: Caracteriza a plasticidade da rede, isto é, sua capacidade de se moldar a novas configurações espaciais por retirada ou adição de ligações ou unidades neuronais. É um resultado indireto do enorme paralelismo da rede; ˆ Armazenamento associativo de informação: A informação é armazenada de forma a relacionar-se com outros dados já presentes na rede. Assim, ao ser exposto a uma entrada identificada qualquer, a rede é capaz de, além de recuperar a informação relativa à entrada, associá-la a outros dados e informações já presentes. Um exemplo biológico para o armazenamento associativo de informação é a capacidade de associar determinados estímulos sensoriais a eventos; ˆ Processamento espaço-temporal de informação: Além da percepção de estímulos capturados pelas conexões de entrada dos neurônios, a rede neural pode processar informação através da associação com informação relativa à posição espacial de um referido dado, e acompanhar sua mudança com relação ao tempo. O comparativo ao cérebro humano é a capacidade de criar uma sucessão de eventos localizados precisamente no espaço e no tempo, um evento histórico ou identificar uma pessoa apesar de não tê-la visto há muito tempo Representação do Conhecimento O termo conhecimento representa, em Inteligência Artificial (IA), os dados que são considerados pelo sistema para criar uma representação verossímil do ambiente à sua volta. Dados sensoriais, de posicionamento, etc, podem ser armazenados de forma ordenada e contextualizada graças à estrutura das redes neurais, tornando-se informação constituinte do conhecimento da máquina. Em aplicações reais de máquinas inteligentes, uma boa solução depende diretamente de como o conhecimento é armazenado e utilizado (Haykin, 1999). Com relação à representação do conhecimento que manipulará, a construção de boas máquinas inteligentes baseadas em redes neurais deve, portanto, nortear-se na definição de qual conhecimento armazenará, tratará e compartilhará (escopo) e como será feito o armazenamento interno deste conhecimento (estrutura do conhecimento) Redes Neurais Artificiais e Inteligência Artificial O grande objetivo da disciplina de IA é o desenvolvimento de algoritmos e paradigmas que possibilitem a execução de tarefas cognitivas por máquinas. Para este fim, um sistema de IA deve ser capaz de realizar três atividades distintas (Haykin, 1999): 33
48 ˆ armazenar conhecimento através de representação de dados; ˆ aplicar o conhecimento armazenado na resolução de problemas, uma forma primordial de raciocínio; ˆ aprendizado de novos conhecimentos através da experiência. A figura 4.2 esquematiza os três itens acima numa estrutura contínua, representando o sistema inteligente. Figura 4.2: A relação entre os três componentes chave de sistemas inteligentes: Representação, Aprendizado e Conhecimento (Haykin, 1999). A representação do conhecimento por uma IA é feita utilizando símbolos compreensíveis a um leitor humano, possibilitando uma interação simplificada entre homem e máquina. A forma simplificada de raciocínio, apesar de restringir a capacidade de solução de problemas, possibilita uma metodologia de solução do problema equiparável a um problema de busca. Uma forma simplificada de resolver tais problemas é usando a metodologia Regras, Dados e Controle, isto é aplicar regras para interpretar os dados, e ajustar as regras conforme o controle preponderante (Haykin, 1999). O aprendizado de máquina em IAs pode envolver duas formas de processamento de informação: processamento indutivo e processamento dedutivo. No processamento indutivo, o controle cria regras e padrões genéricos através do conhecimento adquirido (chamado de experiência) e dos dados recebidos. No processamento dedutivo, o controle atua no processamento de informação através de regras gerais pré-estabelecidas aplicadas diretamente sobre os novos dados captados, num processo inverso ao da indução (Haykin, 1999). 4.2 Arquitetura de Redes Neurais A topologia de uma rede neural e a organização interna do conhecimento que ela faz definem seu funcionamento, sua forma de treinamento e, finalmente, a gama de problemas que pode solucionar (Haykin, 1999). Os nós de uma rede (as unidades neuronais) podem estar conectados a todos os outros nós (totalmente conexo), ou ligados somente a alguns deles (parcialmente conexo) (Kasabov, 1998). 34
49 Alguns neurônios podem direcionar sua conexão de saída para camadas de entrada de neurônios antecessores, configurando a montagem de uma retroalimentação na rede. Redes neurais são categorizadas pela presença ou não de tais mecanismos de retroalimentação, sendo chamadas de redes cíclicas ou recursivas, quando presentes, ou acíclicas, quando não presentes (Kasabov, 1998). A quantidade de camadas que uma rede possui também é fator classificatório de sua arquitetura (Haykin, 1999). Redes de camada simples têm somente uma camada de entrada e uma camada de saída. Conformação utilizada pelas redes de Kohonen (Kohonen, 2001), são organizadas conforme a figura 4.3. Redes com camadas intermediárias entre a camada de entrada e a camada de saída são categorizadas como redes de múltiplas camadas. As camadas intermediárias são ditas camadas ocultas, e sua presença na rede permite um processamento refinado das entradas antes de serem admitidas aos neurônios de saída (Haykin, 1999). Figura 4.3: Arquitetura de uma rede neural acíclica de camada simples. 4.3 Processos de Aprendizado Aprendizado é a capacidade da rede neural de alterar seu comportamento frente aos estímulos recebidos do ambiente (Kasabov, 1998). O processo de aprendizado em uma rede neural artificial consiste no treinamento desta rede com um conjunto de entradas 35
50 selecionado de forma a fazê-la aprender as características e atributos relevantes destes dados. Este conjunto especial é chamado conjunto de treinamento (Kasabov, 1998). Numa etapa de treinamento supervisionado, a rede pode ser apresentada a um conjunto de pares x i de entrada e y i de respostas esperadas. A rede adaptará sua função f(a) para atingir estes valores. Já numa etapa de treinamento não supervisionado, a rede é apresentada somente ao conjunto de entradas x i, absorvendo somente suas características internas. Alguns métodos de aprendizado utilizado são Aprendizado Erro-Correção, Aprendizado baseado em Memória, Aprendizado Hebbiano, Aprendizado de Boltzmann, Aprendizado Não Competitivo e Aprendizado Competitivo (Haykin, 1999; Kasabov, 1998) Aprendizado Competitivo O aprendizado competitivo é uma regra no aprendizado não supervisionado em que vários neurônios receberem estímulos em suas conexões de entrada, mas somente um é efetivamente ativado. A ativação se dá após uma competição pelo maior estímulo entre os neurônios envolvidos: somente o neurônio vencedor é ativado (Kasabov, 1998). 4.4 Perceptrons Figura 4.4: Arquitetura de um perceptron de camada simples. Perceptrons representam uma das primeiras abordagens experimentais para modelagem de processos cognitivos por agentes artificiais inteligentes (Kasabov, 1998). Normalmente são redes acíclicas de camada simples, constituída por neurônios com função de ativação s(u) do tipo limiar, com aprendizado supervisionado, do tipo erro-correção (Kasabov, 1998; Mendes and Oliveira, 2009). 36
51 O processo de aprendizado de um perceptron de camada simples é exibido abaixo. Para esta observação, algumas declarações precisam ser refeitas para se adequar ao algoritmo exibido em 4.1: Declarações: X(n) = vetor de conexões x 1..x m dos neurônios da camada de entrada; W (n) = vetor de pesos das conexões w 1..w k entre os neurônios (k conexões); b(n) = vetor das conexões de entrada fixas w 0 dos neurônios da camada de entrada; y(n) = vetor de respostas reais y i..y l dos neurônios; d(n) = vetor de respostas desejadas d i..d l para os neurônios; e(n) = vetor de erros das saídas e i..e l do perceptron; α(n) = taxa de aprendizagem, uma constante positiva entre 0 e 1; n = o valor da época atual do processo de aprendizado (número de iterações do algoritmo); C = valor mínimo desejado para o erro e(n). Tabela 4.1: Passos do algoritmo de aprendizado por erro-correção de um perceptron (Kasabov, 1998; Mendes and Oliveira, 2009). P1 P2 Inicializar os valores do vetor W (0) e da taxa de aprendizado α(0) Apresentar o vetor de entrada X(n) e a saída desejada d(n), de cada par do conjunto de treinamento T = (x i, d j ) P3 Calcular a resposta real do perceptron, da seguinte forma: y(n) = f(w (n) X(n) + b(n)), onde f(.) é a função de ativação do tipo limiar P4 Calcular o erro da saída da unidade da seguinte forma: e(n) = d(n) y(n); P5 P6 Atualizar o vetor de pesos para cada uma das unidades da rede segundo a regra: W (n + 1) = W (n) + α(n) e(n) X(n); Incrementar o passo de tempo (n) e voltar a P2, até que e(n) = C para todos os elementos do conjunto de treinamento em todas as unidades da rede Os perceptrons de camada simples são máquinas de operação limitada, capazes de resolver problemas de discriminação entre classes linearmente separáveis (Kasabov, 1998). 37
52 (a) Linearmente separáveis. (b) Não linearmente separáveis. Figura 4.5: Exemplos de classes. Classes linearmente separáveis e classes não linearmente separáveis são retratadas na figura 4.5. Perceptrons Multi-Camadas (PMC) são implementações mais complexas, onde, além das duas camadas do perceptron de camada simples, incluem uma ou mais camadas intermediárias (camadas ocultas). 4.5 Auto-Organização O conceito de auto-organização de uma rede neural fundamenta-se num aprendizado não supervisionado de uma rede neural baseado na modificação dos pesos das conexões entre os neurônios da rede, até que uma configuração global se estabeleça Mapas Auto Organizáveis Em uma rede neural baseada em mapas auto organizáveis (também chamados SOMs ou Self Organizing Maps), as unidades neuronais são dispostas como nós de um mapa de coordenadas euclideanas. A rede neural recebe por entrada estímulos sob forma de vetores n-dimensionais e os posiciona em um mapa discreto de dimensões reduzidas, geralmente mono, bi ou tridimensionais (Haykin, 1999; Kohonen, 2001). O modelo de funcionamento de mapas auto-organizáveis proposto por Kohonen é o mais difundido e utilizado, por permitir uma abordagem mais generalizada do problema de criação de mapas computacionais ordenados topologicamente utilizando redução dimensional nos dados de entrada (Haykin, 1999). A figura 4.6 representa um mapa auto organizado bidimensional do modelo de Kohonen. A camada de entrada da SOM, composta por unidades sem capacidade computacional, admite um vetor de entrada x(n) = [x 1 (n)x 2 (n)... x i (n)], que geralmente é uma série de entradas numéricas contínuas ou discretas (Kasabov, 1998). n refere-se ao número de épocas (iterações) do algoritmo de aprendizado da rede. 38
53 Figura 4.6: Arquitetura simplificada de uma rede neural baseada em mapas auto organizáveis de Kohonen e seus principais componentes. A camada de saída é formada pelas saídas y 1 (n), y 2 (n),..., y j (n) das unidades neuronais (nós) que compõem o mapa auto organizado. Os vetores de peso w j (n) = [w 1,1 (n)w 1,2 (n)... w i,j (n)], também chamados vetores de referência, conectam cada vetor de entrada x i (n) com todos os nós y j (n) do mapa. Na figura 4.6, a topologia que ordena os nós sobre o mapa bidimensional é a retangular, evidenciada pelas ligações dos quatro neurônios superiores. Existem várias outras topologias utilizadas em mapas auto organizáveis, com destaque para a topologia hexagonal, muito empregada em máquinas de reconhecimento de padrões visuais (Haykin, 1999). O algoritmo apresentado na tabela 4.2 mostra o processo de aprendizado de um mapa auto organizável de Kohonen. Em suma, o processo de aprendizado de uma rede neural baseada em SOM segue três etapas importantes: a etapa de Competição, a etapa de Cooperação e a etapa de Adaptação dos Pesos Sinápticos. O passo P 2 e P 3 do algoritmo 4.2 correspondem à etapa competitiva de treinamento. Já cálculo de V n no final da etapa P 3 correponde à etapa cooperativa entre os neurônios. No passo de adaptação dos pesos sinápticos (passo 39
54 Tabela 4.2: Passos do algoritmo de aprendizado de um mapa auto organizável de Kohonen (Kasabov, 1998). P0 P1 Designam-se pesos aleatórios pequenos para os vetores de referência w j (0) Apresenta-se um vetor x(n) no respectivo momento n P2 Calcula-se a distância j (num espaço i-dimensional) entre x(n) e w j (n), ou seja, para cada neurônio j. Num espaço Euclideano o cálculo é feito por j = (( x(n) wj (n)) 2 ) P3 P4 O neurônio com função de ativação y k (n) mais próxima de x(n) é declarado o neurônio vencedor. Ele se torna o centro de uma nova área de vizinhança V n Altera-se o peso dos vetores de referência da seguinte forma: w j (n + 1) = w j (n) + hj(n)α(n)( x(n) w j (n)), sej V n w j (n + 1) = w j (n), caso contrário onde hj(n) é a função de vizinhança adotada para a rede P5 Retorna ao passo P1. Os valores de α(n) e V n decrescem com o número crescente de épocas P 4 do algoritmo), a função de vizinhança hj(n) escolhida pode ser de diversos tipos, sendo mais comumente empregado a função gaussiana (Haykin, 1999). A etapa adaptativa do algoritmo pode ser ainda subdividida em uma etapa de ordenação do mapa e uma posterior etapa de convergência. Na etapa de ordenação, os pesos w j (n) são organizados e ordenados no mapa, o que requer em torno de iterações do algoritmo de treinamento (Haykin, 1999). Já na etapa de convergência, ocorre uma verificação detalhada da configuração da rede, utilizando uma quantidade bastante superior de iterações e uma taxa de aprendizado α(n) comparativamente menor do que a utilizada na etapa de ordenação (Kohonen et al., 1996). O erro de quantização médio (average quantization error ou AQE) é uma medida estatística da acurácia do treinamento de SOMs (Kohonen et al., 1996). Seu cálculo, num espaço Euclideano, é dado por n i=1 (( x(i) w k(i)) 2 ), onde w k (i) são os vetores de referência para um neurônio vencedor y k (i) computado na i-ésima etapa de treinamento do algoritmo. 40
55 Capítulo 5 Processamento Distribuído e Projeto EELA-2 As bases teóricas de sistemas e processamento distribuído são rapidamente explanadas neste capítulo. O projeto de cooperação EELA-2 é apresentado, e os principais componentes do middleware glite são brevemente abordados. 5.1 Processamento Distribuído Por ser uma área de recente expressão na comunidade mundial, não há um consenso completo quanto à definição de processamento ou sistema distribuído. Sistemas distribuídos abrangem diversas formas de organização de computadores, desde a Internet e intranets a sistemas ubíqüos (Coulouris et al., 2000), e grids de computadores. A definição usual encontrada relaciona ações coletivas coordenadas de múltiplos elementos de processamento, ou seja, um processamento distribuído de dados. Refinando esse conceito geral, pode-se extrair características intrínsecas a sistemas distribuídos, como segue (Wu, 1999): ˆ Paralelismo de ações conjuntas sobre um conjunto de dados coordenado por uma única thread ou processo de controle; ˆ Concorrência de ações, podendo essas serem executadas em qualquer ordem; ˆ Distribuição computacional, ou seja, a performance de uma tarefa executada em meio distribuído é dependente da comunicação de dados interprocessos e do controle desses dados e processos. Outra abordagem para definir sistemas distribuídos envolve a adição das seguintes características à lista supracitada (Wu, 1999; Coulouris et al., 2000): ˆ Ausência de clock global, pois os processos e recursos de um sistema distribuído se comunicam somente através de troca de mensagens e, portanto, não têm uma noção exata do tempo corrente; 41
56 ˆ Tolerância a falhas, tratamento de falhas e redundância, traduzidas como uma independência de certos componentes do sistema distribuído da finalização correta ou não da tarefa de outros elementos, ou da conectividade da rede; ˆ Plasticidade da rede de comunicação entre os elementos do sistema distribuído, isto é, uma boa capacidade de manter seus serviços apesar da falha de alguns componentes. Finalmente, pode-se considerar um sistema distribuído aquele que (Wu, 1999): ˆ Contém múltiplos elementos de processamento; ˆ Contém um hardware para intercomunicação (uma placa de rede, por exemplo); ˆ Os elementos de processamento falham de forma independente (tolerância a erros e tratamento); ˆ Estados de processamentos são compartilhados. É importante diferenciar as duas formas básicas de organização de computadores, determinadas pela distribuição física da memória no sistema, representadas pela figura 5.1. O modelo representado na figura 5.2(a) representa um sistema fortemente acoplado, em que dados e comandos são compartilhados numa memória global única para todas as CPUs. A comunicação interprocessos é feita através de comandos de escrita e leitura nessa memória compartilhada. Já a figura 5.2(b) representa um sistema fracamente acoplado, onde as várias CPUs contam com uma unidade local de memória para operar, e trocam somente mensagens entre si através de comandos de envio e de recebimento de mensagens. O par CPU - Memória local é denominado elemento de processamento (Wu, 1999) Classificação de Flynn A arquitetura de von Neumann, usual para computadores tanto em sistemas distribuídos como em sistemas não distribuídos, pode ser subcategorizada em uma taxonomia própria para sistemas distribuídos, a classificação de Flynn. Essa classificação toma por base a multiplicidade de fluxos de instruções e de fluxos de dados. A classificação de Flynn define (Wu, 1999): ˆ Single Instruction Single Data (SISD): A visão clássica do modelo Von Neumann, com uma CPU compartilhando um fluxo de instruções e um fluxo de dados com a memória. Mecanismos como pipeline, tratamento de interrupções de dispositivos de entrada/saída são paralelismos possíveis nesta categoria; ˆ Single Instruction Multiple Data (SIMD): Cada CPU executa um fluxo único de instruções em diferentes conjuntos de dados; ˆ Multiple Instruction Single Data (MISD): Múltiplas CPUs executam múltiplos fluxos de instruções, porém, são governadas por uma única e distinta unidade de controle; 42
57 (a) Sistema fortemente acoplado. (b) Sistema fracamente acoplado. Figura 5.1: Tipos de organização básica de computadores (Wu, 1999). ˆ Multiple Instruction Multiple Data (MIMD): A forma mais comumente adotada para sistemas distribuídos, constitui-se de diversas CPUs executando diversos fluxos de instruções em vários conjuntos de dados. Apesar de bastante prática e utilizada, esta classificação não abrange várias decisões de design de redes, tais como tipo de paralelismo utilizado, comunicações interprocesso através de uso de memória compartilhada ou por troca de mensagens, sistemas de uso geral ou sistemas de uso específico, tipo de memória utilizada, controle centralizado ou distribuído do sistema, tipo de acesso a dados e componentes do sistema distribuído, e tipo de conexão utilizada (Coulouris et al., 2000). A denominação grid, mencionada anteriormente, está intrinsecamente relacionada ao conceito de sistemas distribuídos. Grids são agrupamentos de computadores numa relação fracamente acoplada do tipo MIMD, ou seja, contam com diversos fluxos de instruções e diversos dados e recursos compartilhados em seu ambiente distribuído Desafios do processamento distribuído O grande motivo para a construção de sistemas distribuídos reside em sua capacidade ampliada de compartilhamento de recursos. Os desafios para implementação e funcio- 43
58 namento de sistemas distribuídos são vários. Entre eles, pode-se citar (Coulouris et al., 2000): ˆ A heterogeneidade de hardware, de componentes e de software, sistemas operacionais e linguagens de programação utilizadas, entre outros; ˆ A implementação de interfaces reutilizáveis e de fácil compreensão, de documentação detalhada e atualizada para permitir constantes manutenções e melhorias, algo corriqueiro em sistemas distribuídos; ˆ A segurança de sistemas distribuídos, particularmente a privacidade dos dados de usuários armazenados no sistema e medidas adotáveis para evitar ataques de negação de serviço (Denial of Service ou DoS) e execução de código móvel malicioso; ˆ Escalabilidade dos sistemas distribuídos, isto é, sua capacidade de operar de mesma forma em qualquer escala, desde pequenas redes locais até grandes redes, como a Internet. O projeto do sistema distribuído deve minimizar ou evitar situações de gargalos (bottlenecks na execução de tarefas, esgotamento de recursos computacionais e perda de performance dos componentes; ˆ Erros em componentes computacionais de sistemas distribuídos devem ser detectados, mascarados e/ou tratados de forma ágil e eficiente, sem comprometer o funcionamento do sistema. Redundâncias de componentes essenciais devem ser projetadas, neste ínterim; ˆ O projeto de sistemas distribuídos deve possibilitar o acesso a um determinado recurso ou dado compartilhado a vários usuários de forma concomitante, assegurando sua sincronização; ˆ Transparência, ou mascaramento, dos diversos componentes, fisicamente ou logicamente distintos no sistema distribuído, de forma a homogeneizar e facilitar o acesso e utilização do sistema por usuários e programadores Arquiteturas Distribuídas Projetos de arquitetura de sistemas distribuídos levam em conta os vários desafios listados na seção 5.1.2, além de procurar sempre satisfazer as exigências e demandas dos usuários finais do sistema. Para isto, definem uma série de componentes e suas variadas formas de comunicação, e os agrupa em camadas. Camadas de software, no âmbito de sistemas distribuídos, pode ser definido em função de serviços e recursos requeridos e ofertados entre processos localizados num mesmo elemento de processamento ou em diversos elementos de processamento. Esta visão orientada a serviços é representada pela figura 5.2. Na camada superior, os vários serviços disponíveis no sistema distribuído são oferecidos por um ou mais servidores (Coulouris et al., 2000), utilizados pelo próprio sistema e/ou por processos de usuários, de forma coordenada e bem definida. A plataforma do sistema distribuído encapsula as camadas de mais baixo nível de hardware e software, e o middleware é a camada responsável por mascarar a heterogenei- 44
59 Figura 5.2: Camadas de serviços de software e hardware em sistemas distribuídos. dade das diversas plataformas que o sistema distribuído utiliza, e oferecer para a camada acima, de aplicações e serviços, um modelo de programação conveniente de fácil utilização. As estratégias utilizadas em middlewares para atingir esse nível de abstração são as mais diversas: pode-se citar a utilização de chamada de procedimentos remotos (Remote Procedure Call, ou RPC ) e a utilização de interfaces de troca de dados entre processos como a MPI (Message Passing Interface), entre várias outras. As diferentes arquiteturas ou modelos de sistemas distribuídos podem ser classificados, simplificadamente, como arquitetura cliente-servidor ou arquitetura de peers processes (pares de processos) (Coulouris et al., 2000). Mais detalhadamente, a arquitetura cliente-servidor contém outras duas importantes arquiteturas, a de serviços oferecidos por múltiplos servidores e a de servidores de proxy e caches. Arquitetura Cliente-Servidor É a arquitetura mais comumente encontrada em sistemas de computação, por sua configuração e funcionamento bastante simplificados (Coulouris et al., 2000). Os processos na máquina cliente acessam o serviço e seus recursos oferecido pelo servidor. O servidor, por sua vez, pode se comportar como um cliente de outro serviço localizado em outro servidor, como visto na figura 5.3. A troca de informações entre um cliente e vários servidores de serviços independentes muitas vezes é feita sem necessidade de sincronização de dados e estados. Como dito anteriormente, a arquitetura cliente-servidor pode ter duas imple- 45
60 Figura 5.3: Diagrama representativo da arquitetura cliente-servidor. Os quadrados cinzaescuros são os elementos de processamento, e os círculos claros são os processos. mentações variantes para problemas específicos: a arquitetura de serviço disponibilizado por múltiplos servidores e a arquitetura de servidores de proxy e cache. Arquitetura Serviço/Múltiplos Servidores Figura 5.4: Diagrama representativo da arquitetura de um serviço distribuído em múltiplos servidores. Vários servidores podem ser utilizados para prover a um cliente um determinado serviço. Os servidores compartilham dados e recursos necessários ou replicam cópias locais desses em cada servidor. A replicação de servidores para prover um serviço me- 46
61 lhora a performance do serviço, distribuindo a carga de invocações de clientes entre os vários servidores, além de garantir, até certo ponto, a disponibilidade de dados, recursos e do serviço mesmo no caso de falhas. A figura 5.4 esquematiza a arquitetura de um serviço fornecido por vários servidores. Os servidores comunicam-se entre si para sincronizar suas cópias e informações locais. Arquitetura de Servidores de proxy e cache Servidores de proxy avaliam a invocação do cliente, procurando uma cópia local (cache) atualizada do dado requisitado. O processo de recuperação de cache é normalmente muito mais rápido do que proceder à requisição do dado ou serviço a um servidor e recuperar seu resultado. Essa implementação é muito difundida no ambiente web (Coulouris et al., 2000). A figura 5.5 representa esta arquitetura, em que o servidor de proxy, localizado entre os clientes e os servidores dos serviços requisitados pelos clientes, é, normalmente, localizado fisicamente mais próximo dos clientes, permitindo uma recuperação das informações armazenadas em si muito mais rapidamente. Figura 5.5: Diagrama representativo da arquitetura de servidores de proxy. Arquitetura de peer processes Na arquitetura de processos em pares (peer processes) não há distinção hierárquica entre cliente e servidor. Todos os pares compartilham informações e dados sobre o processo executado cooperativamente. O código coordenado incorporado ao processo executado garante a sincronização de ações e métodos da aplicação e a consistência de recursos e dados utilizados pela aplicação. Por não haver distinção entre clientes e servidores, não ocorrem atrasos na comunicação interprocessos (Coulouris et al., 2000). A arquitetura de peers é representada pela figura 5.6. A troca de dados e objetos entre os pares, representadas pelas setas, é feita constantemente através das diretivas determinadas pelo código coordenado. 47
62 Figura 5.6: Diagrama representativo da arquitetura de peers. 5.2 Middleware glite Dada a definição de middleware na seção 5.1.3, pode-se avançar ao detalhamento técnico do middleware glite, base para a confecção da aplicação distribuída do método proposto neste trabalho. Figura 5.7: Camadas de abstração de um grid baseado no middleware glite (Projeto EEGE, 2007). 48
63 O middleware de grids glite, versão 3.1, é construído a partir de vários projetos, entre eles os projetos dos middlewares Globus, DataGrid, entre outros (Burke et al., 2008). A figura 5.7 representa a abstração em camadas de alguns serviços fornecidos pelo middleware. A arquitetura complexa do glite conta com diversos componentes e elementos distintos, fornecendo diversos serviços tanto na camada de mais baixo nível (middleware de base) quanto na camada de mais alto nível, que fornecem os serviços fundamentais para a utilização do grid pelas aplicações de usuários. Os principais componentes utilizados pelo modelo distribuído do método implementado neste trabalho são brevemente explanados na seção abaixo Elementos do glite Os vários serviços oferecidos pelo grid podem ser acessados através de interfaces especialmente criadas (interfaces de usuário) ou pelas aplicações diretamente, utilizando uma variada gama de APIs. Organizações Virtuais Organizações Virtuais são entidades lógicas abstratas que agrupam usuários, instituições e recursos da infraestrutura do grid de acordo com seu domínio administrativo (Burke et al., 2008). Antes que um usuário possa utilizar os recursos computacionais do grid, deve aceitar o termo de uso do projeto que o gerencia e registrar-se num Serviço de Registro. O glite conta com uma infraestrutura básica de segurança (Grid Security Infrastructure ou GSI ) com autenticação através de chaves públicas, criptografia RSA, certificados digitais X.509 e protocolos de comunicação criptografados utilizando Secure Socket Layer (SSL). O certificado digital deve ser emitido por uma Autoridade Certificadora (Certification Authority ou CA) registrada e considerada confiável pelo projeto (Burke et al., 2008). Esse certificado de usuário é utilizado pelo servidor de proxies do grid (MyProxy server) para gerar outro tipo especial de certificado, o certificado de proxy. Esse certificado tem prazo de expiração limitado, e é por meio dele que os vários serviços do grid podem ser utilizados. Interface de Usuário Os pontos de acesso da infraestrutura do grid são as interfaces de usuário (User Interfaces ou UI ). Basicamente, uma UI contém os dados e certificados pessoais do usuário necessários para acesso e utilização do grid. Podem também comportar alguns serviços necessários para operações básicas no grid, tais como envio, gerenciamento e monitoramento de tarefas, transferência de dados de/para o grid e consulta aos recursos do grid. 49
64 Sítios Sítios (sites) são abstrações lógicas do glite que agrupam elementos de processamento (Computing Elements ou CE) e elementos de armazenamento (storage elements ou SE). Normalmente, sítios agrupam CEs e SEs próximos fisicamente. Um elemento de processamento é um conjunto de recursos computacionais, incluindo um cluster de nós de processamento (Worker Nodes ou WN ), uma interface genérica para o cluster (Grid Gate) e um sistema local de gerenciamento de recursos (Local Resource Management System ou LRMS). As tarefas são executadas nos diversos nós de processamento. É importante diferenciar o elemento de processamento descrito para o grid, um agrupamento de WNs, do elemento de processamento descrito na seção 5.1, um computador. Já um elemento de armazenamento (Storage Element ou SE) tem por função uniformizar o acesso a recursos de armazenamento de dados (Burke et al., 2008). Um SE pode ser constituído por vários tipos diferentes de mídias de armazenamento, suportando diversos tipos de protocolos de acesso a dados. A transferência de dados de uma UI para um SE utiliza o protocolo FTP seguro da infraestrutura de segurança do grid (GSIFTP). SEs são usualmente controlados por um Gerenciador de Recursos de Armazenamento (Storage Resources Manager ou SRM ), responsável pela transparência de operações entre diferentes tipos de mídia de armazenamento e monitoramento de espaço em disco, entre vários outros. O gerenciamento de dados, além do SRM, conta com vários serviços e possibilidades. Arquivos no grid podem ter réplicas em vários sítios diferentes, e seu nome lógico (Logical File Name, ou LFN ) é uma das formas de identificação única do arquivo no grid. Através deste identificador, o usuário é capaz de acessar, copiar, mover e remover o arquivo de que é dono. Os sistemas responsáveis pelo gerenciamento de dados mantém um catálogo de réplicas e nomes lógicos dos arquivos no grid, o catálogo de arquivos do grid (LFC ). Sistema de Informações De uma forma geral e abrangente, o Sistema de Informações (Information System ou IS) permite a consulta do estado de recursos do grid. Através de consultas ao IS, recursos são consultados e descobertos (Burke et al., 2008). A informação é publicada no catálogo BDII (Berkeley Database Information Index), e sincronizada através de todo o grid pelo IS. Sistema de Gerenciamento de Tarefas O Sistema de Gerenciamento de Tarefas (Workload Management System ou WMS) é o elemento fundamental para o uso do poder computacional do grid. O sistema é responsável por aceitar tarefas (jobs), designá-las ao CE adequado, registrar seu estado e recuperar seu resultado (Burke et al., 2008). O envio de tarefas ao WMS é descrito por uma linguagem própria, a Linguagem de Descrição de Tarefas (Job Description Language ou JDL), responsável por especificar as exigências para escolha do CE, arquivos a serem 50
65 importados ao sítio para execução da tarefa, além de vários outros parâmetros e diretivas. O serviço de logging e bookkeeping (LB) se responsabiliza, após a submissão da tarefa, de acompanhar seu estado Fluxo de uma tarefa Compreender os principais passos de execução de uma tarefa na infraestrutura glite é fundamental para desenvolver aplicações de execução neste middleware de sistemas distribuídos. A figura 5.8 representa o fluxo de uma tarefa através do WMS, e os estado da tarefa e seu fluxo no grid, explicados nos itens representados por letras. Figura 5.8: Fluxo de uma tarefa através dos componentes integrantes do grid e seus correspondentes estados (Burke et al., 2008). O usuário, munido de certificado digital válido e devidamente registrado numa VO, cria um proxy temporário para as comunicações criptografadas com o grid. a: o usuário submete a tarefa ao WMS, informando, na JDL, os dados de entrada e saída necessários à execução do programa, o executável a ser utilizado para realizar o processamento, entre outros dados. Os dados são copiados temporariamente para o WMS do grid. O evento é registrado no LB e o estado da tarefa é alterado para Submetida (Submitted); 51
66 b: O WMS procura pelo melhor CE disponível para executar a tarefa. O Banco de Informações (cache das informações do IS) é consultado através do BDII. Outro evento é registrado no LB e o estado da tarefa é alterado para Em Espera (Waiting); c: O WMS cria códigos wrappers em torno da tarefa a ser enviada ao CE, junto com outros parâmetros. O evento é registrado no LB e a tarefa passa para o estado Pronta (Ready); d: O CE recebe a tarefa e a envia para execução local. Novamente, o evento é registrado no LB e a tarefa tem seu estado alterado para Agendada (Scheduled); e: O CE gerencia, através do LRMS, a execução das tarefas nos WNs. Os dados de entrada são copiados do WMS para os WNs disponíveis onde serão executados as tarefas. O evento é registrado no LB e a tarefa recebe o estado Em Execução (Running); f: Enquanto a tarefa está sendo executada, arquivos do SE podem ser acessados por protocolos de acesso; g: A tarefa pode produzir grandes arquivos de saída que precisam ser disponibilizados a outros serviços do grid. Estes arquivos podem ser carregados no SE do grid, utilizando os serviços de gerenciamento de dados específicos; h: Terminada a terafa sem erros, seus dados de saída (pequenos dados especificados na JDL) são transferidos de volta ao WMS. O evento é registrado no LB e o estado da tarefa é alterado para Concluída (Done); i: O usuário pode recuperar os dados de saída de volta para sua UI. Caso o faça, o evento é registrado no LB e o estado da tarefa é finalmente alterado para Recuperada (Cleared); j: Consultas ao estado atual da tarefa podem ser feitas ao serviço LB. Também é possível consultar o estado dos recursos de CE e SE do grid, entre outros, através do BDII ; k: Se o sítio ao qual a tarefa foi designada não conseguir aceitar ou executar a tarefa, ela pode ser automaticamente retransmitida a outro CE que satisfaça seus requerimentos. Após um máximo de tentativas especificado pela JDL, a tarefa é cancelada, o evento é registrado no LB e seu estado é alterado para Cancelada (Aborted). 5.3 Projeto EELA-2 O projeto EELA-2 (E-Science Grid facility for Europe and Latin America) tem por objetivo principal construir e manter um ambiente grid de alta capacidade, fornecendo em escala global e de forma ininterrupta acesso aos serviços de processamento e armazenamento distribuídos, através de recursos compartilhados por uma rede de computadores (Gavillet, 2008). O ambiente é desenvolvido para suportar qualquer categoria de aplicação científica, oferecendo todos os serviços necessários à aplicação. 52
67 O projeto EELA-2 é uma sucessão do bem-sucedido projeto EELA, que lançou as bases para a confecção de uma plataforma distribuída sustentável (Gavillet, 2008). O projeto vincula-se, hierarquicamente, ao projeto EGEE (Enabling Grids for E-Science), projetista do middleware glite. Os projetos EELA-2 e EGEE compartilham, por sua vez, vasta porção de suas infraestruturas com o projeto WorldWide LHC Computing Project, ou WLCG, vinculado ao projeto Large Hadron Collider do Laboratório Europeu de Física de Partículas (CERN ). O projeto encerra, atualmente, uma infraestrutura de seu grid com mais de 200 sítios em diversas nações, acumulando cerca de CPUs e 20 PB de espaço de armazenamento (Burke et al., 2008). O projeto EELA-2 tem a participação de 50 instituições, entre universidades, centros de tecnologia e laboratórios de pesquisa (Gavillet, 2008). O Brasil, através da Universidade Federal do Rio de Janeiro (UFRJ ) participa também de uma Unidade de Pesquisa Conjunta (Joint Research Unit ou JRU ), atuando ativamente para a melhoria constante do middleware glite e da infraestrutura do grid. Como histórico da participação da Universidade de Brasília no projeto, pode-se citar a implementação de uma versão distribuída do algoritmo BLAST, batizada de Dist-BLAST, que acumulou bons resultados na diminuição do tempo de processamento do algoritmo (Ciuffo and Mayo, 2009). 53
68 Capítulo 6 Materiais e métodos Com a proposta de discriminar entre sequências mrna e ncrna utilizando o potencial inexplorado de múltiplas classes proporcionado pelas redes de Kohonen, o algoritmo SOM- PORTRAIT - Self-Organizing Maps strategy for Prediction of Transcriptome ncrna by Ab Initio Methods - é proposto. O seguinte capítulo é dividido entre materiais e métodos utilizados para construir o método SOM-PORTRAIT, e, mais adiante, materias e métodos utilizados para construir sua variante distribuída, o método Dist-SOM-PORTRAIT. 6.1 O método SOM-PORTRAIT O método SOM-PORTRAIT utiliza procedimentos ab initio de avaliação de atributos para identificação de ncrnas transcriptômicos. A concepção do método baseia-se na coleta de atributos e características do RNA e confrontação deles a uma rede de Kohonen previamente treinada para classificação em três classes distintas: Coding, Noncoding e Undefined. A classe Coding abarca as sequências de RNA classificadas pelo SOM- PORTRAIT como mrna. A classe Noncoding abarca sequências de RNA classificadas pelo SOM-PORTRAIT como ncrna, e finalmente, a classe Undefined é uma divisão da classificação presumidamente composta por uma subclasse de sequências de RNA pertencente a um grupo específico de ncrna com características próprias Ambiente de trabalho utilizado O ambiente de trabalho utilizado para treinamento e execução dos tesses foi formado por uma máquina com processador de dois núcleos Intel Core 2 Duo (2,0Ghz), Mb de RAM e Sistema Operacional Linux Ubuntu 8.04 (kernel generic) Construção do conjunto de treinamento O resumo das operações referentes à construção do conjunto de treinamento é ilustrado na figura
69 Figura 6.1: Passos para confecção do conjunto de treinamento (Arrial, 2008). O conjunto de treinamento para a rede de Kohonen do SOM-PORTRAIT foi fornecido pelos autores do método PORTRAIT, e utilizado sem modificações. O conjunto de treinamento é formado por dois subconjuntos de sequências de RNA: um subconjunto positivo formado exclusivamente por mrna, nomeado dbcod, e um subconjunto negativo formado exclusivamente por ncrnas, nomeado dbnc. Os dois conjuntos contém sequências dos mais variados organismos, conferindo ao conjunto de treinamento, portanto, uma grande generalidade, pouco observada em métodos classificatórios dessa natureza (Arrial, 2008). A tabela 6.1 contém a composição desse conjunto de treinamento. Tabela 6.1: Composição do conjunto de treinamento (Arrial, 2008). Conjunto Componente Conteúdo Sequências dbcod Sequências do cdnas das proteínas do Swiss EMBL Prot dbnc NONCODE ncrnas estruturais e semelhantes a mrnas, exceto RNAt e rrna dbnc Rfam ncrnas estruturais dbnc RNAdb unificado ncrnas de todas as classes O conjunto positivo dbcod foi obtido diretamente do banco de dados SwissProt, versão 50.8, em Outubro de Sequências com identidade maior do que 70% tiveram somente um representante selecionado, utilizando o programa CD-HIT. O arquivo FASTA 55
70 resultante é constituído somente por proteínas não redundantes. A partir da análise dos códigos identificadores de cada proteína desse arquivo, os seus respectivos nucleotídeos foram recuperados no banco de nucleotídeos do EMBL. As proteínas que não possuiam a correspondente entrada nesse banco foram automaticamente descartadas. Proteínas com mais de uma entrada no banco de nucleotídeos EMBL tiveram todas suas ocorrências incluídas. A partir dessa análise, entradas duplicadas foram eliminadas, e as entradas resultantes foram utilizadas para o download das correspondentes sequências em formato FASTA no banco de dados EMBL (versão de 11 de Outubro de 2006). Sequências pertencentes a cromossomos ou genomas inteiros foram descartadas. Do conjunto de sequências resultante dessas operações, sequências com quantidade de letras maior do que também foram descartadas para evitar problemas de alocação de memória nos programas ANGLE e CAST. Sequências com quantidade de letras menor do que 80 também foram descartadas, pois peptídeos traduzidos a partir desses transcritos são pequenos demais para uma boa análise por algoritmos de aprendizado de máquina (Liu et al., 2006). As sequências foram novamente submetidas a uma nova eliminação de redundância pelo programa BLASTCLUST. Finalmente, as sequências resultantes tiveram suas fases abertas de leitura preditas pelo programa ANGLE (Shimizu et al., 2006). Para a seleção da ORF, um algoritmo de programação dinâmica define o melhor produto protéico entre as seis ORFs encontradas para a seqüẽncia. A partir dessa análise de ORFs, dois subconjuntos de dbcod são definidos: o conjunto de sequências com ORF presente (dbtr OP) e o conjunto de sequências com ORF ausente (dbtr OA). Para o conjunto dbnc, as sequências em formato FASTA foram descarregadas diretamente dos bancos de dados NONCODE, Rfam e RNAdb, em outubro de Sequências redundantes foram eliminadas utilizando novamente o programa BLAST- CLUST. Sequências com número de letras acima de e abaixo de 80 também foram eliminadas pelos mesmos motivos apresentados para o dbcod. Finalmente, as sequências foram traduzidas pelo ANGLE e as sequências com ORF presente foram reunidas e somadas ao conjunto dbtr OP, enquanto que as sequências com ORF ausente foram somadas ao conjunto dbtr OA Descrição do método O método SOM-PORTRAIT envolve a utilização de uma série de procedimentos e ferramentas. O método baseia-se em avaliação ab initio de transcritos para identificação em RNA codificador ou não codificador, por meio de uma rede neural especialmente treinada para esse fim. O método recebe por parâmetro de entrada um arquivo em formato multifasta válido (NCBI, 2007), e retorna um arquivo com os cabeçalhos das sequências submetidas e o resultado da predição, Coding, Noncoding ou Undefined. O diagrama 6.2 resume o workflow do método SOM-PORTRAIT. Cada etapa será explanada abaixo em detalhes. 56
71 Figura 6.2: O workflow do método SOM-PORTRAIT. Validação e conversão O método verifica o formato válido do arquivo multifasta através de um script Perl desenvolvido especialmente para verificação e filtragem de sequências. O script elimina sequências com número de caracteres N, isto é, o nucleotídeo em questão pode ser A,C,T,U ou G, acima de 20% do total de caracteres da sequência. Também elimina sequências que contém caracteres diferentes de A, C, T, G, U e N e sequências que contém número de caracteres menor do que 80 (para restringir o conjunto de ncrnas aos ncrnas estruturais) ou maior do que (por limitações na alocação de memória por parte do programa CAST ). Finalmente, o script verifica o formato correto do cabeçalho FASTA, agrupa a sequência em uma linha contínua de caracteres, eliminando os caracteres especiais CR, LF e espaço, e realiza a transcrição reversa das sequências quando necessário. Após a validação da sequência inicial, o método SOM-PORTRAIT submete todas as sequências à tradução do ANGLE. O programa ANGLE funciona de forma a encontrar todas as seis ORFs da sequência, porém, por meio de um algoritmo de programação 57
72 dinâmica, o método é capaz de discriminar o melhor produto protéico resultante. Caso o ANGLE encontre uma possível ORF, o método SOM-PORTRAIT executa sua tradução, resultando, então, em um arquivo com as seqüencias que possuem ORF, o arquivo com a sequência de aminoácidos de suas respectivas sequências, e um arquivo com as sequências para as quais o ANGLE não conseguiu encontrar ORF. Extração de parâmetros A tabela 6.2 apresenta os nomes e quantidade de variáveis de cada atributo. De acordo com o workflow exibido na tabela 6.2, o método SOM-PORTRAIT extrai atributos de formas diversificadas, de acordo com a presença ou não de ORF predita pelo programa ANGLE. Os atributos extraídos por cada um desses conjuntos de operações são exibidos também na tabela pela sua última coluna. Tabela 6.2: Tabela com o atributo, o programa usado para extraí-lo, seu número total de variáveis de cada atributo e a qual conjunto (dependente de ORF predita ou independente de ORF predita) de operações de extração de parâmetro pertence. Atributo Programa Variáveis Conjunto Composição de nucleotídeos parameter extractor.pl 84 Sequências com ORF e Sequências sem ORF Tamanho de ORF parameter extractor.pl 4 Sequências com ORF Composição de parameter extractor.pl 20 Sequências com aminoácidos ORF Ponto isoelétrico da IEP (EMBOSS) 1 Sequências com proteína ORF Complexidade da CAST 1 Sequências com proteína (entropia ORF composicional) Hidrofobicidade parameter extractor.pl 1 Sequências com média da proteína ORF Tamanho da parameter extractor.pl 4 Sequências sem sequência ORF Após a etapa de tradução das sequências, o método segue para a retirada dos atributos numéricos das sequências, gerando individualmente o vetor de características da sequência. Para o modelo de sequências com ORF predita, são retirados 7 atributos, cada um dos quais se subdivide em vários outros atributos. A composição de nucleotídeos é extraída através da análise quantitativa da sequência de nucleotídeos. A frequência de cada um dos nucleotídeos A, C, T e G na sequência é calculada, resultando em 4 variáveis. A frequência dos dinucleotídeos também é calculada, para cada dinucleotídeo, apenas para a primeira fase de leitura, gerando 16 variáveis. O 58
73 último nucleotídeo da sequência, quando presente, é descartado. Cada possível trinucleotídeo da sequência também é calculado, tendo por base a primeira fase de leitura da sequência, gerando 64 variáveis. Quando presentes, o último nucleotídeos ou os últimos dois nucleotídeos são descartados. Totalizando, 84 variáveis que compõem o atributo composição de nucleotídeos. O tamanho da ORF predita pelo programa ANGLE é calculada a partir da sequência de aminoácidos traduzida do ANGLE. O cálculo quantitativo é baseado em intervalos de tamanho dessa sequência: sequências menores do que 20 aminoácidos, entre 20 e 60 aminoácidos, entre 60 e 100 aminoácidos e maior do que 100 aminoácidos. O atributo totaliza 4 variáveis. A composição de aminoácidos da sequência de peptídeos resultante do ANGLE é calculada de forma similar ao cálculo da composição de nucleotídeos. Cada um dos 20 aminoácidos tem sua frequência calculada em relação ao número de aminoácidos da sequência, totalizando 20 variáveis. A predição do ponto isoelétrico da proteína é realizada por rotina Perl baseada no funcionamento do programa iep da suíte EMBOSS (Rice et al., 2000). O programa analisa a sequência de aminoácidos e retorna um valor normalizado entre 0 e 1. O programa descarta, para sua análise, os caracteres X da sequência. Para o cálculo da entropia composicional da proteína, o programa CAST (Promponas et al., 2000) foi utilizado. O programa recebe a sequência de aminoácidos por entrada, e retorna uma série de informações incorporadas à sequência, referentes à complexidade da estrutura da proteína estimada. Trechos com o caractere X são considerados, pelo programa, trechos de baixa complexidade. O método SOM-PORTRAIT recupera a quantidade de X na sequência resultante do CAST e calcula sua frequência relativa na sequência original de aminoácidos, descontados os caracteres X já presentes na sequência a priori do processamento do CAST. Uma variável é resultante dessa rotina. O cálculo da hidrofobicidade média da proteína é feito a por rotina própria, criada para o programa PORTRAIT (Arrial, 2008) utilizando um método para o cálculo da hidropatia de resíduos (Kyte and Doolittle, 1982). A rotina utiliza uma janela de tamanho três caracteres que desliza um caracter por iteração, desprezando o último ou dois últimos caracteres caso exista. Para cada trinca, consulta a tabela de hidropatia do resíduo, normaliza o valor para constar no intervalo [0, 1] e divide pelo tamanho da janela. Ao final, cada hidrofobicidade individual calculada é somada e seu total dividido pelo total de leituras da janela deslizante, totalizando uma variável. O atributo tamanho de sequência é exclusivo para sequências que não têm ORF presente. Utiliza a mesma lógica de intervalos do atributo tamanho de ORF, usando os intervalos de tamanho de nucleotídeos: 100nt, 400nt, 900nt. O atributo contribui com 4 variáveis. Predição e Treinamento da Rede A rede neural de Kohonen utilizada pelo método SOM-PORTRAIT foi treinada utilizando a biblioteca SOM PAK (Kohonen et al., 1996) versão 3.1. Outras implementações 59
74 como a KNNL - Kohonen Neural Network Library - (Bass and Jakuczun, 2009) mostraramse muito dependentes de configurações de hardware e software, e optou-se por deixar tais implementações em segundo plano nesse primeiro momento do projeto, para manipulações mais simplificadas no ambiente de grid. A biblioteca SOM PAK contém uma implementação em ANSI C do modelo SOM bidimensional proposto por (Kohonen, 2001). A referida biblioteca utiliza uma suíte de programas para treinamento, validação, predição e visualização de dados. A biblioteca é ótima para análise e monitoramento de dados cuja classificação é desconhecida. Após a extração de atributos das sequências, o método SOM-PORTRAIT retorna os dois arquivos de atributos no formato aceito pela SOM PAK com os vários vetores de características de cada sequência para os dois modelos - o modelo de sequências com ORF predita, daqui por diante chamado modelo proteína dependente, e o modelo de sequências que não tiveram ORF predita, daqui por diante chamado modelo proteína independente. Na próxima etapa do método, como é possível visualizar em 6.2, esses dois arquivos são submetidos a modelos diferentes de SOM, um para o modelo proteína dependente, outro para o modelo proteína independente. Os resultados das predições são finalmente agrupados em um único arquivo de resultados, formado pelo cabeçalho da sequência e o nome da única classe a que o modelo atribuiu a sequência: classe Coding, Noncoding ou Undefined. Exemplo de arquivo de entrada da biblioteca SOM PAK (Kohonen et al., 1996). 3 rect 3 1 gaussian A primeira linha, ou cabeçalho do arquivo, é composto por um número indicativo da quantidade de variáveis que compõem cada vetor de características. A segunda informação, rect, diz respeito à topologia utilizada pela rede - retangular nesse exemplo. Os números 3 e 1 são as dimensões, respectivamente, dos eixos x e y de coordenadas do mapa auto-organizável, ou seja, a quantidade de nós (x, y) que compõe o mapa. Finalmente, o valor gaussian reflete o tipo de função de vizinhança aplicado. Nesse exemplo, o tipo de função é gaussiano, e cada um dos três nós, representados pelas três linhas de números no arquivo, são compostos de uma série de valores numéricos representativos dos atributos multidimensionais que compõem os vetores de entrada do mapa. No caso do SOM-PORTRAIT, são 111 atributos numéricos para o modelo de sequências com ORF predita e 88 atributos numéricos para o modelo de sequências sem ORF predita. Para o treinamento, os conjuntos de sequências com ORF presente (dbtr OP) e de sequências sem ORF (dbtr OA) foram submetidas à etapa de extração de atributos, gerando dois respectivos arquivos com os vetores de atributos no formato aceito pela biblioteca, exemplificado por A biblioteca SOM PAK dispõe de várias ferramentas para inicialização randômica dos pesos dos neurônios no mapa, para treinamento, validação e refinamento da rede, e para visualização espacial da rede. O modelo escolhido para representar espacialmente o problema de classificação é constituído por três nós - ou neurônios - cada um dos quais representa dimensionalmente uma 60
75 classe possível para a sequência. Esse mapa simplificado foi escolhido para uma primeira abordagem do método e para possibilitar uma comparação simplificada e direta dos resultados com o método PORTRAIT, que apresenta duas classes de decisão (Coding e Non Coding (Arrial, 2008)). Treinamento do modelo proteína dependente Os arquivos para treinamento da rede de Kohonen para o modelo proteína dependente foram configurados a partir do arquivo dbtr OP, na forma ilustrada pela tabela 6.3. Tabela 6.3: Nomes dos arquivos de treinamento, seu propósito e a quantidade de sequências que o compõe. Nome Função Sequências model.withorf.dat Arquivo de dados para criação do mapa proteína dependente model.withorf.test Arquivo de tesse para estimativa de average quantization error model.withorf.cal Arquivo de calibragem para nomear os nós dos mapas O arquivo model.withorf.dat é o arquivo com vetores de características extraídos de todas as sequências do conjunto dbtr OP, escolhidos de forma aleatória, para reduzir o tempo de execução e evitar a memorização de dados (overfitting) pela rede. O arquivo de tesses utilizado na validação da rede, model.withorf.test, consiste de todos os vetores de características extraídos de dbtr OP. O arquivo de calibragem model.withorf.cal é criado para nomear os nós do mapa de Kohonen treinado. O arquivo contém quantidades idênticas de sequências do conjunto positivo e do conjunto negativo. Os procedimentos a seguir mostram como o mapa para o modelo proteína dependente foi treinado. Inicialmente, o SOM foi construído com os pesos de seus neurônios da camada de entrada atribuídos randomicamente, através do programa randinit. O programa recebe como parâmetros de entrada o arquivo model.withorf.dat, o tamanho do mapa, através das dimensões (parâmetros xdim, igual a 3, e ydim, igual a 1), criando um mapa com três nós (salvo no arquivo model.withorf.cod.1 ). Além disso, o mapa teve sua topologia e função de vizinhança definidos através dos parâmetros topol igual a rect, definindo a topologia retangular, e neigh igual a gaussian, definindo a função de vizinhança igual à função gaussiana. Essa primeira etapa demorou menos de um minuto, utilizando o comando time do shell. A escolha da topologia retangular se baseia na aplicação do problema. Topologias hexagonais são preferíveis em avaliações de imagens e dados gráficos, enquanto que topologias retangulares têm aplicações mais abrangentes (Kohonen et al., 1996). A escolha da função gaussiana em detrimento da função de bolha ou limiar foi definida experimentalmente, os resultados obtidos em tesses preliminares de estimativa de erro com um modelo proteína dependente treinado com função bolha foram piores do que os mesmos resultados com o modelo proteína dependente do SOM com função gaussiana. 61
76 Na segunda etapa, o programa vsom encontrou as coordenadas bidimensionais ótimas dos três nós no mapa em model.withorf.cod.1 para o conjunto de dados apresentado model.withorf.dat. Para o aprendizado da rede, a taxa de aprendizado α(0) foi ajustada para 0, 05, o raio da vizinhança inicial h j (0) para 2 e o número de iterações do aprendizado foi ajustado para passos. A escolha dos valores de α e do raio seguiram as recomendações dos autores da biblioteca SOM PAK. O programa retornou a codificação do mapa de Kohonen encontrado, salva no arquivo model.withorf.cod.2, demorando aproximadamente 5 minutos para construir o mapa. A terceira etapa do treinamento utilizou novamente o programa vsom, dessa vez recebendo o mapa em model.withorf.cod.2 e os mesmos dados model.withorf.dat para a convergência das coordenadas dos nós obtidos na segunda etapa, e refinamento da rede. Nessa etapa, a taxa de aprendizado α(0) foi reduzida para 0, 02, o raio da vizinhança h j (0) para 1 e o número de passos foi aumentado para passos. O programa retornou o mapa treinado para o modelo proteína dependente, nomeado model.withorf.map, em aproximadamente 5 minutos de execução. A tabela 6.4 resume os arquivos criados durante a etapa de treinamento e sua função. Tabela 6.4: Arquivos criados durante a etapa de treinamento e funcionalidade. Nome model.withorf.cod.1 model.withorf.cod.2 model.withorf.map Função Mapa com valores randomizados gerado pelo programa randinit (primeira etapa) Mapa resultante da etapa de aprendizado da rede (segunda etapa) Mapa final resultante da etapa de refinamento da rede (terceira etapa) A quarta etapa de treinamento realizou a verificação estimada do treinamento da rede, através do cálculo do average quantization error. Para esse fim, o programa qerror foi utilizado, recebendo o conjunto de tesses model.withorf.test e o mapa model.withorf.map. O programa estimou o AQE do mapa em 0, 79. Finalmente, na última etapa, os nós do mapa tiveram os nomes das classes desejadas (Coding, Noncoding e Undefined) atribuídos, através do programa vcal. O programa configurou sua calibragem de acordo com o conjunto de sequências utilizado - model.withorfs.cal. O diagrama ilustrado em 6.3 mostra a representação por U-matriz (Hollmen, 2009) do mapa model.withorf.map treinado. Regiões mais escuras representam distâncias maiores entre os nós, e regiões mais claras indicam agrupamentos vicinais de nós. A região mais escura à direita representa uma grande distância entre os nós Coding e Undefined, enquanto que a distância entre os nós Undefined e Noncoding é proporcionalmente muito menor, o que é evidenciado pela cor cinza claro. O tempo de processamento total gasto nas etapas de treinamento e validação do modelo proteína dependente, contabilizados somente os tempos de execução dos programas, foi de aproximadamente 10 minutos. 62
77 Figura 6.3: U-matriz para o mapa do modelo proteína dependente. Treinamento do modelo proteína independente Os arquivos para treinamento da rede de Kohonen para o modelo proteína independente foram configurados a partir do arquivo dbtr OA, na forma ilustrada pela tabela 6.5. Tabela 6.5: Nomes dos arquivos de treinamento, seu propósito e a quantidade de sequências que o compõe. Nome Função Sequências model.withoutorf.dat Arquivo de dados para criação do mapa proteína independente model.withoutorf.test Arquivo de tesse para estimativa de average quantization error model.withoutorf.cal Arquivo de calibragem para nomear os nós dos mapas O arquivo model.withoutorf.dat é um arquivo heterogêneo, composto de vetores de características das sequências do conjunto negativo e os vetores de características de todas as sequências do conjunto positivo. O arquivo de calibragem model.withoutorf.cal contém vetores de características do conjunto negativo e vetores de características do conjunto positivo. Os procedimentos usados para o treinamento do mapa para o modelo proteína independente variam em poucos detalhes dos procedimentos utilizados no modelo proteína dependente. Por causa da escassa quantidade de sequências no conjunto positivo (somente 2.555), o formato do arquivo com os vetores de características foi alterado para incluir pesos no treinamento das sequências. A biblioteca SOM PAK permite o treinamento com pesos através da inclusão do peso relativo da sequência para o treinamento no arquivo de dados model.withoutorf.dat. Ao final das sequências do conjunto positivo, o peso 3, 91, referente à razão entre o número de sequências do conjunto positivo em relação ao número de sequências do conjunto negativo, foi adicionado. No segundo e terceiro passos do treinamento foram usados mesma taxa de aprendizagem, mesmo raio inicial da função de vizinhança e mesmo número de passos. Foram gastos aproximadamente 10 minutos nessas operações e o average quantization error estimado pela biblioteca foi de 0,
78 Figura 6.4: U-matriz para o mapa do modelo proteína independente. A figura 6.4 representa a U-matriz encontrada para o modelo proteína dependente. É bastante nítida a clusterização do mapa, evidenciada pela coloração escura do nó correspondente à classe Coding. Já a coloração acinzentada entre as classes Noncoding e Undefined indicam uma proximidade entre essas duas classes, mas não tão relevante quanto no modelo proteína dependente. Os arquivos resultantes dessa etapas são semelhantes aos arquivos da tabela O método Dist-SOM-PORTRAIT O método Dist-SOM-PORTRAIT é uma alteração do método SOM-PORTRAIT que permite utilizar o poder computacional do grid construído pelo projeto EELA-2. O método per se não é alterado, mas sim sua forma de execução Descrição do método A figura 6.5 representa o workflow diferenciado do método Dist-SOM-PORTRAIT. A rotina de validação do arquivo multifasta de entrada foi mantida em execução local, por motivos de praticidade. De acordo com o tamanho do arquivo fornecido pelo usuário, o método é capaz de encontrar a melhor forma de segmentá-lo em vários arquivos menores. O método pode segmentar o arquivo em até 20 arquivos menores. Essa quantidade de arquivos em que o arquivo de entrada é segmentado foi definida experimentalmente, encontrando um valor ótimo para o número de segmentações, tendo em vista que cada segmento requer upload para o grid, seu monitoramento e download dos resultados, um processo oneroso e muito dependente da qualidade da conexão de rede. O SOM-PORTRAIT, programas externos necessários e bibliotecas são armazenados num Storage Element, e a cada execução, o método Dist-SOM-PORTRAIT se encarrega de criar uma cópia local de trabalho em cada Worker Node do Computing Element do grid escolhido para executar a operação. Como dito, cada segmento precisa ser monitorado pelo método. Para esse fim, a API jlite dispõe de diversos métodos para envio, monitoramento e recuperação das tarefas. Para gerenciar essas tarefas, os métodos utilizam um arquivo que obedece à linguagem descritora de tarefas - JDL ou Job Description Language - utilizada pelo grid. No apêndice A desse trabalho é apresentado um exemplo de arquivo JDL criado automaticamente pelo método Dist-SOM-PORTRAIT. 64
79 Figura 6.5: O workflow do método Dist-SOM-PORTRAIT. Ao final da execução de todas as tarefas no grid, o método recupera os vários arquivos de predições e os reúne em um único arquivo de resultados. O arquivo é disponibilizado via uma URL com um identificador único para o usuário Ambiente operacional do grid As mesmas configurações de máquina utilizadas para o treinamento e execução do método SOM-PORTRAIT foram utilizadas para ser a User Interface do programa Dist- SOM-PORTRAIT. A conexão de rede utilizada para comunicação com o grid teve taxa de upload avaliada em aproximadamente 40Kb/s, enquanto que a taxa de download foi avaliada em aproximadamente 1, 0M b/s. O site utilizado para processamento do programa é sediado na Universidade Federal do Rio de Janeiro (UFRJ), consistindo de uma infraestrutura de 24 núcleos em seu Computing 65
80 Element (CE) e um Storage Element (SE), bem como serviços de Logging and Bookkeeping (LB) e Workload Management System (WMS). O acesso aos recursos do grid exigem um certificado digital no formato X.509 válido. Um certificado pessoal foi utilizado para registro e criação do proxy temporário de conexão com o grid. Todas as máquinas do grid utilizam o middleware glite versão 3.1, operando sobre o sistema operacional Scientific Linux - SL - versão 4.0. Essa versão de sistema operacional utiliza versões de bibliotecas fundamentais especialmente compiladas para ele, portanto, todos os binários e bibliotecas externas utilizados foram compilados com a flag static do gcc para linkagem das bibliotecas dinâmicas Estratégia de distribuição de processamento A estratégia utilizada para distribuição do processamento baseia-se na segmentação do arquivo multifasta de entrada fornecido pelo usuário em vários arquivos de menor porte. Cada arquivo será enviado para execução em uma instância do método SOM- PORTRAIT no correspondente Worker Node do CE do site da UFRJ. Essa instância do SOM-PORTRAIT foi especialmente configurada para execução no sistema operacional utilizado pelo grid, e foi armazenada no SE do site da UFRJ em conjunto com as bibliotecas e programas externos que utiliza. O acesso aos recursos do grid é feito utilizando a API jlite (Sukhoroslov, 2009). A API permite o envio de tarefas, monitoramento de seu estado, envio e recuperação de dados. 66
81 Capítulo 7 Resultados e Discussões No presente capítulo, o método SOM-PORTRAIT é comparado a outros dois métodos, tendo por base sequências do fungo Paracoccidiodes brasiliensis. Finalmente, o método Dist-SOM-PORTRAIT é comparado ao método SOM-PORTRAIT, no quesito tempo de execução, e seus resultados analisados. 7.1 Estudo de caso com o fungo Paracoccidioides brasiliensis Para a validação do SOM-PORTRAIT, optou-se pela comparação com o PORTRAIT, pela grande proximidade dos procedimentos realizados para classificação de sequências por ambos os métodos. O mesmo conjunto de teste foi fornecido como parâmetro de entrada aos dois métodos, e o resultado comparado quantitativamente. Além da comparação com o PORTRAIT, outro mapa de Kohonen, criado com duas classes (Coding e Noncoding) foi treinado, de forma similar à descrita em Esse modelo, nomeado Modelo de Duas Classes (MDC), foi confrontado qualitativamente aos métodos PORTRAIT e SOM-PORTRAIT Construção do conjunto de testes O conjunto de teste utilizado para executar os métodos SOM-PORTRAIT, POR- TRAIT e MDC foram as assembled ESTs do transcriptoma do fungo P. brasiliensis (Felipe et al., 2005). A etapa de validação e formatação do arquivo multifasta de entrada descartou 9 sequências: uma sequência por conter muitos caracteres N, e outras 8 sequências por conterem caracteres considerados inválidos pelo método. As sequências restantes foram submetidas aos testes nos três modelos, e para as comparações foi utilizada uma filtragem relativa à probabilidade da classificação calculada pelo programa PORTRAIT. Foi considerado o conjunto de sequências que obtiveram 67
82 probabilidade de pertencer à classe estimada pelo PORTRAIT (Coding ou Noncoding) maior do que 70%, 80% e 90%. A tabela 7.1 exibe os arquivos de teste criados, o filtro aplicado para selecionar as sequências e a quantidade total de sequências que os compõem. Tabela 7.1: Nome, filtro utilizado e quantidade de sequências dos arquivos de teste. Nome Tipo de Filtro sequências Pb ests.fas Validação e formatação do método Pb ests.fas.70 Probabilidade de classificação 70% Pb ests.fas.80 Probabilidade de classificação 80% Pb ests.fas.90 Probabilidade de classificação 90% Configuração do PORTRAIT Uma versão local do método PORTRAIT foi instalada, e sua rede neural (Support Vector Machine ou SVM ) foi treinada e configurada a partir dos procedimentos descritos por (Arrial et al., 2007; Arrial, 2008). Para esse procedimento, quatro máquinas foram utilizadas, suas configurações descritas abaixo: 1. Processador Intel Core 2 Duo 2.0 Ghz com Mb RAM e sistema operacional Ubuntu 8.04; 2. Processador Intel Core 2 Duo 2.4 Ghz com Mb RAM e sistema operacional OpenSuse 10.3; 3. Processador Intel Core 2 Duo 2.2 Ghz com Mb RAM e sistema operacional Debian 5; 4. Processador Intel Core 2 Duo 2.2 Ghz com Mb RAM e sistema operacional Ubuntu As máquinas 1 e 2 foram utilizadas no treinamento da SVM do modelo proteína dependente, enquanto que as máquinas 3 e 4 desempenharam o treinamento da SVM do modelo proteína independente. O método PORTRAIT utiliza a biblioteca LIBSVM versão 2.84 (Chang and Lin, 2001). Os parâmetros requeridos pela biblioteca foram reproduzidos, e, para diminuir o tempo necessário para o treinamento, que segundo os autores pode demorar semanas, os parâmetros ótimos γ e C encontrados por Arrial e coautores para a função de base radial utilizada no kernel da SVM utilizada pelo PORTRAIT foram fornecidos diretamente para a biblioteca. Assim, obteve-se uma rede com resultados praticamente equivalentes à rede treinada por Arrial após aproximadamente 40 horas de execução do treinamento para o modelo proteína dependente e 30 horas para o modelo proteína independente Testes comparativos: SOM-PORTRAIT PORTRAIT As assembled ESTs do Pb do arquivo Pb ests.fas foram submetidas ao método SOM-PORTRAIT para classificação, demorando 13 minutos de execução, e ao método 68
83 PORTRAIT, demorando 11 minutos de execução. O resultado da classificação do POR- TRAIT relatou possíveis sequências codificadoras e 969 sequências de ncrnas. Esse resultado varia do resultado documentado por Arrial (Arrial, 2008) pois a etapa de validação do método recusou algumas sequências admitidas pelo PORTRAIT originalmente proposto. O teste comparativo consiste do confronto quantitativo de sequências classificadas em uma determinada classe do SOM-PORTRAIT em relação à sua classificação pelo método PORTRAIT. Foram realizados 4 testes, cada qual com um conjunto gradativamente mais criterioso para a qualificação, isto é, com o filtro de probabilidade com corte gradativamente maior. A tabela 7.2 relata os resultados da comparação utilizando o conjunto de testes Pb ests.fas. Já as tabelas 7.3, 7.4 e 7.5 são os resultados da comparação utilizando, respectivamente, os arquivos de teste Pb ests.fas.70, Pb ests.fas.80 e Pb ests.fas.90. Tabela 7.2: Comparação entre o método SOM-PORTRAIT e o método PORTRAIT. A porcentagem é relativa ao total de assembled ESTs no arquivo Pb ests.fas. Sequências Porcentagem SOM-PORTRAIT Sequências codificadoras ,54% ncrnas ,50% Classe Undefined de sequências 419 6,97% PORTRAIT SOM-PORTRAIT Coding/Coding ,87% Noncoding/Noncoding 493 8,20% Coding/ Undefined 38 0,63% Noncoding/ Undefined 380 6,32% Coding/Noncoding 560 9,31% Noncoding/Coding 100 1,66% Tabela 7.3: Comparação entre o método SOM-PORTRAIT e o método PORTRAIT. A porcentagem é relativa ao total de assembled ESTs no arquivo Pb ests.fas.70. Sequências Porcentagem SOM-PORTRAIT Sequências codificadoras ,21% ncrnas ,60% Classe Undefined de sequências 332 6,19% PORTRAIT SOM-PORTRAIT Coding/Coding ,62% Noncoding/Noncoding 308 5,74% Coding/ Undefined 12 0,22% Noncoding/ Undefined 320 5,96% Coding/Noncoding 368 6,86% Noncoding/Coding 32 0,60% 69
84 Tabela 7.4: Comparação entre o método SOM-PORTRAIT e o método PORTRAIT. A porcentagem é relativa ao total de assembled ESTs no arquivo Pb ests.fas.80. Sequências Porcentagem SOM-PORTRAIT Sequências codificadoras ,81% ncrnas 473 9,58% Classe Undefined de sequências 277 5,61% PORTRAIT SOM-PORTRAIT Coding/Coding ,51% Noncoding/Noncoding 207 4,20% Coding/ Undefined 4 0,08% Noncoding/ Undefined 273 5,53% Coding/Noncoding 266 5,39% Noncoding/Coding 15 0,30% Tabela 7.5: Comparação entre o método SOM-PORTRAIT e o método PORTRAIT. A porcentagem é relativa ao total de assembled ESTs no arquivo Pb ests.fas.90. Sequências Porcentagem SOM-PORTRAIT Sequências codificadoras ,55% ncrnas 227 5,40% Classe Undefined de sequências 170 4,05% PORTRAIT SOM-PORTRAIT Coding/Coding ,48% Noncoding/Noncoding 87 2,07% Coding/ Undefined 0 0,00% Noncoding/ Undefined 170 4,05% Coding/Noncoding 140 3,33% Noncoding/Coding 3 0,07% As comparações exibidas nas tabelas 7.2, 7.3, 7.4 e 7.5 são resumidas pela figura 7.1. Na figura, os círculos representam o total de sequências classificadas como Noncoding (esquerda) e Coding (direita) pelo método PORTRAIT, e as faixas coloridas representam o total de sequências classificadas pelo método SOM-PORTRAIT como Noncoding (cinza escuro), Coding (cinza claro) e Undefined (preto). Os dados desse experimento, em conjunto com as U-matriz obtidas para os modelos proteína dependente 6.3 e proteína independente 6.4 do SOM-PORTRAIT, demonstram que a divisão em classes encontrada pela rede de Kohonen para as sequências é coerente com a divisão encontrada pela rede SVM do PORTRAIT. A classe Undefined foi composta em grande parte por sequências classificadas como Noncoding pelo PORTRAIT, o que reforça a visível aproximação das duas classes visualmente identificável pela coloração cinza da U-matriz em ambos os modelos. Presume-se como resultado, então, que 70
85 (a) Arquivo Pb ests.fas (b) Arquivo Pb estas.fas.70 (c) Arquivo Pb estas.fas.80 (d) Arquivo Pb estas.fas.90 Figura 7.1: Comparações entre resultados do SOM-PORTRAIT (faixas) e resultados do PORTRAIT (círculos) para os vários arquivos de teste. a classe Undefined seja uma subclasse especial de ncrnas, identificável através de vários, ou todos, os atributos 6.2 escolhidos para a avaliação pelo classificador. É notável como a conformidade da classificação entre SOM-PORTRAIT e PORTRAIT aumenta significativamente ao aplicar-se o filtro de probabilidade gradativamente maior, chegando a uma conformidade de mais de 92, 00% na comparação utilizando o conjunto de testes com filtro mais exigente, de 90% (tabela 7.5). O método SOM-PORTRAIT, no tocante à classe codificadora, comportou-se similarmente ao método PORTRAIT, em todos os casos de teste. Para a classe não codificadora, a conformidade com o PORTRAIT atingiu, em ordem crescente de rigorosidade do filtro aplicado, 50, 67%, 46, 67%, 41, 82% e 33, 46%, diminuindo, portanto, à medida que a certeza da classificação foi aumentada. Já para a classe Undefined, ocorreu exatamente o inverso: 39, 01%, 48, 48%, 55, 15% e 65, 38%, mostrando que a classe Undefined assumiu várias das sequências classificadas como Noncoding pelo PORTRAIT com boa margem de certeza. Finalmente, a conformidade encontrada para a classificação Coding foi, respectivamente, 88, 10%, 91, 92%, 93, 92% e 96, 45%. Os dados apresentados demonstram a boa conformidade geral do método SOM-PORTRAIT em relação ao método PORTRAIT, e revelam que a terceira classe Undefined criada é realmente uma classe próxima das sequências classificadas como ncrna no PORTRAIT. Além disso, o método revela ser capaz, dados os atributos utilizados para avaliação das sequências, distingüi-las em mais do que duas classes com boa precisão. 71
86 (a) U-matriz do modelo proteína dependente (b) U-matriz do modelo proteína independente Figura 7.2: U-matrizes calculadas para os dois modelos do MDC. Figura 7.3: Comparações entre resultados do SOM-PORTRAIT (faixas) e resultados do MDC (círculos) para as assembled ESTs do Pb Configuração do MDC Um modelo de SOM utilizando somente dois nós foi treinado para comparações e testes. Um mapa treinado dessa forma tende a se comportar como um problema de clusterização simples por K-mean (Kohonen, 2001). Apesar da abordagem simplista, é interessante confrontar os resultados das comparações desse modelo com os métodos SOM-PORTRAIT e PORTRAIT, para exibir o comportamento dos mapas de Kohonen nos dois métodos, e como as classes encontradas se relacionam. A configuração do MDC foi realizada de forma semelhante à descrita em na seção Os arquivos de teste utilizados foram exatamente os mesmos. O valor do AQE encontrado foi 0, 93 para o mapa do modelo proteína dependente, e 0, 64 para o mapa do modelo proteína independente. O treinamento dessa rede demorou aproximadamente 10 minutos. A figura 7.2 representa as U-matrizes do modelo proteína dependente e do modelo proteína independente treinados para o MDC, respectivamente. A cor preta dos nós demonstra que estão separados por uma distância considerável, o que é o comportamento previsto para esse modelo simplificado. 72
87 7.1.5 Testes comparativos: PORTRAIT MDC Os mesmos arquivos de teste usados na seção 7.1 foram submetidos ao MDC, cada predição executando em, aproximadamente, 12 minutos. As tabelas 7.6, 7.7, 7.8 e 7.9 mostram os resultados das comparações. É notável como os resultados percentuais da predição feita pelo MDC se aproximam das percentagens encontradas pelo PORTRAIT. Tabela 7.6: Comparação entre o MDC e o método PORTRAIT. A porcentagem é relativa ao total de assembled ESTs no arquivo Pb ests.fas. Sequências Porcentagem Sequências codificadoras ,50% ncrnas ,50% MDC SVM-PORTRAIT Coding/Coding ,55% Noncoding/Noncoding 491 8,17% Coding/Noncoding 478 7,95% Noncoding/Coding 561 9,33% Tabela 7.7: Comparação entre o MDC e o método PORTRAIT. A porcentagem é relativa ao total de assembled ESTs no arquivo Pb ests.fas.70. Sequências Porcentagem Sequências codificadoras ,40% ncrnas ,60% MDC SVM-PORTRAIT Coding/Coding ,83% Noncoding/Noncoding 308 5,74% Coding/Noncoding 352 6,56% Noncoding/Coding 368 6,86% Tabela 7.8: Comparação entre o MDC e o método PORTRAIT. A porcentagem é relativa ao total de assembled ESTs no arquivo Pb ests.fas.80. Sequências Porcentagem Sequências codificadoras ,42% ncrnas 473 9,58% MDC SVM-PORTRAIT Coding/Coding ,59% Noncoding/Noncoding 207 4,19% Coding/Noncoding 288 5,83% Noncoding/Coding 266 5,39% As comparações feitas nas tabelas 7.6, 7.7, 7.8 e 7.9 demonstram um acréscimo relevante da conformidade entre os dois métodos somente ao se elevar o filtro de probabilidade para 80% de certeza. Aparentemente, o último filtro teve somente o efeito negativo 73
88 Tabela 7.9: Comparação entre o MDC e o método PORTRAIT. A porcentagem é relativa ao total de assembled ESTs no arquivo Pb ests.fas.90. Sequências Porcentagem Sequências codificadoras ,60% ncrnas 227 5,40% MDC SVM-PORTRAIT Coding/Coding ,48% Noncoding/Noncoding 87 2,07% Coding/Noncoding 173 4,12% Noncoding/Coding 140 3,33% (a) Arquivo Pb ests.fas (b) Arquivo Pb estas.fas.70 (c) Arquivo Pb estas.fas.80 (d) Arquivo Pb estas.fas.90 Figura 7.4: Comparações entre resultados do MDC (faixas) e resultados do PORTRAIT (círculos) para os vários arquivos de teste. de descarte de sequências, sem acrescentar muito à conformidade dos métodos. É importante notar que grande parte das sequências classificadas como Noncoding por ambos os métodos foram descartadas, ou, em outras palavras, a probabilidade de serem realmente da mesma classe não é alta para a maioria delas. Isso indica que o método abarcou na classe Noncoding muitas sequências que obtiveram baixa probabilidade no PORTRAIT, o que pode indicar que o método adotou um crivo mais eficiente de classificação, ou também pode indicar que a classe Noncoding agrupa de forma precária várias subclasses bastante distintas de tipos de ncrna. Os resultados mostrados nas tabelas são resumidos na figura 7.4, onde, novamente, os círculos representam o total de sequências classificadas pelo PORTRAIT e as faixas representam a classificação realizada pelo MDC, sendo a classe Coding representada na tonalidade cinza claro, e a classe Noncoding pela cor preta. 74
89 Os resultados mostram que os dois métodos divergiram consideravelmente em sua classificação, especialmente para as sequências não codificadoras. A análise da u-matriz do MDC revela que o treinamento não supervisionado conseguiu individualizar as duas classes com bastante critério. Pode-se supor que, por unir todos os diferentes tipos de ncrna que compõem o conjunto de treinamento 6.1, diversos tipos de ncrna foram agrupados em uma só classe muito heterogênea. Quando confrontados estes resultados aos obtidos pelo SOM-PORTRAIT 7.1.3, a presença dessa terceira classe torna-se evidente. É notável que a quantidade percentual de sequências Noncoding no PORTRAIT classificadas como Coding pelo MDC aumentaram à medida que filtros mais estringentes foram utilizados. Esse comportamento também é sanado pela criação da terceira classe, como é visível nos resultados do SOM-PORTRAIT. Essa divergências na classe Coding do MDC é diminuída, o que leva à hipótese de que a classe Undefined abarca de forma eficiente uma porção dos mrnas preditos pelo MDC erroneamente na classe Noncoding do PORTRAIT. A análise desses comportamentos pode indicar que hajam outras possíveis divisões de classes, que, agregadas, melhorem ainda mais a conformidade das classificações dos métodos SOM-PORTRAIT e PORTRAIT. De toda forma, o método não supervisionado mostrou-se bastante preciso em suas classificações, com a vantagem de requerer muito menos tempo e memória para ser treinado Testes comparativos: SOM-PORTRAIT MDC A comparação entre SOM-PORTRAIT e MDC, abaixo realizada, foi executada sobre as assembled ESTs do transcriptoma do Pb, em aproximadamente 13 minutos, sendo que 9 sequências foram descartadas na etapa de validação do método. A tabela 7.10 e a figura 7.3 exibem os resultados encontrados para a comparação quantitativa. Nessa figura, os círculos representam as classificações no MDC, e as faixas as classificações no modelo SOM-PORTRAIT. A coloração clara refere-se à classe Coding de sequências, a coloração cinza à classe Noncoding e a cor preta à classe Undefined. É notável que, quantitativamente, a classe de sequências Noncoding não se alterou em ambas as classificações. Já a classe Undefined delimita-se totalmente como subconjunto da classe Coding, o que vem reforçar a hipótese de que é uma subclasse distinta de ncrnas com características próximas às de mrnas. Por isso, ao utilizar um mapa com somente dois nós, o método aproxima essa subclasse à classe Coding, causando a divergência vista em Dist-SOM-PORTRAIT: gridificação do método Os testes realizados sobre o método Dist-SOM-PORTRAIT têm por objetivo confirmar a melhoria significativa do tempo de execução necessário ao método SOM-PORTRAIT. 75
90 Tabela 7.10: Comparação entre o método SOM-PORTRAIT e o modelo MDC. A comparação foi realizada sobre as sequências do transcriptoma do Pb. Sequências Porcentagem SOM-PORTRAIT Sequências codificadoras ,54% ncrnas ,45% Classe Undefined de sequências 419 6,92% MDC Sequências codificadoras ,50% ncrnas ,50% MDC SOM-PORTRAIT Coding/Coding ,54% Noncoding/Noncoding ,50% Coding/ Undefined 418 6,95% Noncoding/ Undefined 0 0,00% Coding/Noncoding 0 0,00% Noncoding/Coding 0 0,00% Dados de teste Para a execução dos testes, o arquivo de assembled ESTs do Pb foi replicado quantas vezes necessário, para produzir arquivos com quantidade maior de sequências. O fato de existirem sequências duplicadas no arquivo não influencia de forma alguma na velocidade de execução do método, que não armazena de forma permanente nenhuma informação sobre a sequência. A tabela 7.11 relata o nome do arquivo, seu tamanho em número de sequências e em espaço ocupado em disco, e em quantos fragmentos foi segmentado pelo método Dist-SOM-PORTRAIT. Tabela 7.11: Arquivos de teste criados para a comparação de tempos de execução dos métodos SOM-PORTRAIT e Dist-SOM-PORTRAIT. Pb ests.fas ,7 Mb 5 input.fas ,1 Mb 10 input.fas ,3 Mb 10 input.fas ,4 Mb 15 input.fas ,8 Mb 15 input.fas ,7 Mb 20 O relato do número de fragmentos é importante para compreender os resultados mostrados na figura 7.5, pois, para o envio das tarefas, o método deve realizar o upload do arquivo via protocolo FTP seguro (GSIFTP), o que, na rede utilizada (descrita em 6.2.2), demorou consideravelmente. Os tempos relatados no gráfico comparativo abaixo, portanto, desconsideram o tempo de upload e download dos arquivos. 76
91 7.2.2 Teste comparativo de tempo de execução A tabela 7.12 mostra os tempos de execução de cada arquivo de testes no método SOM-PORTRAIT e no método Dist-SOM-PORTRAIT. A rede, no momento da execução dos testes, apresentou algumas falhas de conexão com o site da UFRJ, o que não impossibilitou a continuidade da execução do método, porém impacta negativamente no resultado dos testes. Para minimizar esse efeito, optou-se por executar o teste com cada arquivo três vezes, e tomar a média aritmética do tempo gasto como resultado, sem descartar o pior resultado. Todos os resultados, ao final, ficaram próximos à média encontrada, variando em torno de 10 minutos para mais ou para menos. Tabela 7.12: Tempos de execução encontrados para cada arquivo de testes. O número de sequências do arquivo denota a coluna, e seu correspondente tempo de execução no método SOM-PORTRAIT e Dist-SOM-PORTRAIT SOM-PORTRAIT 00:14:41 00:23:38 00:50:51 01:16:52 02:30:38 05:53:53 Dist-SOM-PORTRAIT 00:37:58 00:40:14 00:46:50 00:48:53 00:43:07 00:44:16 Figura 7.5: Comparação de tempos de execução entre Dist-SOM-PORTRAIT e SOM- PORTRAIT para o conjunto de arquivos de teste. A curva tracejada corresponde aos valores encontrados para o método SOM-PORTRAIT, enquanto que a curva contínua (em cinza) corresponde aos valores encontrados para o método Dist-SOM-PORTRAIT. Os resultados demonstram uma excelente melhoria no tempo de execução. Os tempos de upload - download dos arquivos, no pior caso do envio dos fragmentos do arquivo input.fas , não superou 50 minutos. Em uma rede com taxas de upload - download melhores, esse gargalo praticamente não existirá. 77
92 Capítulo 8 Conclusões Neste trabalho, um método inédito foi proposto para a identificação de RNAs não codificadores baseado na análise composicional de RNA transcriptômico e utilizando mapas auto organizáveis de Kohonen. O método, nomeado SOM-PORTRAIT, foi totalmente implementado e a rede neural treinada com um conjunto de treinamento constituído de sequências de vasta gama de organismos. Os mapas auto organizáveis treinados foram avaliados por meio da análise de suas u- matrizes, e confrontados experimentalmente ao método PORTRAIT, utilizando, para este fim, as assembled ESTs do fungo Paracoccidioides brasiliensis. Além disto, outro método similar, utilizando um mapa auto organizável de dois nós e duas possíveis classificações, foi concebido, para confronto de seus resultados com os obtidos pelo PORTRAIT e pelo SOM-PORTRAIT. Os resultados obtidos confirmaram, de forma consistente, a boa acurácia do método SOM-PORTRAIT, e dá fortes constatações de sua capacidade de subdivisão em várias outras classificações, pela análise da composição da classe hipotética criada, a classe Undefined. As vantagens em termos de tempo de processamento e exigência de memória necessários para treinamento e execução do método também foram notáveis em comparação com métodos similares de identificação baseados em SVMs. Finalmente, uma implementação em sistema distribuído do método foi desenvolvida, e batizada de Dist-SOM-PORTRAIT, utilizando o middleware glite e os diversos serviços da infraestrutura de grid do projeto EELA-2. Testes de performance entre os métodos SOM-PORTRAIT e Dist-SOM-PORTRAIT revelaram uma grande vantagem na utilização do método distribuído para arquivos com número expressivo de sequências. Aliaram-se, portanto, a possibilidade de identificação e categorização de ncrnas nas suas diversas classes, tornada viável pelo inédito método SOM-PORTRAIT, com sua capacidade de utilização veloz e eficiente em larga escala por projetos biológicos, descortinando várias novas possibilidades nesta incipiente área de pesquisa. Em resumo, os objetivos propostos ao início do trabalho foram concluídos com sucesso: ˆ O método SOM-PORTRAIT foi proposto e implementado; 78
93 ˆ Os estudos de caso com o organismo P. brasiliensis confirmaram a boa acurácia do método; ˆ A versão distribuída do método SOM-PORTRAIT, Dist-SOM-PORTRAIT, foi implementada e implantada com sucesso na infraestrutura do projeto EELA Perspectivas Futuras São perpectivas futuras para o método SOM-PORTRAIT e Dist-SOM-PORTRAIT: ˆ Um novo conjunto de teste será criado com excertos de RNA codificador e não codificador escolhidos de forma aleatória, aplicado ao método e, através dos resultados coletados, a acurácia e sensibilidade do SOM-PORTRAIT serão calculadas; ˆ Comparações com outros métodos além do PORTRAIT serão executadas; ˆ Adaptações ao método estão em procedimento, com a redução do número de atributos que compõem o vetor de características; ˆ Incluir probabilidades para determinar o nível de confiança da identificação e classificação das sequências; ˆ A remoção do passo de predição de ORFs utilizando o programa ANGLE está sendo analisada em termos de sua viabilidade e das melhorias em performance que pode trazer para o método; ˆ O desenvolvimento de uma interface web de fácil acesso está em progresso, integrando os métodos SOM-PORTRAIT e Dist-SOM-PORTRAIT ; ˆ Alterações no método podem ser realizadas para incluir novas classes e avaliar a capacidade classificatória do método, além de poderem ser incluídas de acordo com a necessidade de usuários do método; ˆ Experiências com treinamento de mapas utilizando um número reduzido de atributos e características têm convergido para bons resultados, espera-se adaptá-los ao método; ˆ Em relação ao método Dist-SOM-PORTRAIT, refinar o tratamento de erros de conexão, para maior facilidade e transparência para o usuário final. 79
94 Apêndice A Exemplo de um arquivo JDL Exemplo de um arquivo JDL criado automaticamente pelo Dist-SOM-PORTRAIT para execução do script run-som.sh tendo por parâmetro o arquivo segmentado arquivo1.fasta. O arquivo define o CE do site de UFRJ para executar a tarefa (campo Requirements) e, ao término da execução da tarefa, recupera os arquivos de saída de erros (run-som.err)e de saída padrão (run-som.out), arquivos criados obrigatoriamente pelo glite, e o arquivo com as predições (arquivo1.result). O script run-som.sh foi criado para preparar o ambiente do worker node para a execução do método SOM-PORTRAIT. Mais especificamente, o script descarrega do SE do site da UFRJ o arquivo compactado com o método SOM-PORTRAIT, bibliotecas e arquivos essenciais, além de executar algumas rotinas de configuração do ambiente. Type="Job"; JobType="Normal"; Executable="/bin/sh"; StdOutput="run-som.out"; StdError="run-som.err"; ShallowRetryCount=10; Rank=other.GlueCEStateFreeCPUs; InputSandbox={"run-som.sh","arquivo1.fas"}; Arguments="run-som.sh arquivo1.fas"; OutputSandbox={"run-som.err","run-som.out","arquivo1.results"}; Requirements=(other.GlueCEUniqueID=="ce01.eela.if.ufrj.br:2119/jobmanager-lcgpbs-prod") 80
95 Anexo I Artigo de descrição do método SOM-PORTRAIT (BSB 2009) 81
96 SOM-PORTRAIT: Identifying non-coding RNAs using Self-Organizing Maps T. C. Silva 1, P. A. Berger 1, R. T. Arrial 2, R. C. Togawa 3, M. M. Brigido 2, M. E. M. T. Walter 1 1 Department of Computer Science - Institute of Exact Sciences 2 Laboratory of Molecular Biology - Institute of Biology Campus Universitario Darcy Ribeiro, University of Brasilia, Zip Code Bioinformatics Laboratory, EMBRAPA Genetic Resources and Biotechnology, Zip Code Brasilia-Brazil Abstract Recent experiments have shown that some types of RNA may control gene expression and phenotype by themselves, besides their traditional role of allowing the protein synthesis. Roughly speaking, RNAs can be divided into two classes: mrnas, that are translated into proteins, and non-coding RNAs (ncrnas), which play several cellular important roles besides protein coding. In recent years, many computational methods based on different theories and models have been proposed to distinguish mrnas from ncrnas. Particularly, Self-Organizing Maps (SOM), a neural network model, is time efficient for the training step, and present a straightforward implementation that allow easily increasing of the number of classes for clustering the input data. In this work, we propose a method for identifying non-coding RNAs using Self Organizing Maps, named SOM-PORTRAIT. We implemented the method and applied it to a data set containing Assembled ESTs of the Paracoccidioides brasiliensis fungus transcriptome. The obtained results were promising, with the advantage that the time and memory requirements needed to our SOM-PORTRAIT are much less than those needed for methods based on the Support Vector Machine (SVM) paradigm, like PORTRAIT. 1 Introduction The usual view of the central dogma of molecular biology [25] predicts that genetic information flow from DNA to proteins using RNA as intermediate. DNA is responsible for the genotype of a cell while protein is responsible for the cell s phenotype. This orthodox view of the central dogma suggests that RNA is an auxiliary molecule involved in all stages of protein synthesis and gene expression. But recent experiments have shown that some types of RNA may indeed control gene expressing and phenotype by themselves. Many other biological functions of RNAs are already known, and new functions are continuously being discovered. Roughly speaking, RNAs can be divided into two classes, mrnas - which are
97 translated into proteins, and non-coding RNAs (ncrnas) - which play several cellular important roles besides protein coding. In recent years, many computational methods have been proposed to distinguish mrnas from ncrnas. It is noteworthy that traditional methods that successfully identify mrnas in general fail when used to identify ncrnas, although they can be somewhat combined with other methods to identify a few number of well conserved RNAs, like rrna. For example, BLAST [1] with customized parameters together with covariance models approaches correctly identified snr- NAs [20]. Then, methods based on different theories have been developed, such as theory of probability like Infernal [6], thermodynamics [26,11], or Support Vector Machine (SVM) like CONC [17], CPC [14] and PORTRAIT [4]. Particularly, although CONC [17] and PORTRAIT [4] had achieved good results, a potential drawback of an algorithm based on the SVM model when dealing with large number of RNA sequences is both training time and memory requirement. For example, when n training instances must be held in memory, the best-known SVM implementation takes O(n a ) time, with a typically between 1.8 and 2. But there is another neural network model, the Self-Organizing Maps (SOM), that takes O(n) time for the training step, and has a straightforward implementation which allow easily increasing of the number of classes for grouping the input data, while this is far more complicated for the SVM approach. In this context, the objective of this work is to propose a method for identifying ncrnas using Self Organizing Maps, named SOM-PORTRAIT. In Section 2, we briefly discuss ncrnas and our previous program PORTRAIT, that is based on the SVM model. In Section 3 we shortly describe the SOM model. In Section 4 we present our SOM model to identify ncrnas, and show some implementation details. In Section 5, we present the results of applying our method to the Paracoccidioides brasiliensis fungus and compare them with SVM-PORTRAIT. Finally, in Section 6, we conclude and suggest future work. 2 About ncrnas and SVM-PORTRAIT As said before, experimental evidences have been suggesting that most of RNA transcribed throughout the genome does not code for proteins [19]. These RNAs have been called ncrnas, a heterogeneous category of RNAs that includes regulatory molecules, conserved molecules with unknown function and transcriptional noise [24]. So, ncrnas is an expanding class that includes different types of RNA involved in several cell activities, such as the well known ribosomal RNA (rrna) and the transfer RNA (trna), both involved in the protein biosynthetic machinery, but it also includes small nuclear RNA (snrna), small nucleolar RNA (snorna) and micro RNA (mirna), among others. A special group is composed by a mrna like ncrna, that is, these molecules act very alike mrna, but does not code for proteins, and frequently contaminate mrna preparation [8]. By definition, ncrna is characterized by the absence of an open reading frame (ORF), but this premise is misleading, since the fortuitous presence of small ORFs is quite frequent. On the other side, the absence of a detectable
98 ORF may not be observed in low quality transcripts, as those normally found in expressed sequence tag (ESTs). Therefore, to detect ncrna from a set of ESTs composing a transcriptome is a challenging task. In order to distinguish coding and non-coding RNA from an EST data set, we had previously proposed the PORTRAIT program [4], based on the SVM model, from here after named SVM-PORTRAIT. This program focused on small transcriptome projects, based in EST derived from poorly characterized organisms, for which there is just a little genetic information. SVM-PORTRAIT relies on intrinsic features of RNA sequence and represents an improvement for the CONC [18] and CPC [14] programs, since it does not use homology derived information associated to an error prone translator for ORF definition. SVM-PORTRAIT uses the LIBSVM [5] v2.84 implementation, with Radial Basis Function kernel, set as C-SVM and binary classification problem, and creates two classes, coding (positive set) and non-coding (negative set) RNA. Two models were induced separately: a protein-dependent one, induced with dbtr OP set as training data, and a nucleotide-only using dbtr OA for training. 3 Self-Organizing Maps A Self-Organizing Map (SOM) is an Artificial Neural Network first described by T. Kohonen [12], also called Kohonen Map or Kohonen Neural Network. A SOM represents an open set of multivariate items by a finite set of model items, which makes it useful for classifying high-dimensional data [22]. A SOM has a simple organization (Figure 1) composed by only two layers: the input layer (not computational) and the output layer, also known as the Kohonen layer. x 1 (t) w 11 (t) v 1 (t) I 1 O 1 x 2 (t) I 2 O 2 v 2 (t) x i (t) I i w ij (t) O j v j (t) Input Layer Output Layer Figure 1. The layers and vectors of a SOM: input vector x(t) = [x 1(t)x 2(t)... x i(t)], weight vector w j(t) = [w 1,1w 1,2... w i,j] and activation level v j(t).
99 When a stimulus x i (t) is presented, neurons compete by mutual lateral inhibition. The winner neuron has the higher activation level v j (t). So, the activation level v j (t) can be expressed as follows, in which w i,j is the i th element of the weight vector w j (t), v j (t) is the activation level of the j th neuron, and x i (t) is the i th element of the input vector x(t): v j (t) = w 1,1 (t)x 1 (t) + w 2,1 (t)x 2 (t) w i,j (t)x i (t) The training formula for a neuron with weight vector w j (t) is given as follows, in which α(t) is the learning coefficient that is gradually reduced and h j (t) is the neighborhood function: w j (t + 1) = w j (t) + h j (t)α(t) [ x(t) w j (t)] During the training, the neighborhood function h j (t) depends on the distance between the winner neuron and the j th neuron. This function could be as simple as a constant for a number of neurons close enough to the winner neuron, but an widely used choice is a gaussian function [10]. The neighborhood function shrinks from one training epoch to another. The training process is repeated for all input vectors for a number of interactions. The network associate output nodes with clusters of input data sharing some characteristics, and each output node is associated with a class of data. 4 The SOM-PORTRAIT Method In this section, we present our method, showing how the training set was built, describing our SOM-PORTRAIT method, and giving details of the model configuration and implementation characteristics. 4.1 Training set construction The training set for SOM-PORTRAIT was built following exactly the same steps designed for the SVM-PORTRAIT method [3], which allowed us to compare the results obtained from both methods. The SOM-PORTRAIT training set consists of two sets, one composed by known protein-coding sequences (mrnas) and the other formed from known non-coding sequences (ncrnas). The mrnas set was primarily downloaded from the SwissProt database [2], version 50.8, in october Redundant sequences were eliminated using CD- HIT [15] with sequence similarity above 70% and running BLASTCLUST over the remaining sequences. The ORF prediction was done by ANGLE [23], and the obtained results were separated into three distinct files: a file containing nucleotide sequences with predicted ORFs, a file containing the corresponding amino acid sequences as predicted by ANGLE, and another file containing nucleotide sequences without predicted ORFs. The ncrnas set was also downloaded in october 2006 and contained sequences from NONCODE [16], Rfam [9] and RNAdb [21] databases, with redundant sequences eliminated using BLASTCLUST. The ANGLE prediction was done exactly in the same way as described for the mrnas set.
100 4.2 The method description The SOM-PORTRAIT method uses ab initio steps to predict ncrna in a transcriptome. The method was originally conceived for two classes (coding RNA and non-coding RNA), but it could be easily modified to create more classes. We also worked with a three classes model, as discussed later. The method used ANGLE to identify nucleotide sequences with and without predictable ORFs, but another ORF predictor could be used as well. Besides, attributes from the sequences with predicted ORFs were extracted separately from those without predicted ORFs. Sequence attributes must be converted to numerical data, that are the real input for the SOM-PORTRAIT. These attributes were chosen based on their hypothetical relevance to distinguish mrnas from ncr- NAs, and they are exactly the same as those adopted for the SVM-PORTRAIT experiments. We developed PERL scripts to extract, from each sequence, 111 variables, which were grouped into the following 7 main attributes, listed with the number of the variables generated by each attribute: nucleotide composition (84 variables), ORF length (4 variables), amino acid composition (20 variables), protein isoeletric point (1 variable), protein complexity (1 variable), mean protein hidropathy (1 variable), length (4 variables). Note that attributes 2 to 6 refer only to sequences with predicted ORFs. Attributes 1 and 7 are extracted from both sequences with and without ORFs. So, the protein-dependent model consists of 111 variables comprised in 6 attributes, and the protein-independent model consists of 88 variables comprised in 2 attributes. The extracting attributes step generates two input data files, each one containing numerical data relative to all the nucleotide sequences. The first file, containing sequences with predicted ORFs, is sent to prediction using a proteindependent SOM (called model.withorf.map), and the second file, containing sequences without predicted ORFs, is sent to prediction using the proteinindependent SOM (called model.withoutorf.map). Details for the two maps configuration are explained in the following section. Figure 2 shows the SOM- PORTRAIT workflow, together with the main input/output files. 4.3 Model configuration and implementation details We used the SOM-PAK [13] library (version 3.1) to configure the two models, one with ORFs and the other without ORFs. The machine used for training was a PC with a dual processor (Core 2 Duo 2.0 Ghz) and a 2,024 MHz RAM, executing Linux Ubuntu 8.04 (kernel generic). First, we configured a two classes model to classify the transcripts in coding RNA (mrna) and non-coding RNA (ncrna). Following, we configured a three classes model, creating an additional class labeled undefined, trying to refine the classification of transcripts in another RNA class. For each of these experiments, two SOMs were configured, respectively, model.withorf.map and proteindependent, for sequences with predicted ORFs, and model.withoutorf.map and protein-independent, for sequences without predicted ORFs.
101 Figure 2. The workflow of the SOM-PORTRAIT program. The execution of the SOM-PAK library created many files from the original training sets, and the desired attributes were extracted using a PERL script (parameter extractor.pl). This script ran approximately 11 hours for the proteindependent model and 3 hours for the protein-independent model, for both SOM models - with two and three classes. Also, both SOM-PORTRAIT models executed in less than 10 minutes of real CPU time, with time estimated using the time shell command, in the machines described in Section 4.3. Two Classes For the two classes experiment, the attribute files were further subdivided (Table 1). We note that in general a greater number of classes are used for SOM models. This experiment was developed in order to compare the results produced by SOM-PORTRAIT and the two classes SVM-PORTRAIT. The configuration of the two classes SOM model was performed on three steps. First, the map was initialized as follows. SOM-PAK has a random initial distribution of the weights in the map. We chose a map with two nodes (representing the two classes), a rectangular topology and the gaussian function as the neighborhood function h j (t). The input data were the.dat files shown in Table 1. The second step was to organize the values of the.dat files between the two nodes initialized in the first step. We set the training rate α to 0.05, the radius for the initial neighborhood to 1, and the iteration value to 20, 000 steps. These parameters were chosen following the recommendation for SOM training
102 Table 1. Each column shows, for each sequence file, respectively, its name, contents and number of sequences. Name Purpose Sequences model.withorf.dat data file for ORF map creation 20,000 model.withoutorf.dat data file for non-orf map creation 12,555 model.withorf.test test file for quantization error estimate 76,827 model.withoutorf.test test file for quantization error estimate 47,151 of SOM-PAK [13] and took about 10 minutes. On the third step, a smaller training rate α(0) = 0.02 was adopted, the radius for the initial neighborhood was h j (0) = 1, and the number of iterations t was fixed in 200, 000 steps. The trained map was submitted to a large test file, containing all the attributes extracted from the training set sequences (.test files), to estimate the average quantization error (AQE). AQE is a statistical measure of the SOM training accuracy, and is calculated as the mean of the Euclidean distance x i (t) v c (t), where v c (t) is the winner node computed by SOM. In our experiments, the protein-dependent map AQE was 0.93, while the protein-independent map AQE was Three Classes Then, we configured a three classes SOM model, performing training and configuration analogously to the two classes SOM-PORTRAIT (Table 1). In the first step, we chose a map with three nodes (representing the three classes), a rectangular topology and the gaussian function as the neighborhood function, analogously to the two classes model. The input data was the.dat files in Table 1. In the second step of organizing the map, we set the training rate α to 0.05, the radius for the initial neighborhood to 2, and the iteration value to 20, 000 steps. We used the same parameters adopted for the two classes experiment. For the last step, we used a smaller training rate of α(0) = 0.02, the radius for the initial neighborhood was set to 1, and the number of iterations was fixed in 200, 000 steps. SOM-PAK estimated 0.79 for the protein-dependent map AQE, and 0.49 for the protein-independent map AQE. 5 A case study: the Paracoccidioidis brasiliensis fungus The testing set used to validate the SOM-PORTRAIT method was the transcriptome of the Paracoccidioidis brasiliensis fungus, named Pb transcriptome, that has 6,022 Assembled ESTs [7]. A PERL script was developed to filter this data set, accepting a sequence if it had length with at least 80 nucleotides and at most 20% of characters different from A, C, G and T. This filter script discarded 9 sequences, from the 6,022 Pb Assembled ESTs, generating a final testing set containing 6,013 Assembled ESTs. The machines used for testing and validation were the same described in Section 4.3. SOM-PORTRAIT workflow (Figure 2) was implemented in PERL. The testing set was executed by the program in 13 minutes of real CPU time.
103 A local version of the SVM-PORTRAIT was completely reimplemented. Training, testing and validation were done exactly as specified by Arrial and co-authors [3,4]. We used four machines for training and validation, all with processor Core 2 Duo 2.2 GHz, three with 2,024 Mhz RAM (Machines 1, 3 e 4), and one with 3,036 Mhz RAM (Machine 2). Machines 1 and 2 were used for training and validating the protein-dependent SVM model, while machines 3 and 4 were used for training and validating the protein-independent SVM model. For training and validation, the parameters and optimal values were adjusted following the specifications of the SVM-PORTRAIT. These two steps executed in approximately 40 hours for the protein-dependent model and 30 hours for the protein-independent model of real CPU time. Now, we show two experiments with SOM-PORTRAIT, the first one with two classes and the second one with three classes, comparing the obtained results with the SVM-PORTRAIT output. Finally, we compare the results obtained by the two SOM-PORTRAIT models. For these comparisons, we used as input the sequences of SVM-PORTRAIT presenting classification probability above 70%, which discarded 645 sequences, or 10, 78% from the above 6,013 sequences. So, for the experiments, we considered a total of 5, 365 Assembled ESTs from the Pb transcriptome. 5.1 Experiment 1: Two Classes The same test steps done by SVM-PORTRAIT to classify the Assembled ESTs of the Pb transcriptome were repeated for the two classes SOM-PORTRAIT. The classification step executed 12 minutes of real CPU time. Table 2 shows that the percentages of coding sequences and ncrnas found by SVM-PORTRAIT and the two classes SOM-PORTRAIT are very close. The third portion of this table shows a comparison between the classification as coding and non-coding sequences done by both methods. Table 2. Comparisons between the two classes SOM-PORTRAIT and SVM- PORTRAIT. The percentage is relative to the 5,365 sequences of the Pb transcriptome. Sequences Percentage SVM-PORTRAIT Coding sequences 4, % ncrnas % SOM-PORTRAIT Coding sequences 4, % ncrnas % SOM-PORTRAIT SVM-PORTRAIT coding/coding 4, % non-coding/non-coding % coding/non-coding % non-coding/coding %
104 Comparisons of Table 2 are shown in Figure 3, in which each circle represents the total of sequences classified as non-coding (left) and coding (right) by SVM- PORTRAIT, and inside each circle we represent the total of sequences classified by SOM-PORTRAIT as non-coding (black) and coding (light gray). Figure 3. Classifications produced for the Assembled ESTs of the Pb transcriptome by the SVM-PORTRAIT and the two classes SOM-PORTRAIT. These results show that the two methods found very different classification for the input data, specially for non-coding sequences. This could be explained by the fact that the class of ncrnas is very heterogeneous. SVM-PORTRAIT defined two classes, which means that the training step of this method included all ncrnas in just one class. For the SOM-PORTRAIT model, we did not force the classification, that is, each class is built by the method taking the closest sequences. This analysis led us to develop another experiment including more classes in SOM-PORTRAIT, described in the next section. In fact, for the three classes SOM model, these results were improved, as we will see. Nonetheless, the non-supervised learning algorithm adopted in our SOM-PORTRAIT method reached a data classification accuracy comparable to that obtained by the SVM- PORTRAIT supervised learning method, with the clear advantages of reducing training time and needing less computational memory. 5.2 Experiment 2: Three Classes The 5,365 sequences of the Pb transcriptome were submitted as input for a three classes SOM-PORTRAIT. The classification step executed in 13 minutes of real CPU time. Table 3 shows that the percentages of ncrnas found by SVM-PORTRAIT and the three classes SOM-PORTRAIT remain very close. Comparisons of Table 3 are shown in Figure 4, in which each circle represents the total of sequences classified as non-coding (left) and coding (right) by SVM- PORTRAIT, and inside each circle we represent the total of sequences classified by the three classes SOM-PORTRAIT as non-coding (dark gray), coding (light gray) and undefined (black). We can note that the undefined class was composed most by the SVM- PORTRAIT non-coding class. The sequences classified by SOM-PORTRAIT as
105 Table 3. Comparisons between the three classes SOM-PORTRAIT and SVM- PORTRAIT. The percentage is relative to the 5,365 sequences of the Pb transcriptome. Sequences Percentage SOM-PORTRAIT Coding sequences 4, % ncrnas % Undefined sequences class % SVM-PORTRAIT SOM-PORTRAIT coding/coding 4, % non-coding/non-coding % coding/ undefined % non-coding/ undefined % coding/non-coding % non-coding/coding % Figure 4. Comparisons between the classification for the Pb transcriptome produced by the SVM-PORTRAIT and the three classes SOM-PORTRAIT. coding when compared to those found by the SVM-PORTRAIT non-coding class slightly shrink, indicating that the new undefined class grouped some of the sequences that were differently classified in the first experiment. This might indicate new possibilities for classifying, that could be done by the SOM model. Furthermore, it could be directly correlated to the great number of different classifications of the non-coding sequences found by the two classes SOM-PORTRAIT with respect to the SVM-PORTRAIT, which indicates that we could create more classes in the SOM model to increase the classification accuracy. 5.3 Comparing two and three classes SOM-PORTRAIT Figure 5 shows a comparison between both SOM-PORTRAIT models, in which each circle represents the total of sequences classified as non-coding (left) and coding (right) by the two classes SOM-PORTRAIT, and the portions inside each circle shows the total of sequences classified as non-coding (dark gray), coding (light gray) and undefined (black) by the three classes SOM-PORTRAIT. Notice that the non-coding class remains almost the same on both models. It is interesting to note that the non-coding class of SOM-PORTRAIT remains constant between two and three classes classifier, but a fraction of the
106 Figure 5. Comparisons of the classifications for the 6,013 Assembled ESTs of the Pb transcriptome produced by two classes and three classes SOM-PORTRAIT. coding sequences of the two classes SOM-PORTRAIT was transferred to the undefined class of the three classes classifier. But, this undefined fraction was classified as non-coding by the SVM-PORTRAIT. Therefore, it is tempting to explain this undefined class as a bona fide ncrna quite close to coding RNA, that could be wrongly classified when the classification is restricted to two classes. 6 Conclusions and future work In this work we proposed a method to identify non-coding RNAs using Self Organizing Maps (SOM), named SOM-PORTRAIT. We implemented the method and applied it to a data set containing Assembled ESTs of the Paracoccidioides brasiliensis fungus transcriptome. The obtained results were reliable, when compared to a method based on the SVM paradigm, noting that the time and memory requirements needed to our SOM-PORTRAIT is much less than those needed for methods based on the SVM paradigm, like PORTRAIT. The following step is to assess the sensitivity and accuracy of our method, which could be done by applying SOM-PORTRAIT to randomly chosen known coding and non-coding RNAs and comparing the results to other ncrna predictor methods. We also could test how much ANGLE affects the accuracy of the SOM-PORTRAIT method, removing ANGLE from the method and considering only the protein independent parameters to analyze how much the accuracy would be changed. Other interesting works are to develop WEB interfaces for the SOM-PORTRAIT allowing the user to select features to create classes according to his needs, to create more classes to see if specific sets of ncrnas could be found, to use training pruners, and to include confidence level to the classification. References 1. S. F. Altschul, T. L. Madden, A. A. Schäffer, J. Zhang, Z. Zhang, W. Miller, and D. J. Lipman. Gapped BLAST and PSI-BLAST: a new generation of protein
- Ácido ribonucléico (ARN ou RNA): participa do processo de síntese de proteínas.
 1- TIPOS DE ÁCIDO NUCLÉICO: DNA E RNA Existem dois tipos de ácidos nucléicos: - Ácido desoxirribonucléico (ADN ou DNA): é o principal constituinte dos cromossomos, estrutura na qual encontramos os genes,
1- TIPOS DE ÁCIDO NUCLÉICO: DNA E RNA Existem dois tipos de ácidos nucléicos: - Ácido desoxirribonucléico (ADN ou DNA): é o principal constituinte dos cromossomos, estrutura na qual encontramos os genes,
Bioinformática. Conceitos Fundamentais de Biologia Molecular. Paulo Henrique Ribeiro Gabriel phrg@ufu.br
 Bioinformática Conceitos Fundamentais de Biologia Molecular Paulo Henrique Ribeiro Gabriel phrg@ufu.br Faculdade de Computação Universidade Federal de Uberlândia 24 de agosto de 2015 Paulo H. R. Gabriel
Bioinformática Conceitos Fundamentais de Biologia Molecular Paulo Henrique Ribeiro Gabriel phrg@ufu.br Faculdade de Computação Universidade Federal de Uberlândia 24 de agosto de 2015 Paulo H. R. Gabriel
Criado e Desenvolvido por: RONNIELLE CABRAL ROLIM Todos os direitos são reservados 2015. www.tioronni.com
 Criado e Desenvolvido por: RONNIELLE CABRAL ROLIM Todos os direitos são reservados 2015. www.tioronni.com ÁCIDOS NUCLEICOS ÁCIDOS NUCLÉICOS: são substâncias formadoras de genes, constituídas por um grande
Criado e Desenvolvido por: RONNIELLE CABRAL ROLIM Todos os direitos são reservados 2015. www.tioronni.com ÁCIDOS NUCLEICOS ÁCIDOS NUCLÉICOS: são substâncias formadoras de genes, constituídas por um grande
Princípios moleculares dos processos fisiológicos
 2012-04-30 UNIVERSIDADE AGOSTINHO NETO FACULDADE DE CIÊNCIAS DEI-BIOLOGIA ---------------------------------------------- Aula 5: Princípios moleculares dos processos fisiológicos (Fisiologia Vegetal, Ano
2012-04-30 UNIVERSIDADE AGOSTINHO NETO FACULDADE DE CIÊNCIAS DEI-BIOLOGIA ---------------------------------------------- Aula 5: Princípios moleculares dos processos fisiológicos (Fisiologia Vegetal, Ano
Hoje estudaremos a bioquímica dos ácidos nucléicos. Acompanhe!
 Aula: 2 Temática: Ácidos Nucléicos Hoje estudaremos a bioquímica dos ácidos nucléicos. Acompanhe! Introdução: Os ácidos nucléicos são as moléculas com a função de armazenamento e expressão da informação
Aula: 2 Temática: Ácidos Nucléicos Hoje estudaremos a bioquímica dos ácidos nucléicos. Acompanhe! Introdução: Os ácidos nucléicos são as moléculas com a função de armazenamento e expressão da informação
ÁCIDOS NUCLEÍCOS RIBOSSOMO E SÍNTESE PROTEÍCA
 ÁCIDOS NUCLEÍCOS RIBOSSOMO E SÍNTESE PROTEÍCA ÁCIDOS NUCLÉICOS: Moléculas orgânicas complexas, formadas polimerização de nucleotídeos (DNA e RNA) pela Contêm a informação que determina a seqüência de aminoácidos
ÁCIDOS NUCLEÍCOS RIBOSSOMO E SÍNTESE PROTEÍCA ÁCIDOS NUCLÉICOS: Moléculas orgânicas complexas, formadas polimerização de nucleotídeos (DNA e RNA) pela Contêm a informação que determina a seqüência de aminoácidos
Núcleo Celular. Biomedicina primeiro semestre de 2012 Profa. Luciana Fontanari Krause
 Núcleo Celular Biomedicina primeiro semestre de 2012 Profa. Luciana Fontanari Krause Núcleo Celular Eucarioto: núcleo delimitado por membrana nuclear (carioteca) Portador dos fatores hereditários e controlador
Núcleo Celular Biomedicina primeiro semestre de 2012 Profa. Luciana Fontanari Krause Núcleo Celular Eucarioto: núcleo delimitado por membrana nuclear (carioteca) Portador dos fatores hereditários e controlador
MEDICINA VETERINÁRIA. Disciplina: Genética Animal. Prof a.: D rd. Mariana de F. Gardingo Diniz
 MEDICINA VETERINÁRIA Disciplina: Genética Animal Prof a.: D rd. Mariana de F. Gardingo Diniz TRANSCRIÇÃO DNA A transcrição é o processo de formação de uma molécula de RNA a partir de uma molécula molde
MEDICINA VETERINÁRIA Disciplina: Genética Animal Prof a.: D rd. Mariana de F. Gardingo Diniz TRANSCRIÇÃO DNA A transcrição é o processo de formação de uma molécula de RNA a partir de uma molécula molde
Equipe de Biologia. Biologia
 Aluno (a): Série: 3ª Turma: TUTORIAL 5B Ensino Médio Equipe de Biologia Data: Biologia Ácidos nucléicos Os ácidos nucléicos são moléculas gigantes (macromoléculas), formadas por unidades monoméricas menores
Aluno (a): Série: 3ª Turma: TUTORIAL 5B Ensino Médio Equipe de Biologia Data: Biologia Ácidos nucléicos Os ácidos nucléicos são moléculas gigantes (macromoléculas), formadas por unidades monoméricas menores
Ácidos nucléicos. São polímeros compostos por nucleotídeos. Açúcar - pentose. Grupo fosfato. Nucleotídeo. Base nitrogenada
 ÁCIDOS NUCLÉICOS Ácidos nucléicos São polímeros compostos por nucleotídeos Açúcar - pentose Nucleotídeo Grupo fosfato Base nitrogenada Composição dos Ácidos nucléicos pentoses: numeração da pentose: pentose
ÁCIDOS NUCLÉICOS Ácidos nucléicos São polímeros compostos por nucleotídeos Açúcar - pentose Nucleotídeo Grupo fosfato Base nitrogenada Composição dos Ácidos nucléicos pentoses: numeração da pentose: pentose
BASES NITROGENADAS DO RNA
 BIO 1E aula 01 01.01. A determinação de como deve ser uma proteína é dada pelos genes contidos no DNA. Cada gene é formado por uma sequência de códons, que são sequências de três bases nitrogenadas que
BIO 1E aula 01 01.01. A determinação de como deve ser uma proteína é dada pelos genes contidos no DNA. Cada gene é formado por uma sequência de códons, que são sequências de três bases nitrogenadas que
O fluxo da informação é unidirecional
 Curso - Psicologia Disciplina: Genética Humana e Evolução Resumo Aula 3- Transcrição e Tradução Dogma central TRANSCRIÇÃO DO DNA O fluxo da informação é unidirecional Processo pelo qual uma molécula de
Curso - Psicologia Disciplina: Genética Humana e Evolução Resumo Aula 3- Transcrição e Tradução Dogma central TRANSCRIÇÃO DO DNA O fluxo da informação é unidirecional Processo pelo qual uma molécula de
Estrutura e função dos ácidos nucléicos. Profa. Melissa de Freitas Cordeiro-Silva
 Estrutura e função dos ácidos nucléicos Profa. Melissa de Freitas Cordeiro-Silva > Polímeros de nucleotídeos Funções: DNA (ácido desoxirribonucléico) : > Armazenar as informações necessárias para a construção
Estrutura e função dos ácidos nucléicos Profa. Melissa de Freitas Cordeiro-Silva > Polímeros de nucleotídeos Funções: DNA (ácido desoxirribonucléico) : > Armazenar as informações necessárias para a construção
Genética Humana. Prof. João Ronaldo Tavares de Vasconcellos Neto
 Genética Humana Prof. João Ronaldo Tavares de Vasconcellos Neto JAN/2012 Princípios Básicos As proteínas são vinculo entre genótipo e fenótipo; A expressão gênica é o processo pelo qual o DNA coordena
Genética Humana Prof. João Ronaldo Tavares de Vasconcellos Neto JAN/2012 Princípios Básicos As proteínas são vinculo entre genótipo e fenótipo; A expressão gênica é o processo pelo qual o DNA coordena
As bactérias operárias
 A U A UL LA As bactérias operárias Na Aula 47 você viu a importância da insulina no nosso corpo e, na Aula 48, aprendeu como as células de nosso organismo produzem insulina e outras proteínas. As pessoas
A U A UL LA As bactérias operárias Na Aula 47 você viu a importância da insulina no nosso corpo e, na Aula 48, aprendeu como as células de nosso organismo produzem insulina e outras proteínas. As pessoas
Bioinformática Aula 01
 Bioinformática Aula 01 Prof. Ricardo Martins Ramos * * Doutorando em Genética e Toxicologia Aplicada CEFET-PI/ULBRA-RS Linha de Pesquisa Bioinformática Estrutural E-mail: ricardo@cefetpi.br Visão Holística
Bioinformática Aula 01 Prof. Ricardo Martins Ramos * * Doutorando em Genética e Toxicologia Aplicada CEFET-PI/ULBRA-RS Linha de Pesquisa Bioinformática Estrutural E-mail: ricardo@cefetpi.br Visão Holística
DNA E SÍNTESE PROTEICA
 Genética Animal DNA e síntese proteica 1 DNA E SÍNTESE PROTEICA Estrutura do DNA: -Molécula polimérica, cujos monômeros denominam-se nucleotídeos. -Constituição dos nucleotídeos: açúcar pentose (5 -desoxirribose)
Genética Animal DNA e síntese proteica 1 DNA E SÍNTESE PROTEICA Estrutura do DNA: -Molécula polimérica, cujos monômeros denominam-se nucleotídeos. -Constituição dos nucleotídeos: açúcar pentose (5 -desoxirribose)
Os primeiros indícios de que o DNA era o material hereditário surgiram de experiências realizadas com bactérias, sendo estas indicações estendidas
 GENERALIDADES Todo ser vivo consiste de células, nas quais está situado o material hereditário. O número de células de um organismo pode variar de uma a muitos milhões. Estas células podem apresentar-se
GENERALIDADES Todo ser vivo consiste de células, nas quais está situado o material hereditário. O número de células de um organismo pode variar de uma a muitos milhões. Estas células podem apresentar-se
BANCO DE QUESTÕES - BIOLOGIA - 1ª SÉRIE - ENSINO MÉDIO ==============================================================================================
 PROFESSOR: Leonardo Mariscal BANCO DE QUESTÕES - BIOLOGIA - 1ª SÉRIE - ENSINO MÉDIO ============================================================================================== Ácidos Nucleicos 01- Os
PROFESSOR: Leonardo Mariscal BANCO DE QUESTÕES - BIOLOGIA - 1ª SÉRIE - ENSINO MÉDIO ============================================================================================== Ácidos Nucleicos 01- Os
Replicação Quais as funções do DNA?
 Replicação Quais as funções do DNA? Aula nº 4 22/Set/08 Prof. Ana Reis Replicação O DNA é a molécula que contém a informação para todas as actividades da célula. Uma vez que as células se dividem, é necessário
Replicação Quais as funções do DNA? Aula nº 4 22/Set/08 Prof. Ana Reis Replicação O DNA é a molécula que contém a informação para todas as actividades da célula. Uma vez que as células se dividem, é necessário
O DNA é formado por pedaços capazes de serem convertidos em algumas características. Esses pedaços são
 Atividade extra Fascículo 2 Biologia Unidade 4 Questão 1 O DNA é formado por pedaços capazes de serem convertidos em algumas características. Esses pedaços são chamados de genes. Assinale abaixo quais
Atividade extra Fascículo 2 Biologia Unidade 4 Questão 1 O DNA é formado por pedaços capazes de serem convertidos em algumas características. Esses pedaços são chamados de genes. Assinale abaixo quais
COMUNICAÇÃO DA INFORMAÇÃO NAS MOLÉCULAS DE DNA E RNA
 COMUNICAÇÃO DA INFORMAÇÃO NAS MOLÉCULAS DE DNA E RNA Andréia Cristina Hypólito José 11075810 Fernando Caldas Oliveira 11085410 Giovana Zaninelli 11017210 Renato Fernandes Sartori 11061110 Rodrigo de Mello
COMUNICAÇÃO DA INFORMAÇÃO NAS MOLÉCULAS DE DNA E RNA Andréia Cristina Hypólito José 11075810 Fernando Caldas Oliveira 11085410 Giovana Zaninelli 11017210 Renato Fernandes Sartori 11061110 Rodrigo de Mello
MEDICINA VETERINÁRIA. Disciplina: Genética Animal. Prof a.: Drd. Mariana de F. G. Diniz
 MEDICINA VETERINÁRIA Disciplina: Genética Animal Prof a.: Drd. Mariana de F. G. Diniz Gene, é a unidade fundamental da hereditariedade. Cada gene é formado por uma sequência específica de ácidos nucléicos
MEDICINA VETERINÁRIA Disciplina: Genética Animal Prof a.: Drd. Mariana de F. G. Diniz Gene, é a unidade fundamental da hereditariedade. Cada gene é formado por uma sequência específica de ácidos nucléicos
> ESTUDO DO RNA. (C) O ácido nucléico I é DNA e o II, RNA. (D) O ácido nucléico I é RNA e o II, DNA. (E) I é exclusivo dos seres procariontes.
 Biologia > Citologia > Sintese Protéica > Alunos Prof. Zell (biologia) (C) O ácido nucléico I é DNA e o II, RNA. (D) O ácido nucléico I é RNA e o II, DNA. (E) I é exclusivo dos seres procariontes. > ESTUDO
Biologia > Citologia > Sintese Protéica > Alunos Prof. Zell (biologia) (C) O ácido nucléico I é DNA e o II, RNA. (D) O ácido nucléico I é RNA e o II, DNA. (E) I é exclusivo dos seres procariontes. > ESTUDO
14/02/2017. Genética. Professora Catarina
 14/02/2017 Genética Professora Catarina 1 A espécie humana Ácidos nucleicos Tipos DNA ácido desoxirribonucleico RNA ácido ribonucleico São formados pela união de nucleotídeos. 2 Composição dos nucleotídeos
14/02/2017 Genética Professora Catarina 1 A espécie humana Ácidos nucleicos Tipos DNA ácido desoxirribonucleico RNA ácido ribonucleico São formados pela união de nucleotídeos. 2 Composição dos nucleotídeos
Estrutura e Função de Ácidos Nucléicos
 UNIVERSIDADE DE SÃO PAULO INSTITUTO DE QUÍMICA DEPARTAMENTO DE BIOQUÍMICA QBQ0313 Estrutura e Função de Ácidos Nucléicos Flavia Carla Meotti Os Ácidos Nucléicos Função: armazenamento e transmissão da informação
UNIVERSIDADE DE SÃO PAULO INSTITUTO DE QUÍMICA DEPARTAMENTO DE BIOQUÍMICA QBQ0313 Estrutura e Função de Ácidos Nucléicos Flavia Carla Meotti Os Ácidos Nucléicos Função: armazenamento e transmissão da informação
ACESSO VESTIBULAR QUESTÕES DE PROCESSAMENTO DE RNA OU SPLICING 01. (MAMA 2007.1) PÁGINAS OCULTAS NO LIVRO DA VIDA
 ACESSO VESTIBULAR QUESTÕES DE PROCESSAMENTO DE RNA OU SPLICING 01. (MAMA 2007.1) PÁGINAS OCULTAS NO LIVRO DA VIDA Os biólogos supunham que apenas as proteínas regulassem os genes dos seres humanos e dos
ACESSO VESTIBULAR QUESTÕES DE PROCESSAMENTO DE RNA OU SPLICING 01. (MAMA 2007.1) PÁGINAS OCULTAS NO LIVRO DA VIDA Os biólogos supunham que apenas as proteínas regulassem os genes dos seres humanos e dos
Transcrição e Tradução. Profa. Dra. Juliana Garcia de Oliveira Disciplina: Biologia Celular e Molecular Turmas: Biologia, enfermagem, nutrição e TO.
 Transcrição e Tradução Profa. Dra. Juliana Garcia de Oliveira Disciplina: Biologia Celular e Molecular Turmas: Biologia, enfermagem, nutrição e TO. Tópicos abordados na aula Dogma Central da Biologia Molecular;
Transcrição e Tradução Profa. Dra. Juliana Garcia de Oliveira Disciplina: Biologia Celular e Molecular Turmas: Biologia, enfermagem, nutrição e TO. Tópicos abordados na aula Dogma Central da Biologia Molecular;
DO GENE À PROTEÍNA ALGUNS CONCEITOS BASICOS COMO SE ORGANIZAM OS NUCLEÓTIDOS PARA FORMAR O DNA?
 DO GENE À PROTEÍNA O processo de formação das proteínas no ser humano pode ser difícil de compreender e inclui palavras e conceitos que possivelmente nos são desconhecidos. Assim, vamos tentar explicar
DO GENE À PROTEÍNA O processo de formação das proteínas no ser humano pode ser difícil de compreender e inclui palavras e conceitos que possivelmente nos são desconhecidos. Assim, vamos tentar explicar
A natureza química do material genético Miescher nucleínas. ácidos nucleicos. ácido desoxirribonucleico ácido ribonucleico Avery MacLeod McCarty
 UNIVERSIDADE FEDERAL DO RIO GRANDE DO SUL COLÉGIO DE APLICAÇÃO Departamento de Ciências Exatas e da Natureza Disciplina: Biologia Professora: Lauren Valentim A natureza química do material genético A natureza
UNIVERSIDADE FEDERAL DO RIO GRANDE DO SUL COLÉGIO DE APLICAÇÃO Departamento de Ciências Exatas e da Natureza Disciplina: Biologia Professora: Lauren Valentim A natureza química do material genético A natureza
Bases Moleculares da Hereditariedade
 UNIVERSIDADE FEDERAL DO PIAUÍ CENTRO DE CIÊNCIAS AGRÁRIAS PROG. DE PÓS GRAD. EM GENET. E MELHORAMENTO NÚCLEO DE ESTUDOS EM GENET. E MELHORAMENTO Bases Moleculares da Hereditariedade Ministrante: João Paulo
UNIVERSIDADE FEDERAL DO PIAUÍ CENTRO DE CIÊNCIAS AGRÁRIAS PROG. DE PÓS GRAD. EM GENET. E MELHORAMENTO NÚCLEO DE ESTUDOS EM GENET. E MELHORAMENTO Bases Moleculares da Hereditariedade Ministrante: João Paulo
1. (Unesp) A ilustração apresenta o resultado de um teste de paternidade obtido pelo método do DNA-Fingerprint, ou "impressão digital de DNA".
 Ácidos Nuclêicos 1. (Unesp) A ilustração apresenta o resultado de um teste de paternidade obtido pelo método do DNA-Fingerprint, ou "impressão digital de DNA". a) Segundo o resultado acima, qual dos homens,
Ácidos Nuclêicos 1. (Unesp) A ilustração apresenta o resultado de um teste de paternidade obtido pelo método do DNA-Fingerprint, ou "impressão digital de DNA". a) Segundo o resultado acima, qual dos homens,
Criado e Desenvolvido por: Todos os direitos são reservados 2015. www.tioronni.com
 Criado e Desenvolvido por: Todos os direitos são reservados 2015. www.tioronni.com O NÚCLEO E A SÍNTESE PROTEÍCA O núcleo celular, descoberto em 1833 pelo pesquisador escocês Robert Brown, é uma estrutura
Criado e Desenvolvido por: Todos os direitos são reservados 2015. www.tioronni.com O NÚCLEO E A SÍNTESE PROTEÍCA O núcleo celular, descoberto em 1833 pelo pesquisador escocês Robert Brown, é uma estrutura
O processo fisiológico que está representado no gráfico é
 Questão 01) Analise o gráfico a seguir. Disponível em: . Acesso em: 22 set. 2014. O processo fisiológico que está representado no gráfico é a) o efeito do aumento
Questão 01) Analise o gráfico a seguir. Disponível em: . Acesso em: 22 set. 2014. O processo fisiológico que está representado no gráfico é a) o efeito do aumento
Genes. Menor porção do DNA capaz de produzir um efeito que pode ser detectado no organismo. Região do DNA que pode ser transcrita em moléculas de RNA.
 Genes Menor porção do DNA capaz de produzir um efeito que pode ser detectado no organismo. Região do DNA que pode ser transcrita em moléculas de RNA. Ácidos nucleicos Os ácidos nucléicos são macromoléculas
Genes Menor porção do DNA capaz de produzir um efeito que pode ser detectado no organismo. Região do DNA que pode ser transcrita em moléculas de RNA. Ácidos nucleicos Os ácidos nucléicos são macromoléculas
23/03/2015. Moléculas orgânicas - Carboidratos
 Moléculas orgânicas - Carboidratos São formados por C, H, O. São Conhecidos como: Hidratos de Carbono Glucídios Glicídios Açúcares Sacarídeos Funções: Energética (glicose); Glicogênio : reserva energética
Moléculas orgânicas - Carboidratos São formados por C, H, O. São Conhecidos como: Hidratos de Carbono Glucídios Glicídios Açúcares Sacarídeos Funções: Energética (glicose); Glicogênio : reserva energética
Introdução à Biologia Celular e Molecular
 Introdução à Biologia Celular e Molecular Este texto foi retirado do anexo de [Lem00], revisado por [Bas00], e tem como objetivo principal apresentar alguns conceitos básicos de biologia celular e molecular.
Introdução à Biologia Celular e Molecular Este texto foi retirado do anexo de [Lem00], revisado por [Bas00], e tem como objetivo principal apresentar alguns conceitos básicos de biologia celular e molecular.
BIOLOGIA MOLECULAR. Ácidos Nucléicos e Síntese de Proteínas
 BIOLOGIA MOLECULAR Ácidos Nucléicos e Síntese de Proteínas Nucleotídeos São moléculas formadas pela união de um açúcar ou pentose, uma base nitrogenada e um grupo fosfato. Os Ácidos Nucléicos (DNA e RNA)
BIOLOGIA MOLECULAR Ácidos Nucléicos e Síntese de Proteínas Nucleotídeos São moléculas formadas pela união de um açúcar ou pentose, uma base nitrogenada e um grupo fosfato. Os Ácidos Nucléicos (DNA e RNA)
A Molécula da Vida. Estrutura
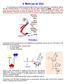 A Molécula da Vida Os cromossomos de células eucarióticas são formado por DNA associado a moléculas de histona, que são proteínas básicas. É na molécula de DNA que estão contidos os genes, responsáveis
A Molécula da Vida Os cromossomos de células eucarióticas são formado por DNA associado a moléculas de histona, que são proteínas básicas. É na molécula de DNA que estão contidos os genes, responsáveis
Genética e Evolução: Profa. Gilcele
 Genética e Evolução: Profa. Gilcele Genética É o estudo dos genes e de sua transmissão para as gerações futuras. É o estudo da hereditariedade, a transmissão de traços de genitores para filhos. É dividida
Genética e Evolução: Profa. Gilcele Genética É o estudo dos genes e de sua transmissão para as gerações futuras. É o estudo da hereditariedade, a transmissão de traços de genitores para filhos. É dividida
BIOLOGIA MOLECULAR. Prof. Dr. José Luis da C. Silva
 BIOLOGIA MOLECULAR Prof. Dr. José Luis da C. Silva BIOLOGIA MOLECULAR A Biologia Molecular é o estudo da Biologia em nível molecular, com especial foco no estudo da estrutura e função do material genético
BIOLOGIA MOLECULAR Prof. Dr. José Luis da C. Silva BIOLOGIA MOLECULAR A Biologia Molecular é o estudo da Biologia em nível molecular, com especial foco no estudo da estrutura e função do material genético
Projeto Genoma e Proteoma
 Projeto Genoma e Proteoma Grupo 3: *Artur S. Nascimento *Bárbara S. Costa *Beatrice Barbosa *Tamyres S. E. Guimarães *Yara Cavalcante O que é genoma? O genoma é o conjunto de todo o material genético que
Projeto Genoma e Proteoma Grupo 3: *Artur S. Nascimento *Bárbara S. Costa *Beatrice Barbosa *Tamyres S. E. Guimarães *Yara Cavalcante O que é genoma? O genoma é o conjunto de todo o material genético que
RNA: transcrição e processamento
 Universidade Federal do Piauí Centro de Ciências Agrárias Programa de Pós-graduação em Genética e Melhoramento Núcleo de Estudos em Genética e Melhoramento Bases Moleculares da Hereditariedade RNA: transcrição
Universidade Federal do Piauí Centro de Ciências Agrárias Programa de Pós-graduação em Genética e Melhoramento Núcleo de Estudos em Genética e Melhoramento Bases Moleculares da Hereditariedade RNA: transcrição
BIOLOGIA - 1 o ANO MÓDULO 08 RIBOSSOMOS E SÍNTESE PROTEICA
 BIOLOGIA - 1 o ANO MÓDULO 08 RIBOSSOMOS E SÍNTESE PROTEICA Fixação 1) (UNICAMP) Considere um fragmento de DNA com a seguinte sequência de bases: GTA GCC TAG E responda: a) Qual será a sequência
BIOLOGIA - 1 o ANO MÓDULO 08 RIBOSSOMOS E SÍNTESE PROTEICA Fixação 1) (UNICAMP) Considere um fragmento de DNA com a seguinte sequência de bases: GTA GCC TAG E responda: a) Qual será a sequência
O processo da Expressão Gênica
 Coordenadoria de Educação e Difusão de Ciências Rua 9 de Julho, 1205 - São Carlos - São Paulo e-mail: cbme@if.sc.usp.br- Telefone: (16) 3373-9159 http://cbme.ifsc.usp.br http://cbme.usp.br O processo da
Coordenadoria de Educação e Difusão de Ciências Rua 9 de Julho, 1205 - São Carlos - São Paulo e-mail: cbme@if.sc.usp.br- Telefone: (16) 3373-9159 http://cbme.ifsc.usp.br http://cbme.usp.br O processo da
ESTRUTURA DAS PROTEÍNAS
 ESTRUTURA DAS PROTEÍNAS Aminoácidos ligam-se por ligações peptídicas = reação de condensação entre: OH do grupo carboxila de um aminoácido H do grupo amina do outro aminoácido ( liberação de uma molécula
ESTRUTURA DAS PROTEÍNAS Aminoácidos ligam-se por ligações peptídicas = reação de condensação entre: OH do grupo carboxila de um aminoácido H do grupo amina do outro aminoácido ( liberação de uma molécula
DNA, RNA E INFORMAÇÃO
 DNA, RNA E INFORMAÇÃO OS ÁCIDOS NUCLEICOS Embora descobertos em 1869, por Miescher, no pus das bandagens de ferimentos, o papel dos ácidos nucleicos na hereditariedade e no controle da atividade celular
DNA, RNA E INFORMAÇÃO OS ÁCIDOS NUCLEICOS Embora descobertos em 1869, por Miescher, no pus das bandagens de ferimentos, o papel dos ácidos nucleicos na hereditariedade e no controle da atividade celular
BIOTECNOLOGIA. 2. Conceito de clonagem molecular
 BIOTECNOLOGIA 1. Introdução Até a década de 70, o DNA era o componente celular mais difícil de ser analisado. Sua seqüência de nucleotídeos de enorme tamanho e monotonia química era geralmente analisada
BIOTECNOLOGIA 1. Introdução Até a década de 70, o DNA era o componente celular mais difícil de ser analisado. Sua seqüência de nucleotídeos de enorme tamanho e monotonia química era geralmente analisada
Programa de Pós-Graduação Stricto Sensu em Biologia Computacional e Sistemas. Seleção de Mestrado 2012-B
 Programa de Pós-Graduação Stricto Sensu em Biologia Computacional e Sistemas Seleção de Mestrado 2012-B INSTRUÇÕES (LEIA ATENTAMENTE ANTES DE PREENCHER A PROVA): a. Identifique sua prova unicamente com
Programa de Pós-Graduação Stricto Sensu em Biologia Computacional e Sistemas Seleção de Mestrado 2012-B INSTRUÇÕES (LEIA ATENTAMENTE ANTES DE PREENCHER A PROVA): a. Identifique sua prova unicamente com
CÓDIGO GENÉTICO E SÍNTESE PROTEICA
 CÓDIGO GENÉTICO E SÍNTESE PROTEICA Juliana Mara Stormovski de Andrade As proteínas são as moléculas mais abundantes e funcionalmente diversas nos sistema biológicos. Praticamente todos os processos vitais
CÓDIGO GENÉTICO E SÍNTESE PROTEICA Juliana Mara Stormovski de Andrade As proteínas são as moléculas mais abundantes e funcionalmente diversas nos sistema biológicos. Praticamente todos os processos vitais
Dra. Kátia R. P. de Araújo Sgrillo. Sgrillo.ita@ftc.br
 Dra. Kátia R. P. de Araújo Sgrillo Sgrillo.ita@ftc.br São macromoléculas gigantescas, com massa molecular maior que 100 milhões. Os ácidos nucléicos foram isolados pela primeira vez a partir do núcleo
Dra. Kátia R. P. de Araújo Sgrillo Sgrillo.ita@ftc.br São macromoléculas gigantescas, com massa molecular maior que 100 milhões. Os ácidos nucléicos foram isolados pela primeira vez a partir do núcleo
Aula 4 Estrutura do RNA
 Biologia Molecular Básica Módulo I Básico Aula 4 Estrutura do RNA O RNA é uma molécula intermediária na síntese de proteínas. Ela faz a intermediação entre o DNA e as proteínas. As principais diferenças
Biologia Molecular Básica Módulo I Básico Aula 4 Estrutura do RNA O RNA é uma molécula intermediária na síntese de proteínas. Ela faz a intermediação entre o DNA e as proteínas. As principais diferenças
48 Como produzimos a insulina?
 A U A UL LA Como produzimos a insulina? Na aula passada você estudou a importância da insulina no nosso organismo. Dá para imaginar o que aconteceria conosco se não fabricássemos esse hormônio ou se o
A U A UL LA Como produzimos a insulina? Na aula passada você estudou a importância da insulina no nosso organismo. Dá para imaginar o que aconteceria conosco se não fabricássemos esse hormônio ou se o
Mitocôndrias e Cloroplastos
 Universidade Federal de Sergipe Centro de Ciências Biológicas e da Saúde Departamento de Morfologia Biologia Celular Mitocôndrias e Cloroplastos Características gerais de mitocôndrias e cloroplastos Mitocôndrias
Universidade Federal de Sergipe Centro de Ciências Biológicas e da Saúde Departamento de Morfologia Biologia Celular Mitocôndrias e Cloroplastos Características gerais de mitocôndrias e cloroplastos Mitocôndrias
COLÉGIO PEDRO II CAMPUS TIJUCA II. DEPARTAMENTO DE BIOLOGIA E CIÊNCIAS COORD.: PROFa. CRISTIANA LIMONGI
 COLÉGIO PEDRO II CAMPUS TIJUCA II DEPARTAMENTO DE BIOLOGIA E CIÊNCIAS COORD.: PROFa. CRISTIANA LIMONGI 1º & 2º TURNOS 3ª SÉRIE / ENSINO MÉDIO REGULAR & INTEGRADO ANO LETIVO 2015 PROFESSORES: FRED & PEDRO
COLÉGIO PEDRO II CAMPUS TIJUCA II DEPARTAMENTO DE BIOLOGIA E CIÊNCIAS COORD.: PROFa. CRISTIANA LIMONGI 1º & 2º TURNOS 3ª SÉRIE / ENSINO MÉDIO REGULAR & INTEGRADO ANO LETIVO 2015 PROFESSORES: FRED & PEDRO
GABARITO BIOLOGIA REVISÃO 01 3 ANO A/B ENSINO MÉDIO
 GABARITO BIOLOGIA REVISÃO 01 3 ANO A/B ENSINO MÉDIO Resolução: 01. B 02. E 03. No alantóide da ave há uma rede de capilares sangüíneos onde ocorre a respiração. O principal excreta nitrogenado da ave é
GABARITO BIOLOGIA REVISÃO 01 3 ANO A/B ENSINO MÉDIO Resolução: 01. B 02. E 03. No alantóide da ave há uma rede de capilares sangüíneos onde ocorre a respiração. O principal excreta nitrogenado da ave é
Painéis Do Organismo ao Genoma
 Painéis Do Organismo ao Genoma A série de 5 painéis do organismo ao genoma tem por objetivo mostrar que os organismos vivos são formados por células que funcionam de acordo com instruções contidas no DNA,
Painéis Do Organismo ao Genoma A série de 5 painéis do organismo ao genoma tem por objetivo mostrar que os organismos vivos são formados por células que funcionam de acordo com instruções contidas no DNA,
TRANSCRICAO E PROCESSAMENTO DE RNA
 TRANSCRICAO E PROCESSAMENTO DE RNA Número de genes para RNA RNA ribossômico - rrna Os rrnas correspondem a 85 % do RNA total da célula, e são encontrados nos ribossomos (local onde ocorre a síntese proteíca).
TRANSCRICAO E PROCESSAMENTO DE RNA Número de genes para RNA RNA ribossômico - rrna Os rrnas correspondem a 85 % do RNA total da célula, e são encontrados nos ribossomos (local onde ocorre a síntese proteíca).
CONTROLE DO METABOLISMO GENES
 CONTROLE DO METABOLISMO GENES 10/06/15 1º ANO - BIOLOGIA 1 ESTRUTURA DO GENE Segmentos (pedaços) da molécula de DNA, o constituinte dos nossos cromossomos, onde estão inscritas receitas (códigos genéticos)
CONTROLE DO METABOLISMO GENES 10/06/15 1º ANO - BIOLOGIA 1 ESTRUTURA DO GENE Segmentos (pedaços) da molécula de DNA, o constituinte dos nossos cromossomos, onde estão inscritas receitas (códigos genéticos)
BIOTECNOLOGIA E ENGENHARIA GENÉTICA. Profa. Maria Paula
 BIOTECNOLOGIA E ENGENHARIA GENÉTICA Profa. Maria Paula FERRAMENTAS Enzimas: de restrição, DNA-ligase, DNA-polimerase, transcriptase Vetores: plasmídeos, vírus 1) PGH O número de genes é muito menor do
BIOTECNOLOGIA E ENGENHARIA GENÉTICA Profa. Maria Paula FERRAMENTAS Enzimas: de restrição, DNA-ligase, DNA-polimerase, transcriptase Vetores: plasmídeos, vírus 1) PGH O número de genes é muito menor do
BIOLOGIA EXERCÍCIOS. Anabolismo Nuclear
 Anabolismo Nuclear EXERCÍCIOS 1. mesmo responsável pela decodificação do genoma humano em 2001, o presidente dos EUA, Barack Obama, pediu a seus conselheiros especializados em biotecnologia para analisarem
Anabolismo Nuclear EXERCÍCIOS 1. mesmo responsável pela decodificação do genoma humano em 2001, o presidente dos EUA, Barack Obama, pediu a seus conselheiros especializados em biotecnologia para analisarem
DNA A molécula da vida. Prof. Biel Série: 9º ano
 DNA A molécula da vida Prof. Biel Série: 9º ano DNA FINGER-PRINTING A expressão DNA "Finger-Print" (ou Impressões Genéticas) designa uma técnica de separação de segmentos de DNA que permite a identificação
DNA A molécula da vida Prof. Biel Série: 9º ano DNA FINGER-PRINTING A expressão DNA "Finger-Print" (ou Impressões Genéticas) designa uma técnica de separação de segmentos de DNA que permite a identificação
EXAME DE BIOLOGIA Prova de Acesso - Maiores 23 Anos (21 de Abril de 2009)
 INSTITUTO POLITÉCNICO DE BEJA EXAME DE BIOLOGIA Prova de Acesso - Maiores 23 Anos (21 de Abril de 2009) Nome do Candidato Classificação Leia as seguintes informações com atenção. 1. O exame é constituído
INSTITUTO POLITÉCNICO DE BEJA EXAME DE BIOLOGIA Prova de Acesso - Maiores 23 Anos (21 de Abril de 2009) Nome do Candidato Classificação Leia as seguintes informações com atenção. 1. O exame é constituído
COLÉGIO XIX DE MARÇO excelência em educação
 OLÉIO XIX DE MRÇO excelência em educação 1ª PROV DE REPERÇÃO DE BIOLOI luno: Nº Série: 2º Turma: Data: Nota: Professor: Regina Volpato Valor da Prova: 40 pontos Orientações gerais: 1) Número de questões
OLÉIO XIX DE MRÇO excelência em educação 1ª PROV DE REPERÇÃO DE BIOLOI luno: Nº Série: 2º Turma: Data: Nota: Professor: Regina Volpato Valor da Prova: 40 pontos Orientações gerais: 1) Número de questões
UNIVERSIDADE FEDERAL DE ALAGOAS INSTITUTO DE CIÊNCIAS BIOLÓGICAS E DA SAÚDE SETOR DE BIOLOGIA CELULAR E MOLECULAR
 UNIVERSIDADE FEDERAL DE ALAGOAS INSTITUTO DE CIÊNCIAS BIOLÓGICAS E DA SAÚDE SETOR DE BIOLOGIA CELULAR E MOLECULAR DISCIPLINA: BIOLOGIA CELULAR E MOLECULAR ESTUDO DIRIGIDO FLUXO DA INFORMAÇÃO GÊNICA págs:
UNIVERSIDADE FEDERAL DE ALAGOAS INSTITUTO DE CIÊNCIAS BIOLÓGICAS E DA SAÚDE SETOR DE BIOLOGIA CELULAR E MOLECULAR DISCIPLINA: BIOLOGIA CELULAR E MOLECULAR ESTUDO DIRIGIDO FLUXO DA INFORMAÇÃO GÊNICA págs:
TRANSCRIÇÃO DO DNA: Tipos de RNA
 TRANSCRIÇÃO DO DNA: Síntese do mrna Gene (Unidades transcricionais) Tipos de RNA Tipos de RNA polimerase Tipos de RNA polimerase DNA dependente Transcrição em Procariotos Transcrição em Eucariotos Mecanismos
TRANSCRIÇÃO DO DNA: Síntese do mrna Gene (Unidades transcricionais) Tipos de RNA Tipos de RNA polimerase Tipos de RNA polimerase DNA dependente Transcrição em Procariotos Transcrição em Eucariotos Mecanismos
Composição química celular
 Natália Paludetto Composição química celular Proteínas Enzimas Ácidos nucléicos Proteínas Substâncias sólidas; Componente orgânico mais abundante da célula. Podem fornecer energia quando oxidadas, mas
Natália Paludetto Composição química celular Proteínas Enzimas Ácidos nucléicos Proteínas Substâncias sólidas; Componente orgânico mais abundante da célula. Podem fornecer energia quando oxidadas, mas
NÚCLEO e DIVISÃO CELULAR
 NÚCLEO e DIVISÃO CELULAR CÉLULA EUCARIONTE Cláudia Minazaki NÚCLEO Único; Normalmente: central Formato: acompanha a forma da célula Tamanho: varia com o funcionamento da célula Ciclo de vida da célula
NÚCLEO e DIVISÃO CELULAR CÉLULA EUCARIONTE Cláudia Minazaki NÚCLEO Único; Normalmente: central Formato: acompanha a forma da célula Tamanho: varia com o funcionamento da célula Ciclo de vida da célula
Profa Estela Rossetto
 Profa Estela Rossetto Síntese de Proteínas: Um trabalho em grupo dos RNA! ATP RNAt RNAm enzimas RNAr aminoácidos Ribossomo: Organela onde ocorre a síntese de proteínas. Organela não delimitada por membrana,
Profa Estela Rossetto Síntese de Proteínas: Um trabalho em grupo dos RNA! ATP RNAt RNAm enzimas RNAr aminoácidos Ribossomo: Organela onde ocorre a síntese de proteínas. Organela não delimitada por membrana,
UNIVERSIDADE FEDERAL DE SANTA CATARINA GRADUAÇÃO EM SISTEMAS DE INFORMAÇÃO DEPARTAMENTO DE INFORMÁTICA E ESTATÍSTICA DATA MINING EM VÍDEOS
 UNIVERSIDADE FEDERAL DE SANTA CATARINA GRADUAÇÃO EM SISTEMAS DE INFORMAÇÃO DEPARTAMENTO DE INFORMÁTICA E ESTATÍSTICA DATA MINING EM VÍDEOS VINICIUS DA SILVEIRA SEGALIN FLORIANÓPOLIS OUTUBRO/2013 Sumário
UNIVERSIDADE FEDERAL DE SANTA CATARINA GRADUAÇÃO EM SISTEMAS DE INFORMAÇÃO DEPARTAMENTO DE INFORMÁTICA E ESTATÍSTICA DATA MINING EM VÍDEOS VINICIUS DA SILVEIRA SEGALIN FLORIANÓPOLIS OUTUBRO/2013 Sumário
Introdução à genética quantitativa usando os recursos do R
 Introdução à genética quantitativa usando os recursos do R Marisa R. Cantarino 1 Julia M. P. Soler (orientadora) 2 1 Introdução Um dos principais desafios da pesquisa genética atualmente é estabelecer
Introdução à genética quantitativa usando os recursos do R Marisa R. Cantarino 1 Julia M. P. Soler (orientadora) 2 1 Introdução Um dos principais desafios da pesquisa genética atualmente é estabelecer
Resposta: Interbits SuperPro Web
 1. (Fuvest 2012) Uma mutação, responsável por uma doença sanguínea, foi identificada numa família. Abaixo estão representadas sequências de bases nitrogenadas, normal e mutante; nelas estão destacados
1. (Fuvest 2012) Uma mutação, responsável por uma doença sanguínea, foi identificada numa família. Abaixo estão representadas sequências de bases nitrogenadas, normal e mutante; nelas estão destacados
GENÉTICA VII APLICAÇÕES DO CONHECIMENTO GENÉTICO
 GENÉTICA VII APLICAÇÕES DO CONHECIMENTO GENÉTICO Prof. Jose Amaral/2012/2013 Metabolismo de controle O metabolismo é controlado pelos ácidos nucléicos, compostos que coordenam uma série de reações em que
GENÉTICA VII APLICAÇÕES DO CONHECIMENTO GENÉTICO Prof. Jose Amaral/2012/2013 Metabolismo de controle O metabolismo é controlado pelos ácidos nucléicos, compostos que coordenam uma série de reações em que
Aula: 16 Temática: Estrutura dos aminoácidos e proteínas parte I. Iremos iniciar o estudo da estrutura dos aminoácidos e proteínas.
 Aula: 16 Temática: Estrutura dos aminoácidos e proteínas parte I Acompanhe! Iremos iniciar o estudo da estrutura dos aminoácidos e proteínas. Introdução: A proteína é o composto orgânico mais abundante
Aula: 16 Temática: Estrutura dos aminoácidos e proteínas parte I Acompanhe! Iremos iniciar o estudo da estrutura dos aminoácidos e proteínas. Introdução: A proteína é o composto orgânico mais abundante
A descoberta da célula
 A descoberta da célula O que são células? As células são a unidade fundamental da vida CITOLOGIA A área da Biologia que estuda a célula, no que diz respeito à sua estrutura e funcionamento. Kytos (célula)
A descoberta da célula O que são células? As células são a unidade fundamental da vida CITOLOGIA A área da Biologia que estuda a célula, no que diz respeito à sua estrutura e funcionamento. Kytos (célula)
UFABC Bacharelado em Ciência & Tecnologia
 UFABC Bacharelado em Ciência & Tecnologia Transformações Bioquímicas (BC0308) Prof Luciano Puzer http://professor.ufabc.edu.br/~luciano.puzer/ Propriedades, funções e transformações de aminoácidos e proteínas
UFABC Bacharelado em Ciência & Tecnologia Transformações Bioquímicas (BC0308) Prof Luciano Puzer http://professor.ufabc.edu.br/~luciano.puzer/ Propriedades, funções e transformações de aminoácidos e proteínas
Complemento II Noções Introdutória em Redes Neurais
 Complemento II Noções Introdutória em Redes Neurais Esse documento é parte integrante do material fornecido pela WEB para a 2ª edição do livro Data Mining: Conceitos, técnicas, algoritmos, orientações
Complemento II Noções Introdutória em Redes Neurais Esse documento é parte integrante do material fornecido pela WEB para a 2ª edição do livro Data Mining: Conceitos, técnicas, algoritmos, orientações
Técnicas para Programação Inteira e Aplicações em Problemas de Roteamento de Veículos 14
 1 Introdução O termo "roteamento de veículos" está relacionado a um grande conjunto de problemas de fundamental importância para a área de logística de transportes, em especial no que diz respeito ao uso
1 Introdução O termo "roteamento de veículos" está relacionado a um grande conjunto de problemas de fundamental importância para a área de logística de transportes, em especial no que diz respeito ao uso
Como a vida funciona? O processo de Transcrição. Prof. Dr. Francisco Prosdocimi
 Como a vida funciona? O processo de Transcrição Prof. Dr. Francisco Prosdocimi Dogma central O fluxo da informação é unidirecional Refutação definitiva da herança dos caracteres adquiridos Transcrição
Como a vida funciona? O processo de Transcrição Prof. Dr. Francisco Prosdocimi Dogma central O fluxo da informação é unidirecional Refutação definitiva da herança dos caracteres adquiridos Transcrição
1.1. Organização de um Sistema Computacional
 1. INTRODUÇÃO 1.1. Organização de um Sistema Computacional Desde a antiguidade, o homem vem desenvolvendo dispositivos elétricoeletrônicos (hardware) que funciona com base em instruções e que são capazes
1. INTRODUÇÃO 1.1. Organização de um Sistema Computacional Desde a antiguidade, o homem vem desenvolvendo dispositivos elétricoeletrônicos (hardware) que funciona com base em instruções e que são capazes
Conteúdo. Disciplina: INF 02810 Engenharia de Software. Monalessa Perini Barcellos. Centro Tecnológico. Universidade Federal do Espírito Santo
 Universidade Federal do Espírito Santo Centro Tecnológico Departamento de Informática Disciplina: INF 02810 Prof.: (monalessa@inf.ufes.br) Conteúdo 1. Introdução 2. Processo de Software 3. Gerência de
Universidade Federal do Espírito Santo Centro Tecnológico Departamento de Informática Disciplina: INF 02810 Prof.: (monalessa@inf.ufes.br) Conteúdo 1. Introdução 2. Processo de Software 3. Gerência de
Algoritmos Genéticos
 UNIVERSIDADE PRESBITERIANA MACKENZIE Laboratório de Computação Natural LCoN I ESCOLA DE COMPUTAÇÃO NATURAL Algoritmos Genéticos Rafael Xavier e Willyan Abilhoa Outubro/2012 www.computacaonatural.com.br
UNIVERSIDADE PRESBITERIANA MACKENZIE Laboratório de Computação Natural LCoN I ESCOLA DE COMPUTAÇÃO NATURAL Algoritmos Genéticos Rafael Xavier e Willyan Abilhoa Outubro/2012 www.computacaonatural.com.br
Bioinformática. Licenciaturas em Biologia, Bioquímica, Biotecnologia, Ciências Biomédicas, Engenharia Biológica. João Varela jvarela@ualg.
 Bioinformática Licenciaturas em Biologia, Bioquímica, Biotecnologia, Ciências Biomédicas, Engenharia Biológica João Varela jvarela@ualg.pt Docentes Paulo Martel (alinhamentos, pesquisas de sequências em
Bioinformática Licenciaturas em Biologia, Bioquímica, Biotecnologia, Ciências Biomédicas, Engenharia Biológica João Varela jvarela@ualg.pt Docentes Paulo Martel (alinhamentos, pesquisas de sequências em
CÓDIGO GENÉTICO E SÍNTESE PROTEICA
 Terceirão Biologia 1 Professor João CÓDIGO GENÉTICO E SÍNTESE PROTEICA Dogma central da Biologia Descreve o fluxo unidirecional de informações, do DNA à síntese de proteínas. Duplicação/Replicação Síntese
Terceirão Biologia 1 Professor João CÓDIGO GENÉTICO E SÍNTESE PROTEICA Dogma central da Biologia Descreve o fluxo unidirecional de informações, do DNA à síntese de proteínas. Duplicação/Replicação Síntese
16/04/2015 ÁCIDOS NUCLEICOS DNA E RNA DNA E RNA DNA E RNA BREVE HISTÓRICO DA DESCOBERTA DO DNA BREVE HISTÓRICO DA DESCOBERTA DO DNA
 ÁCIDOS NUCLEICOS E RNA E RNA Plano de Aula -Componentes básicos de e RNA -Características estruturais e funcionais -Tipos de RNA Profª Dra. Juliana Schmidt Medicina 2014 E RNA BREVE HISTÓRICO DA DESCOBERTA
ÁCIDOS NUCLEICOS E RNA E RNA Plano de Aula -Componentes básicos de e RNA -Características estruturais e funcionais -Tipos de RNA Profª Dra. Juliana Schmidt Medicina 2014 E RNA BREVE HISTÓRICO DA DESCOBERTA
SÍNTESES NUCLEARES. O DNA éo suporte da informação genética. Parte 1 Replicação
 SÍNTESES NUCLEARES O DNA éo suporte da informação genética Parte 1 Replicação Estrutura do DNA Replicação do DNA Nucleótidos A informação genética das células é armazenada sob a forma de 2 moléculas similares:
SÍNTESES NUCLEARES O DNA éo suporte da informação genética Parte 1 Replicação Estrutura do DNA Replicação do DNA Nucleótidos A informação genética das células é armazenada sob a forma de 2 moléculas similares:
PRÓ-MATATEMÁTICA NA FORMAÇÃO DE PROFESSORES
 PRÓ-MATATEMÁTICA NA FORMAÇÃO DE PROFESSORES Regina Luzia Corio de Buriasco * UEL reginaburiasco@sercomtel.com.br Magna Natália Marin Pires* UEL magna@onda.com.br Márcia Cristina de Costa Trindade Cyrino*
PRÓ-MATATEMÁTICA NA FORMAÇÃO DE PROFESSORES Regina Luzia Corio de Buriasco * UEL reginaburiasco@sercomtel.com.br Magna Natália Marin Pires* UEL magna@onda.com.br Márcia Cristina de Costa Trindade Cyrino*
ÁCIDOS NUCLEICOS DNA - ÁCIDO DESOXIRRIBONUCLEICO RNA - ÁCIDO RIBONUCLEICO
 ÁCIDOS NUCLEICOS DNA - ÁCIDO DESOXIRRIBONUCLEICO RNA - ÁCIDO RIBONUCLEICO 1 Funções dos ácidos nucleicos Armazenar e expressar a informação genética Replicação Cópia da mensagem contida no DNA, que será
ÁCIDOS NUCLEICOS DNA - ÁCIDO DESOXIRRIBONUCLEICO RNA - ÁCIDO RIBONUCLEICO 1 Funções dos ácidos nucleicos Armazenar e expressar a informação genética Replicação Cópia da mensagem contida no DNA, que será
Química do material genético
 1 O NÚCLEO No núcleo estão os cromossomos, onde estão "armazenadas" as informações genéticas de cada espécie. Os seguintes componentes constituem o núcleo celular: Membrana Nuclear: também chamada de carioteca
1 O NÚCLEO No núcleo estão os cromossomos, onde estão "armazenadas" as informações genéticas de cada espécie. Os seguintes componentes constituem o núcleo celular: Membrana Nuclear: também chamada de carioteca
UNIDADE 4. Introdução à Metodologia de Desenvolvimento de Sistemas
 UNIDADE 4. Introdução à Metodologia de Desenvolvimento de Sistemas 4.1 Motivação Sistemas de Informação são usados em diversos níveis dentro de uma organização, apoiando a tomada de decisão; Precisam estar
UNIDADE 4. Introdução à Metodologia de Desenvolvimento de Sistemas 4.1 Motivação Sistemas de Informação são usados em diversos níveis dentro de uma organização, apoiando a tomada de decisão; Precisam estar
 DNA barcoding é um método que utiliza um trecho do DNA de cerca de 650 nucleotídeos como marcador para caracterizar espécies. Trata-se de uma sequência extremamente curta em relação à totalidade do genoma,
DNA barcoding é um método que utiliza um trecho do DNA de cerca de 650 nucleotídeos como marcador para caracterizar espécies. Trata-se de uma sequência extremamente curta em relação à totalidade do genoma,
Ácidos Nucleicos 22/12/2011. Funções do Material Genético. informação genética.
 Ácidos Nucleicos Profa. Dra. Juliana Garcia de Oliveira Disciplina: Biologia Celular e Molecular Turmas: Ciências Biológicas, enfermagem, nutrição e TO. Funções do Material Genético Mendel, 1865: genes
Ácidos Nucleicos Profa. Dra. Juliana Garcia de Oliveira Disciplina: Biologia Celular e Molecular Turmas: Ciências Biológicas, enfermagem, nutrição e TO. Funções do Material Genético Mendel, 1865: genes
Tradução Modificando o alfabeto molecular. Prof. Dr. Francisco Prosdocimi
 Tradução Modificando o alfabeto molecular Prof. Dr. Francisco Prosdocimi Tradução em eukarya e prokarya Eventos pós-transcricionais Processo de síntese de proteínas RNAm contém o código do gene RNAt é
Tradução Modificando o alfabeto molecular Prof. Dr. Francisco Prosdocimi Tradução em eukarya e prokarya Eventos pós-transcricionais Processo de síntese de proteínas RNAm contém o código do gene RNAt é
Do Corpo Humano ao DNA. Noções de Biologia Molecular. Nucleotídeos - DNA RNA. Dogma central. Prof a. Dr a. Mônica B.
 Do Corpo Humano ao DNA Noções de Biologia Molecular Prof a. Dr a. Mônica B. Melo FCM - SCSP - Estrutura dos ácidos nucléicos (DNA, RNA) - Replicação - Transcrição - Processamento - Tradução -Mutações -
Do Corpo Humano ao DNA Noções de Biologia Molecular Prof a. Dr a. Mônica B. Melo FCM - SCSP - Estrutura dos ácidos nucléicos (DNA, RNA) - Replicação - Transcrição - Processamento - Tradução -Mutações -
Organização do Material Genético nos Procariontes e Eucariontes
 Organização do Material Genético nos Procariontes e Eucariontes Organização do Material Genético nos Procariontes e Eucariontes Procariontes Eucariontes Localização Organização Forma Disperso no citoplasma
Organização do Material Genético nos Procariontes e Eucariontes Organização do Material Genético nos Procariontes e Eucariontes Procariontes Eucariontes Localização Organização Forma Disperso no citoplasma
Universidade de Brasília
 Universidade de Brasília Instituto de Ciências Exatas Departamento de Ciência da Computação Pipelines para transcritomas obtidos por sequenciadores de alto desempenho Paulo Antonio Alvarez Monografia apresentada
Universidade de Brasília Instituto de Ciências Exatas Departamento de Ciência da Computação Pipelines para transcritomas obtidos por sequenciadores de alto desempenho Paulo Antonio Alvarez Monografia apresentada
FACULDADE DE ENGENHARIA DE COMPUTAÇÃO. PROJETO FINAL I e II PLANO DE TRABALHO <NOME DO TRABALHO> <Nome do Aluno> <Nome do Orientador>
 FACULDADE DE ENGENHARIA DE COMPUTAÇÃO PROJETO FINAL I e II PLANO DE TRABALHO O Trabalho de Conclusão de Curso (TCC) a ser desenvolvido
FACULDADE DE ENGENHARIA DE COMPUTAÇÃO PROJETO FINAL I e II PLANO DE TRABALHO O Trabalho de Conclusão de Curso (TCC) a ser desenvolvido
Biologia Professor Vianna 1ª série / 1º trimestre
 Biologia Professor Vianna 1ª série / 1º trimestre Módulo 3 ÁCIDOS NUCLEICOS E CITOLOGIA 1 Os itens abaixo referem-se à estrutura, composição e função dos ácidos nucleicos. Estrutura: I) Dupla hélice; II)
Biologia Professor Vianna 1ª série / 1º trimestre Módulo 3 ÁCIDOS NUCLEICOS E CITOLOGIA 1 Os itens abaixo referem-se à estrutura, composição e função dos ácidos nucleicos. Estrutura: I) Dupla hélice; II)
A partícula viral infectante, chamada vírion, consiste de um ácido nucléico e de uma capa protéica externa (capsídeo). O conjunto do genoma mais o
 1 A partícula viral infectante, chamada vírion, consiste de um ácido nucléico e de uma capa protéica externa (capsídeo). O conjunto do genoma mais o capsídeo de um vírion é denominado de nucleocapsídeo.
1 A partícula viral infectante, chamada vírion, consiste de um ácido nucléico e de uma capa protéica externa (capsídeo). O conjunto do genoma mais o capsídeo de um vírion é denominado de nucleocapsídeo.
