TATIANA FERREIRA DE ALMEIDA. Análise da dispersão das populações nativas americanas: uma abordagem genético-fisiográfica
|
|
|
- João Vítor Campelo Domingos
- 7 Há anos
- Visualizações:
Transcrição
1 TATIANA FERREIRA DE ALMEIDA Análise da dispersão das populações nativas americanas: uma abordagem genético-fisiográfica Dispersion analysis of the native american populations: a geneticfisiographyc approach São Paulo 2011
2 TATIANA FERREIRA DE ALMEIDA Análise da dispersão das populações nativas americanas: uma abordagem genético-fisiográfica Dispersion analysis of the native american populations: a geneticfisiographyc approach Dissertação apresentada ao Departamento de Genética e Biologia Evolutiva do Instituto de Biociências da Universidade de São Paulo, para a obtenção de Título de Mestre em Ciências, na Área de Genética e Biologia Evolutiva Orientador: Walter Alves Neves São Paulo 2011
3 Ficha Catalográfica Almeida, Tatiana Ferreira Análise da dispersão das populações nativas americanas: uma abordagem genéticofisiográfica 374 Páginas Dissertação (Mestrado) - Instituto de Ciências da Universidade de São Paulo. Departamento de Genética e Biologia Evolutiva 1. Biogeografia 2. Nativos Americanos 3. Marcadores Moleculares I. Universidade de São Paulo. Instituto de Biociências, Departamento de Genética e Biologia Evolutiva Comissão Julgadora: Prof(a). Dr.(a). Prof(a). Dr.(a). Prof(a). Dr.(a). Orientador(a)
4 Dedicatória Dedico este trabalho a minha mãe que mais uma vez abdicou de tanto por um sonho meu
5 Epígrafe Take a bone from a dog: what remains? Alice considered. The bone wouldn t remain, of course, if I took it - and the dog wouldn t remain: it would come to bite me - and I m sure I shouldn t remain! Then you think nothing would remain? said the Red Queen. I think that s the answer. Wrong, as usual, said the Red Queen: the dog s temper would remain. Lewis Carrol, Through the looking glass
6 Agradecimentos Ao meu orientador, Prof. Dr. Walter Alves Neves, pela paciência, dedicação, perseverança e pelo exemplo científico; Ao meu co-orientador, Prof. Dr. Diogo Meyer, pelo tempo disposto e fundamental auxílio durante todo o processo de mestrado; Aos professores que me orientaram no percurso desta pesquisa; À Prof. Dr. Regina Célia Mingroni e Dr. Nadia de Moraes Barros, pelas orientações durante a qualificação que proporcionaram maior robustez à minha pesquisa; À FAPESP pelo fundamental apoio financeiro; À minha família, que mesmo longe tem papel fundamental em minhas conquistas; À minha amiga Mariana, que de perto viu todos os meus melhores e piores momentos e esteve sempre comigo; Aos meus amigos distantes, Clarissa, Raul e Cíntia que sempre me apoiaram e mesmo durante os momentos mais atribulados não me negaram nunca sua amizade; Ao meu pai Airton e à minha mãe Flávia, que sempre me apoiaram e me dedicaram um amor incondicional durante todas as minhas decisões; E ao meu marido, Danilo, grande amor da minha vida, sem ele nada disso teria sido possível.
7 1 INTRODUÇÃO! 3 Saída da África e teorias de dispersão continentais! 4 Genética e Biogeografia! 17 Uso de marcadores em estudos populacionais! 19 Introdução à Biogeografia e seus objetivos! 24 Biogeografia da América! 33 MATERIAL E MÉTODOS! 35 Banco de Dados! 35 Microssatélites! 36 DNA mitocondrial! 37 Localizações geográficas! 40 Banco de dados fisiográficos (biomas)! 44 Construção das matrizes! 54 Matrizes de distância genética! 57 Matrizes de distâncias geográficas/físicas! 62 Matrizes de distâncias fisiográficas! 66 Testes estatísticos! 68 Testes de correlação! 69 Teste de significância de Mantel! 70 Algoritmo de Monmonier! 71 RESULTADOS! 73 DNA mitocondrial! 73 Conjuntos menores! 73 América global! 84
8 América do Norte! 86 América do Sul! 87 Patagônia! 89 Região centro norte da América do Sul! 92 Microssatélites! 94 América global! 94 América do Norte! 96 América Central! 98 América do Sul! 99 Algoritmo de Monmonier! 102 DISCUSSÃO E CONCLUSÕES! 104 Validade dos Métodos! 104 Distância genética e geografia! 108 Distância genética e fisiográfica! 111 RESUMO! 114 ABSTRACT! 115 REFERÊNCIAS! 116 ANEXOS 141 2
9 3 1. INTRODUÇÃO A questão norteadora deste trabalho consiste na investigação da possível relação entre a estrutura genética das populações nativas americanas e os ambientes por elas ocupados, procurando compreender qual o papel das fronteiras ecológicas no assentamento dessas populações. Ela é formada a partir da suposição de que essas fronteiras, aqui representadas por biomas, podem ser consideradas como parte do caráter formador da estrutura genética dos nativos americanos. Se a hipótese de que as barreiras fisiográficas foram importantes para a estruturação da diversidade genética desses povos estiver correta, espera-se encontrar uma menor distância genética entre as populações que ocupam um mesmo ambiente, em comparação a populações assentadas em ambientes diferentes. No entanto, se a hipótese não for verdadeira espera-se encontrar uma correlação não diferente daquela representada pelo acaso entre as distâncias genética e fisiográfica. Este problema foi primeiramente levantado por James Mosimann e Paul Martin, em 1973 (Dixon 2001), quando propuseram um modelo de ocupação da América, no sentido norte-sul, em linhas arqueadas e paralelas (Figura 1), provocada pela busca de megamamíferos como recursos alimentares (Martin 1973), cunhado depois o termo blitzkrieg 1 para descrever a ocupação que provocou o extermínio da megafauna (O Rourke 2010). Em 1991, John M. Beaton propôs, em contraponto a teoria de Mosimann & Martin, que as populações nativas do Novo Mundo ocuparam o território americano obedecendo os limites de grandes biomas como costas, desertos, florestas e montanhas. Essa proposta teve grande influência para o modelo defendido por James Dixon (2001), segundo o qual, os ambientes foram ocupados por populações com ferramentas adaptadas a eles, sendo necessária uma readaptação da tecnologia e da subsistência para a ocupação de um novo ambiente (Figura 2). 1 Nome importando de um termo de guerra, que faz alusão ao método de ataques rápidos e brutais do exército alemão durante a segunda guerra mundial.
10 4 Este trabalho procura testar de forma quantitativa qual modelo explica melhor a diversidade genética nativa americana atual, lançando mão de ferramentas estatísticas e de modelos fisiográficos. Nesta introdução serão apresentados os pontos teóricos necessários para o desenvolvimento da hipótese e a interpretação dos resultados obtidos ao longo da pesquisa. A introdução foi dividida em dois grandes tópicos: o primeiro com descrições dos modelos de dispersão humana desde a origem da espécie e o segundo sobre as bases técnicas de biologia molecular e biogeografia, que subsidiarão teoricamente este trabalho Saída da África e teorias de dispersão continentais Este primeiro tópico foi dividido em duas partes: uma, sobre a saída da África e a ocupação de territórios adjacentes, até o momento de entrada na América e outra, sobre as teorias de dispersão mais relevantes dentro do próprio continente americano. O Homo sapiens é atualmente a espécie conhecida com mais ampla dispersão no mundo (Foley 1993), ocupando diversas regiões do planeta, abarcando, com isso, uma imensa gama de variações ecológicas. É uma espécie que apresenta uma boa performance em diversas situações ambientais e tem como marca a capacidade de adequar suas tecnologias conforme as exigências do meio em que vive (Klein 2005). Devido à distribuição cosmopolita dos humanos, uma questão fundamental a ser respondida diz respeito a como ocorreu essa distribuição, onde se originou e de que forma ocupou tantas regiões diversas. Existe atualmente um consenso sobre o local aproximado do surgimento do Homo sapiens. Este ponto de origem está localizado no continente africano, muito provavelmente no leste da África, na região de Omo Kibbish, na atual Etiópia, onde o
11 mais antigo crânio de humano anatomicamente moderno foi encontrado (McDougall et al. 2005). Outra forte evidência da saída do homem moderno a partir da África pode ser vista 5 Figura 1: Mapa adaptado do trabalho de Martin (1973) através da distribuição da diversidade genética humana, que aponta a África como o local de maior diversidade (Bowcock et al. 1994; Jin et al. 2000), além de ser o local onde se encontra o ramo mais basal (quando se trata de microssatélites e polimorfismos de inserção Alu) em análises de filogenia (Batzer et al. 1994; Bowcock et al. 1994). Também estudos de morfologia craniana (Betti et al. 2009) e de biologia molecular (Ramachandran et al. 2005) demonstraram também uma perda progressiva de diversidade genética à medida que as populações se afastam da África, podendo ser interpretada como consequência da migração a partir do continente africano, seguindo pela Europa, Oceania e Ásia até chegar às Américas (Handley et al. 2007). Para outras teorias de surgimento da espécie humana ver Wolpoff et al. (2000). A partir do surgimento do Homo sapiens anatomicamente moderno houve três tentativas bem caracterizadas de saída da África. A primeira, por volta de 115 mil anos
12 6 Figura 2: Mapa adaptado de Dixon (2001) antes do presente (AP), que alcançou o Levante, região onde hoje se localiza Israel (Stringer 2003), considerada naquela época apenas uma extensão do território africano, sem distinção da fauna e da flora africanas (Shea 2003). A segunda tentativa de saída da África, há aproximadamente 85 mil anos AP, teve sucesso em chegar, pela porção sul da Ásia e Europa, até a Austrália, porém sem alcançar as regiões de maiores latitudes ao norte da Ásia (Klein 2005). A terceira e maior expansão a partir da África, que chegou a latitudes extremas e povoou inclusive a América, deu-se por volta de 55 mil anos AP (Forster & Matsumura 2005), concomitante à explosão do Paleolítico Superior (Bar-Yosef 2002). Existem teorias de que foi uma resposta rápida às exigências do meio que permitiu ao ser humano
13 7 explorar ambientes nunca antes dominados por nenhum outro hominíneo, como o extremo nordeste da Ásia e a América (Hall 2004). Após a terceira saída da África, o Homo sapiens passou a ter representantes em todos os continentes do planeta, e para cada região ocupada existiu uma história particular de chegada e colonização (Pavlov et al. 2001; Salas et al. 2002; Atkinson et al. 2008). A distribuição genética das populações européias indica que desde a saída da África até tempos atuais houve cinco episódios distintos de colonização: o primeiro durante o Paleolítico Superior, de populações vindas da África; o segundo, representado por uma re-colonização de populações que se encontravam numa região de refúgio ao sul da Europa, após o último período de máximo glacial (LGM); o terceiro, constituído de populações provindas de áreas desérticas após o final do Younger Dryas 2 (marcando o final do Pleistoceno); o quarto, consistindo na dispersão de populações localizadas mais a leste; e o quinto período formado por pequenas migrações em todo o continente devido às redes econômicas a partir da Idade do Cobre (Soares et al. 2010). A Oceania, assim como o Oriente Médio e a Europa, forma um dos primeiros pontos de estabelecimento das populações humanas após a saída da África. Na Austrália e na Nova Guiné existem sítios arqueológicos de aproximadamente 50 mil e 40 mil anos, respectivamente. Nessa época, esses territórios estavam unidos, devido à retração dos oceanos, como consequência do sequestro de água marinha nos pólos (Clark 2001), e sua junção foi denominada Sahul. Alguns cientistas acreditam que a ocupação da Australia e da Nova Guiné se deu de forma independente, outros acreditam que as populações que saíram da África se estabeleceram em Sahul, ocupando os dois lugares ao mesmo tempo. As ilhas mais distantes da Australia e da Nova Guiné foram 2 Mantido o termo Younger Dryas, por falta de uma tradução para o português que seja tão adequada quanto o original. Na tradução de Marcos Santarrita, da obra de Steven Mithen (2003), Depois do Gelo: uma história humana global a.c., o termo foi traduzido por Jovem Dryas.
14 8 posteriormente ocupadas, tendo um componente melanésico (do sudeste da Ásia) bastante importante. Ainda não se tem certeza de que rota foi seguida para se chegar à Oceania, mas existem fortes evidências de que a ocupação desta região se deu por uma rota ao sul da Ásia, entrando pela Índia (Kayser 2010). Neste mesmo sentido, após a saída da África, houve também a colonização da Ásia, a partir de sua porção sul, na região onde atualmente se encontra a Índia, o Paquistão, o Nepal, o Butão, Bangladesh, o Sri Lanka e Mianma. Ainda não está claro quantas migrações foram responsáveis pela estruturação das populações na região, mas acredita-se que existiram duas rotas principais a partir da África, uma vinda pelo norte, através do Vale do Nilo e outra pelo sul, passando pela Península da Somália, através do Mar Vermelho, pela costa da Índia, sendo esta via pelo sul a mais antiga (Majumder 2010). Após a ocupação do sul da Ásia, sua região leste foi naturalmente ocupada pelas populações humanas em decorrência dos deslocamentos posteriores. Acredita-se que foram dois grandes eventos de expansão: o primeiro, há mil anos pela rota sul, atravessando a Arábia Saudita e a costa da Índia e o segundo, entre mil anos atrás pelo norte do Himalaia. A ocupação do leste da Ásia ocorreu, conforme mostra grande parte dos resultados em biologia molecular, de sul para norte, até atingir altas latitudes no extremo nordeste (Stoneking & Delfin 2010). Em torno de 27 mil anos atrás começaram os primeiros movimentos para a ocupação da Sibéria. Esta data é baseada tanto nos achados de sítios arqueológicos na região do Rio Yana, na Sibéria (Pitulko et al. 2004), quanto no relógio molecular de haplótipos de DNA mitocondrial (Achilli et al. 2008; Fagundes et al. 2008a). Após ocupar o extremo nordeste da Ásia, o homem permaneceu na região por um determinado período antes da entrada na América (Waguespack 2007). Na época da ocupação humana, por volta de anos AP, a paisagem nesta região era bastante
15 9 diferente da que é hoje (Alfimov 2001). A retração dos oceanos, que teve por consequência a exposição de porções de terra atualmente submersas, além de Sahul, formou também a Beríngia, região entre o nordeste da Ásia e o noroeste da América, que cobria desde Kolyma, próximo ao Rio Lena, a oeste, até o território de Yukon, a leste (Guthrie 2001; Tamm et. al. 2007). Essa exposição de terra resultou em regiões de refúgio com um clima mais ameno para as populações de animais que ali viviam, formando um oásis de flora e fauna em meio a regiões permanentemente congeladas, viabilizando possibilidades de caça e de coleta aos humanos (Guthrie 2006). Porém, paradoxalmente, o ambiente frio, ao mesmo tempo em que expôs um caminho terrestre que ligava a Ásia e a América, também impediu a migração para dentro da América, com barreiras de gelo nas altas latitudes, obrigando assim que as populações migrantes permanecessem na Beríngia (Kitchen et al. 2008). Durante esta permanência na Beríngia, que ocorreu entre 30 a 20 mil anos atrás (Kitchen et al. 2008; Tamm et al. 2007), as populações se diferenciaram de seus ancestrais tanto por processos de deriva populacional, quanto por processos seletivos relacionados principalmente ao clima (Roseman & Weaver 2004). Os sítios arqueológicos mais antigos encontrados na América, fora da região da Beríngia, datam de aproximadamente C 3 anos atrás (Waguespack 2007), sendo a evidência mais antiga de humanos na América do Norte estabelecida pela presença de coprólitos de aproximadamente C anos atrás, no estado do Oregon nos EUA (Thomas et al. 2008). O mais antigo sítio arqueológico da América do Sul encontrado no Chile (Dillehay 2009), data de aproximadamente C AP (Lewin 1998). Adicionalmente, o esqueleto humano mais antigo, diretamente datado, encontrado na América é de aproximadamente C AP (Waters et al. 2007). Poucas são as controvérsias nesse sentido, porém, pesquisadores no Brasil acreditam que possa ter 3 Datações marcadas com 14 C denotam os anos pela datação da queda do isótopo de carbono 14, sem a correção devida à perda não contínua de carbono 14.
16 10 havido uma colonização muito anterior, marcada pela entrada das populações na América por volta de 100 mil anos atrás, estimativa essa, baseada na datação de uma pintura rupestre na cidade de São Raimundo Nonato no Piauí de 34 mil anos atrás (Guidon 2008). Uma vez discutidas as possíveis datas de entrada para os primeiros habitantes da América, outro ponto importante a ser esclarecido para a compreensão da ocupação do continente é o caminho por onde entraram. É senso comum que todas as populações nativas americanas entraram pelo norte, através do estreito de Bering (Powell & Neves 1999; Kitchen et al. 2008). Porém, existem hipóteses, que não podem ser totalmente refutadas, de rotas alternativas. Uma dessas hipóteses sugere que a entrada na América se deu por via marítima vinda do centro-leste da Ásia, tendo como apoio a presença de um haplogrupo mitocondrial presente nesta região da Ásia, na Austrália e na América, porém ausente no extremo nordeste da Ásia (Wilson 2008). Uma hipótese alternativa de rota, para a chegada às Américas, seria através da Polinésia. Os achados que sugerem esta rota são de parasitas humanos presentes em esqueletos antigos, que não seriam capazes de sobreviver à migração pela Sibéria devido às baixas temperaturas (Araujo et al. 2008), e também pela existência do sítio arqueológico em São Raimundo Nonato, citado anteriormente, no qual seria impossível chegar pelo norte do continente, que até AP estava coberto por gelo (Brigham-Grette 2001). Tendo em mente quando e por onde as populações que saíram da África, e que povoaram a Europa, a Ásia e a Oceania, chegaram ao continente americano é natural que surja a pergunta: em que condições essas populações entraram no novo continente? De acordo com os achados da genética, principalmente baseados em haplogrupos de DNA mitocondrial, chegou-se a pensar em diversos números de ondas migratórias para a entrada no Novo Mundo (Schurr 2004), sendo que atualmente o grande embate se dá
17 11 entre modelos que propõem uma única migração (Fagundes et al. 2008b) e outros que sugerem mais de um evento migratório (Hubbe et al e Perego et al. 2009). O cenário genético atual das populações nativas americanas é usado como base para prever como era este cenário no momento da entrada no Novo Mundo. Os primeiros estudos feitos em populações americanas, com o intuito de compreender os processos de migração, utilizaram marcadores clássicos, que são proteínas geralmente polimórficas e que permitem inferir genótipos facilmente (O Rourke 1992; Callegari-Jacques et al. 1994). Para O Rourke (1992), os marcadores clássicos de antígenos de células sanguíneas revelaram um padrão evidente de cline de norte para sul na América do Norte, um padrão menos forte para a América Central, e nenhuma correlação, ao contrário de Salzano e Callegari-Jacques (Salzano 1988), entre geografia e genética para as populações da América do Sul. O DNA mitocondrial para as populações nativas americanas é representado por cinco haplogrupos A, B, C, D e X (Merriwether 1995), sendo os autóctones achados nas populações nativas americanas: A2, B2, C1, D1 e X2a (Achilli et al. 2008; Fagundes et al. 2008a). Conforme descrito mais recentemente, os haplogrupos fundadores das populações nativas americanas são: B2, X2g, X2a, A2, A2a, A2b, C1b, C1c, C1d, C1d1, C4c, D1, D2a, D3 e D4h3a (Perego et al. 2010; Malhi et al. 2010). Todos esses haplogrupos são provenientes das linhagens ancestrais, N, M e R, que são, por sua vez, provindas da linhagem L3, africana. (Perego et al. 2010). Estudos de biologia molecular em esqueletos antigos evidenciaram a presença do haplogrupo M na América, que atualmente não está mais presente entre os nativos americanos (Malhi et al. 2007). Em termos de distribuição dos haplogrupos na América, o haplogrupo A diminui de frequência de norte para sul, enquanto C e D aumentam nessa direção (Schurr 2004). O haplogrupo B apresenta outro padrão de dispersão, estando presente na América do Sul,
18 12 Central e no sudoeste da América do Norte (Wilson 2008). Todos estes haplogrupos são encontrados em populações asiáticas (Fagundes et al. 2008). O cromossomo Y é representado na América pelos haplogrupos Q-M3, R1a1-M17, P-M45, F-M89 e CM130, sendo QM3 e P-M45 representantes da maioria dos haplogrupos nativos americanos. O haplogrupo QM3 está presente em todas as populações nativas americanas estudadas e apresenta um aumento de frequência de norte para sul (Schurr & Sherry 2004). O uso de microssatélites autossômicos traz algumas vantagens sobre as análises com cromossomo Y, DNA mitocondrial e marcadores clássicos. Os microssatélites são informativos sobre a herança materna e paterna simultaneamente, além de apresentar um padrão neutro de evolução e um alto grau de resolução de polimorfismos, já que apresenta a maior taxa de polimorfismos dentre os três marcadores citados acima (quando não se utilizam abordagens de sequenciamento). As análises com esses marcadores demonstram que as populações nativas americanas experimentaram sucessivos gargalos populacionais a partir do estreito de Bering até o sul da América (Wang et al. 2007). Estudos de inserções Alu demonstraram afinidades das populações das Américas com as populações asiáticas e colocaram os nativos americanos em uma posição intermediária ao sudeste e o nordeste asiático (Battilana et al. 2006). Novick e colaboradores (1998) encontraram uma menor variabilidade nos sítios de inserção Alu para as populações nativas americanas quando comparadas com o restante do mundo, sugerindo uma forte ação de gargalo populacional e deriva genética nas populações que entraram no continente (Novick et al. 1998). Tendo em vista este cenário geral de distribuição da diversidade genética nas populações nativas americanas atuais, pode-se pensar sobre como essa distribuição foi atingida. Sendo importante compreender em qual arranjo essas populações iniciaram o
19 13 processo de povoamento para depois entender como se deu a dispersão deste arranjo inicial e mais tarde por qual motivo as populações nativas americanas estão alocadas da forma como são vistas hoje. A explicação mais econômica para a ocupação das Américas é a de que todos os nativos americanos são originários de um único evento migratório que continha todos os haplogrupos encontrados entre os nativos atuais, tanto de DNA mitocondrial, quanto de cromossomo Y, a partir da Ásia (Bonatto & Salzano 1997; Novick et al. 1998; Fagundes et al. 2008a). Esta afirmação é baseada nos achados de tempo de dispersão comum entre os 5 haplogrupos de DNA mitocondrial (Fagundes et al. 2008b) e na presença de um mesmo alelo de microssatélite compartilhado por todas as populações nativas do Novo Mundo (Schroeder et al. 2007; Schroeder et al. 2009). Porém, o modelo de uma única migração não é, para alguns, a melhor explicação para os achados genéticos em nativos americanos, levando muitos pesquisadores a propor modelos alternativos (Ray et al. 2010; Perego et al. 2009; Volodko et al. 2008). Karafet e colaboradores (1999), estudando a variabilidade do cromossomo Y, propuseram um modelo de mais de uma migração paternal a partir da região do Lago Baikal. Estudos baseados em DNA mitocondrial também questionam a migração única confirmando a possibilidade de mais de uma migração. Perego e colaboradores (2009), num estudo de sequências de DNA mitocondrial, retomaram a idéia de duas rotas de entrada na América, uma mais antiga pela costa oeste, responsável pela presença de um haplogrupo raro (D4h3) nas populações que atualmente ocupam a costa do Pacífico, e outra mais recente, pelo interior da América do Norte, entre os lençóis de gelo Laurentidea e Cordileriana, devido à presença de outro haplogrupo raro X2a, exclusivamente nas populações desta região (Perego et al. 2009). Outro grupo de pesquisadores sugere ao menos duas migrações distintas para a formação do pool genético das populações de esquimós da Groenlândia (Gilbert et al. 2008). Ainda em 2008, Volodko e colaboradores
20 14 publicaram um artigo sugerindo duas fontes populacionais de origem Siberiana para a formação da estrutura genética dos nativos americanos (Volodko et al. 2008). Os dados de microssatélites autossômicos não contradizem a teoria de migração única, porém não testaram explicitamente as outras possibilidades (Wang et al. 2007). Mais recentemente, Ray e colaboradores, também baseados em dados de microssatélites e utilizando métodos de estatística Bayesiana, testaram três modelos de migração para as Américas: um, consistindo em um modelo com uma onda migratória; outro, com duas ondas migratórias; e um terceiro, sem um número fixo de ondas migratórias, mas que permitiria um fluxo gênico contínuo entre as populações asiáticas e americanas. Este último modelo explicou melhor os dados genéticos atuais (Ray et al. 2010). Além da biologia molecular, a paleoantropologia e a arqueologia também discutem como o Novo Mundo foi inicialmente ocupado e, com base em dados de morfologia craniana, propõem modelos que explicam esta entrada. Um modelo proposto em 1989 por Neves & Pucciarelli, com base no estudo de crânios antigos de nativos americanos, sugere a ocupação das Américas por dois grupos humanos diferentes, o que indicaria ao menos duas migrações para o continente. O modelo paleoantropológico foi depois reafirmado por outros pesquisadores (Gonzalez-Jose et al. 2001) e apesar de não se preocupar a fundo com o número exato de migrações, afirma que dois padrões distintos de morfologia humana povoaram as Américas (Neves & Hubbe 2005). Uma população com morfologia mais generalizada, crânio longo e faces baixas, habitou o continente antes da chegada dos povos com morfologia mongolóide, de crânio curto e faces altas (Neves et al. 2003). Os crânios de morfologia generalizada foram encontrados nas regiões de Lagoa Santa, no Brasil, Terra do Fogo, no Chile, Baixa Califórnia, no México, nos Estados Unidos e arredores de Bogotá, na Colômbia (Gonzalez-Jose et al. 2003; Neves et al. 2007a e Neves et al 2007b). Esses crânios datam de C a C anos atrás (Neves et al. 1999; Neves et al. 2007a) e
21 15 alguns acreditam que ainda possam existir remanescentes desse povo entre os nativos americanos atuais (Lévi-Strauss 2004; Atuí 2005). Recentemente Gonzáles-José et al. (2008), numa tentativa de combinar os conhecimentos gerados até hoje pela biologia molecular e pela paleoantropologia de forma multidisciplinar, chegaram à conclusão que a América recebeu o influxo de várias migrações de uma mesma população que sofreu processos microevolutivos e passou de uma morfologia mais generalizada para uma mongolóide mais especializada, na Sibéria. Apesar de não haver consenso sobre como os primeiros americanos vieram para o Novo Mundo, é fato indiscutível que chegaram às Américas e povoaram o continente com as mais diversas culturas e línguas. Para uniformizar as descrições e discussões serão usados os pontos de maior consenso sobre a entrada e as datações dos primeiros ocupantes do continente. Neste trabalho será adotada a rota de chegada pela Beríngia, por ser a mais provável, desconsiderando as demais possibilidades. As datas de até anos para a entrada no continente serão também adotadas pelos mesmos motivos. A visão mais atual sobre a dispersão dos nativos americanos mostra que uma rota costeira partindo do estreito de Bering até a América do Sul, talvez tenha sido a primeira forma de entrada e ocupação do continente (Waguespack 2007; Perego et al. 2009). Há indícios de que a costa oeste da América estava livre de gelo desde aproximadamente anos atrás (Hall 2004), o que proporcionou uma rota de dispersão relativamente rápida (Fix 2005), como indicam os achados em Monte Verde, no Chile, Quebrada Jaguay, no Peru, Arlington Springs, na California e On-Your-Knees Cave, no Alaska, datados de a C anos AP (Dillehay 1997; Waguespack 2007), além de simulações de percurso com microssatélites que demonstraram que entre uma rota costeira e outra pelo interior, a primeira se encaixa melhor na distribuição genética atual das populações nativas americanas (Wang et al. 2007). Contudo, essas evidências não
22 16 descartam a possibilidade de ocupação por rotas interioranas, através do corredor livre de gelo, com presença de sítios arqueológicos nesta região datados de aproximadamente C (Waguespack 2007) e recentemente retomada por Perego et al. (2009) com análises de alelos raros de DNA mitocondrial. Tanto a rota costeira quanto a rota interiorana assumem uma entrada pelo norte do continente com posterior ocupação até chegada ao extremo sul, padrão este confirmado por dados de microssatélites (Wang et al. 2007). Além disso, existe uma discussão sobre a distribuição das populações em um sentido longitudinal. Tarazona-Santos et al. (2001), com base em dados de cromossomo Y, observaram uma diversidade genética maior para os povos localizados a oeste quando comparados às populações a leste. De acordo com esses autores, esse padrão poderia ser explicado por um número de indivíduos fundadores maior, e maior fluxo gênico, nas populações a oeste de que a leste da América do Sul. Já para Pucciarelli et al. (2006 & 2008), os dados de morfologia craniana mostraram um padrão de diferenciação entre as populações localizadas no oeste e no leste, e indicam que, ao contrário do visto por Tarazona-Santos et al., essa diferença entre as duas localidades não é resultado de uma divergência na quantidade de variação presente em cada lado, mas sim que ocorreu padrões diferentes de fluxo gênico, de tamanhos de populações fundadoras e de intensidade de deriva genética entre as regiões leste e oeste do continente, assim como parcialmente sugerido por Fuselli et al. (2003). O cenário genético das populações nativas americanas atuais é bastante diferente daquele de populações em outros pontos do mundo e as explicações de como se deu a ocupação do território americano podem ser de extremo auxílio para entender essa singularidade. As populações nativas americanas são o grupo mais homogêneo do planeta, constituído de baixa heterozigosidade, com populações muito uniformes internamente e que por consequência aparentam uma grande distância genética entre si (Rosenberg et al. 2002).
23 17 Para explicar este caráter peculiar das populações nativas americanas Neel e Salzano propuseram na década de 1960 a teoria de fissão e fusão (Neel & Salzano, 1967). Esta teoria trabalha com a idéia de que as populações nativas se uniriam em situação de abundância, mas se separariam em pequenas populações endogâmicas em situações de escassez (Steward 1963; Hunley et al. 2008). O mesmo caráter também poderia ser explicado como uma consequência de deriva extrema, devido ao pequeno tamanho populacional, baixo fluxo gênico entre as populações fundadoras e pelos inúmeros gargalos populacionais sofridos durante a ocupação (Monsalve et al. 1999). No entanto, foi observado, ao menos para as populações nativas do nordeste da América do Norte, um grande fluxo gênico entre populações em regiões próximas, o que diminuiria o efeito da deriva (Shook & Smith 2008). Em resumo, as populações nativas americanas apresentam um padrão de variação gênica muito singular, que pode ser resultado de uma história demográfica particular em que uma grande região foi povoada de forma rápida por grupos de tamanho populacional pequeno e que permaneceram de certa forma endogâmicos, evitando assim a homogenização entre populações. A colonização da América está longe de ser completamente entendida e ainda apresenta muitas controvérsias quanto ao tempo de entrada, quais e quantas populações foram responsáveis pela formação do pool genético dos nativos americanos atuais e quais rotas foram utilizadas por essas populações para chegarem ao Novo Mundo (Schurr & Sherry 2004) Genética e Biogeografia Este segundo tópico foi dividido em três partes; a primeira, dedicada à descrição de métodos de biologia molecular utilizados para a compreensão dos processos evolutivos e
24 18 estudos populacionais. A sua importância é bastante clara ao se perceber que, sem conhecimentos básicos sobre alguns conceitos em genética, não se pode obter de forma correta a total compreensão do texto. Além disso, a falta de conhecimento sobre as técnicas aplicadas nos estudos de genética impossibilita uma correta leitura dos resultados, pois intrinsecamente a cada resultado obtido existem limitações inerentes aos marcadores e aos métodos empregados. A segunda parte enfatiza, através de um resumo histórico da Biogeografia, como as relações entre as espécies e os indivíduos com o ambiente estão diretamente relacionadas à teoria evolutiva e como as metodologias criadas para a avaliação dessas relações influenciam profundamente os resultados. Sua relevância para o presente trabalho ainda é bastante discreta, pois não foram usadas metodologias específicas, mas sim, análises preliminares. A terceira parte apresenta o cenário ambiental em que se encontrava a América desde o momento da colonização até o momento atual. Tal caracterização é importante do ponto de vista técnico e teórico, pois a entrada e a colonização do continente americano se deram durante um período de grande mudança ambiental, em uma região que inclusive atualmente abarca uma grande variedade de ambientes. A ocupação da savana em detrimento da floresta que estava diminuindo, a exploração de novos territórios com a saída da África, a relação com os outros animais ao passar de carniceiro a caçador são exemplos de como o ambiente é importante para a história da evolução humana e como assinaturas genéticas e fenotípicas podem ajudar a reconstruir a inserção das linhagens humanas nos cenários evolutivos passados (Martinón-Torres et al. 2007; Thorpe et al. 2007). Para tanto é fundamental conhecer as teorias da Biologia Molecular e da Ecologia que se ocupam em descrever e explicar quais mudanças ocorreram na espécie humana e por quais motivos elas aconteceram.
25 Uso de marcadores em estudos populacionais Nos primórdios dos estudos dos mecanismos de evolução, especialmente a partir da proposição dos mecanismos da Seleção Natural, descobriu-se que a grande variabilidade nos seres vivos contribuiu enormemente para a possibilidade de mudança e formação de novas espécies (Darwin 2004). Porém, não se tinha idéia, à época, como essas diferenças eram passadas aos descendentes e por quais meios elas permaneciam na nova população ou espécie (Zimmer 2001). Mesmo sem compreender corretamente como os mecanismos de herança funcionavam, Darwin foi capaz de descrever o fenômeno de forma tão completa que ele é usado até hoje praticamente da mesma forma em que foi proposto. A Seleção Natural descreve um dos meios pelos quais ocorrem as mudanças nas populações ao longo do tempo e tem como consequência a evolução biológica. A partir de uma enorme gama de variações dentro das populações locais, os indivíduos que, devido a forças exercidas pelo ambiente, conseguem deixar um maior número de descendentes, quando comparados a outros indivíduos da mesma população, deixam suas características mais frequentemente representadas na próxima geração e com o passar do tempo a população em questão se modifica (Darwin 2004). Atualmente, também se acredita que essas mudanças não ocorrem apenas de forma gradual, mas também por grandes saltos de mudanças drásticas (Gould & Eldredge 1993). Com os trabalhos de Gregor Mendel, redescobertos em torno de 1900, começouse a entender como as características são passadas geração a geração e o que as faziam se apresentar como eram (Griffiths 2006). Atualmente, os conceitos de genótipo e fenótipo ficaram mais complexos e para se entender a fundo o que significam é necessário ter em mente outros conceitos mais básicos como os de alelo e de locus. O alelo pode ser definido como uma variante de DNA (seja por sequência ou tamanho) presente em um locus cromossômico, sendo este último entendido pelo local
26 20 físico no cromossomo que a sequência de DNA em questão ocupa. No DNA de todos os organismos vivos, para cada locus existe um ou mais alelos correspondentes. Naqueles seres constituídos por um par de cromossomos o locus é o mesmo para os dois, podendo os alelos em um locus do mesmo indivíduo serem iguais (indivíduo classificado como homozigoto) ou distintos (indivíduo classificado como heterozigoto) (Griffiths 2006; Ridley 2006). Apesar de o número de alelos não exceder o número de loci em um indivíduo é possível que haja na população mais alelos do que loci naquela espécie (Griffiths 2006). Tendo em mente os conceitos de locus e alelo, os atuais conceitos de genótipo e fenótipo podem ser caracterizados como: genótipo sendo a composição específica de alelo(s) de uma célula ou indivíduo respeitando a divisão por loci e fenótipo como a manifestação externa dos genótipos, por meio de características físicas, comportamentais e bioquímicas de um indivíduo (Griffiths 2006; Ridley 2006). Primeiramente, o exame do genótipo era feito com base em fenótipos discretos, assim como os primeiros trabalhos de Mendel sobre a herança genética (Griffiths 2006). Contudo, essas características, além de serem difíceis de ocorrer, pois a maioria dos caracteres fenotípicos é determinada por interações entre mais de um gene (Ridley 2006), muitas vezes não apresentavam variações (polimorfismos) suficientes. Além disso, fenótipos podem ter sido fixados por forças seletivas e não indicar, portanto, ancestralidade comum. A força seletiva e a baixa quantidade de polimorfismos dificultam as análises de caracteres quando se tratam de populações. Para suprimir tais dificuldades técnicas foram desenvolvidas as análises de proteínas. A separação de enzimas em gel de polímero é uma delas. Após a separação, as diferentes posições das enzimas são reveladas pela reação com seu substrato. Presume-se também que essas alterações teriam caráter neutro, uma vez que a enzima continua com a sua função preservada, mesmo ocupando pontos diferentes no gel de polímero (Murphy 1996). Em meados de
27 , foram obtidos os primeiros sequenciamentos de proteínas, que são fruto da transcrição e da tradução direta da sequência de DNA (Avise 1994). Entretanto, ainda não tinha sido encontrada uma fonte confiável de informações sobre o material de transmissão genética, pois as proteínas que apresentam a mesma velocidade de corrida em gel, ou a mesma sequência, não garantem de forma alguma a certeza de ancestralidade e muito menos que os polimorfismos encontrados sejam evolutivamente neutros (Raybould et al. 1996). O desenvolvimento de técnicas que avaliassem diretamente a molécula de DNA foi o passo consequente dos esforços para se compreender as variações populacionais. Com a idealização da técnica de reação em cadeia de polimerase (polimerase chain reaction - PCR) tornou-se possível amplificar a porção do DNA desejada em quantidade suficiente para se fazer análises em gel de trama polímera ou até mesmo sequenciamento das bases nucleotídicas da fita de DNA. Apenas a porção desejada estaria presente na análise, sem contaminações com outras porções que poderiam levar a conclusões equivocadas (Palumbi 1996). Outro recurso desenvolvido para a observação direta do material genético é a análise de fragmentos obtidos por enzimas de restrição. Esta técnica consiste em cortar a fita de DNA em pontos específicos marcados por tais enzimas. O DNA digerido é colocado em um gel de trama polímera que permite movimentação dos fragmentos e sua separação por tamanho e/ou conformação. As variações nos padrões de fragmentos são chamados pela sigla RFLP (Restriction Fragments Lentgh Polymorphisms) 4. Apesar do grande avanço esta técnica apresenta, mesmo que em menor escala e com um olhar direto sobre o material genético, os mesmos problemas descritos anteriormente para as proteínas (Dowling 1996). 4 Traduzido diretamente do inglês os RFLP são polimorfirmos de comprimento gerados por enzimas de restrição.
28 22 Parte desses problemas pode ser contornada com a amplificação de regiões específicas do DNA, que são altamente polimórficas e sabidamente não estão sob seleção. Essas regiões denominadas de microssatélites e minissatélites são porções de repetição de nucleotídeos que são contabilizados pelo tamanho de cada fragmento (Li et al. 2002). Outras regiões não codificadoras presentes ao longo do DNA nuclear e mitocondrial também podem ser usadas com o mesmo objetivo. O sequenciamento direto do DNA, apesar de mais custoso e demorado em relação aos métodos discutidos anteriormente, é também bastante utilizado, por permitir uma alta precisão na contabilização de mutações e na quantidade de polimorfismos de qualquer região do DNA. Ele pode ser feito de diversas maneiras, sendo a técnica de sequenciamento de Sanger a mais utilizada atualmente por sequenciadores automáticos (Hillis 1996). Mesmo com os dados brutos sobre genótipos e fenótipos em mãos, é necessário ainda o uso de ferramentas estatísticas para que seja possível explicar como e porque a distribuição das variantes se encontram nas populações da forma como estão. O conhecimento da herança genética e dos mecanismos de determinação de características herdadas não é suficiente para inferir as consequências dos mecanismos de ação da evolução para as populações. Para tanto faz-se necessária uma disciplina que contabilize as heranças parentais no âmbito populacional através de gerações e permita a formulação de modelos microevolutivos, para que os efeitos da evolução possam ser detectados e previstos. Tal disciplina é conhecida como Genética de Populações (Templeton 2006), e tenta solucionar questões específicas de um grande problema geral, como as espécies e as populações deixaram de ser como eram no passado e se tornaram como são no presente (Gillespie 2004; Templeton 2006)? Os estudos de genética de populações mostraram a importância de outro mecanismo evolutivo, a deriva genética. Neste mecanismo, os genes que formam uma
29 23 geração são antes de tudo uma amostra aleatória dos genes que estavam na geração anterior. Assim, além do valor adaptativo da característica ou do indivíduo, os efeitos de amostragem para a formação da geração seguinte teriam também grande importância na fixação, na extinção e na alteração de frequências de características e na formação de novas espécies (Ridley 2006). Na década de 1930 e 1940, as teorias evolutivas foram unidas na síntese moderna que abarcava os eventos randômicos, como a deriva genética, e os eventos determinísticos como a seleção natural. A nova síntese moderna, além dessas forças descritas, acrescentou a resposta randômica, as limitações mutacionais e as epigenéticas (Mayr 1970). O primeiro modelo estatístico sobre estrutura populacional pelo ponto de vista evolutivo foi o modelo de Equilíbrio de Hardy-Weinberg (Ridley 2006). De acordo com ele, as frequências gênicas de uma população permanecem as mesmas ao longo das gerações e as frequências genotípicas devem estar nas proporções p 2, 2pq e q 2 (p 2 e q 2 para os genótipos homozigotos e 2pq para os genótipos heterozigotos, considerando uma população com indivíduos diplóides com apenas 2 alelos polimórficos (p e q) no locus em questão). Para que este equilíbrio seja mantido ao longo das gerações é preciso que não haja qualquer mecanismo de evolução atuando na população (Falconer 1996). A partir desse modelo é possível fazer previsões de como os mecanismos evolutivos afetarão as frequências gênicas e genotípicas das populações e quais frequências serão as esperadas ao longo das gerações (Beiguelman 1994). Algumas forças evolutivas, como a mutação, o endocruzamento, o fluxo gênico, a deriva genética e a seleção natural são bem compreendidas teoricamente e podem ser analisadas matematicamente a partir do modelo de Equilíbrio de Hardy-Weinberg (Gillespie 2004). Um ponto fundamental na aplicação das teorias evolutivas, ao se analisar a história e a estrutura de uma população, consiste em observar a porção mais adequada do material genético a ser investigado (Avise 1994). Dentro destes aspectos, os marcadores
30 24 mais adequados para revelar a história demográfica e a estrutura de uma população são os que apresentam padrões evolutivos neutros e grandes polimorfismos. Sem a atuação da Seleção Natural de forma primordial é possível obter uma boa estimativa da história demográfica da população, através da teoria de coalescência. Porém, quando a seleção está presente ela altera expectativas e frequências, podendo ser estudada para um gene específico, tornando difícil a análise da história demográfica populacional (Templeton 2006). Os marcadores sob seleção também podem revelar detalhes da história populacional, mas devem ser usados com cautela, uma vez que duas populações diferentes podem apresentar as mesmas frequências gênicas devido a uma convergência seletiva e não por compartilhar uma história recente comum (Gillespie 2004). Já o polimorfismo é necessário para se ter material para comparação. Um mesmo alelo presente em duas populações, de um marcador com poucos alelos, não é necessariamente um indicativo de história demográfica comum, mas apenas uma consequência de se ter poucas variantes. Em contrapartida, um excesso de polimorfismos pode tornar difícil a análise populacional devido à possibilidade de divisão excessiva da população, criando identidades familiares e não populacionais. Os estudos evolutivos dos diferentes taxa podem ser feitos, em princípio, com qualquer marcador genético, mas as conclusões retiradas dos resultados obtidos devem ser bastante cuidadosas, no sentido de não fazer formulações teóricas para as quais os marcadores em questão não são adequados Introdução à Biogeografia e seus objetivos A Biogeografia é o estudo da distribuição dos seres vivos no ambiente e de como a diversidade biológica varia na superfície da Terra, tanto no passado quanto no presente. É uma disciplina sintética que envolve outras áreas da ciência como a genética, a
31 25 sistemática, a paleontologia e a geologia. O desenvolvimento dessas disciplinas influencia diretamente o andamento das pesquisas em Biogeografia (Lomolino 2005). O conhecimento da distribuição dos animais e das plantas no planeta sempre esteve presente na história humana, pois é de grande importância conseguir identificar no ambiente quais os seres vivos que co-habitam e que podem ser usados como recursos (Lévi-Strauss 2004). Os primeiros estudos envolvendo os conceitos gerais da Biogeografia são de Aristóteles, no século IV AC. Na época da colonização das Américas pelos europeus, mesmo não sendo uma disciplina formal, a Biogeografia ganhou muita força devido à necessidade de se compreender os novos seres presentes nas terras recém descobertas (Lomolino 2005). Antes da publicação da Origem das Espécies em 1859 e da consolidação das teorias evolutivas, acreditava-se que os seres vivos eram imutáveis e muitas teorias foram elaboradas para explicar a distribuição deles no planeta (Nelson 1978). No século XVIII, Carolus Linnaeus publicou trabalhos em que descrevia o padrão de distribuição da diversidade biológica conhecida na época, adotando uma teoria de que os seres vivos, criados por Deus, eram dispersos a partir de um único ponto de origem (Linnaeus 2004; Lomolino 2005). Após Linnaeus, ainda no século XVIII, George-Louis Leclerc, conhecido como conde de Buffon, também havia observado que as espécies eram diferentes dependendo do lugar onde estivessem e que teriam condições próprias para suportar as pressões do meio (Leclerc 2004). Ele postulou uma lei, conhecida como o primeiro princípio da Biogeografia a lei de Buffon estabelecendo que regiões ambientais similares, porém isoladas, apresentam diferentes tipos de mamíferos e pássaros. O que foi demonstrado mais tarde ser aplicável para os outros seres vivos (Lomolino 2005). Christian Bergman e Joel Asaph Allen, no século XVIII, criaram postulados que relacionavam a morfologia dos corpos dos animais com o clima. Estes postulados
32 26 prediziam que animais de clima frio teriam corpos maiores com membros mais curtos se comparados a animais de clima quente (Lomolino 2005), o que foi confirmado pelos estudos em animais de climas quente e frio e pode ser percebido inclusive entre os hominíneos (Jacobs 1985). Em 1820, de Candole publicou o Géographie Botanique que definiu parte das ideias gerais da disciplina de Biogeografia. Em 1855 reconheceu, pela primeira vez, três tipos fisiológicos diferentes de plantas relacionados ao clima (de Candolle 2004). James Cowles Prichard e William Swainson, na primeira metade do século XIX, construíram um mapa de distribuição global de animais, separando-os em seis regiões concordantes com os continentes (Cox 2005). Entretanto, a biogeografia de plantas avançou mais rápido do que a de animais, pela facilidade de coleta e determinação da área de distribuição (Lomolino 2005). Até aquele momento, o estudo da distribuição dos organismos na Terra estava restrito à descrição das observações feitas. Mesmo com esta limitação, ainda no século XIX, emergiram novas teorias demonstrando que tanto os seres vivos quanto a Terra apresentavam mudanças no decorrer do tempo e estas poderiam ser a chave da explicação de como os organismos estavam distribuídos no planeta (Lomolino 2005). Com os trabalhos de Charles Lyell, Charles Darwin e Alfred Russel Wallace inaugurou-se um marco nos estudos de Biogeografia, porquanto se propuseram a explicar como as mudanças na superfície terrestre e nos seres vivos ocorrem através do tempo e do espaço (Darwin 2004; Wallace 2004). A partir de então, os trabalhos de Biogeografia concentraram-se em explicações de como e por que os seres vivos se distribuem no planeta da maneira como estão e passaram a criar modelos e testar hipóteses mais rigorosas. Wallace, considerado hoje o pai da Zoogeografia, desenvolveu um mapa de regiões biogeográficas da Terra bastante preciso e detalhado, baseado na distribuição dos
33 27 animais. Ele percebeu que existiam duas faunas distintas nas ilhas da Indonésia, sendo a do oeste mais semelhante à da Ásia e a do leste à australiana; tal linha divisória nas ilhas da Indonésia leva o seu nome (Wallace 2004; Cox 2005). Em 1879, Engler desenhou um mapa do mundo que mostrava as floras regionais, muito semelhante ao padrão atualmente aceito de distribuição (Cox 2005). Apesar do início tão remoto do estudo da distribuição dos seres vivos no planeta, a Biogeografia foi aceita como disciplina somente nos anos 1950 e 1960, pois alguns dos maiores desenvolvimentos para o estudo da Biogeografia aconteceram nos últimos 50 anos. Dentre eles, a aceitação da teoria das placas tectônicas, o desenvolvimento de novos métodos filogenéticos, novas abordagens científicas no estudo da ecologia e a investigação dos mecanismos que limitam as distribuições dos organismos vivos (Lomolino 2005). As primeiras ferramentas que a Biogeografia dispunha para estudar a diversidade entre os seres vivos eram a morfologia e a ecologia (Mayr 2004). No início dos anos 1960, iniciaram-se os trabalhos de cladística que, apesar de promover um grande avanço nos estudos de Biogeografia, por trazer à tona relações evolutivas mais claras e rigorosas, ainda apresentava o problema de trabalhar somente com características morfológicas (Cox 2005). Com os trabalhos de Dobzhansky, na primeira metade do século XX, as variações genéticas e fisiológicas puderam entrar como mais uma fonte de dados para os estudos sobre a distribuição geográfica das populações (Dobzhansky 1982). A importância de se utilizar dados genéticos para entender a distribuição dos seres vivos, principalmente quando envolve estudos de filogenia, consiste também no fato de que os efeitos de convergência nos genes são mais discretos e controláveis do que na morfologia, uma vez que a proporção de sequências idênticas por convergência é menor do que a proporção de estruturas morfológicas pelo mesmo motivo.
34 28 Inicialmente, as populações eram classificadas considerando-se sua distribuição geográfica e suas semelhanças morfológicas, dados que podem ter um grande viés por não acessar a real proximidade genética dessas populações. Com o aperfeiçoamento e a especialização da Genética de Populações, alguns problemas puderam ser sanados permitindo acessar, de fato, a distância genética entre as populações e separá-las com base na quantidade de fluxo gênico que ainda persistiam entre elas (Pritchard et al. 2000). Tendo em vista a diversidade de temas abarcados pela Biogeografia, é mais conveniente para fins didáticos separá-los em duas grandes áreas, a Biogeografia Ecológica e a Biogeografia Histórica (Wiens & Donoghue 2004). A Biogeografia Ecológica visa ao estudo da distribuição atual dos organismos, acessada por dados ecológicos, genéticos e morfológicos. É, por exemplo, mais utilizada em estudos de conservação. A Biogeografia Histórica procura entender como ocorreu a dispersão dos organismos no passado, por um longo período de tempo e quais suas relações evolutivas. Trabalha geralmente com áreas grandes e globais, com taxa acima do nível de espécie e utiliza larga escala de tempo que abarcam grandes mudanças climáticas dentro das regiões estudadas (Morrone 1995). A Paleontologia é fundamental para a Biogeografia Histórica (Wiens & Donoghue 2004). O estudo da Biogeografia está bastante avançado e existem muitas teorias que auxiliam a descobrir as relações evolutivas e ecológicas que ocorrem entre os taxa. Entender que os seres vivos se relacionam não só com seu ambiente, mas também com outros seres vivos foi de primordial importância para se construir teorias sobre a dispersão dos organismos. Um ponto de especial importância nessa abordagem são as barreiras de dispersão. A área de distribuição de uma espécie é cercada por regiões que ela não conseguiria ocupar e manter populações viáveis. Tais regiões devem ser vistas como barreiras que
35 29 precisam ser ultrapassadas para se poder colonizar outro ambiente. Sob este ponto de vista cada espécie torna-se prisioneira de sua própria história evolutiva (Cox 2005). A dispersão de uma espécie está relacionada à sua história evolutiva e à história do local em que se encontra. Os estudos biogeográficos preconizam que há três principais maneiras para a consolidação da distribuição de populações ao longo da geografia e do tempo. Na primeira, a extinção, populações que previamente ocupavam um habitat se extinguem e passam a não estar mais presentes no local. Isso afetaria não somente a distribuição desta população em particular, mas também das populações que com ela interagem. A segunda, chamada dispersão, explica como uma população pode ser encontrada em dois pontos distintos separados por uma barreira. Através da migração ao acaso de indivíduos da população original, que conseguiram ultrapassar a barreira e chegar ao outro ponto e estabelecer uma nova população. A terceira, denominada de vicariância, explica a presença de populações semelhantes em locais diferentes separados por barreiras que se instalaram posteriormente à dispersão da população por essas regiões (Cox 2005). Existem muitas razões para que populações transponham barreiras ecológicas. A principal é a restrição ao seu crescimento. Os habitats geralmente apresentam restrição de recursos, o que gera competição entre os indivíduos, conferindo vantagem àqueles que conseguem transpor uma barreira e estabelecerem-se em um novo local, com novos recursos para ele e seus descendentes (Pulliam 1991; Cox 2005). Os biogeógrafos acreditam que existem três caminhos para se transpor uma barreira. O primeiro, por meio de um corredor com características semelhantes aos dos habitats envolvidos, que une duas localizações ecologicamente semelhantes, cercados por diferentes condições. As espécies de cada extremo passam livremente pelo corredor e chegam ao outro extremo. O segundo, comparável a um filtro, em que haveria um corredor unindo habitats semelhantes em suas extremidades, mas o corredor em si
36 30 constituído de apenas algumas amostras da totalidade dos habitats presentes nas extremidades e apenas as espécies que estivessem adaptadas aos ambientes presentes no corredor poderiam ser encontradas nas duas extremidades. O terceiro, em que o habitat inicial está completamente envolto por ambientes diferentes, sem a presença de corredores com ambientes total ou parcialmente semelhantes, tornando a travessia dos ambientes a que não estão primariamente adaptados um evento primordialmente ao acaso (Cox 2005). Na maioria das vezes essas barreiras não são claras, como, por exemplo, quando um deserto ou montanhas separam duas regiões semelhantes, mas se apresentam como diferenças discretas na distribuição de espécies e de microclima, impondo um impedimento à presença dos indivíduos de uma população qualquer. Apesar de as barreiras interferirem na distribuição dos organismos, muitas vezes podem não existir limites bem definidos e seu papel como fator biogeográfico é difícil de ser detectado (Templeton 2006). Um exemplo desse caso se dá quando se observa os ambientes graduais, regiões em que ocorre uma transição progressiva de um ambiente para outro (Ridley 2006), tendo como consequência para os seres que ali vivem um estresse fisiológico à medida que a paisagem muda e com isso populações menos numerosas acompanham a mudança do habitat (Cox 2005), resultando em espécies que podem ser vistas geneticamente como cline 5 ou cline escalonada 6 (Templeton 2006). Além do padrão de dispersão dos indivíduos, é também de primordial importância para os estudos de Biogeografia a classificação correta desses indivíduos quanto à sua espécie, gênero e taxa mais abrangentes, pois com esta informação sabe-se 5 Cline é uma gradação geográfica na frequência de um gene ou no valor médio de um caracter (Ridley, 2006) 6 Cline escalonada é uma cline em que ocorre uma mudança brusca na frequência de um gene ou de uma característica (Ridley 2006)
37 31 corretamente quais as características dos indivíduos em questão e as inferências sobre sua relação com o habitat tornam-se mais precisas. É preciso ter em mente que muitas teorias e análises biogeográficas trabalham com taxa acima do nível de espécie, o que significa que não há fluxo gênico entre os diversos agrupamentos e que a teoria de base não precisaria, na maioria das vezes, considerar essa troca de informações gênicas nem precisaria lidar com o problema de quais seriam os grupos discretos (Katinas 2003). Esses problemas tornam-se evidentes quando se tenta aplicar as teorias da Biogeografia em populações de uma mesma espécie, sendo o primeiro deles reconhecer quais são os limites das populações, para depois abordar o quão discretas elas de fato são e quais as implicações disso para a dispersão da espécie. A primeira disciplina a estudar as relações biogeográficas dentro das espécies é a Filogeografia. Ela foi originalmente proposta por John C. Avise em 1987 e utiliza como marcador genético o DNA mitocondrial (Avise 1987; Katinas 2003), que permite fazer inferências sobre as relações genealógicas entre genes que, por sua vez, permitem inferências sobre a história evolutiva e demográfica da população em questão e também por apresentar a tendência de ser mais variável entre as diversas populações do que dentro delas (Katinas 2003). O estudo das populações pela Filogeografia parte de alguns princípios básicos que podem ser resumidos como: (a) na maioria das populações o DNA mitocondrial apresenta uma estrutura filogeográfica, (b) populações com pouca estrutura filogeográfica estavam em áreas onde o fluxo gênico não era impedido e (c) as populações que apresentam uma marcada estrutura filogeográfica devem ter tido uma longa história de barreira ao fluxo gênico entre elas (Avise 2000). Para que a análise de comparação entre a árvore filogenética e a geográfica seja considerada informativa é preciso que a estrutura inter-populacional se mantenha mesmo
38 32 depois que muitas técnicas de reamostragem sejam utilizadas, e que permaneça inalterada quando diferentes conjuntos de genes são analisados. A partir desses dados pode-se analisar se outras espécies apresentam os mesmos padrões de estrutura e se podem estar relacionados com as mesmas barreiras geográficas. Esta técnica é concentrada basicamente na comparação entre a árvore filogenética das populações e a distribuição geográfica de cada uma delas, com o intuito de se encontrar relações entre barreiras genéticas e geográficas (Avise 1987). Em 1992, Kare Bremer propôs a técnica de áreas ancestrais para definir quais as áreas de ocupação atual que são as mais prováveis de terem sido a origem de dispersão daquelas espécies. Essa técnica utiliza cladogramas como matéria prima que podem ser obtidos tanto com informações morfológicas, quanto com informações genéticas ou por ambas. Até aquele momento, as técnicas de estudo das populações requeriam, ou um conhecimento a priori de quais eram as populações a serem estudadas, ou separavam as populações em árvores filogenéticas que podem não indicar a real estruturação da população em questão (Hunley & Long 2005). Por conta disso, houve a necessidade de se procurar entender quais são as reais barreiras genéticas em uma espécie e quais são de fato as populações que compõem essa espécie (Barbujani & Belle 2006). Para tanto foi desenvolvida a disciplina denominada de Landscape Genetics 7, em que os principais objetivos são detectar as descontinuidades genéticas e correlacioná-las com o ambiente. Idealmente, na Landscape Genetics trabalha-se com os próprios indivíduos para que as barreiras genéticas possam ser melhor detectadas, mas também é possível trabalhar com as frequências gênicas das populações, pois a técnica é capaz de avaliar, mesmo nessas condições, se as divisões espaciais entre as populações são coincidentes com as barreiras genéticas (Manel 2003). 7 Mantida a expressão Landscape Genetics por não se ter conhecimento de uma tradução formal do nome do modelo em português.
39 33 No âmbito da Landscape Genetics existem várias técnicas, entre elas a interpolação, o Wombling, o algoritmo de Monmonier (Dupanloup et al. 2002) e o mapa sintético (Cavalli-Sforza et al. 1994). A principal diferença entre a Landscape Genetics e as técnicas empregadas pela Biogeografia é que com aquela é possível visualizar a distribuição genética no próprio mapa geográfico em que ela ocorre, sendo possível identificar padrões como cline, cline escalonada, isolamento por distância, barreiras ao fluxo gênico e metapopulações (Manel 2003). Com este apanhado geral histórico é possível perceber que a indagação de como as espécies estão distribuídas no espaço acompanha a questão de como as espécies surgiram. Entender a distribuição física dos seres vivos é um grande passo para a compreensão dos processos evolutivos que os influenciam e de sua história natural, sendo a linhagem humana parte dessa conformação e não uma exceção Biogeografia da América Considerando que a proposta deste trabalho compreende a investigação, através do uso de marcadores genéticos, de como se deu a dispersão dos nativos americanos nos últimos anos, não se pode ignorar o fato de que as populações humanas ocuparam a região em franca transformação ecológica, devido ao fim da era glacial. Nesse sentido, a descrição do cenário biogeográfico daquela época até o presente é de fundamental importância para o entendimento da ocupação do Novo Mundo pelo homem. O último período glacial durou de até anos atrás (Ahn & Brook 2008), com algumas interrupções por períodos mais quentes chamados de interstadiais, mas o período da colonização inicial da América ocorreu no final do Pleistoceno, durante a transição entre o período glacial e o primeiro período interglacial desde a saída do homem da África (Fagundes et al. 2008; Tamm et al. 2007).
40 34 As regiões de altas latitudes foram mais afetadas pelo frio e tiveram suas florestas, atualmente consideradas temperadas, substituídas por florestas boreais (Cox 2005). As florestas tropicais sofreram retração e foram permeadas por regiões mais áridas de savana, o que provavelmente ocasionou a retenção dos animais adaptados ao clima tropical úmido em zonas de refúgio, regiões de floresta tropical preservada e permeada por regiões mais secas (Whitmore 1987). É possível que no período em que os animais de floresta tropical estavam limitados a pequenas ilhas microclimáticas tenha ocorrido a grande diversificação da vida que é vista hoje nas florestas tropicais (Haffer 1969; Vanzolini 1992). Porém, esta não é uma visão consensual entre a comunidade científica (Lessa et al. 2003). Pouco antes do fim da era glacial há anos, as temperaturas começaram a subir de maneira não uniforme em diferentes regiões oceânicas e terrestres, o que provocou um pequeno intervalo de aquecimento, que foi seguido por um período de intenso resfriamento, principalmente nas porções do planeta banhadas pelo Oceano Atlântico entre aproximadamente e anos atrás (entre e C anos atrás) que foi chamado de Younger Dryas (Broecker 2006). Em torno de anos atrás, iniciou-se de fato o período interglacial e as temperaturas subiram consideravelmente, mudando completamente a paisagem do mundo vista pelos humanos (Cox 2005). Durante a colonização da porção nordeste da Ásia, atualmente conhecida como Sibéria, o clima glacial, ao provocar a retração das águas oceânicas, expôs porções de terra que ligaram o nordeste da Ásia ao noroeste da América por faixas contínuas de terra (Tamm et al. 2007). Há anos ocorreu o último período de máximo glacial, chegando a temperaturas nos trópicos de 2 a 3 ºC menores do que as atuais e de 4 a 5ºC nas regiões subtropicais (Shin 2003). Esse período coincide com dados moleculares e arqueológicos das primeiras tentativas dos homens de colonizarem a América. Naquele
41 35 momento os lençóis de gelo que cobriam a América do Norte não permitiam a entrada para o interior do continente, havia apenas uma porção sem gelo na costa do Pacífico, próximo ao Alaska e Ilhas Aleutas (Hetherington 2003). Ainda atualmente a América apresenta particularidades para a dispersão de populações humanas, porque, além de ter sido altamente modificada devido às alterações climáticas no momento em que as populações entravam no continente, é o continente que mais abarca variações latitudinais e por consequência apresenta uma vasta extensão e diversidade de climas e biomas. Compreende desde regiões bastante frias nas altas latitudes (norte e sul) até regiões de floresta tropical sempre úmidas, permeadas com regiões desérticas em áreas de baixas latitudes. Presentes também nos trópicos estão as regiões de intensa mudança climática durante as estações do ano, mudando radicalmente sua paisagem no período de um ano (Whitmore 1987, Kottek 2006). É neste cenário bastante dinâmico que a dispersão e a ocupação humana devem ser interpretadas. Assim como os genes nas populações estavam mudando, pelo processo de deriva genética e isolamento por distância, também é possível que estivessem acompanhando as mudanças ocorridas na paisagem enquanto estas populações adentravam o Novo Mundo, já que os indivíduos poderiam responder de forma variada a essas alterações deixando a marca de sua passagem em seus descendentes. 2. MATERIAL E MÉTODOS 2.1. Banco de Dados Para o cumprimento dos objetivos foram utilizados dois tipos de marcadores genéticos, os microssatélites e o DNA mitocondrial. O primeiro, apesar de sua amostragem estar restrita a poucas populações, apresenta maior precisão de informação genética, uma vez que os indivíduos apresentam vários loci compostos de diversos
42 36 polimorfismos. Já o segundo, apresenta a vantagem de ser representado por mais populações, mas com menor precisão, pois distingue-se por apenas um locus, com quatro variáveis. Além disso, existe uma diferença em termos de representação da história demográfica, uma vez que microssatélites são herdados de ambos os pais e podem sofrer recombinação. O DNA mitocondrial é transmitido apenas por herança materna, tendo um grau de recombinação entre moléculas desprezível (Oliveira et al. 2010). Os bancos aqui analisados provêm de estudos anteriores. Neste trabalho não foram obtidos originalmente dados de sequência ou de genotipagem para os marcadores descritos. Por se tratar de uma proposta inédita, não houve a necessidade de angariar mais informações ao banco de dados porque ainda não se sabe qual a resolução do método proposto. Também foram utilizadas informações de geoprocessamento sobre as localizações espaciais das populações, para que pudesse ser acessada a distância física entre elas e a sua inserção nos diferentes ecossistemas Microssatélites O banco de dados utilizado compreende 530 indivíduos, de 29 populações nativas americanas, sendo três da América do Norte, oito da América Central, dez do oeste da América do Sul e oito do leste. Todos os genótipos trabalhados foram obtidos diretamente do material suplementar Dataset S1 (doi: /journal.pgen sd001) de Wang et al. (2007). Conforme descrito por esses autores, os genótipos dos 678 loci, espalhados pelos 22 cromossomos autossômicos, de cada indivíduo de 24 populações (Chipewyan, Ojibwa, Cree, Zapotec, Mixtec, Mixe, Kachikel, Cabecar, Guaymi, Wayuu, Arhuaco, Kogi, Zenu, Embera, Waunana, Ingá, Ticuna Tarapaca, Ticuna Arara, Quéchua, Aymara, Huilliche, Ache, Guarani e Kaingang) foram obtidos pelo Mammalian Genotyping Service. Este serviço faz genotipagem pelo sistema SCAFUD (Scanning Fluorescence Detector), que
43 37 permite que os fragmentos de DNA sejam separados por eletroforese em gel e detectados por fluorescência em vez de filmes radioativos. A leitura dos alelos é feita com um software que analisa os dados diretamente do SCAFUD ( research.marshfieldclinic.org/genetics/technology/technology_index.asp). Os marcadores dos microssatélites foram retirados de Marshfield Screening Set 16 e 54. As cinco populações restantes (Pima, Maya, Piapoco, Karitiana e Suruí) tiveram seus genótipos caracterizados para os mesmos 678 loci descritos no banco de dados do Human Genome Diversity Panel (HGDP-CEPH). O banco de dados foi adaptado da plataforma Linux para a plataforma Windows sendo necessária a busca de um programa que trabalhasse na plataforma Windows e que permitisse um número infinito de colunas, uma vez que o Bloco de Notas, programa oficial do Windows, não permite. Originalmente o arquivo estava estruturado para uso no software STRUCTURE (Figura 3) (Pritchard et al. 2000) e teve de ser reformulado para uso no ARLEQUIN (Figura 4) (Excoffier 1995). Essa transformação foi realizada com o auxílio dos programas Windows Office Excell e TextPad. Após a mudança de uma estrutura para outra todos os parâmetros foram revistos e os indivíduos foram conferidos um a um, quanto à sua alocação nas respectivas populações DNA mitocondrial Ao contrário do banco de dados para microssatélites, o de DNA mitocondrial não foi inteiramente obtido de uma única fonte. A partir de Mendes (2005), foram obtidas as referências para a composição do banco de dados de DNA mitocondrial. As frequências de DNA mitocondrial presentes no trabalho foram conferidas e outras populações foram incluídas.
44 38 O banco de dados final consiste em trezentas e trinta e duas populações, com descrição das frequências de quatro haplogrupos (A-D) da região hipervariável I do DNA mitocondrial. O quinto haplogrupo (X) não foi contabilizado pelo fato de a maioria dos artigos não trazer a pesquisa deste haplogrupo nas populações descritas, sendo na maior parte das vezes relatado como pertencentes a outros os haplogrupos que não se encaixam em nenhum dos outros quatro A-D. Como não se pode ter certeza que esses outros sejam todos pertencentes ao X, a posição mais conservadora foi excluir essas frequências das análises. A consolidação do banco final foi baseada em cinquenta e duas referências. As frequências de haplogrupos foram obtidas por duas técnicas principais, o RFLP e o sequenciamento direto da região hipervariável I. A técnica de RFLP definia os haplogrupos conforme a presença ou ausência de determinado sítio de restrição (Tabela 1). Mais recentemente, passou-se a estudar a sequência de nucleotídeos diretamente, o que permitia também o acesso aos haplogrupos já descritos. A Tabela 2 traz a informação da metodologia empregada para quarenta e sete artigos usados na construção do banco de dados. As populações, suas frequências de haplogrupos e os respectivos artigos de referência estão descritos no Apêndice I. Tabela 1: Marcadores específicos de haplogrupos mitocondriais humanos (Merriwether & Ferrel 1996) Haplogrupo Sítio Condição A Hae III np 663 ganho B deleção de 9 pb perda C Alu I np 5176 perda D Hinc II np ganho
45 39 Tabela 2: Relação dos artigos e métodos empregados para a classificação dos indivíduos dentro dos haplogrupos de DNA mitocondrial. Cada referência está associada a uma cor única. Referência Carlyle et al 2000 Bailliet et al Bert et al 2001 Bolnick & Smith 2003 Brown et al Demarchi et al Derbeneva et al Easton et al Eshleman et al Forster et al Fuselli et al Gonzalez-Oliver et al Horai et al Kaestle & Smith 2001 Keyeux et al Kolman & Bermingham 1997 Kolman et al Lalueza-Fox et al Lewis et al Lorenz & Smith 1996 Malhi et al Malhi et al Malhi et al Merriwether & Ferrell 1996 Merriwether et al Merriwether et al Mesa et al Moraga et al O'Rourke et al Mendes, 2005 Rickards et al Saillard et al Schmitt et al Schurr et al Shields et al Shinoda et al Smith et al Starikovskaya et al Stone & Stoneking 1998 Torroni 1993 Torroni et al Torroni et al. 1994a Torroni et al. 1994b Vona et al RFLP RFLP RFLP RFLP RFLP RFLP sequenciamento RFLP sequenciamento sequenciamento sequenciamento Método empregado RFLP e sequenciamento sequenciamento RFLP RFLP e sequenciamento RFLP e sequenciamento RFLP e sequenciamento sequenciamento RFLP e sequenciamento RFLP RFLP e sequenciamento RFLP e sequenciamento sequenciamento RFLP RFLP RFLP RFLP sequenciamento RFLP e sequenciamento RFLP sequenciamento RFLP e sequenciamento RFLP e sequenciamento RFLP sequenciamento RFLP e sequenciamento RFLP e sequenciamento RFLP e sequenciamento RFLP e sequenciamento RFLP RFLP RFLP RFLP sequenciamento
46 40 Tabela 2 (continuação) Referência Carlyle et al 2000 Ward et al Ward et al Williams et al Método empregado RFLP RFLP sequenciamento RFLP e sequenciamento Localizações geográficas As localizações geográficas para as vinte e nove populações de microssatélites também foram acessadas através dos dados suplementares de Wang et al (2007) e estão descritas na Tabela 3 e plotadas no mapa da Figura 5. Para as populações de DNA mitocondrial, as coordenadas foram obtidas de três formas distintas. A primeira, a partir das coordenadas descritas nos artigos originais, de mapas presentes nos artigos e de dados etnográficos. As coordenadas obtidas diretamente dos artigos foram agregadas ao banco de dados exatamente como estão dispostas nos artigos e foram marcadas com a cor verde na tabela (Apêndice II). A segunda, obtida por uma aproximação em mapa, são marcadas pela cor amarela na tabela (Apêndice II), foram encontradas com o auxílio da ferramenta Google Earth 5.0. Um marcador foi colocado na localização aproximada pelo mapa descrito no artigo original, para ser posteriormente conferida a real possibilidade de ocupação da localização encontrada. Aqueles marcadores que recaíram sobre regiões de cursos d água, lagos, picos de montanhas e cobertas totalmente por gelo foram realocados de forma a estarem de acordo com a proximidade proposta pelo mapa do artigo original e fora dessas regiões de difícil ocupação. A terceira forma de obtenção das coordenadas, marcadas em vermelho na tabela (Apêndice II), foi por pesquisa indireta na literatura etnográfica da população descrita e passaram pelo mesmo teste de alocação com a ferramenta Google Earth 5.0 descrito anteriormente (Figura 6).
47 41 Tabela 3: Populações de microssatélites e suas respectivas coordenadas geográficas em decimais. População Latitude Longitude Chipewyan 107,3W 59,55N Cree 102,5W 50,33N Ojibwa 81W 46,5N Pima 108W 29N Mixtec 97W 17N Zapotec 97W 16N Mixe 96W 17N Maya 91W 19N Kaqchikel 91W 15N Cabecar 84W 9,5N Guaymi 82W 8,5N Kogi 74W 11N Arhuaco 73,8W 11N Waunana 77W 5N Embera 76W 7N Zenu 75W 9N Inga 77W 1N 74W 14S Aymara 70W 22S Huilliche 73W 41S Kaingang 52,5W 24S Guarani 54W -23S Wayuu 73W 11N Piapoco 68W 3N TicunaTarapaca 70W 4S TIcunaArara 70W 4S Karitiana 63W 10S Surui 62W 11S Ache 56W 24S
48 Figura 5: Localização geográfica das populações de microssatélites 42
49 Figura 6: Populações de DNA mitocondrial plotadas em mapa do Google Earth
50 44 A partir do banco de dados original foram retiradas as populações que estariam ocupando coordenadas geográficas idênticas (que poderiam ser consideradas amostras diferentes da mesma população) e foram mantidas as populações com o maior número de indivíduos amostrados, restando das 332 populações originais apenas 237. No Apêndice II estão representadas as populações que foram usadas para as análises, trazendo também as informações sobre o número de indivíduos amostrados em cada população, a frequência absoluta dos haplogrupos e as coordenadas geográficas. Os padrões de cores são os mesmos descritos anteriormente. O passo seguinte para a concretização do banco de dados de coordenadas geográficas para DNA mitocondrial consistiu na conferência dos pontos obtidos pelo Google Earth 5.0 no mapa em que seriam usados pelo programa de geoprocessamento para o cálculo das distâncias geográficas entre as populações. Na passagem de dados de um local para outro poderia ocorrer um desvio na localização das populações mesmo sendo as coordenadas no Google Earth 5.0 e no mapa de relevo utilizado no trabalho (Amante & Eakins, 2009) frutos de uma projeção cilíndrica simples com o datum WGS84. Para que não houvesse risco de as populações estarem alocadas em lugares diferentes daqueles primeiramente obtidos, cada população foi conferida quanto à sua localização no novo mapa (Figura 7) Banco de dados fisiográficos (biomas) Como o objetivo principal deste trabalho é a análise do papel das barreiras ecológicas na composição genética dos nativos americanos, a escolha de qual divisão ecológica adotada foi de suma importância. Neste caso foi escolhida a divisão por biomas. O conceito de biomas, criado por Frederick E. Clements, em 1949, pode ser traduzido como uma comunidade de plantas e animais que ocupam uma determinada área em que as relações climáticas essenciais são semelhantes ou idênticas (Coutinho
51 2006). Essa divisão foi escolhida por definir regiões em que os meios de subsistência fossem semelhantes devido à constância de tipos vegetais e animais. 45 Figura 7: Localizações geográficas das populações de DNA mitocondrial. A divisão de biomas adotada neste trabalho foi adaptada de um mapa oferecido pela WWF (World Wildlife Fund) construído por Olson et al. (2001) (Figuras 8 e 9). Este mapa apresenta catorze biomas distintos para o mundo todo. É de extrema importância destacar que a América apresenta todos os catorze biomas. Destes, onze foram ocupados pelas populações aqui amostradas: florestas úmidas tropicais e subtropicais,
52 46 florestas de coníferas tropicais e subtropicais, florestas temperadas de folhas largas, florestas de coníferas temperadas, florestas boreais e taiga, savanas e pradarias tropicais e subtropicais, savanas e pradarias temperadas, pradarias montanhosas e arbustos, tundra, desertos secos de arbustos e florestas mediterrânicas cobertas por árvores e arbustos (Tabelas 4 e 5). Figura 8: Populações nativas americanas com dados de microssatélites plotadas no mapa de biomas.
53 Figura 9: Populações nativas americanas com dados de DNA mitocondrial plotadas no mapa de biomas. 47
54 48 Tabela 4: Biomas nos quais as populações com dados de microssatélites estão inseridas Populações Chipewyan Cree Ojibwa Pima Mixtec Zapotec Mixe Maya Kaqchikel Cabecar Guaymi Kogi Arhuaco Waunana Embera Zenu Inga Aymara Huilliche Kaingang Guarani Wayuu Piapoco Ticuna Tarapaca TIcuna Arara Karitiana Surui Ache Biomas Floresta boreal/taiga Savanas e pradarias temperadas Florestas temperadas de folhas largas Florestas de coníferos tropical e subtropical Florestas de coníferos tropical e subtropical Florestas de coníferos tropical e subtropical Florestas de coníferos tropical e subtropical Florestas úmidas tropicais e subtropicais Florestas de coníferos tropical e subtropical Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Deserto e arbustos secos Florestas temperadas de folhas largas Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Deserto e arbustos secos Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais
55 Tabela 5: Biomas nos quais as populações com dados de DNA mitocondrial estão inseridas 49 População Ache Akimal O odham Aleuts Aleuts ancient Aleuts St. Paul Anasazi ancient Ancash Aónikenk Apache Arara Arequipa Peru Assurini Assurini Atacameno Atacameno Atacamenos Awá-Guajá Aymara Aymara Baníwa Bella Coola Bella Coola Bella Coola Bella Coola Boruca Bribi-Cabecar Cayapa Cayapa Ecuador Cherokee Cherokee Oklahoma Red Cross Cherokee Stillwell Cheyenne/Arapahoe Cheyenne/Araphoe Chickasaw Chickasaw/Choctaw Chimane Chimila Chippewa Chippewa Turtle Muntain Chippewa Wisconsin Chippewa/Kickapoo Choctaw Choroti Choroti Chumash Ciboneys Bioma Florestas úmidas tropicais e subtropicais Florestas mediterrânicas cobertas por árvores e arbustos Tundra Tundra Tundra Florestas mediterrânicas cobertas por árvores e arbustos Pradarias montanhosas e de arbustos Savanas e pradarias temperadas Florestas mediterrânicas cobertas por árvores e arbustos Florestas úmidas tropicais e subtropicais Florestas mediterrânicas cobertas por árvores e arbustos Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Pradarias montanhosas e de arbustos Pradarias montanhosas e de arbustos Florestas mediterrânicas cobertas por árvores e arbustos Florestas úmidas tropicais e subtropicais Pradarias montanhosas e de arbustos Pradarias montanhosas e de arbustos Florestas úmidas tropicais e subtropicais Florestas de coníferos tempradas Florestas de coníferos tempradas Florestas de coníferos tempradas Florestas de coníferos tempradas Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas temperadas de folhas largas Savanas e pradarias temperadas Florestas temperadas de folhas largas Savanas e pradarias temperadas Savanas e pradarias temperadas Florestas temperadas de folhas largas Florestas temperadas de folhas largas Florestas úmidas tropicais e subtropicais Florestas secas tropicais e subtropicais Florestas temperadas de folhas largas Florestas temperadas de folhas largas Florestas temperadas de folhas largas Savanas e pradarias temperadas Florestas de coníferos tempradas Savanas e pradarias tropicais e subtropicais Florestas úmidas tropicais e subtropicais Desertos e arbustos secos Florestas úmidas tropicais e subtropicais
56 Tabela 5 (continuação) 50 População Cochimi Coreguaje Creek (Muskoke) Creek/Seminole Curripaco Dogrib Dogrib Embera Embera Embera Ammassalik (Angmagssalik) Gambel Ilulissat (Jakobshavn) Ittoqqortoormiitt (Scoresbysund) Nanortalik Nuuk (Godthab) Savoonga Siberian Upernarik Uummannaq (Umanak) Yupik s s (Savoonga e Gambel) s Old Harbor s Ouzinkie Fueguian Argentina Gaviao Guahibo Guahibo-Sikuani Guambiano Guane-Butaregua Guatuso Guayabero Guaymi Haida Havasupai/Hualapai/Yavapai/ Mojave hokan (central coast) Hokan Northern Hopi Huetar Huetar Huilliche Huilliche Huilliche Chile Bioma Florestas mediterrânicas cobertas por árvores e arbustos Florestas úmidas tropicais e subtropicais Florestas temperadas de folhas largas Florestas de coníferos tempradas Florestas úmidas tropicais e subtropicais Floresta boreal/taiga Tundra Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Tundra Tundra Tundra Tundra Tundra Tundra Tundra Tundra Tundra Tundra Tundra Tundra Tundra Tundra Tundra Savanas e pradarias temperadas Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Savanas e pradarias tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Savanas e pradarias tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas de coníferos tempradas Florestas mediterrânicas cobertas por árvores e arbustos Desertos e arbustos secos Florestas de coníferos tempradas Florestas mediterrânicas cobertas por árvores e arbustos Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas temperadas de folhas largas Florestas temperadas de folhas largas Florestas temperadas de folhas largas
57 51 Tabela 5 (continuação) População Huitoto Hupa Ignaciano Ijka-Arhuaco Ingano Ingano Colombia Inuit eskimo Jemez Kanamarí Katuena Katukina Katukina Kawéskar Kaxináwa Kayapó Kiliwa Kiliwa/Paipai Kiowa Kogui Kraho Kumiai Kuna Kuna Kuna Macushi Makiritare Mapuche Mapuche Mapuche (Chile) Mapuche Anecón Grande Mapuche Argentina Mapuche Chile Marúbo Marúbo Brasil Mataco (Chaco) Mataco (Salta) Mataco (Wichí) Argentina Mataco (Wichí) Formosa Maya Maya Contenporary Micmac Micmac/Narragansett Mixe Mixtec Mohawk Bioma Florestas úmidas tropicais e subtropicais Florestas de coníferos tempradas Florestas úmidas tropicais e subtropicais Florestas mediterrânicas cobertas por árvores e arbustos Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Tundra Florestas mediterrânicas cobertas por árvores e arbustos Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas temperadas de folhas largas Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas mediterrânicas cobertas por árvores e arbustos Desertos e arbustos secos Savanas e pradarias temperadas Florestas mediterrânicas cobertas por árvores e arbustos Florestas secas tropicais e subtropicais Desertos e arbustos secos Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Savanas e pradarias temperadas Savanas e pradarias temperadas Florestas temperadas de folhas largas Savanas e pradarias temperadas Savanas e pradarias temperadas Florestas temperadas de folhas largas Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Savanas e pradarias tropicais e subtropicais Savanas e pradarias tropicais e subtropicais Florestas úmidas tropicais e subtropicais Savanas e pradarias tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Floresta boreal/taiga Florestas temperadas de folhas largas Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas temperadas de folhas largas
58 52 Tabela 5 (continuação) População Mohawk Mosetén Bolivia Movima Bolívia Murui-Muinane Muskoke Oklahoma Nahua Nahua/Cora Navajo Ngöbé Nukak Nuu-Chah-Nulth Ojibwa Manitoulin Island Ojibwa North Ontario Ojibwa/Chippewa Old Harbor Ouzinkie Paez Paiute N./Shoshoni Paiute/Shoshone Parakanã Pasto Pawnee Pehuenche Butalelbum Pehuenche Trapa Trapa Peneunche Chile 2 Penutian California Penutian California Piaroa Piaroa Colombia Pilagá Formosa Pima Pima Pima Poturujara (Zoé) Pyramid Lake ancient Quechan/Cocopa (Arequipa) (Tayacaja) Bolivia /Nmatsiquenga (San Martin) Sahaptian Plateau Aleut Saint Paul Salinan/Chumash Bioma Florestas temperadas de folhas largas Florestas úmidas tropicais e subtropicais Savanas e pradarias tropicais e subtropicais Florestas úmidas tropicais e subtropicais Savanas e pradarias temperadas Florestas mediterrânicas cobertas por árvores e arbustos Florestas secas tropicais e subtropicais Florestas mediterrânicas cobertas por árvores e arbustos Florestas úmidas tropicais e subtropicais Savanas e pradarias tropicais e subtropicais Florestas de coníferos tempradas Florestas temperadas de folhas largas Floresta boreal/taiga Floresta boreal/taiga Tundra Tundra Florestas úmidas tropicais e subtropicais Florestas mediterrânicas cobertas por árvores e arbustos Florestas mediterrânicas cobertas por árvores e arbustos Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Savanas e pradarias temperadas Desertos e arbustos secos Florestas temperadas de folhas largas Florestas temperadas de folhas largas Savanas e pradarias temperadas Savanas e pradarias temperadas Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Savanas e pradarias tropicais e subtropicais Desertos e arbustos secos Florestas mediterrânicas cobertas por árvores e arbustos Florestas mediterrânicas cobertas por árvores e arbustos Florestas úmidas tropicais e subtropicais Florestas mediterrânicas cobertas por árvores e arbustos Florestas de coníferos tempradas Florestas mediterrânicas cobertas por árvores e arbustos Pradarias montanhosas e de arbustos Florestas úmidas tropicais e subtropicais Florestas secas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas mediterrânicas cobertas por árvores e arbustos Tundra Desertos e arbustos secos
59 53 Tabela 5 (continuação) População Salish Plateau Selknam Seminole Seri Siona Siouan Sioux Sisseton/Wapheton Sioux Stillwater Marsh ancient Takik Taono O odham (Papago) Tayacaja Peru Tehuelche Argentina Teribe Teribe Tikúna Tikúna Brasil Tikúna Colombia Toba Toba (chaco) Toba (Formosa) Trinitario Bolivia Tucano Tule-Cuna Uto-Aztecan California Uto-Aztecan Central Uto-Aztecan Northern Waiãpi Wapishana Brasil Washo Waunana Wayuu Wayuu Colombia Wiwa Wounan Xavante Yaghan Chile Yakama Yakima Yamamadi Yámana (Yaghan) Yaminawa Yanomama Yanomama Brasil Yanomami Yanomamö Bioma Florestas de coníferos tempradas Florestas temperadas de folhas largas Florestas de coníferos tempradas Florestas mediterrânicas cobertas por árvores e arbustos Florestas úmidas tropicais e subtropicais Savanas e pradarias temperadas Savanas e pradarias temperadas Florestas mediterrânicas cobertas por árvores e arbustos Florestas mediterrânicas cobertas por árvores e arbustos Florestas mediterrânicas cobertas por árvores e arbustos Florestas mediterrânicas cobertas por árvores e arbustos Savanas e pradarias temperadas Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Savanas e pradarias tropicais e subtropicais Savanas e pradarias tropicais e subtropicais Savanas e pradarias tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Savanas e pradarias temperadas Florestas mediterrânicas cobertas por árvores e arbustos Florestas mediterrânicas cobertas por árvores e arbustos Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas de coníferos tempradas Florestas úmidas tropicais e subtropicais Florestas mediterrânicas cobertas por árvores e arbustos Florestas mediterrânicas cobertas por árvores e arbustos Florestas secas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Savanas e pradarias tropicais e subtropicais Florestas temperadas de folhas largas Florestas mediterrânicas cobertas por árvores e arbustos Florestas de coníferos tempradas Florestas úmidas tropicais e subtropicais Florestas temperadas de folhas largas Florestas úmidas tropicais e subtropicais Savanas e pradarias tropicais e subtropicais Florestas úmidas tropicais e subtropicais Savanas e pradarias tropicais e subtropicais Florestas úmidas tropicais e subtropicais
60 54 Tabela 5 (continuação) População Yok-Utian Yuko-Yukpa Yuman Yuman (Hokan) Yuman Delta Yuman Pai Yuman River Yuracaré Zapotec Zenu Zoro Zuni Zuni (penutian) Bioma Florestas de coníferos tempradas Florestas úmidas tropicais e subtropicais Florestas mediterrânicas cobertas por árvores e arbustos Florestas mediterrânicas cobertas por árvores e arbustos Florestas mediterrânicas cobertas por árvores e arbustos Florestas mediterrânicas cobertas por árvores e arbustos Florestas mediterrânicas cobertas por árvores e arbustos Florestas úmidas tropicais e subtropicais Florestas úmidas tropicais e subtropicais Savanas e pradarias tropicais e subtropicais Florestas úmidas tropicais e subtropicais Florestas mediterrânicas cobertas por árvores e arbustos Florestas mediterrânicas cobertas por árvores e arbustos 2.2. Construção das matrizes A comparação de matrizes tornou-se um método importante de análise de variáveis par a par (Smouse et al. 1986; Legendre, 2000), sendo largamente utilizadas para o contexto desenvolvido neste trabalho, as matrizes de distâncias, que são por definição quadradas e simétricas. Os valores da matriz utilizada expressam as distâncias entre as populações, que podem ser atribuídas quantitativa ou qualitativamente. Essas matrizes apresentam como propriedades: a) o fato de sua diagonal (elementos da matriz que obedecem a condição aij 8 sendo i igual a j) ser igual a zero, b) os elementos de fora da diagonal, que obedecem à condição aij em que i é diferente de j apresentam os mesmos valores nos triângulos inferior e superior, pois nessas posições os pares são os mesmos, por isso são chamadas simétricas. Devido à dificuldade de saber em que nível de estruturação genética poderia aparecer a assinatura das barreiras ecológicas, os bancos de dados foram analisados no todo e em partes. O banco de dados de microssatélites foi analisado com as vinte e nove 8 Por definição aij é a distância entre duas populações sendo i representada por uma e j por outra.
61 55 populações (denominadas no decorrer do texto como micro ) e também parcionado em três sub-conjuntos correspondentes à divisão política atual de América do Sul ( micros ), América Central ( microc ) e América do Norte ( micron ). Optou-se por essas divisões pelo fato de as populações amostradas respeitarem-nas, não apresentando populações nas fronteiras (Figura 10). Figura 10: Mapa da América com as divisões usadas para análise de microssatélites. (micron) populações representantes da América do Norte, (microc) populações representantes da América Central e (micros) populações representantes da América do Sul.
62 56 Para os dados de DNA mitocondrial foram criados onze bases de dados. Uma seleção foi escolhida para conferência do método, em que seis grupos de 3 a 6 populações obedecessem ao padrão de maior proximidade física entre aquelas presentes em biomas distintos ( conj1-6 ) (Figura 11 a 13). O sétimo grupo consistiu na exclusão das populações com tamanho amostral menor que dez indivíduos ( n10 ). As outras divisões obedeceram à separação natural das amostragens, divididas em um conjunto na Patagonia ( biom31 ), outro no centro norte da América do Sul ( biom42 ), outro unindo todas as populações dos biom31 e biom42 ( biom74 ) e ainda uma quarta divisão com América do Norte e norte da América Central ( biom63 ) (Figura 14 a 16). Essas divisões foram escolhidas por serem os lugares com amostras mais homogêneas visualmente, evitando assim, grandes distâncias fisiográficas e físicas sem populações amostradas. Figura 11: Populações presentes nos conjuntos 1 (oito populações marcadas com a cor vermelha) e 2 (três populações marcadas com a cor azul)
63 Matrizes de distância genética As distâncias genéticas entre as populações nativas americanas foram obtidas com a estatística Fst. O Fst é uma derivação do coeficiente de endocruzamento (F), sendo este último definido como a probabilidade de um indivíduo ser homozigoto, em decorrência do encontro, nele, de um par de alelos idênticos, derivados de um único gene herdado de um de seus ancestrais (Beiguelman 1994). Já o Fst mede a redução da heterozigosidade em relação à esperada se ocorressem apenas cruzamentos aleatórios em uma população (Hartl 2008). Figura 12: Populações presentes nos conjuntos 3 (três populações marcadas com a cor lilás), 5 (três populações marcadas com a cor amarela) e 6 (quatro populações marcadas com a cor preta). O Fst indica a média de endocruzamento em uma subpopulação em relação à população total (Falconer 1996). Esta estatística desenvolvida por Sewal Wright, em 1951
64 (Hedrick 1999), como uma razão da variância observada nas frequências dos genes nas subpopulações até a variância máxima possível dadas as frequências alélicas, sem levar 58 Figura 13: Populações presentes no conjunto 4 (três populações Figura 14: Populações referentes ao biom31
65 59 Figura 14: Populações referentes ao biom42 Figura 16: Populações referentes ao biom63
66 60 em conta sua própria estrutura (Wright 1965); também pode ser vista como uma medida da diferença entre a probabilidade de que dois alelos retirados de uma subpopulação sejam idênticos e a probabilidade de que dois alelos retirados da espécie sejam idênticos (Gillespie 2004). Esse conceito pode ser usado para comparar populações entre si, como um índice de diversidade (Nei 1973), pois coloca em números a porcentagem da diversidade que pode ser atribuída à divergência entre as populações estudadas, gerando um índice de distância entre elas. Neste trabalho, como as populações são tomadas par a par, o Fst foi escolhido, pois pode ser usado como uma medida de distância, e como medida indireta da quantidade de fluxo gênico entre elas. Para o cálculo de Fst é necessário que as populações ou subpopulações sejam definidas a priori, além de implicitamente supor o modelo de mutação de alelos infinitos, que é baseado em mutações que ocorrem raramente e quando ocorrem não trazem nenhuma informação sobre a sequência ancestral (Beiguelman, 1994). Para microssatélites o modelo de mutação de alelos infinitos não é o mais adequado, pois este modelo não leva em consideração a possibilidade de intercâmbio entre as formas alélicas. Já o modelo de step-wise descreve melhor os processos mutacionais que envolvem os microssatélites. Slatkin (1995) formulou um índice baseado no Fst, que considera parâmetros mais adequados para microssatélites e o batizou de Rst. Este novo índice, segundo Slatkin, teria maior correspondência com os acontecimentos históricos na estimativa de valores demográficos das populações analisadas (Slatkin 1995). As distâncias entre as populações foram calculadas como índices de fixação (Fst e Rst) geradas pelo programa Arlequim 3.11 (Excoffier 2005), com mil permutações dos haplótipos das populações e nível de significância em 0,05, o que significa que em apenas 5% das permutações, que embaralha as populações entre os grupos, é
67 61 encontrado um valor para o Fst ou Rst entre as populações igual ou acima do observado. Foi permitido 10% de dados faltantes por locus. Ao final da análise de microssatélites restaram 647 loci. O Fst para o DNA mitocondrial foi calculado segundo os mesmos parâmetros, mas com base apenas nas frequências alélicas absolutas das populações de apenas um locus com quatro ou cinco variantes. Os quatro polimorfismos são as frequências dos haplogrupos A-D. Porém, para algumas populações essas frequências não somaram 100%, porque não foi possível determinar para todas as populações quais eram as frequências exatas do quinto haplogrupo nativo americano, o haplogrupo X. Atribuiu-se, então, um quinto polimorfismo, fictício, que englobasse todas essas frequências faltantes. Adotou-se esta alternativa porque o programa usado para o cálculo de Fst não calcula as distâncias com dados faltantes. Para suprir a falta nos dados originais o programa automaticamente altera os valores das amostras para que as frequências relativas somem 100%. Por exemplo, em uma população com 10 indivíduos, se 20% fossem haplogrupo A, 30% B, 10% C e 10% D, o programa mudaria o número amostral para 7 indivíduos e as frequências relativas passariam a ser 28,6% para A, 42,8% para B, 14,3% para C e 14,3% para D. Com a criação do quinto haplogrupo fictício, as frequências de A-D estariam corretas apesar de unir em um haplogrupo combinações de alelos/haplogrupos que poderiam ser muito distintas. Essa abordagem foi escolhida pelo desconhecimento de um programa ou de uma forma de calcular o Fst sem precisar adaptar o número da amostra inicial. Neste trabalho foram construídas uma matriz de Fst e outra de Rst para os dados de microssatélites e apenas de Fst para os dados de DNA mitocondrial (Apêndices III a XXXII).
68 Matrizes de distâncias geográficas/físicas As distâncias geográficas, ou físicas, entre as populações foram obtidas com o auxílio do programa PATHMATRIX, desenvolvido em 2005, por Nicholas Ray. O PATHMATRIX usa como base o programa de geoprocessamento Arcview 3.2 GIS software. Com esta ferramenta foi possível calcular as distâncias entre as populações de duas formas distintas. A primeira, chamada aqui de distância euclidiana, calcula as distâncias mais próximas entre dois pontos, sem considerar desvios de rotas de difícil acesso. Para o cálculo desta distância todos os pontos do mapa apresentam um mesmo valor e esse valor aumenta homogeneamente à medida que se afasta do ponto de partida para qualquer direção (Figura 17). A B Figura 17: (A) Mapa de fragmento da América do Norte sem atribuição de custo aos diferentes pontos do percurso. (B) Representação gráfica dos valores de distâncias a medida que se afastam de um ponto inicial.
69 63 A segunda forma, chamada aqui de distância de custos, atribuiu custos a percursos que seriam de difícil travessia, obrigando o algoritmo a calcular rotas mais longas, mas que não atravessassem regiões de difícil acesso, como formações montanhosas ou grandes porções de água (Figura 18). A B Figura 18: (A) Mapa de fragmento da América do Norte com atribuição de custo aos diferentes pontos do percurso. (B) Representação gráfica dos valores de distâncias à medida que se afasta de um ponto inicial(quanto mais forte a cor, maior o custo atribuído). Neste trabalho, os dois tipos de rotas serão comparados aos dados genéticos, de forma a testar qual impacto o uso de rotas com custos pode exercer em análises quando comparado às rotas sem custos (Wang et al. 2007). Para a construção dos mapas para uso no PATHMATRIX foi utilizada a ferramenta ArcMap GIS. Obteve-se um mapa (Figura 19) com a topografia mundial (Amante & Eakins, 2009) em que cada valor de altitude estava associado a uma célula de
70 64 Figura 19: Mapa topográfico da América
71 65 aproximadamente um quilômetro quadrado. Este tipo de mapa, chamado de Raster map 9, é a base para o cálculo das distâncias no programa PATHMATRIX. Sua característica principal é que um mapa para ser considerado raster, precisa apresentar um valor único associado a uma célula. Este valor pode ser alterado para um valor de custo desejado. Nos atributos do mapa o valor aparece como value e a célula a que ele está associado aparece como count. Para o mapa aqui utilizado os valores de value foram transformados em 3 categorias distintas. Para as altitudes entre -431 e 0m foi atribuído o custo de cinco; de 0 a 2.000m, o custo de zero, e acima de 2.000m, o custo de vinte. Para as regiões oceânicas, marcadas neste mapa como sem dados (no data), atribuiu-se o custo de vinte (Figura 20). Os valores foram atribuídos de maneira a dificultar substancialmente a travessia dessas regiões, uma vez que cada quilometro percorrido nestas áreas conta como vinte quilômetros. Figura 20: Mapa mundial com custos atribuídos O segundo passo foi acrescentar as regiões de grandes lagos ao custo 20. O mapa de grandes lagos foi transformado em Raster map com o auxílio de ferramentas no próprio ArcMap, para os lagos com mais de 70km 2 atribuiu-se um custo vinte e para 9 Mantido termo em inglês pelo desconhecimento de similarem português
72 66 aqueles com menos de 70km 2, um custo zero, seguindo o mesmo protocolo. À soma do mapa de lagos com o mapa anterior novos custos foram atribuídos seguindo a mesma lógica. O mapa final de custos está representado na Figura 23. Com esses valores de custo, as rotas que ligam diferentes populações evitam atravessar oceanos, regiões montanhosas e regiões com grandes porções de água (Figura 22). Após a construção final do mapa foi necessário diminuir a sua precisão em 10 vezes e transformar uma célula de aproximadamente um quilometro quadrado em dez quilômetros quadrados, devido às limitações do algoritmo do programa PATHMATRIX. A transformação foi feita usando-se como valor final para os dez quilômetros quadrados a média das células presentes neste grupo. Após essas modificações finais, submeteu-se o mapa ao programa PATHMATRIX para cálculo das distâncias (Figura 23). Ao final, as análises foram feitas usando grupos de 25 e 50 populações (pois o programa não comportava mais do que 150 populações concomitantemente) e depois foram unidas em duas matrizes, conforme se distâncias euclidianas ou de custo com o auxílio do programa Excell. As matrizes prontas podem ser encontradas nos Apêndices XXXIII a XXXIV Matrizes de distâncias fisiográficas As distâncias fisiográficas foram computadas de duas formas distintas. A primeira, com escores binários de zero e um, sendo zero usado para pares de populações que se encontram dentro de um mesmo bioma e um para pares que se encontram em biomas distintos. A segunda, com escores cumulativos, em que cada população iniciou com escore um e a cada bioma atravessado, pelo caminho com menor número de biomas envolvidos, somou-se mais um ao escore inicial.
73 Figura 21: Mapa mundial final com custos atribuídos 67
74 68 Figura 22: Exemplo de caminhos de menor custo gerados pelo programa para 25 populações aleatórias de DNA mitocondrial. Para a construção das matrizes, tanto as populações representadas por microssatélites quanto por DNA mitocondrial foram plotadas no mapa de biomas (Figuras 8 e 9) sendo atribuídos os valores visualmente, conforme descrito acima. As matrizes estão presentes nos Apêndices XXXV a XXXVII Testes estatísticos Os testes estatísticos consistiram na comparação entre as matrizes. Todos os testes foram feitos utilizando o programa R (R Development Core Team 2006) e o pacote estatístico Vegan versão (Oksanen et al. 2010).
75 69 Figura 23: Exemplo com três populações das matrizes construídas para as distâncias euclidianas (acima) e as distâncias por custo (abaixo) Testes de correlação As correlações entre os pares de matrizes foram obtidas pelos métodos estatísticos Pearson, Kendall e Spearman. A correlação de Pearson (r) é feita a partir da covariância padronizada, sendo baseada no somatório das diferenças entre os valores observados (xi, yi) e a média para cada grupo (x, y), dividido pelo produto dos desvios padrões (sx, sy), representado pela fórmula (3.1). (3.1)
76 70 O teste de correlação de Pearson consiste em um teste paramétrico no qual é preciso que as variáveis tenham uma distribuição normal e valores intervalares. O teste de correlação de Spearman e de Kendall são testes não-paramétricos e podem ser usados com variáveis categóricas não intervalares. Ambos trabalham com a ordenação dos dados antes de aplicá-los à fórmula de Pearson. O teste de Kendall é mais adequado quando se tem um número pequeno de dados com grande número de postos empatados (Field 2009). Devido ao fato de os dados analisados apresentarem uma matriz binária, que por definição é uma matriz categórica, as três formas de correlação foram utilizadas (Verzani 2004; Field 2009) Teste de significância de Mantel A principal dificuldade em analisar correlações entre distâncias par a par é testar a significância das correlações encontradas, devido à falta de independência entre os dados (Smouse 1986). Porém, para transpor este problema, Nathan Mantel desenvolveu uma metodologia para testar a significância atribuída às correlações, que se baseia na hipótese nula de que a correlação encontrada entre os pares não seria diferente da correlação encontrada entre os pares distribuídos ao acaso (Mantel 1967). A solução proposta por ele consiste em permutar as linhas e colunas de uma das matrizes enquanto a outra permanece constante, criando uma distribuição de valores de correlações gerados ao acaso. A partir dessa distribuição é possível quantificar a probabilidade de se obter o valor da correlação testada sob o modelo nulo. Em situações em que o número de permutações é muito grande pode-se lançar mão do recurso de amostragem Monte Carlo para se obter a curva de valores de correlação (Mantel 1967, Smouse 1986).
77 71 O teste parcial de Mantel, aplicado aqui, foi feito para três matrizes. O teste consiste em controlar os efeitos de uma matriz na correlação de outras duas. Por exemplo, dado um conjunto de dados A e outros dois conjuntos B e C que apresentam uma correlação positiva com A e entre si é difícil saber qual a parcela da correlação entre A e B que é consequência da influência que C tem em B e que, por conseguinte se correlaciona com A ou vice-versa. O teste parcial retira o efeito de um conjunto de dados, digamos C, para que a correlação apresentada por A e B não tenha nenhum efeito residual de C (Field 2009, Smouse 1986, Marroig et al 2001) Algoritmo de Monmonier O algoritmo de Monmonier consiste em uma análise de Landscape Genetics (Manel 2003) em que as populações são unidas usando uma rede de Delauney (método gráfico para definir pontos adjacentes em um mapa) (Figura 24). Em seguida, traça-se uma perpendicular entre as populações adjacentes com maior índice de Fst (distância genética) e segue-se a perpendicular, sempre associada às mais altas distâncias genéticas, até que se chegue a uma borda ou forme um círculo em torno de uma ou mais A B Figura 24: (A) Populações distribuídas espacialmente. (B) Rede de Delauney para as mesmas populações.
78 72 localidades (Figura 25). Com o uso deste algoritmo tem-se a definição de grupos em ambos os lados de uma barreira genética (Dupanloup et al. 2002). Este algoritmo foi utilizado como mais uma ferramenta para investigar as possíveis barreiras genéticas entre as populações nativas americanas. Figura 25: Perpendicular entre as populações de maior distância genética formando dois grupos distintos separados por uma barreira genética.
79 73 3. RESULTADOS Os resultados obtidos no curso deste trabalho podem ser divididos em duas categorias principais: de DNA mitocondrial e de microssatélite, havendo dez subcategorias, seis para DNA mitocondrial, consistindo em onze conjuntos de populações e quatro para microssatélites, consistindo em quatro conjuntos de populações. Todas as subcategorias são compostas por resultados de correlação entre duas matrizes, envolvendo as matrizes de distâncias genética, geográficas e fisiográficas e também envolvendo três matrizes, distância genética e geográfica corrigindo para distância fisiográfica e entre distância genética e fisiográfica corrigindo o efeito de distância geográfica. Além dessas análises o conjunto de todas as populações de microssatélites também foi analisado de acordo com o o algoritmo de Monmonier. Os resultados serão apresentados conforme as abreviações a seguir: correlação de Pearson (rp), correlação de Kendall (rk), correlação de Spearman (rs), matriz de fst (fst), matriz de fst com 5 haplogrupos de DNA mitocondrial (fsthap5), matriz de rst (rst), matriz de distância geográfica euclidiana (eucl), matriz de distância geográfica de custo (cust), matriz de distância fisiográfica binária (eco), matriz de distância fisiográfica cumulativa (eco2) DNA mitocondrial Conjuntos menores Foram criados seis conjuntos menores, o primeiro (conj1) envolvendo as populações Salish, Yakama, Yakima, Nuu-Chah-Nulth, Bella Coola, Sahapatian, Paiute N./Shoshoni e Uto-aztecan do norte. Para essas populações rp(eucl, cust) = 0,9633 (p=0,0002); rp(fst, eucl) = 0,3986 (p=0,042); rp(fst, cust) = 0,4483 (p=0,044); rp(fst, eco) = 0,2365; rk(fst, eco) = 0,1930 e rs(fst, eco) = 0,2323; as três não significativas. Ainda para este primeiro conjunto, rp(fsthap5, eucl) = 0,4086 (p=0,052); rp(fsthap5, cust) = 0,4554 (p=0,066); rp
80 74 (fsthap5, eco) = 0,2391 (p=0,144); rk(fsthap5, eco) = 0,1708 (p=0,171) e rs(fsthap5, eco) = 0,2055 (p=0,170). Para as correlações entre distâncias geográficas e fisiográficas: rp(eucl, eco) = 0,3863 (p=0,031); rk(eucl, eco) = 0,3118 (p=0,029); rs(eucl, eco) = 0,3752 (p=0,027); rp(cust, eco) = 0,2921 (p=0,028); rk(cust, eco) = 0,2673 (p=0,029) e rs(cust, eco) = 0,3216 (p=0,026). As correlações parciais de Mantel para este conjunto não foram significativas para nenhuma comparação. As correlações entre a distância genética e euclidiana controlando o efeito de fisiografia são representadas pela notação (fst, eucl eco). Os resultados obtidos foram: rp(fst, eucl eco) = 0,3428 (p=0,060); rk(fst, eucl eco) = 0,2192 (p=0,074); rs (fst, eucl eco) = 0,3441 (p=0,66); rp(fsthap5, eucl eco) = 0,3531 (p=0,103); rk(fsthap5, eucl eco) = 0,2201 (p=0,99) e rs(fsthap5, eucl eco) = 0,3693 (p=0,77). De correlação genética e fisiográfica controlando para o efeito de distância geográfica euclidiana (fst, eco eucl) com resultados rp(fst, eco eucl) = 0,0976 (p=0,293); rk(fst, eco eucl) = 0,1206 (p=0,224); rs(fst, eco eucl) = 0,0976 (p=0,304); rp(fsthap5, eco eucl) = 0,0965 (p=0,292); rk(fsthap5, eco eucl) = 0,0980 (p=0,250) e rs(fsthap5, eco eucl) = 0,0602 (p=0,331). Os resultados de correlação genética e distância de custo controlando para o efeito fisiográfico (fst, cust eco) foram: rp(fst, cust eco) = 0,4070 (p=0,069); rk(fst, cust eco) = 0,2308 (p=0,104); rs(fst, cust eco) = 0,3896 (p=0,080); rp(fsthap5, cust eco) = 0,4152 (p=0,098); rk(fsthap5, cust eco) = 0,2863 (p=0,085) e rs(fsthap5, cust eco) = 0,4248 (p=0,087). As distâncias genéticas e fisiográficas controlando para o efeito de distâncias de custo (fst, eco cust) foram: rp(fst, eco cust) = 0,1238 (p=0,242); rk(fst, eco cust) = 0,1303 (p=0,226); rs(fst, eco cust) = 0,1088 (p=0,289); rp(fsthap5, eco cust) = 0,1246 (p=0,235); rk(fsthap5, eco cust) = 0,0940 (p=0,252) e rs(fsthap5, eco cust) = 0,0685 (p=0,326). Resultados descritos na Tabela 6.
81 75 Tabela 6: Valores das correlações para o conjunto 1 entre as matrizes e suas respectivas significâncias. Marcado em negrito estão as correlações significativas com p<0,05. mantel Valor p eucl, cust (rp) fst, eucl (rp) fst, cust (rp) fst, eco (rp) fst, eco (rk) fst, eco (rs) fsthap5, eucl (rp) fsthap5, cust (rp) fsthap5, eco (rp) fsthap5, eco (rk) fsthap5, eco (rs) eucl, eco (rp) eucl, eco (rk) eucl, eco (rs) cust, eco (rp) cust, eco (rk) cust, eco (rs) 0,9633 0,3986 0,4473 0,2365 0,1930 0,2323 0,4086 0,4554 0,2391 0,1708 0,2055 0,3863 0,3118 0,3752 0,2921 0,2673 0,3216 0,0002 0,042 0,044 0,140 0,169 0,168 0,052 0,066 0,144 0,171 0,170 0,031 0,029 0,027 0,028 0,029 0,026 parcial de mantel fst, eucl eco (rp) fst, eucl eco (rk) fst, eucl eco (rs) fst, eco eucl (rp) fst, eco eucl (rk) fst, eco eucl (rs) fsthap5, eucl eco (rp) fsthap5, eucl eco (rk) fsthap5, eucl eco (rs) fsthap5, eco eucl (rp) fsthap5, eco eucl (rk) fsthap5, eco eucl (rs) fst, cust eco (rp) fst, cust eco (rk) fst, cust eco (rs) fst, eco cust (rp) fst, eco cust (rk) fst, eco cust (rs) fsthap5, cust eco (rp) fsthap5, cust eco (rk) fsthap5, cust eco (rs) fsthap5, eco cust (rp) fsthap5, eco cust (rk) fsthap5, eco cust (rs) 0,3428 0,060 0,2192 0,074 0,3441 0,066 0,0976 0,293 0,1206 0,224 0,0976 0,304 0,3531 0,103 0,2201 0,099 0,3693 0,077 0,0965 0,292 0,0980 0,250 0,0602 0,331 0,4070 0,069 0,2308 0,104 0,3896 0,080 0,1238 0,242 0,1303 0,226 0,1088 0,289 0,4152 0,098 0,2863 0,085 0,4248 0,087 0,1246 0,235 0,0940 0,252 0,0685 0,326
82 76 O segundo conjunto (conj2) envolveu as populações Chippewa, Chippewa/Kickapoo e Chickasaw. Nenhuma correlação foi significativa para este conjunto, os valores serão descritos adiante: rp(eucl, cust) = 0,9991 (p=0,171), rp(fst, eucl) = 0,2807 (p=0,171), rp(fst, cust) = 0,2389 (p=0,508), rp(fst, eco) = -0,1814 (p=0,673), rk(fst, eco) e rs(fst, eco) = 0 (p=0,661), rp(fsthap5, eucl) = 0,4350 (p=0,505), rp(fsthap5, cust) = 0,3956 (p=0,498), rp (fsthap5, eco) = -0,3408 (p=0,670), rk(fsthap5, eco) = 0,000 (p=0,669), rs(fsthap5, eco) = 0,000 (p=0,660), rp(eucl, eco) = -0,9948 (p=1), rk(eucl, eco) = -0,8165 (p=1), rs(eucl, eco) = -0,8660 (p=1), rp(cust, eco) = -0,9983 (p=1), rk(cust, eco) = -0,8165 (p=1), rs(cust, eco) = -0,8660 (p=1). Para as correlações parciais de Mantel também não houve significância para nenhuma correlação, rp(fst, eucl eco) = 1 (p=0,338), rk(fst, eucl eco) = 0,5774 (p=0,338), rs(fst, eucl eco) = 1 (p=0,333), rp(fsthap5, eucl eco) = 1 (p=0,326), rk(fsthap5, eucl eco) = 0,5774 (p=0,498), rs(fsthap5, eucl eco) = 1 (p=0,334), rp(fst, eco eucl) = 1 (p=0,331), as correlações de rk e rs não foram calculadas pelo programa, rp(fsthap5, eco eucl) = 1 (p=0,333), as correlações de rk e rs não foram calculadas pelo programa, rp(fst, cust eco) = 1 (p=0,333), rk(fst, cust eco) = 0,5774 (p=0,505), rs(fst, cust eco) = 1 (p=0,328), rp(fsthap5, cust eco) = 1 (p=0,331), rk(fsthap5, cust eco) = 0,5774 (p=0,499) e rs(fsthap5, cust eco) = 1 (p=0,332), rp(fst, eco cust) = 1 (p=0,331), as correlações de rk e rs não foram calculadas pelo programa, rp(fsthap5, eco cust) = 1 (p=0,328), as correlações de rk e rs não foram calculadas pelo programa (Tabela 7).
83 77 Tabela 7: Valores das correlações para o conjunto 2 entre as matrizes e suas respectivas significâncias. Não houve nenhuma correlação significativa eucl, cust (rp) fst, eucl (rp) fst, cust (rp) fst, eco (rp) fst, eco (rk) fst, eco (rs) fsthap5, eucl (rp) fsthap5, cust (rp) fsthap5, eco (rp) fsthap5, eco (rk) fsthap5, eco (rs) eucl, eco (rp) eucl, eco (rk) eucl, eco (rs) cust, eco (rp) cust, eco (rk) cust, eco (rs) mantel Valor p 0,9991 0,171 0,2807 0,501 0,2389 0,508-0,1814 0,673 0,0000 0,661 0,0000 0,661 0,4350 0,505 0,3956 0,498-0,3408 0,670 0,0000 0,669 0,0000 0,660-0, , , , , , parcial de mantel fst, eucl eco (rp) fst, eucl eco (rk) fst, eucl eco (rs) fst, eco eucl (rp) fsthap5, eucl eco (rp) fsthap5, eucl eco (rk) fsthap5, eucl eco (rs) fsthap5, eco eucl (rp) fst, cust eco (rp) fst, cust eco (rk) fst, cust eco (rs) fst, eco cust (rp) fsthap5, cust eco (rp) fsthap5, cust eco (rk) fsthap5, cust eco (rs) fsthap5, eco cust (rp) 1 0,338 0,5774 0, , , ,326 0,5774 0, , , ,333 0,5774 0, , , ,331 0,5774 0, , ,328 O terceiro conjunto (conj3) envolveu as populações Kayapó, Xavante e Kraho. Nenhuma correlação foi significativa para este conjunto, os valores são: rp(eucl, cust) = 1
84 (p=0,168), rp(fst, eucl) = 0,4425 (p=0,505), rp(fst, cust) = 0,4425 (p=0,496), rp(fst, eco) = -0,0581 (p=0,668), rk(fst, eco) e rs(fst, eco) = 0 (p=0,663), rp(fsthap5, eucl) = 0, (p=0,499), rp(fsthap5, cust) = 0,4425 (p=0,502), rp(fsthap5, eco) = -0,0581 (p=0,669), rk (fsthap5, eco) = 0,000 (p=0,668), rs(fsthap5, eco) = 0,000 (p=0,676), rp(eucl, eco) = -0,9209 (p=1), rk(eucl, eco) = -0,8165 (p=1), rs(eucl, eco) = -0,8660 (p=1), rp(cust, eco) = -0,9210 (p=1), rk(cust, eco) = -0,8165 (p=1), rs(cust, eco) = -0,8660 (p=1). Para as correlações parciais de Mantel também não houve significância para nenhuma correlação, rp(fst, eucl eco) = 1 (p=0,494), rk(fst, eucl eco) = 0,5774 (p=0,504), rs(fst, eucl eco) = 1 (p=0,333), rp(fsthap5, eucl eco) = 1 (p=0,491), rk(fsthap5, eucl eco) = 0,5774 (p=0,502) rs (fsthap5, eucl eco) = 1 (p=0,327), rp(fst, eco eucl) = 1 (p=0,335), as correlações de rk e rs não foram calculadas pelo programa, rp(fsthap5, eco eucl) = 1 (p=0,327), as correlações de rk e rs não foram calculadas pelo programa, rp(fst, cust eco) = 1 (p=0,333), rk(fst, cust eco) = 0,5774 (p=0,497), rs(fst, cust eco) = 1 (p=0,338), rp(fsthap5, cust eco) = 1 (p=0,325), rk(fsthap5, cust eco) = 0,5774 (p=0,501) e rs(fsthap5, cust eco) = 1 (p=0,335), rp (fst, eco cust) = 1 (p=0,498), as correlações de rk e rs não foram calculadas pelo programa, rp(fsthap5, eco cust) = 1 (p=0,505), as correlações de rk e rs não foram calculadas pelo programa (Tabela 8). O quarto conjunto (conj4) envolveu as populações Fueguian, Mapuche e Mapuche Chile. Nenhuma correlação foi significativa para este conjunto, os valores são: rp(eucl, cust) = 0,9967 (p=0,166), rp(fst, eucl) = -0,1248 (p=0,503), rp(fst, cust) = -0,2049 (p=0,497), rp(fst, eco) = -0,7355 (p=1), rk(fst, eco) = -0,8165 (p=1), rs(fst, eco) = -0,8660 (p=1), rp(fsthap5, eucl) = -0,1841 (p=0,501), rp(fsthap5, cust) = -0,2633 (p=0,502), rp (fsthap5, eco) = -0,6935 (p=1), rk(fsthap5, eco) = 0,8165 (p=1), rs(fsthap5, eco) = 0,8660 (p=1), rp(eucl, eco) = -0,5804 (p=1), rk(eucl, eco) = -0,8165 (p=1), rs(eucl, eco) = -0,8660 (p=1), rp(cust, eco) = -0,5124 (p=1), rk(cust, eco) = -0,8165 (p=1), rs(cust, eco) = -0,8660 (p=1). Para as correlações parciais de Mantel também não houve significância para
85 79 nenhuma correlação, rp(fst, eucl eco) = -1 (p=0,831), rk(fst, eucl eco) = -1 (p=1), rs(fst, eucl eco) = -1 (p=1), rp(fsthap5, eucl eco) = -1 (p=1), rk(fsthap5, eucl eco) = -1 (p=1) rs(fsthap5, eucl eco) = -1 (p=1), rp(fst, eco eucl) = -1 (p=0,832), as correlações de rk e rs não foram calculadas pelo programa, rp(fsthap5, eco eucl) = -1 (p=1), as correlações de rk e rs não foram calculadas pelo programa, rp(fst, cust eco) = -1 (p=0,826), rk(fst, cust eco) = -1 (p=1), rs(fst, cust eco) = -1 (p=1), rp(fsthap5, cust eco) = -1 (p=1), rk(fsthap5, cust eco) = -1 (p=1) e rs(fsthap5, cust eco) = -1 (p=1), rp(fst, eco cust) = -1 (p=0,676), as correlações de rk e rs não foram calculadas pelo programa, rp(fsthap5, eco cust) = -1 (p=0,833), as correlações de rk e rs não foram calculadas pelo programa (Tabela 9). Tabela 8: Valores das correlações para o conjunto 3 entre as matrizes e suas respectivas significâncias. Não houve nenhuma correlação significativa mantel Valor p eucl, cust (rp) fst, eucl (rp) fst, cust (rp) fst, eco (rp) fst, eco (rk) fst, eco (rs) fsthap5, eucl (rp) fsthap5, cust (rp) fsthap5, eco (rp) fsthap5, eco (rk) fsthap5, eco (rs) eucl, eco (rp) eucl, eco (rk) eucl, eco (rs) cust, eco (rp) cust, eco (rk) cust, eco (rs) parcial de mantel fst, eucl eco (rp) fst, eucl eco (rk) fst, eucl eco (rs) fst, eco eucl (rp) fsthap5, eucl eco (rp) fsthap5, eucl eco (rk) fsthap5, eucl eco (rs) fsthap5, eco eucl (rp) fst, cust eco (rp) fst, cust eco (rk)
86 80 Tabela 8 (continuação) mantel Valor p fst, cust eco (rs) 1 0 fst, eco cust (rp) 1 0 fsthap5, cust eco (rp) 1 0 fsthap5, cust eco (rk) 1 1 fsthap5, cust eco (rs) 1 0 fsthap5, eco cust (rp) 1 1 Tabela 9: Valores das correlações para conjunto 4 entre as matrizes e suas respectivas significâncias. Não houve nenhuma correlação significativa mantel Valor p eucl, cust (rp) fst, eucl (rp) fst, cust (rp) fst, eco (rp) fst, eco (rk) fst, eco (rs) fsthap5, eucl (rp) fsthap5, cust (rp) fsthap5, eco (rp) fsthap5, eco (rk) fsthap5, eco (rs) eucl, eco (rp) eucl, eco (rk) eucl, eco (rs) cust, eco (rp) cust, eco (rk) cust, eco (rs) parcial de mantel fst, eucl eco (rp) fst, eucl eco (rk) fst, eucl eco (rs) fst, eco eucl (rp) fsthap5, eucl eco (rp) fsthap5, eucl eco (rk) fsthap5, eucl eco (rs) fsthap5, eco eucl (rp) fst, cust eco (rp) fst, cust eco (rk) fst, cust eco (rs) fst, eco cust (rp) fsthap5, cust eco (rp) fsthap5, cust eco (rk) fsthap5, cust eco (rs) fsthap5, eco cust (rp)
87 81 O quinto conjunto (conj5) envolveu as populações Atacameno, Atacamenos e Aymara. Nenhuma correlação foi significativa para este conjunto, os valores são: rp(eucl, cust) = 0,8885 (p=0,337), rp(fst, eucl) = 0,9994 (p=0,167), rp(fst, cust) = 0,9040 (p=0,334), rp(fst, eco) = -0,4553 (p=0,667), rk(fst, eco) = 0 (p=0,662) e rs(fst, eco) = 0 (p=0,668), rp (fsthap5, eucl) = 0,9994 (p=0,162), rp(fsthap5, cust) = 0,9040 (p=0,329), rp(fsthap5, eco) = -0,4553 (p=0,673), rk(fsthap5, eco) = 0 (p=0,671), rs(fsthap5, eco) = 0 (p=0,670), rp(eucl, eco) = -0,4239 (p=0,668), rk(eucl, eco) = 0 (p=0,663), rs(eucl, eco) = 0 (p=0,662), rp(cust, eco) = -0,7923 (p=1), rk(cust, eco) = -0,8165 (p=1), rs(cust, eco) = -0,8660 (p=1). Para as correlações parciais de Mantel também não houve significância para nenhuma correlação, rp(fst, eucl eco) = 1 (p=0,324), rk(fst, eucl eco) = 1 (p=0,168), rs(fst, eucl eco) = 1 (p=0,496), rp(fsthap5, eucl eco) = 1 (p=0,333), rk(fsthap5, eucl eco) = 1 (p=0,168), rs (fsthap5, eucl eco) = 1 (p=0,496), rp(fst, eco eucl) = -1 (p=0,502), as correlações de rk e rs não foram calculadas pelo programa, rp(fsthap5, eco eucl) = -1 (p=0,499), as correlações de rk e rs não foram calculadas pelo programa, rp(fst, cust eco) = 1 (p=0,500), rk(fst, cust eco) = 0,5774 (p=0,507), rs(fst, cust eco) = 1 (p=0,336), rp(fsthap5, cust eco) = 1 (p=0,500), rk(fsthap5, cust eco) = 0,5774 (p=0,501) e rs(fsthap5, cust eco) = 1 (p=0,342), rp (fst, eco cust) = 1 (p=0,330), as correlações de rk e rs não foram calculadas pelo programa, rp(fsthap5, eco cust) = 1 (p=0,338), as correlações de rk e rs não foram calculadas pelo programa (Tabela 10). O sexto conjunto (conj6) envolveu as populações Movima, Murui-Muinane, Pilagá Formosa e Zoro. As correlações encontradas para este conjunto foram rp(eucl, cust) = 0,9987 (p=0,044), rp(fst, eucl) = -0,8844 (p=0,959), rp(fst, cust) = -0,8881 (p=0,959), rp(fst, eco) = -0,0779 (p=0,673), rk(fst, eco) = 0 (p=1) e rs(fst, eco) = 0 (p=1), rp(fsthap5, eucl) = -0,8908 (p=0,960), rp(fsthap5, cust) = -0,8938 (p=0,958), rp(fsthap5, eco) = -0,0649 (p=0,667), rk(fsthap5, eco) = 0 (p=1), rs(fsthap5, eco) = 0 (p=1), rp(eucl, eco) = 0,1297
88 82 (p=0,336), rk(eucl, eco) = 0 (p=1), rs(eucl, eco) = 0 (p=1), rp(cust, eco) = 0,1313 (p=0,332), rk(cust, eco) = 0 (p=1), rs(cust, eco) = 0 (p=1). Para as correlações parciais de Mantel também não houve significância para nenhuma correlação, rp(fst, eucl eco) = -0,8845 (p=0,956), rk(fst, eucl eco) = -0,7333 (p=0,961), rs(fst, eucl eco) = -0,8857 (p=0,956), rp (fsthap5, eucl eco) = -0,8918 (p=0,955), rk(fsthap5, eucl eco) = -0,7333 (p=0,960), rs (fsthap5, eucl eco) = -0,8857 (p=0,964), rp(fst, eco eucl) = 0,0796 (p=0,365), as correlações de rk e rs não foram calculadas pelo programa, rp(fsthap5, eco eucl) = 0,1124 (p=0,382), as correlações de rk e rs não foram calculadas pelo programa, rp(fst, cust eco) = -0,8883 (p=0,960), rk(fst, cust eco) = -0,7333 (p=0,962), rs(fst, cust eco) = -0,8857 (p=0,961), rp(fsthap5, cust eco) = 0,8949 (p=0,957), rk(fsthap5, cust eco) = -0,7333 (p=0,958) e rs(fsthap5, cust eco) = -0,8857 (p=0,959), rp(fst, eco cust) = 0,0851 (p=0,367), as correlações de rk e rs não foram calculadas pelo programa, rp(fsthap5, eco cust) = 0,1181 (p=0,373), as correlações de rk e rs não foram calculadas pelo programa (Tabela 11). Tabela 10: Valores das correlações para o conjunto 5 entre as matrizes e suas respectivas significâncias. Não houve nenhuma correlação significativa mantel Valor p eucl, cust (rp) fst, eucl (rp) fst, cust (rp) fst, eco (rp) fst, eco (rk) fst, eco (rs) fsthap5, eucl (rp) fsthap5, cust (rp) fsthap5, eco (rp) fsthap5, eco (rk) fsthap5, eco (rs) eucl, eco (rp) eucl, eco (rk) eucl, eco (rs) cust, eco (rp) cust, eco (rk) cust, eco (rs) 0,8885 0,9994 0,9040-0, ,9994 0,9040-0, , ,7923-0,8165-0,8660 0,337 0,167 0,334 0,667 0,662 0,668 0,162 0,329 0,673 0,671 0,670 0,668 0,663 0, parcial de mantel fst, eucl eco (rp) 1 0,324
89 83 Tabela 10 (continuação) mantel Valor p fst, eucl eco (rk) 1 0,168 fst, eucl eco (rs) 1 0,496 fst, eco eucl (rp) 1 0,333 fsthap5, eucl eco (rp) 1 0,168 fsthap5, eucl eco (rk) 1 0,502 fsthap5, eucl eco (rs) -1 0,502 fsthap5, eco eucl (rp) -1 0,499 fst, cust eco (rp) 1 0,500 fst, cust eco (rk) 0,5774 0,507 fst, cust eco (rs) 1 0,336 fst, eco cust (rp) 1 0,500 fsthap5, cust eco (rp) 0,5774 0,501 fsthap5, cust eco (rk) 1 0,342 fsthap5, cust eco (rs) 1 0,330 fsthap5, eco cust (rp) 1 0,338 Tabela 11: Valores das correlações para o conjunto 6 entre as matrizes e suas respectivas significâncias. Marcado em negrito estão as correlações significativas com p<0,05. mantel Valor p eucl, cust (rp) fst, eucl (rp) fst, cust (rp) fst, eco (rp) fst, eco (rk) fst, eco (rs) fsthap5, eucl (rp) fsthap5, cust (rp) fsthap5, eco (rp) fsthap5, eco (rk) fsthap5, eco (rs) eucl, eco (rp) eucl, eco (rk) eucl, eco (rs) cust, eco (rp) cust, eco (rk) cust, eco (rs) 0,9987-0,8844-0,8881-0, ,8908-0,8938-0, , , ,044 0,959 0,959 0, ,960 0,958 0, , , parcial de mantel fst, eucl eco (rp) fst, eucl eco (rk) fst, eucl eco (rs) fst, eco eucl (rp) fsthap5, eucl eco (rp) fsthap5, eucl eco (rk) -0,8845 0,956-0,7333 0,961-0,8857 0,956-0,8918 0,955-0,7333 0,960-0,8857 0,964
90 84 Tabela 11 (continuação) mantel Valor p fsthap5, eucl eco (rs) 0,0796 0,365 fsthap5, eco eucl (rp) 0,1124 0,382 fst, cust eco (rp) -0,8883 0,960 fst, cust eco (rk) -0,7333 0,962 fst, cust eco (rs) -0,8857 0,961 fst, eco cust (rp) -0,8949 0,957 fsthap5, cust eco (rp) -0,7333 0,958 fsthap5, cust eco (rk) -0,8857 0,959 fsthap5, cust eco (rs) 0,0851 0,367 fsthap5, eco cust (rp) 0,1181 0, América global Para este conjunto de dados foram usadas todas as populações do banco de dados final com número de indivíduos em cada população maior que dez, num total de 209 populações (Apêndice XXXVIII). As correlações entre duas matrizes para este conjunto foram todas significativas. Os valores encontrados para este conjunto foram rp(eucl, cust) = 0,9391 (p=0,000), rp(fst, eucl) = 0,2462 (p=0,000), rp(fst, cust) = 0,2801 (p=0,000), rp(fst, eco) = 0,0992 (p=0,000), rk não foi calculado pelo programa e rs(fst, eco) = 0,1046 (p=0,000), rp(fsthap5, eucl) = 0,2336 (p=0,000), rp(fsthap5, cust) = 0,2738 (p=0,000), rp(fsthap5, eco) = 0,0931 (p=0,000), rk não foi calculado pelo programa, rs(fsthap5, eco) = 0,1021 (p=0,000), rp(eucl, eco) = 0,3624 (p=0,000), rk não foi calculado pelo programa, rs(eucl, eco) = 0,4088 (p=0,000), rp(cust, eco) = 0,3477 (p=0,000), rk não foi calculado pelo programa, rs(cust, eco) = 0,4141 (p=0,000). Para as correlações parciais de Mantel não houve significância para as correlações com fisiografia corrigidas para distância geográfica, rp(fst, eucl eco) = 0,2267 (p=0,000), rk não foi calculado pelo programa, rs(fst, eucl eco) = 0,2084 (p=0,000), rp(fsthap5, eucl eco) = 0,2154 (p=0,000), rk não foi calculado pelo programa, rs(fsthap5, eucl eco) = 0,1994 (p=0,000), rp(fst, eco eucl) = 0,0110 (p=0,292), rk não foi calculado pelo
91 85 programa, rs(fst, eco eucl) = 0,0110 (p=0,267), rs(fsthap5, eco eucl) = 0,0930 (p=0,327), rk não foi calculado pelo programa, rs(fsthap5, eco eucl) 0,0123 (p=246), rp(fst, cust eco) = 0,2633 (p=0,000), rk não foi calculado pelo programa, rs(fst, cust eco) = 0,2154 (p=0,000), rp(fsthap5, cust eco) = 0,2586 (p=0,000), rk não foi calculado pelo programa e rs(fsthap5, cust eco) = 0,2093 (p=0,000), rp(fst, eco cust) = 0,0020 (p=0,457), rk não foi calculado pelo programa, rs(fst, eco cust) = 0,0053 (p=0,3812), rp(fsthap5, eco cust) = - 0,0024 (p=0,534), rk não foi calculado pelo programa, rs(fsthap5, eco cust) = 0,0055 (p=0,384) (Tabela 12). Tabela 12: Valores das correlações para n10 entre as matrizes e suas respectivas significâncias. Marcado em negrito estão as correlações significativas com p<0,05. mantel Valor p eucl, cust (rp) fst, eucl (rp) fst, cust (rp) fst, eco (rp) fst, eco (rs) fsthap5, eucl (rp) fsthap5, cust (rp) fsthap5, eco (rp) fsthap5, eco (rs) eucl, eco (rp) eucl, eco (rs) cust, eco (rp) cust, eco (rs) 0,9391 0,2462 0,2801 0,0992 0,1046 0,2336 0,2738 0,0931 0,1021 0,3624 0,4088 0,3477 0,4141 0, ,0001 0,0001 0,0002 0,0001 0,0001 0,0001 0,0001 0,0001 0,0001 0,0001 0,0001 0,0001 parcial de mantel fst, eucl eco (rp) fst, eucl eco (rs) fsthap5, eucl eco (rp) fsthap5, eucl eco (rs) fst, eco eucl (rp) fst, eco eucl (rs) fsthap5, eco eucl (rp) fsthap5, eco eucl (rs) fst, cust eco (rp) fst, cust eco (rs) fsthap5, cust eco (rp) fsthap5, cust eco (rs) fst, eco cust (rp) fst, eco cust (rs) fsthap5, eco cust (rp) fsthap5, eco cust (rs) 0,2267 0,0001 0,2084 0,0001 0,2154 0,0001 0,1994 0,0001 0,0110 0,2924 0,0110 0,2671 0,093 0,3267 0,0123 0,2461 0,2633 0,0001 0,2154 0,0001 0,2586 0,0001 0,2093 0,0001 0,0020 0,4566 0,0053 0,3812-0,0024 0,5338 0,0055 0,3844
92 América do Norte Para esta subcategoria foram usadas 73 populações localizadas na América do Norte (Figura 16). As correlações entre duas matrizes para estas populações foram todas significativas. Os valores encontrados para este conjunto foram rp(eucl, cust) = 0,9837 (p=0,000), rp(fst, eucl) = 0,2008 (p=0,000), rp(fst, cust) = 0,2107 (p=0,000), rp(fst, eco) = 0,1299 (p=0,000), rk não foi calculado pelo programa e rs(fst, eco) = 0,1327 (p=0,000), rp (fsthap5, eucl) = 0,1950 (p=0,000), rp(fsthap5, cust) = 0,1993 (p=0,000), rp(fsthap5, eco) = 0,1244 (p=0,000), rk não foi calculado pelo programa, rs(fsthap5, eco) = 0,1320 (p=0,000), rp(eucl, eco) = 0,4139 (p=0,000), rk não foi calculado pelo programa, rs(eucl, eco) = 0,4542 (p=0,000), rp(cust, eco) = 0,4133 (p=0,000), rk não foi calculado pelo programa, rs(cust, eco) = 0,4444 (p=0,000). Para as correlações parciais de Mantel não houve significância apenas para algumas correlações calculadas pelo método de Spearman, rp(fst, eucl eco) = 0,1629 (p=0,000), rk não foi calculado pelo programa, rs(fst, eucl eco) = 0,1908 (p=0,000), rp(fsthap5, eucl eco) = 0,1589 (p=0,000), rk não foi calculado pelo programa rs (fsthap5, eucl eco) = 0,1934 (p=0,000), rp(fst, eco eucl) = 0,0525 (p=0,036), rk não foi calculado pelo programa, rs(fst, eco eucl) = 0,0332(p=0,106), rp(fsthap5, eco eucl) = 0,0489 (p=0,046), rk não foi calculado pelo programa, rs(fsthap5, eco eucl) = 0,0314 (p=0,116), rp(fst, cust eco) = 0,1739 (p=0,000), K não foi calculado pelo programa, rs(fst, cust eco) = 0,1976 (p=0,000), rp(fsthap5, cust eco) = 0,1637 (p=0,000), rk não foi calculado pelo programa e rs(fsthap5, cust eco) = 0,1947 (p=0,000), rp(fst, eco cust) = 0,0481 (p=0,047), rk não foi calculado pelo programa, rs(fst, eco cust) = 0,0328 (p=0,109), rp (fsthap5, eco cust) = 0,0471 (p=0,053), rk não foi calculado pelo programa, rs(fsthap5, eco cust) = 0,0334 (p=0,108) (Tabela 13).
93 América do Sul Para esta subcategoria foram usadas 123 populações localizadas na América do Sul e porção sul da América Central (biom74). Tabela 13: Valores das correlações para biom63 entre as matrizes e suas respectivas significâncias. Marcado em amarelo estão as correlações significativas com p<0,05. mantel Valor p eucl, cust (rp) fst, eucl (rp) fst, cust (rp) fst, eco (rp) fst, eco (rs) fsthap5, eucl (rp) fsthap5, cust (rp) fsthap5, eco (rp) fsthap5, eco (rs) eucl, eco (rp) eucl, eco (rs) cust, eco (rp) cust, eco (rs) 0,9837 0,2008 0,2107 0,1299 0,1327 0,1950 0,1993 0,1244 0,1320 0,4139 0,4542 0,4133 0,4444 0,0001 0,0001 0,0001 0,0001 0,0002 0,0001 0,0001 0,0002 0,0001 0,0001 0,0001 0,0001 0,0001 parcial de mantel fst, eucl eco (rp) fst, eucl eco (rs) fsthap5, eucl eco (rp) fsthap5, eucl eco (rs) fst, eco eucl (rp) fst, eco eucl (rs) fsthap5, eco eucl (rp) fsthap5, eco eucl (rs) fst, cust eco (rp) fst, cust eco (rs) fsthap5, cust eco (rp) fsthap5, cust eco (rs) fst, eco cust (rp) fst, eco cust (rs) fsthap5, eco cust (rp) fsthap5, eco cust (rs) 0,1629 0,0001 0,1908 0,0001 0,1589 0,0003 0,1934 0,0001 0,0525 0,0365 0,0332 0,106 0,0489 0,0465 0,0314 0,1159 0,1739 0,0001 0,1976 0,0001 0,1637 0,0002 0,1947 0,0001 0,0481 0,0474 0,0328 0,1093 0,0471 0,0528 0,0334 0,1085
94 88 As correlações entre duas matrizes para estas populações não foram significativas quando comparadas com distância fisiográfica. Os valores encontrados para este conjunto foram rp(eucl, cust) = 0,9175 (p=0,000), rp(fst, eucl) = 0,2027 (p=0,000), rp(fst, cust) = 0,1845 (p=0,000), rp(fst, eco) = 0,0212 (p=0,337), rk não foi calculado pelo programa e rs(fst, eco) = 0,0367 (p=0,202), rp(fsthap5, eucl) = 0,2007 (p=0,000), rp (fsthap5, cust) = 0,1859 (p=0,000), rp(fsthap5, eco) = 0,0133 (p=0,383), rk não foi calculado pelo programa, rs(fsthap5, eco) = 0,0320 (p=0,231), rp(eucl, eco) = 0,3751 (p=0,000), rk não foi calculado pelo programa, rs(eucl, eco) = 0,3703 (p=0,000), rp(cust, eco) = 0,4621 (p=0,000), rk não foi calculado pelo programa, rs(cust, eco) = 0,4707 (p=0,000). Para as correlações parciais de Mantel não houve significância para as correlações com fisiografia corrigidas para distância geográfica, rp(fst, eucl eco) = 0,2101 (p=0,000), rk não foi calculado pelo programa, rs(fst, eucl eco) = 0,2197 (p=0,000), rp (fsthap5, eucl eco) = -0,0604 (p=0,901), rk não foi calculado pelo programa rs(fsthap5, eucl eco) = -0,0483 (p=0,875), rp(fst, eco eucl) = 0,2111 (p=0,000), rk não foi calculado pelo programa, rs(fst, eco eucl) = 0,2217(p=0,000), rp(fsthap5, eco eucl) = -0,0682 (p=0,927), rk não foi calculado pelo programa, rs(fsthap5, eco eucl) = -0,0537 (p=0,896), rp (fst, cust eco) = 0,1971 (p=0,000), rk não foi calculado pelo programa, rs(fst, cust eco) = 0,1935 (p=0,000), rp(fsthap5, cust eco) = 0,2027 (p=0,000), rk não foi calculado pelo programa e rs(fsthap5, cust eco) = 0,1990 (p=0,000), rp(fst, eco cust) = -0,0735 (p=0,947), rk não foi calculado pelo programa, rs(fst, eco cust) = -0,0596 (p=0,932), rp(fsthap5, eco cust) = -0,0833 (p=0,967), rk não foi calculado pelo programa, rs(fsthap5, eco cust) = -0,0666 (p=0,950) (Tabela 14)
95 89 Tabela 14: Valores das correlações para biom74 entre as matrizes e suas respectivas significâncias. Marcado em negrito estão as correlações significativas com p<0,05. mantel Valor p eucl, cust (rp) fst, eucl (rp) fst, cust (rp) fst, eco (rp) fst, eco (rs) fsthap5, eucl (rp) fsthap5, cust (rp) fsthap5, eco (rp) fsthap5, eco (rs) eucl, eco (rp) eucl, eco (rs) cust, eco (rp) cust, eco (rs) 0,9175 0,2027 0,1845 0,0212 0,0367 0,2007 0,1859 0,0133 0,0320 0,3751 0,3703 0,4621 0,4707 0,0001 0,0001 0,0001 0,337 0,202 0,0001 0,0001 0,383 0,231 0,0001 0,0001 0,0001 0,0001 parcial de mantel fst, eucl eco (rp) fst, eucl eco (rs) fst, eco eucl (rp) fst, eco eucl (rs) fsthap5, eucl eco (rp) fsthap5, eucl eco (rs) fsthap5, eco eucl (rp) fsthap5, eco eucl (rs) fst, cust eco (rp) fst, cust eco (rs) fsthap5, cust eco (rp) fsthap5, cust eco (rs) fst, eco cust (rp) fst, eco cust (rs) fsthap5, eco cust (rp) fsthap5, eco cust (rs) 0,2101 0,0001 0,2197 0,0001-0,0604 0,901-0,0483 0,875 0,2111 0,0001 0,2217 0,0001-0,0682 0,927-0,0537 0,896 0,1971 0,0001 0,1935 0,0001 0,2027 0,0001 0,1990 0,0001-0,0735 0,947-0,0596 0,932-0,0833 0,967-0,0666 0, Patagônia Para esta subcategoria foram usadas 17 populações localizadas na América do Sul na região da Patagônia (Figura 14). Para estas populações foi calculada a matriz fisiográfica binária e com escores cumulativos.
96 90 As correlações entre duas matrizes para estas populações não foram significativas quando comparadas com distância fisiográfica. Os valores encontrados para este conjunto foram rp(eucl, cust) = 0,9968 (p=0,000), rp(fst, eucl) = 0,2930 (p=0,014), rp(fst, cust) = 0,3164 (p=0,010), rp(fst, eco) = 0,0108 (p=0,406), rk(fst, eco) = 0,0142 (p=0,369) e rs(fst, eco) = 0,0174 (p=0,378), rp(fst, eco2) = -0,0041 (p=0,459), rp(fsthap5, eucl) = 0,2668 (p=0,028), rp(fsthap5, cust) = 0,2918 (p=0,019), rp(fsthap5, eco) = 0,0083 (p=0,422), rk (fsthap5, eco) = 0,0102 (p=0,384), rs(fsthap5, eco) = 0,0124 (p=0,383), rp(fsthap5, eco2) = -0,0056 (p=0,475), rp(eucl, eco) = -0,0502 (p=0,733), rk(eucl, eco) = -0,0186 (p=0,531), rs (eucl, eco) = -0,0226 (p=0,530), rp(cust, eco) = -0,0517 (p=0,742), rk(cust, eco) = -0,0210 (p=0,535), rs(cust, eco) = -0,0256 (p=0,532), rp(eucl, eco2) = -0,0923 (p=0,793) e rp(cust, eco2) = -0,1014 (p=0,815). Para as correlações parciais de Mantel não houve significância para as correlações com fisiografia corrigidas para distância geográfica, rp (fst, eucl eco) = 0,2939 (p=0,012), rk(fst, eucl eco) = 0,1950 (p=0,009), rs(fst, eucl eco) = 0,2881 (p=0,009), rp(fsthap5, eucl eco) = 0,2675 (p=0,024), rk(fsthap5, eucl eco) = 0,1760 (p=0,017), rs(fsthap5, eucl eco) = 0,2666 (p=0,014), rp(fst, eco eucl) = 0,0027 (p=0,359), rk (fst, eco eucl) = 0,0182 (p=0,359), rs(fst, eco eucl) = 0,0249 (p=0,346), rp(fsthap5, eco eucl) = 0,0226 (p=0,362), rk(fsthap5, eco eucl) = 0,0137 (p=0,374), rs(fsthap5, eco eucl) = 0,0192 (p=0,359), rp(fst, cust eco) = 0,3174 (p=0,008), rk(fst, cust eco) = 0,2094 (p=0,006), rs(fst, cust eco) = 0,3041 (p=0,007), rp(fsthap5, cust eco) = 0,2927 (p=0,017), rk(fsthap5, cust eco) = 0,1916 (p=0,010), rs(fsthap5, cust eco) = 0,2846 (p=0,010), rp(fst, eco cust) = 0,0287 (p=0,343), rk(fst, eco cust) = 0,0190 (p=0,343), rs(fst, eco cust) = 0,0264 (p=0,340), rp(fsthap5, eco cust) = 0,0245 (p=0,363), rk(fsthap5, eco cust) = 0,0145 (p=0,369), rs (fsthap5, eco cust) = 0,0206 (p=0,359), rp(fst, eucl eco2) = 0,2939 (p=0,015), rp(fsthap5, eucl eco2) = 0,2674 (p=0,024), rp(fst, eco2 eucl) = 0,0241 (p=0,375), rp(fsthap5, eco2 eucl) = 0,0196 (p=0,384), rp(fst, cust eco2) = 0,3177 (p=0,011), rp(fsthap5, cust eco2) = 0,2928
97 (p=0,013), rp(fst, eco2 cust) = 0,0297 (p=0,358) e rp(fsthap5, eco2 cust) = 0,0250 (p=0,367) (Tabela 15). 91 Tabela 15: Valores das correlações para biom31 entre as matrizes e suas respectivas significâncias. Marcado em negrito estão as correlações significativas com p<0,05. eucl, cust (rp) fst, eucl (rp) fst, cust (rp) fst, eco (rp) fst, eco (rk) fst, eco (rs) fst, eco2 (rp) fsthap5, eucl (rp) fsthap5, cust (rp) fsthap5, eco (rp) fsthap5, eco (rk) fsthap5, eco (rs) fsthap5, eco2 (rp) eucl, eco (rp) eucl, eco (rk) eucl, eco (rs) cust, eco (rp) cust, eco (rk) cust, eco (rs) eucl, eco2 (rp) cust, eco2 (rp) mantel Valor p 0,9968 0,0001 0,2930 0,014 0,3164 0,010 0,0108 0,406 0,0142 0,369 0,0174 0,378-0,0041 0,459 0,2668 0,028 0,2918 0,019 0,0083 0,422 0,0102 0,384 0,0124 0,383-0,0056 0,475-0,0502 0,733-0,0186 0,531-0,0226 0,530-0,0517 0,742-0,0210 0,535-0,0256 0,532-0,0923 0,793-0,1014 0,815 parcial de mantel fst, eucl eco (rp) fst, eucl eco (rk) fst, eucl eco (rs) fsthap5, eucl eco (rp) fsthap5, eucl eco (rk) fsthap5, eucl eco (rs) fst, eco eucl (rp) fst, eco eucl (rk) fst, eco eucl (rs) fsthap5, eco eucl (rp) fsthap5, eco eucl (rk) fsthap5, eco eucl (rs) fst, cust eco (rp) fst, cust eco (rk) fst, cust eco (rs) fsthap5, cust eco (rp) fsthap5, cust eco (rk) fsthap5, cust eco (rs) fst, eco cust (rp) fst, eco cust (rk) fst, eco cust (rs) 0,2939 0,012 0,1950 0,009 0,2881 0,009 0,2675 0,024 0,1760 0,017 0,2666 0,014 0,0027 0,359 0,0182 0,359 0,0249 0,346 0,0226 0,362 0,0137 0,374 0,0192 0,359 0,3174 0,008 0,2094 0,006 0,3041 0,007 0,2927 0,017 0,1916 0,010 0,2846 0,010 0,0287 0,343 0,0190 0,343 0,0264 0,340
98 92 Tabela 15 (continuação) mantel Valor p fsthap5, eco cust (rp) fsthap5, eco cust (rk) fsthap5, eco cust (rs) fst, eucl eco2 (rp) fsthap5, eucl, eco2 (rp) fst, eco2 eucl (rp) fsthap5, eco2 eucl (rp) fst, cust eco2 (rp) fsthap5, cust eco2 (rp) fst, eco2 cust (rp) fsthap5, eco2 cust (rp) 0,0245 0,363 0,0145 0,369 0,0206 0,359 0,2939 0,015 0,2674 0,024 0,0241 0,375 0,0196 0,384 0,3177 0,011 0,2928 0,013 0,0297 0,358 0,0250 0, Região centro norte da América do Sul Para esta subcategoria foram usadas 106 populações localizadas na América do Sul (Figura 15). As correlações entre duas matrizes para estas populações não foram significativas quando comparadas a distância genética e distância fisiográfica. Os valores encontrados para este conjunto foram rp(eucl, cust) = 0,8322 (p=0,0001), rp(fst, eucl) = 0,1856 (p=0,000), rp(fst, cust) = 0,1243 (p=0,004), rp(fst, eco) = 0,0136 (p=0,384), rk não foi calculado pelo programa e rs(fst, eco) = 0,0260 (p=0,286), rp(fsthap5, eucl) = 0,1900 (p=0,000), rp(fsthap5, cust) = 0,1321 (p=0,003), rp(fsthap5, eco) = 0,0047 (p=0,454), rk não foi calculado pelo programa, rs(fsthap5, eco) = 0,0211 (p=0,327), rp(eucl, eco) = 0,1817 (p=0,000), rk não foi calculado pelo programa, rs(eucl, eco) = 0,1796 (p=0,000), rp(cust, eco) = 0,3494 (p=0,000), rk não foi calculado pelo programa, rs(cust, eco) = 0,3404 (p=0,000). Para as correlações parciais de Mantel não houve significância para as correlações com fisiografia corrigidas para distância geográfica, rp(fst, eucl eco) = 0,1862 (p=0,000), rk não foi calculado pelo programa, rs(fst, eucl eco) = 0,2060 (p=0,000), rp (fsthap5, eucl eco) = 0,1923 (p=0,000), rk não foi calculado pelo programa rs(fsthap5, eucl
99 93 eco) = 0,2115 (p=0,000), rp(fst, eco eucl) = -0,0208 (p=0,633), rk não foi calculado pelo programa, rs(fst, eco eucl) = -0,0116 (p=0,579), rp(fsthap5, eco eucl) = -0,0309 (p=0,708), rk não foi calculado pelo programa, rs(fsthap5, eco eucl) = -0,0177 (p=0,624), rp(fst, cust eco) = 0,1276 (p=0,002), rk não foi calculado pelo programa, rs(fst, cust eco) = 0,1396 (p=0,000), rp(fsthap5, cust eco) = 0,1392 (p=0,000), rk não foi calculado pelo programa e rs (fsthap5, cust eco) = 0,1490 (p=0,000), rp(fst, eco cust) = -0,0321 (p=0,723), rk não foi calculado pelo programa, rs(fst, eco cust) = -0,0232 (p=0,684), rp(fsthap5, eco cust) = -0,0446 (p=0,797), rk não foi calculado pelo programa, rs(fsthap5, eco cust) = -0,0313 (p=0,738) (Tabela 16). Tabela 16: Valores das correlações para biom42 entre as matrizes e suas respectivas significâncias. Marcado em negrito estão as correlações significativas com p<0,05. mantel Valor p eucl, cust (rp) fst, eucl (rp) fst, cust (rp) fst, eco (rp) fst, eco (rs) fsthap5, eucl (rp) fsthap5, cust (rp) fsthap5, eco (rp) fsthap5, eco (rs) eucl, eco (rp) eucl, eco (rs) cust, eco (rp) cust, eco (rs) 0,8322 0,1856 0,1243 0,0136 0,0260 0,1900 0,1321 0,0047 0,0211 0,1817 0,1796 0,3494 0,3404 0,0001 0,0001 0,004 0,384 0,286 0,0001 0,003 0,454 0,327 0,0002 0,0001 0,0001 0,0001 parcial de mantel fst, eucl eco (rp) fst, eucl eco (rs) fsthap5, eucl eco (rp) fsthap5, eucl eco (rs) fst, eco eucl (rp) fst, eco eucl (rs) fsthap5, eco eucl (rp) fsthap5, eco eucl (rs) fst, cust eco (rp) fst, cust eco (rs) fsthap5, cust eco (rp) fsthap5, cust eco (rs) fst, eco cust (rp) 0,1862 0,0001 0,2060 0,0001 0,1923 0,0001 0,2115 0,0001-0,0208 0,633-0,0116 0,579-0,0309 0,708-0,0177 0,624 0,1276 0,002 0,1396 0,0002 0,1392 0,0004 0,1490 0,0002-0,0321 0,723
100 Tabela 16 (continuação) 94 mantel Valor p fst, eco cust (rs) -0,0232 0,684 fsthap5, eco cust (rp) -0,0446 0,797 fsthap5, eco cust (rs) -0,0313 0, Microssatélites Para estas populações foram calculadas a matriz fisiográfica binária e com escores cumulativos, além terem sido também calculadas a distâncias genéticas por rst América global Para esta subcategoria foram usadas as 29 populações localizadas na América (Figura 8). A correlação entre as duas matrizes de distância genética rp(fst, rst) = 0,9599 (p=0,000), correlação alta e significativa. As correlações entre distância genética e geográfica e distância genética e fisiográfica não geraram valores significativos. Os valores encontrados para este conjunto foram rp(eucl, cust) = 0,9968 (p=0,000), rp(fst, eucl) = -0,0086 (p=0,502), rp(fst, cust) = -0,0488 (p=0,614), rp(fst, eco) = -0,3234 (p=0,995), rk(fst, eco) = -0,2560 (p=0,994) e rs(fst, eco) = -0,3132 (p=0,994), rp(fst, eco2) = -0,0671 (p=0,637), rp(rst, eucl) = 0,0410 (p=0,360), rp(rst, cust) = 0,0127 (p=0,425), rp(rst, eco) = -0,2689 (p=0,985), rk(rst, eco) = -0,2210 (p=0,983), rs(rst, eco) = -0,2704 (p=0,986), rp(rst, eco2) = -0,0202 (p=0,507), rp (eucl, eco) = 0,4782 (p=0,000), rk(eucl, eco) = 0,4113 (p=0,000), rs(eucl, eco) = 0,5029 (p=0,000), rp(cust, eco) = 0,5103 (p=0,000), rk(cust, eco) = 0,4440 (p=0,000), rs(cust, eco) = 0,5430 (p=0,000), rp(eucl, eco2) = 0,8292 (p=0,000) e rp(cust, eco2) = 0,8288 (p=0,000). Para as correlações parciais de Mantel apenas as correlações entre rst e distância geográfica corrigidas para fisiografia foram significativas. Os valores encontrados foram: rp (fst, eucl eco) = 0,1757 (p=0,100), rk(fst, eucl eco) = 0,1026 (p=0,093), rs(fst, eucl eco) =
101 95 0,1606 (p=0,090), rp(rst, eucl eco) = 0,2005 (p=0,070), rk(rst, eucl eco) = 0,1348 (p=0,038), rs(rst, eucl eco) = 0,2105 (p=0,039), rp(fst, eco eucl) = -0,3635 (p=0,999), rk(fst, eco eucl) = -0,2742 (p=0,999), rs(fst, eco eucl) = -0,3475 (p=0,999), rp(rst, eco eucl) = -0,3288 (p=0,999), rk(rst, eco eucl) = -0,2556 (p=0,997), rs(rst, eco eucl) = -0,3359 (p=0,999), rp(fst, cust eco) = 0,1429 (p=0,131), rk(fst, cust eco) = 0,0881 (p=0,115), rs(fst, cust eco) = 0,1317 (p=0,121), rp(rst, cust eco) = 0,1810 (p=0,086), rk(rst, cust eco) = 0,1253 (p=0,049), rs(rst, cust eco) = 0,1971 (p=0,050), rp(fst, eco cust) = -0,3475 (p=0,999), rk(fst, eco cust) = -0,2674 (p=0,998), rs(fst, eco cust) = -0,3343 (p=0,999), rp(rst, eco cust) = -0,3203 (p=0,998), rk(rst, eco cust) =-0,2523 (p=0,997), rs(rst, eco cust) = -0,3301 (p=0,999), rp(fst, eucl eco2) = 0,0843 (p=0,200), rp(rst, eucl eco2) = 0,1034 (p=0,150), rp(fst, eco2 eucl) = -0,1072 (p=0,778), rp(rst, eco2 eucl) = -0,0971 (p=0,758), rp(fst, cust eco2) = 0,0122 (p=0,449), rp(rst, cust eco2) = 0,0526 (p=0,294), rp(fst, eco2 cust) = -0,0477 (p=0,614) e rp (rst, eco2 cust) = -0,0549 (p=0,639) (Tabela 17). Tabela 17: Valores das correlações para micro entre as matrizes e suas respectivas significâncias. Marcado em negrito estão as correlações significativas com p<0,05. fst, rst (rp) eucl, cust (rp) fst, eucl (rp) fst, cust (rp) fst, eco (rp) fst, eco (rk) fst, eco (rs) fst, eco2 (rp) rst, eucl (rp) rst, cust (rp) rst, eco (rp) rst, eco (rk) rst, eco (rs) rst, eco2 (rp) eucl, eco (rp) eucl, eco (rk) eucl, eco (rs) cust, eco (rp) cust, eco (rk) cust, eco (rs) eucl, eco2 (rp) cust, eco2 (rp) mantel Valor p 0,9599 0,0001 0,9810 0,0001-0,0086 0,502-0,0488 0,614-0,3234 0,995-0,2560 0,994-0,3132 0,994-0,0671 0,637 0,0410 0,360 0,0127 0,425-0,2689 0,985-0,2210 0,983-0,2704 0,986-0,0202 0,507 0,4782 0,0001 0,4113 0,0001 0,5029 0,0001 0,5103 0,0001 0,4440 0,0001 0,5430 0,0001 0,8292 0,0001 0,8288 0,0001
102 96 Tabela 19 (continuação) mantel Valor p parcial de mantel fst, eucl eco (rp) fst, eucl eco (rk) fst, eucl eco (rs) rst, eucl eco (rp) rst, eucl eco (rk) rst, eucl eco (rs) fst, eco eucl (rp) fst, eco eucl (rk) fst/eco/eucl spe rst, eco eucl (rp) rst, eco eucl (rk) rst, eco eucl (rs) fst, cust eco (rp) fst, cust eco (rk) fst, cust eco (rs) rst, cust eco (rp) rst, cust eco (rk) rst, cust eco (rs) fst, eco cust (rp) fst, eco cust (rk) fst, eco cust (rs) rst, eco cust (rp) rst, eco cust (rk) rst, eco cust (rs) fst, eucl eco2 (rp) rst, eucl eco2 (rp) fst, eco2 eucl (rp) rst, eco2 eucl (rp) fst, cust eco2 (rp) rst, cust eco2 (rp) fst, eco2 cust (rp) rst, eco2 cust (rp) 0,1757 0,100 0,1026 0,093 0,1606 0,090 0,2005 0,070 0,1348 0,038 0,2105 0,039-0,3635 0,999-0,2742 0,999-0,3475 0,999-0,3288 0,999-0,2556 0,997-0,3359 0,999 0,1429 0,131 0,0881 0,115 0,1317 0,121 0,1810 0,086 0,1253 0,049 0,1971 0,050-0,3475 0,999-0,2674 0,998-0,3343 0,999-0,3203 0,998-0,2523 0,997-0,3301 0,999 0,0843 0,200 0,1034 0,150-0,1072 0,778-0,0971 0,758 0,0122 0,449 0,0526 0,294-0,0477 0,614-0,0549 0, América do Norte Para esta subcategoria foram usadas as 3 populações localizadas na América do Norte ((micron) Figura 10). Não houve significância para nenhuma correlação. Devido à redundância da matriz fisiográfica binária o programa não gerou valores de correlação para as comparações que envolvessem esta matriz.
103 97 Os valores encontrados para este conjunto foram rp(fst, rst) = 0,9080 (p=0,168), rp (eucl, cust) = 0,9997 (p=0,166), rp(fst, eucl) = -0,0058 (p=0,503), rp(fst, cust) = 0,0204 (p=0,507), rp(fst, eco2) = 0,9936 (p=0,334), rp(rst, eucl) = 0,4136 (p=0,500), rp(rst, cust) = 0,4373 (p=0,500), rp(rst, eco2) = 0,8550 (p=0,332), rp(eucl, eco2) = -0,1186 (p=0,664) e rp (cust, eco2) = -0,0924 (p=0,673). Para as correlações parciais de Mantel os valores encontrados foram: rp(fst, eucl eco2) = 1 (p=0,165), rp(rst, eucl eco2) = 1 (p=0,342), rp(fst, eco2 eucl) = 1 (p=0,166), rp(rst, eco2 eucl) = 1 (p=0,510), rp(fst, cust eco2) = 1 (p=0,165), rp(rst, cust eco2) = 1 (p=0,334), rp(fst, eco2 cust) = 1 (p=0,338) e rp(rst, eco2 cust) = 1 (p=0,333) (Tabela 18). Tabela 18: Valores das correlações para micron entre as matrizes e suas respectivas significâncias. Não houve nenhuma correlação significativa fst, rst (rp) eucl, cust (rp) fst, eucl (rp) fst, cust (rp) fst, eco2 (rp) rst, eucl (rp) rst, cust (rp) rst, eco2 (rp) eucl, eco2 (rp) cust, eco2 (rp) mantel Valor p 0,9080 0,168 0,9997 0,1662-0,0058 0,503 0,0204 0,507 0,9936 0,334 0,4136 0,500 0,4373 0,500 0,8550 0,332-0,1186 0,664-0,0924 0,673 parcial de mantel fst, eucl eco2 (rp) rst, eucl eco2 (rp) fst, eco2 eucl (rp) rst, eco2 eucl (rp) fst, cust eco2 (rp) rst, cust eco2 (rp) fst, eco2 cust (rp) rst, eco2 cust (rp) 1 0, , , , , , , ,333
104 América Central Para esta subcategoria foram usadas as 8 populações localizadas na América Central ((microc) Figura 10). A correlação entre as duas matrizes de distância genética rp (fst, rst) = 0,9740 (p=0,000) foi alta e extremamente significativa. As correlações entre distância genética e geográfica geraram valores significativos de correlação e entre distância genética e fisiográfica apenas quando comparadas com eco2 para fst e eco rk e eco rs para rst. Os valores encontrados para este conjunto foram rp(eucl, cust) = 0,9877 (p=0,000), rp(fst, eucl) = 0,8466 (p=0,000), rp(fst, cust) = 0,8073 (p=0,000), rp(fst, eco) = P 0,2220 (p=0,105), rk(fst, eco) = 0,1363 (p=0,178) e rs(fst, eco) = 0,1640 (p=0,180), rp(fst, eco2) = 0,1169 (p=0,027), rp(rst, eucl) = 0,8577 (p=0,000), rp(rst, cust) = 0,8163 (p=0,000), rp(rst, eco) = 0,2573 (p=0,072), rk(rst, eco) = 0,2026 (p=0,036), rs (rst, eco) = 0,2438 (p=0,038), rp(rst, eco2) = 0,1431 (p=0,055), rp(eucl, eco) = 0,3270 (p=0,049), rk(eucl, eco) = 0,2626 (p=0,055), rs(eucl, eco) = 0,3148 (p=0,055), rp(cust, eco) = 0,2760 (p=0,092), rk(cust, eco) = 0,1952 (p=0,087), rs(cust, eco) = 0,2349 (p=0,094), rp (eucl, eco2) = 0,3615 (p=0,028) e rp(cust, eco2) = 0,3718 (p=0,033). Para as correlações parciais de Mantel apenas as correlações entre distância genética e geográfica corrigida para fisiografia foram significativas. Os valores encontrados foram: rp(fst, eucl eco) = 0,8400 (p=0,000), rk(fst, eucl eco) = 0,5822 (p=0,001), rs(fst, eucl eco) = 0,7601 (p=0,001), rp(rst, eucl eco) = 0,8470 (p=0,000), rk(rst, eucl eco) = 0,5818 (p=0,001), rs(rst, eucl eco) = 0,7949 (p=0,001), rp(fst, eco eucl) = -0,1090 (p=0,697), rk(fst, eco eucl) = -0,0247 (p=0,520), rs(fst, eco eucl) = -0,1245 (p=0,705), rp(rst, eco eucl) = -0,0477 (p=0,583), rk(rst, eco eucl) = 0,0575 (p=0,352), rs(rst, eco eucl) = -0,0192 (p=0,512), rp(fst, cust eco) = 0,7922 (p=0,000), rk(fst, cust eco) = 0,5716 (p=0,001), rs(fst, cust eco) = 0,7533 (p=0,000), rp(rst, cust eco) = 0,8024 (p=0,000), rk(rst, cust eco) = 0,5813 (p=0,001), rs(rst, cust eco) = 0,7939 (p=0,000), rp(fst, eco cust) = 0,0004 (p=0,492), rk(fst, eco cust) = 0,0284 (p=0,384), rs(fst, eco cust) = -0,0234
105 99 (p=0,513), rp(rst, eco cust) = 0,0577 (p=0,390), rk(rst, eco cust) = 0,1092 (p=0,251), rs(rst, eco cust) = 0,0947 (p=0,328), rp(fst, eucl eco2) = 0,8686 (p=0,002), rp(rst, eucl eco2) = 0,8734 (p=0,003), rp(fst, eco2 eucl) = -0,3811 (p=0,986), rp(rst, eco2 eucl) = -0,3481 (p=0,987), rp(fst, cust eco2) = 0,8246 (p=0,002), rp(rst, cust eco2) = 0,8306 (p=0,004), rp (fst, eco2 cust) = -0,3294 (p=0,980) e rp(rst, eco2 cust) = -0,2991 (p=0,966) (Tabela 19) América do Sul Para esta subcategoria foram usadas as 18 populações localizadas na América do Sul ((micros) Figura 10). A correlação entre as duas matrizes de distância genética rp(fst, rst) = 0,9634 (p=0,000), correlação alta e significativa. As únicas correlações significativas para este conjunto foram entre distância geográfica e fisiográfica. Os valores encontrados para este conjunto foram rp(eucl, cust) = 0,9668 (p=0,000), rp(fst, eucl) = -0,0639 (p=0,630), rp(fst, cust) = -0,1186 (p=0,769), rp(fst, eco) = -0,3978 (p=0,993), rk(fst, eco) = -0,3465 (p=0,990) e rs(fst, eco) = -0,4229 (p=0,993), rp(fst, eco2) = -0,0860 (p=0,629), rp(rst, eucl) = -0,0370 (p=0,553), rp(rst, cust) = -0,0846 (p=0,692), rp(rst, eco) = -0,3366 (p=0,977), rk(rst, eco) = -0,2998 (p=0,978), rs(rst, eco) = -0,3660 (p=0,978), rp(rst, eco2) = -0,0576 (p=0,578), rp(eucl, eco) = 0,2777 (p=0,061), rk(eucl, eco) = 0,2126 (p=0,057), rs(eucl, eco) = 0,2590 (p=0,059), rp(cust, eco) = 0,3654 (p=0,013), rk(cust, eco) = 0,2685 (p=0,023), rs(cust, eco) = 0,3275 (p=0,025), rp (eucl, eco2) = 0,7857 (p=0,000) e rp(cust, eco2) = 0,8057 (p=0,000). Para as correlações parciais de Mantel não houve valores significativos em nenhuma análise. Os valores encontrados foram: rp(fst, eucl eco) = 0,0528 (p=0,333), rk (fst, eucl eco) = 0,0428 (p=0,297), rs(fst, eucl eco) = 0,0351 (p=0,365), rp(rst, eucl eco) = 0,0624 (p=0,308), rk(rst, eucl eco) = 0,0525 (p=0,253), rs(rst, eucl eco) = 0,0683 (p=0,287), rp(fst, eco eucl) = P -0,3965 (p=0,993), rk(fst, eco eucl) = -0,3473 (p=0,993), rs(fst, eco eucl) = -0,4181 (p=0,993), rp(rst, eco eucl) = -0,3399 (p=0,981), rk(rst, eco eucl) = 0,3037
106 100 (p=0,982), rs(rst, eco eucl) = -0,3702 (p=0,984), rp(fst, cust eco) = 0,0313 (p=0,395), rk(fst, cust eco) = 0,0293 (p=0,358), rs(fst, cust eco) = 0,0138 (p=0,437), rp(rst, cust eco) = 0,0439 (p=0,361), rk(rst, cust eco) = 0,0432 (p=0,297), rs(rst, cust eco) = 0,0499 (p=0,328), rp(fst, eco cust) = -0,3835 (p=0,992), rk(fst, eco cust) = -0,3419 (p=0,993), rs(fst, eco cust) = -0,4070 (p=0,990), rp(rst, eco cust) = -0,3296 (p=0,981), rk(rst, eco cust) = -0,3001 (p=0,980), rs(rst, eco cust) = -0,3621 (p=0,983), rp(fst, eucl eco2) = 0,0059 (p=0,487), rp (rst, eucl eco2) = 0,0132 (p=0,462), rp(fst, eco2 eucl) = -0,0580 (p=0,617), rp(rst, eco2 eucl) = -0,0046 (p=0,581), rp(fst, cust eco2) = -0,0835 (p=0,002), rp(rst, cust eco2) = -0,0646 (p=0,784), rp(fst, eco2 cust) = 0,0162 (p=0,429) e rp(rst, eco2 cust) = -0,0179 (p=0,432) (Tabela 20). Tabela 19: Valores das correlações para microc entre as matrizes e suas respectivas significâncias. Marcado em negrito estão as correlações significativas com p<0,05. fst, rst (rp) eucl, cust (rp) fst, eucl (rp) fst, cust (rp) fst, eco (rp) fst, eco (rk) fst, eco (rs) fst, eco2 (rp) rst, eucl (rp) rst, cust (rp) rst, eco (rp) rst, eco (rk) rst, eco (rs) rst, eco2 (rp) eucl, eco (rp) eucl, eco (rk) eucl, eco (rs) eucl, eco2 (rp) cust, eco (rp) cust, eco (rk) cust, eco (rs) cust, eco2 (rp) mantel Valor p 0,9740 0,0001 0,9877 0,0001 0,8466 0,0005 0,8073 0,0005 0,2220 0,105 0,1363 0,178 0,1640 0,180 0,1169 0,027 0,8577 0,0002 0,8163 0,0002 0,2573 0,072 0,2026 0,036 0,2438 0,038 0,1431 0,055 0,3270 0,049 0,2626 0,55 0,3148 0,055 0,3615 0,028 0,2760 0,092 0,1952 0,087 0,2349 0,094 0,3718 0,033 parcial de mantel fst, eucl eco (rp) fst, eucl eco (rk) fst, eucl eco (rs) rst, eucl eco (rp) rst, eucl eco (rk) rst, eucl eco (rs) 0,8400 0,0005 0,5822 0,001 0,7601 0,0008 0,8470 0,0002 0,5818 0,001 0,7949 0,0008
107 101 Tabela 19 (continuação) fst, eco eucl (rp) fst, eco eucl (rk) fst, eco eucl (rs) rst, eco eucl (rp) rst, eco eucl (rk) rst, eco eucl (rs) fst, cust eco (rp) fst, cust eco (rk) fst, cust eco (rs) rst, cust eco (rp) rst, cust eco (rk) rst, cust eco (rs) fst, eco cust (rp) fst, eco cust (rk) fst, eco cust (rs) rst, eco cust (rp) rst, eco cust (rk) rst, eco cust (rs) fst, eucl eco2 (rp) rst, eucl eco2 (rp) fst, eco2 eucl (rp) rst, eco2 eucl (rp) fst, cust eco2 (rp) rst, cust eco2 (rp) fst, eco2 cust (rp) rst, eco2 cust (rp) mantel Valor p -0,1090 0,697-0,0247 0,520-0,1245 0,705-0,0477 0,583 0,0575 0,352-0,0192 0,512 0,7922 0,0005 0,5716 0,001 0,7533 0,0002 0,8024 0,0002 0,5813 0,001 0,7939 0,0005 0,0004 0,492 0,0284 0,384-0,0234 0,513 0,0577 0,390 0,1092 0,251 0,0947 0,328 0,8686 0,002 0,8734 0,003-0,3811 0,986-0,3481 0,987 0,8246 0,002 0,8306 0,004-0,3294 0,980-0,2991 0,966 Tabela 20: Valores das correlações para micros entre as matrizes e suas respectivas significâncias. Marcado em negrito estão as correlações significativas com p<0,05. fst, rst (rp) eucl, cust (rp) fst, eucl (rp) fst, cust (rp) fst, eco (rp) fst, eco (rk) fst, eco (rs) fst, eco2 (rp) rst, eucl (rp) rst, cust (rp) rst, eco (rp) rst, eco (rk) rst, eco (rs) rst, eco2 (rp) eucl, eco (rp) eucl, eco (rk) eucl, eco (rs) eucl, eco2 (rp) cust, eco (rp) mantel Valor p 0,9634 0,0001 0,9668 0,0001-0,0639 0,630-0,1186 0,769-0,3978 0,993-0,3465 0,990-0,4229 0,993-0,0860 0,629-0,0370 0,553-0,0846 0,692-0,3366 0,977-0,2998 0,978-0,3660 0,978-0,0576 0,578 0,2777 0,061 0,2126 0,057 0,2590 0,059 0,7857 0,0001 0,3654 0,013
108 102 Tabela 20 (continuação) cust, eco (rk) cust, eco (rs) cust, eco2 (rp) mantel Valor p 0,2685 0,023 0,3275 0,025 0,8057 0,0001 parcial de mantel fst, eucl eco (rp) fst, eucl eco (rk) fst, eucl eco (rs) rst, eucl eco (rp) rst, eucl eco (rk) rst, eucl eco (rs) fst, eco eucl (rp) fst, eco eucl (rk) fst, eco eucl (rs) rst, eco eucl (rp) rst, eco eucl (rk) rst, eco eucl (rs) fst, cust eco (rp) fst, cust eco (rk) fst, cust eco (rs) rst, cust eco (rp) rst, cust eco (rk) rst, cust eco (rs) fst, eco cust (rp) fst, eco cust (rk) fst, eco cust (rs) rst, eco cust (rp) rst, eco cust (rk) rst, eco cust (rs) fst, eucl eco2 (rp) rst, eucl eco2 (rp) fst, eco2 eucl (rp) rst, eco2 eucl (rp) fst, cust eco2 (rp) rst, cust eco2 (rp) fst, eco2 cust (rp) rst, eco2 cust (rp) 0,0528 0,333 0,0428 0,297 0,0351 0,365 0,0624 0,308 0,0525 0,253 0,0683 0,287-0,3965 0,993-0,3473 0,993-0,4181 0,993-0,3399 0,981-0,3037 0,982-0,3702 0,984 0,0313 0,395 0,0293 0,358 0,0138 0,437 0,0439 0,361 0,0432 0,297 0,0499 0,328-0,3835 0,992-0,3419 0,993-0,4070 0,990-0,3296 0,981-0,3001 0,980-0,3621 0,983 0,0059 0,487 0,0132 0,462-0,0580 0,617-0,0046 0,581-0,0835 0,840-0,0646 0,784 0,0162 0,429-0,0179 0, Algoritmo de Monmonier Não foi encontrada nenhuma estruturação geográfica para as populações da América com marcador de microssatélites pelo algoritmo de Monmonier (Figura 26).
109 Figura 26: (A) Desenho da rede de Delaunay para as populações de microssatélites. (B) Traço vermelho entre as populações ligadas pela rede com maior distância genética entre si. 103
110 DISCUSSÃO E CONCLUSÕES As principais discussões deste trabalho podem ser divididas em três categorias: (1) quanto à validade dos métodos; (2) quanto às correlações entre genética e geografia e (3) quanto às correlações entre genética e fisiografia 4.1. Validade dos Métodos A comparação entre matrizes tem sua validade reconhecida como método de pesquisa (Dow & Cheverud, 1985; Marroig & Cheverud, 2001) e é utilizada em muitos estudos, tanto envolvendo humanos, quanto outras espécies (Kemp et al. 2010; Hutsemekers et al. 2010; Liu et al. 2009). Apesar de ser amplamente utilizado, o método traz algumas limitações potencialmente problemáticas para o teste proposto neste trabalho (Smouse & Long, 1992; Smouse et al., 1986; Legengre 2000). O primeiro desses problemas consiste no fato de uma das matrizes de ecossistemas ser binária e, portanto, essencialmente categórica, o que a torna, a priori, não recomendável para o cálculo da correlação de Pearson (Field 2009). Porém, as matrizes categóricas de zero e um constituem a única exceção para este problema e podem ser usadas para o cálculo da correlação de Pearson (Field 2009). As correlações não paramétricas de Kendall e Spearman, mais adequadas para variáveis categóricas, tampouco são as mais adequadas para este trabalho, pois utilizam como base de cálculo a hierarquização das categorias (Field 2009) e no que se refere à matriz de biomas não existe uma hierarquia entre as variáveis. Além disto, não geram resultados confiáveis quando aplicadas ao teste parcial de Mantel (Oksanen et al., 2010). Devido a tais problemas, os cálculos foram feitos com todos os tipos de correlações para comparação. Aqueles com grandes discrepâncias entre si, foram descartados.
111 105 A correlação de Kendall não foi calculada para a maior parte dos casos devido a alguma dificuldade no algoritmo do programa, portanto esta correlação foi imediatamente descartada para a discussão dos resultados. Os resultados de correlação por Spearman foram obtidos para todos os conjuntos com mais de 8 populações. Neste contexto não houve diferenças marcantes entre os resultados obtidos com Pearson e Spearman, sendo, em geral, menores os valores obtidos para a correlação de Spearman quando comparados aos resultados obtidos pela correlação de Pearson, mas mantendo, em grande parte das vezes, os mesmos níveis de significância. Portanto, as correlações consideradas para a discussão baseiam-se nos resultados gerados pela correlação de Pearson. Tal escolha se deu por três razões: a primeira, porque esta correlação pode ser usada com segurança nos testes parciais de Mantel; a segunda, porque não houve resultados tão discrepantes entre as técnicas; e por fim, porque a correlação de Pearson também pode ser usada em casos de matrizes binárias de zero e um. Existem vários métodos para o cálculo das correlações parciais de Mantel (Cheverud et al., 1989; Legendre 2000; Smouse 1986). O método escolhido aqui foi o de condicionamento da terceira matriz, segundo o qual só a primeira matriz é permutada (Oksanen et al., 2010; Legendre 2000). Um possível problema desta ospção é a dificuldade em se saber se o cálculo com uma matriz binária impossibilita o teste parcial de Mantel. Este problema não foi discutido por Legendre (2000) ao formular o método. Porém, existem artigos na literatura que utilizam o método desta forma (Boizard et al, 2010; Manica et al., 2005). Como teste preliminar para a validação do método, foram criados os conjuntos menores de populações (conj1 a conj6). Entretanto, o pequeno número de populações não foi suficiente para dar poder estatístico à comparação, especialmente com a fisiografia, gerando resultados inconclusivos sobre a validação dos métodos. De toda maneira, como este método já foi usado antes em situações análogas (Kemp et al., 2010)
112 106 ele foi mantido acreditando-se que sua eficácia comprovada na literatura seria mantida para os parâmetros deste estudo. Outro problema importante que envolve matrizes binárias reside no fato de que as especificações de ausência e presença podem não explorar totalmente as relações entre as populações, tornando próximas populações que, mesmo para o critério aqui adotado, seriam distantes. Exemplos deste problema são os casos dos Siouan na América do Norte e dos Mapuche na América do Sul. Essas populações receberam escore zero na matriz de fisiografia, pois estão localizadas em um mesmo bioma: savanas e pradarias temperadas. Entretanto, estão muito distantes entre si fisicamente e fisiograficamente, uma vez que os ancestrais da população Mapuche tiveram de passar obrigatoriamente por um ambiente totalmente novo (de florestas na América Central) para chegar ao bioma hoje ocupado. Com o objetivo de resolver este problema, três estratégias foram aqui empregadas: a primeira, separar as populações em grupos menores, nos quais não estariam tão distantes fisicamente. Esta estratégia traz como problema o fato de não abarcar a totalidade de biomas que compõe a América. No entanto, foi a única que mostrou resultados positivos e significativos. A segunda estratégia, a matriz cumulativa de biomas, foi proposta pensando-se em manter a integridade dos biomas do continente. Um teste foi feito com microssatélites e com a região da Patagônia, ambos com menor número de populações. Não houve diferença entre os resultados apresentados pelo método cumulativo de biomas em relação ao binário, por isto a tentativa foi abandonada. O algoritmo de Monmonier foi uma terceira solução ao problema de matrizes binárias. Consiste em uma forma de avaliar a estruturação das populações sem necessidade de comparação entre matrizes, apenas com a localização das populações em um mapa e suas distâncias genéticas (Manel 2003). O método foi testado para os
113 107 dados de microssatélites, mas não apresentou melhor resolução do que os cálculos feitos com a matriz binária de biomas. Ainda assim, parece ser um método bastante promissor que poderá ser aplicado futuramente. Outra questão importante foi a escolha do sistema de classificação de biomas, uma vez que as proposições de Beaton (1991) e Dixon (2001), apesar de os mencionarem, não dividiram as regiões continentais de acordo com os 14 biomas reconhecidos na literatura, mas sim reunidas em 5 grandes áreas: ártico, costa oeste, cordilheira, interior e costa leste. Esta separação em 5 grandes regiões traz consigo alguns problemas para a construção da matriz fisiográfica proposta neste trabalho. A região do ártico abarca poucas populações estudadas quanto ao DNA mitocondrial e nenhuma quanto aos microssatélites. Além disto, não existe uma delimitação clara entre costa oeste e cordilheira, tornando difícil a alocação visual das populações. Por fim, os bancos de dados tanto de DNA mitocondrial quanto de microssatélites, não apresentam populações para a costa leste. Devido a essas dificuldades, a divisão em ambientes proposta por Dixon acabaria se resumindo em uma divisão entre costa oeste e cordilheira versus interior. Esta abordagem foi substituída pela abordagem em biomas por dois motivos principais: primeiro, porque a comparação entre costa oeste e interior já foi exaustivamente estudada (Wang et a., 2007; Pucciarelli et al., 2006, 2008; Fuselli et al., 2003) mostrando uma diferenciação entre as populações dessas localidades, sugerindo que os nativos americanos usaram uma rota costeira mais rápida para povoar as Américas, anterior ou concomitantemente à ocupação do interior (Waguespack, 2007). Porém, não se pode tirar conclusões sobre adaptações ao meio por esta abordagem, uma vez que para as Américas a costa oeste é separada do interior não somente pela diversidade ambiental,
114 108 mas também por uma cadeia montanhosa que pode ter separado essas populações mais do que suas tecnologias. Segundo, porque os diversos ambientes do interior da América seriam descartados como fonte de diversidade adaptativa. Seguindo o argumento de Beaton e Dixon, os primeiros americanos tiveram que adaptar seus estilos de vida e tecnologias aos diversos tipos de ambientes encontrados. As populações adaptadas a ambientes marítimos, por exemplo, teriam que se adaptar a um ambiente interiorano (Dixon 2001). Do ponto de vista adaptativo, uma alteração substancial no ambiente poderia provocar a necessidade de nova adaptação, promovendo uma permanência preferencial no local de origem ao qual já se estava adaptado (Kormondy & Brown 2002, Trigger 2004 e Diamond 2005). Neste sentido, a diferenciação por biomas fornece um parâmetro mais aceitável de diversidade ambiental, uma vez que agregam em cada um conjuntos semelhantes de flora e fauna Distância genética e geografia De forma geral, as correlações entre distâncias de custo e euclidianas 10 foram muito altas, superiores a 0.95, com apenas três exceções para o DNA mitocondrial: r=0.93 para todas as populações da América juntas; r=0.83 para as populações da região descrita como centro norte da América do Sul; e r=0.92 para as populações da América do Sul, o que mostra que não existe grande diferença entre os dois tipos de medidas de distância geográfica. Diante destas pequenas diferenças dos valores de correlação entre as distâncias geográficas, seria esperado que as distâncias de custo tivessem maior correlação com as distâncias genéticas, pois refletem possibilidades migratórias mais plausíveis, uma vez 10 Distâncias de custo são aquelas em que regiões de difícil transposição são evitadas pelos caminhos escolhidos pelo programa. Distâncias euclidianas são aquelas de caminhos diretos em linha reta entre as populações.
115 109 que obedecem barreiras naturais de difícil transposição (enquanto as distâncias euclidianas traçam linhas retas entre as populações). Apesar de teoricamente ser esperada uma maior correlação entre distâncias de custo e distâncias genéticas, este padrão não foi encontrado em todas as análises: de 7 agrupamentos populacionais com resultados significativos entre distância genética e geográfica, em apenas 4 as distâncias de custo obtiveram maior correlação, contra 3 com maior correlação para distância euclidiana. Dos conjuntos maiores de populações, a América do Sul como um todo (biom74), a região centro norte da América do Sul (biom42) (analisadas por DNA mitocondrial), e a América Central (por microssatélites) apresentaram uma maior correlação com distâncias euclidianas do que com distâncias de custo. Surpreendentemente, essas exceções referem-se a populações com as mais altas correlações entre distâncias genéticas e geográficas (América Central para microssatélites r=0.8466) e com a menor correlação entre distâncias genéticas e geográficas (região centro norte da América do Sul para DNA mitocondrial r=0.1856). A região centro norte da América do Sul foi, entre todos os agrupamentos populacionais, o que apresentou a menor correlação entre as distâncias geográficas (correlação entre custo e euclidiana r=0.8322), o que significa que o caminho construído pelo programa para evitar acidentes naturais (distâncias de custo) foi o mais diferenciado do caminho direto, ignorando tais acidentes (distâncias euclidianas). Estes resultados podem ser explicados pelo custo atribuído às altitudes entre -431m e 0m o que pode ter gerado uma diferença no caminho tomado por essas populações. Estas localizações cortam o nordeste brasileiro até próximo à Amazônia e são condizentes com leitos de rios da bacia hidrográfica amazônica, incluindo o Rio Amazonas. As rotas por rios podem ter facilitado percursos não computados nas análises e os dados para custo podem ter feito as populações desviarem especificamente dessas regiões, dando a impressão de que os
116 110 caminhos mais lineares seriam mais adequados, mesmo quando nenhum dos dois caminhos é o mais apropriado. Já para as populações da América Central (por microssatélites), a explicação pode estar correlacionada com o meio que essas populações usaram para locomoção. O fato de a correlação entre as matrizes de distância geográfica ser bastante alta (r=0.9877), indica que os caminhos traçados pelo programa, um evitando completamente as regiões oceânicas e outro ignorando o obstáculo marítimo, não foram tão distintos. Porém, as diferenças neles contidas foram suficientes para que o caminho de distâncias euclidianas obtivesse maior correlação. Isto leva a crer que uma ocupação usando os meios marítimos, como por exemplo por cabotagem, pode ter sido uma realidade para essas populações. Com os resultados aqui obtidos não se pode fazer nenhuma afirmação quanto aos benefícios do uso de distâncias com custos em detrimento das distâncias euclidianas, uma vez que os resultados não apontaram claramente uma preferência entre as duas distâncias, deixando uma grande margem de discussão. A correlação entre distâncias genéticas e distâncias geográficas para dados de DNA mitocondrial é baixa, entre r=0.31 para a região da Patagônia e r=0.12 para a região descrita como centro norte da América do Sul. Para a América como um todo o valor é intermediário (r=0.28). Esses baixos valores podem ser fruto de duas possibilidades: a primeira, de que o isolamento por distância não foi o principal fator estruturador das populações nativas americanas pelo menos pela herança materna. Neste caso a variação genética entre as populações poderia ser explicada por outros fatores que não a deriva genética (como exemplo do papel da língua na estruturação das populações nativas americanas ver Fagundes et al., 2002). A outra explicação reside no fato de que devido às realocações dessas populações e constantes transferências para reservas indígenas (Ribeiro 1996), elas podem não estar em seu local de origem e, portanto, sua relação
117 111 genética com seus vizinhos pode estar subestimada, levando a uma baixa correlação com distância geográfica. A análise de microssatélites mostrou uma correlação não significativa entre distância genética e distâncias geográficas entre as populações da América do Norte e do Sul. Estes resultados são compatíveis com os obtidos com DNA mitocondrial, indicando que a hipótese de ser um problema de descendência materna exclusiva é menos provável, uma vez que microssatélites apresentam ancestralidade biparental. Porém, em desacordo com os resultados anteriores, as populações da América Central, com microssatélites, apresentaram uma alta correlação (r=0.84) entre distância geográfica euclidiana e distância genética, o que indica o isolamento por distância como principal fonte estruturadora, diminuindo a probabilidade de as populações na América Central terem sofrido grandes realocações. Mesmo com este resultado não é possível fazer inferências sobre os motivos que levaram à baixa correlação nas outras localidades, uma vez que este resultado apresentou-se como exceção e não como regra. Ele pode indicar ainda que o isolamento por distância foi um fator pontual para as populações da América Central analisadas por microssatélites, não sendo necessariamente verdadeiro para outras regiões e populações do Novo Mundo Distância genética e fisiográfica De modo geral é possível afirmar que os biomas, como estão divididos atualmente, tiveram pouca ou nenhuma influência na estruturação da diversidade genética das populações nativas americanas, levando-se em consideração sua distribuição atual. A única correlação significativa ocorreu entre as populações da América do Norte, usando marcadores de DNA mitocondrial, e mostrou-se bastante fraca. Apesar de a América como um todo com análises de DNA mitocondrial (n10) ter mostrado uma correlação significativa entre distância genética e fisiográfica, essas
118 112 correlações não se sustentaram depois de retirado o efeito da distância geográfica. Portanto, esses resultados não são indicativos do efeito do bioma na estruturação genética das populações, devido à possibilidade de uma correlação cruzada com distância geográfica. Depois de aplicado o teste parcial de Mantel, a única correlação entre genética e fisiografia que permaneceu foi aquela entre as populações da América do Norte, através do DNA mitocondrial. Ainda assim, a correlação, apesar de significativa, é bastante fraca, indicando que para a conformação atual das populações, se houve um papel do meio ambiente ele foi muito pequeno e explica pouco da variação genética dos nativos americanos. Isso indica que deve haver outros fatores mais expressivos que o meio ambiente e o isolamento por distância para explicar a estruturação dessa variação. Outra possibilidade é que, como discutido anteriormente, essas populações estejam vivendo atualmente em regiões muito distintas daquelas da ocupação original. Uma explicação para o padrão diferenciado de distribuição de haplogrupos na América do Norte, Central e Sul baseia-se nos resultados encontrados por Perego et al. (2009), que sugerem que a ocupação do continente americano se deu por duas rotas migratórias principais, uma pelo interior da América do Norte e outra pela costa até atingir as Américas Central e do Sul. A proposta de Perego e colaboradores é reforçada pela presença de um alelo quase exclusivo no interior da América do Norte em contrapartida a um alelo presente na América Central e do Sul. Este padrão de uma distribuição diferenciada de haplogrupos para a América do Norte versus América Central e do Sul também pode ser visto na distribuição dos haplogrupos A e B (Schurr 2004), em que B está presente apenas na porção sudeste da América do Norte, na América Central e na do Sul, enquanto A está presente principalmente na região centro norte da América do Norte. Esses padrões podem significar que as regiões da América Central e do Sul receberam fluxos populacionais diferentes. Segundo os resultados obtidos neste trabalho,
119 113 as populações que ocuparam o Norte poderiam apresentar um padrão cultural mais especializado, enquanto as populações que ocuparam a América Central e do Sul apresentaram tecnologia mais generalizada, facilitando a ocupação de diversos ambientes concomitantemente. Em resumo, apesar de haver uma pequena participação dos biomas na estruturação das populações do Novo Mundo, esta divisão pode ser apenas mais um fator que contribuiu para a estruturação populacional, mas está longe de ser o principal. De qualquer maneira, os resultados apresentados neste trabalho falam contra a hipótese de que o ambiente, pelo menos como se apresenta atualmente, teve um papel fundamental no processo de ocupação das Américas. Entretanto, existe a possibilidade de o modelo estar correto, mesmo com os resultados aqui apresentados, devido ao fato do ambiente na América estar em contínuo processo de diferenciação desde o fim da última era glacial. Permanece em aberto, contudo, quais outros fatores, junto com isolamento por distância, seriam os responsáveis pela estruturação genética dos nativos americanos atuais, ou ainda, se existe um fator principal, atualmente desconhecido, por trás dessa estruturação. Talvez esta pergunta jamais venha a ser respondida devido ao grande remanejamento das populações indígenas nas Américas desde a entrada dos europeus no Novo Mundo. Este trabalho traz também como contribuição o fato de ter sido o primeiro teste formal para a ideia de que os biomas tiveram alguma influência na estruturação da diversidade genética das populações nativas americanas. Os resultados são ainda tímidos, mas abrem caminho para pesquisas futuras.
120 RESUMO Até recentemente, o povoamento das Américas era visto como um produto de uma expansão em linhas paralelas do norte para o sul do continente. Sob este cenário, os sítios arqueológicos dos primeiros americanos deveria obedecer um gradiente cronológico seguindo a mesma lógica, independente de sua longitude. Recentemente, no entanto, especialistas começaram a reconhecer que certas características dos diferentes biomas poderiam favorecer diferentes taxas de expansão populacional. Beaton (1991), por exemplo, sugeriu que as expansões humanas em escala continental seriam mais condicionadas às características do ambiente (biomas) de que a distâncias geográficas lineares, ideia esta, também suportada por Dixon (2001). Neste estudo foi testada a hipótese de Beaton e Dixon, aplicada às Américas, investigando se a estrutura genética dos nativos americanos atuais é influenciada pelos biomas que elas ocupam. Para fazer isso, três diferentes tipo de matrizes foram construídas baseadas em dados de DNA mitocondrial e microssatélites de grupos de nativos americanos: uma, formada por distâncias genéticas (Fst) entre as populações, outra formada pelas distâncias geográficas entre as mesmas populações em quilômetros, e uma última formada pelas distâncias fisiográficas. Essas matrizes foram comparadas pela correlação de Pearson seguida de testes de Mantel e parciais de Mantel. Os resultados obtidos mostraram que em geral os diferentes biomas não tiveram um papel significativo na estruturação genética das populações nativas americanas, ao menos como estão distribuídas hoje.
121 ABSTRACT Until recently, the settlement of the Americas was seen as the product of a "bow wave" human expansion from north do south. Under this scenario, the archaeological sites of the first americans should obey a chronological gradient following the same logic, independent of their longitude. Recently, however, specialists began to recognize that certain characteristics of different biomes could have favored different rates of demic expansion. Beaton (1991), for instance, suggested that human expansions in continental scales are much more conditioned by the ecological attributes of the macro environmental zones (biomes) involved than by linear geographic distances, an idea also spoused by Dixon (2001). In this study we test Beaton s and Dixon s ideas, as applied to the Americas, by investigating if the genetic structure of recent native american populations is influenced by the biomes they occupy. In order to do this, three different kinds of matrices were constructed based on the frequency of mtdna and microsatelites from native american groups: one formed by the genetic distances (Fst) among the populations, a second one formed by the geographic distances among the same populations in kilometers, and a last one formed by their "physiographic" distances. These matrices were compared by Pearson s correlation followed by Mantel and partial Mantel tests. The results obtained showed that in general the different biomes did not play a significant role in the native american genetic structuring, at least as they are distributed today.
122 REFERÊNCIAS ACHILLI, A.; PEREGO, U.A.; BRAVI, C.M.; COBLE, M.D.; KONG, Q.; WOODWARD, S.R.; SALAS, A.; TORRONI, A.; BANDELT, H. The phylogeny of the four pan-american MtDNA haplogroups: implications for evolutionary and disease studies. PLoS One 3 (3): e1764, AHN, J.; BROOK, E. Atmospheric CO2 and climate on millennial time scales during the last glacial period. Science 322:83-85, ALFIMOV, A.V.; BERMAN, D.I. Beringian Climate During the Late Pleistocene and Holocene. Quaternary Science Reviews 20: 8, AMANTE, C.; EAKINS, B.W. ETOPO1 1 Arc-Minute Global Relief Model: Procedures, Data Sources and Analysis. NOAA Technical Memorandum NESDIS NGDC-24, 19 pp., ARAUJO, A.; REINHARD, K.J.; FERREIRA, L.F. Parasites as probes for prehistoric human migrations? Trends Parasitology 24(3): 112-5, ATKINSON, Q.D.; Gray, R.D.; DRUMMOND, A.J. mtdna variation predicts population size in humans and reveals a major Southern Asian chapter in human prehistory. Mol Biol Evol 25(2): , ATUÍ, J.P.V. Morfologia craniana de ameríndios brasileiros recentes e suas implicações para a questão da ocupação do Novo Mundo: uma análise exploratória. Dissertação de Mestrado do Instituto de Biociências, AVISE, J.C. Molecular Markers, Natural History and Evolution. Chapman & Hall Inc. Nova York, AVISE, J.C. Phylogeography: The History and Formation of Species. Harvard University Press. Cambridge, AVISE, J.C.; ARNOLD, J.; BALL, R.M.; BERMINGHAM, E.; LAMB, T.; NEIGEL, J.E.; REEB, C.A.; Saunders, N.C. Intraspecific Phylogeography: The Mitochondrial DNA
123 117 Bridge Between Population Genetics and Systematics. Ann. Rev. Ecol. Syst. 18: 34, BAILLIET, G.; ROTHHAMMER, F.; CARNESE, F.R.; BRAVI, C.M; BIANCHI, N.O. Founder mitochondrial haplotypes in Amerindian populations. American Journal of Human Genetics 55:27-33, 1994 BAR-YOSEF, O. The Upper Paleolithic Revolution. Ann. Rev. Anthropol. 31: 31, BARBUJANI, G.; BELLE, E.M. Genomic boundaries between human populations. Hum Hered 61(1): 15-21, BATTILANA, J.; FAGUNDES, N.J.R; HELLER, A.H.; GOLDANI, A.; FREITAS, L.B.; TARAZONA-SANTOS, E.; MUNKHBAT, B; MUNKHTUVSHIN, N.; KRYLOV, M.; BENEVOLENSKAIA, L.; ARNETT, F.C.; BATZER, M.A.; DEININGER, P.L.; SALZANO, F.M.; BONATTO, S.L. Alu insertion polymorphisms in Native Americans and related Asian populations. Ann Hum Biol 33(2): , BATZER, M.A.; STONEKING, M.; ALEGRIA-HARTMAN, M.; BAZAN, H.; KASS, D.H.; SHAIKH, T.H.; NOVICK, G.E.; IOANNOU, P.A.; SCHEER, W.D.; HERRERA, R.J.; DEININGER, P.L. African origin of human-specific polymorphic Alu insertions. Proc Natl Acad Sci U S A 91(25): , BEATON, J.M. Colonizing continents: some problems for Australia and the Americas. In: Dillehay, T. D., Meltzer, D. J. (Eds). The first americans: search and research. CRP Press, Boca Raton, FL: 11, BEIGUELMAN, B. Dinâmica dos genes nas famílias e nas populações. 2 ed. Sociedade Brasileira de Genética. Ribeirão Preto, BERT, F.; CORELLA, A.; GENE, M.; PEREZ-PEREZ, A.; TURBON BORREGA, D. Major mitochondrial DNA haplotype heterogeneity in highland and lowland Amerindian Poulation from Bolivia Human Biology 73(1):1-16, 2001.
124 118 BETTI, L.; BALLOUX, F.; AMOS, W.; HANIHARA, T.; MANICA, A. Distance from Africa, not climate, explains within-population phenotypic diversity in humans. Proc Biol Sci 276(1658): , BIANCHI, N.O.; BAILLIET, G.; BRAVI, C.M. Peopling of the Americas as inferred through the analysis of mitochondrial DNA. Brazilian Journal of Genetics 18(4): , BOIZARD, J.; MAGNAN, P.; ANGERS, B. Effects of dynamic landscape elements on fish dispersal: the example of creek chub (Semotilus atromaculatus). Molecular Ecology vol. 18 (3) pp , BOLNICK, D.A.W.; Smith, D.G. Unexpected patterns of mitochondrial DNA variation among Native Americans from the Southeastern United Stades. American journal of physical anthropology 122: , BONATTO, S.L.; SALZANO, F.M. A single and early migration for the peopling of the Americas supported by mitochondrial DNA sequence data. Proc Natl Acad Sci U S A 94(5): , BOWCOCK, A.M.; RUIZ-LINARES, A.; TOMFOHRDE, J.; MINCH, E,; KIDD, J.R.; CAVALLI-SFORZA, L.L. High resolution of human evolutionary trees with polymorphic microsatellites. Nature 368(6470): 455-7, BRIGHAM-GRETTE, J. New Perspectives on Beringian Quaternary Paleogeography, Stratigraphy, and Glacial History. Quaternary Science Reviews 20: 10, BROECKER, W.S. Geology. Was the Younger Dryas triggered by a flood? Science 312 (5777): , BROWN, M.D.; HOSSEINI, S.H.; TORRONI, A.; BANDELT, H.; ALLEN, J.C.; SCHURR, T.G.; SCOZZARI, R.; CRUCIANI, F.; WALLACE, D.C. mtdna haplogroup X: an ancient link between Europe/Western Asia and North America?. The American Journal of Human Genetics 63: , 1998.
125 119 CALLEGARI-JACQUES, S.M.; SALZANO, F.M.; WEIMER, T.A.; HUTZ, M.H.; BLACK, F.L.; SANTOS, S.E.B.; GUERREIRO, J.F.; MESTRINER, M.A.; PANDEY, J.P. Further blood genetic studies on Amazonian diversity--data from four Indian groups. Ann Hum Biol 21(5): , CANDOLLE, A. In: Lomolino, M. V., Sax, D. F., Brown, J. H. Foundations of Biogeography. Essai Élémentaire de Géographie Botanique. 1 ed. University of Chicago Press. Chicago, CARLYLE, S.W.; PARR, R.L.; HAYES, G.; O ROURKE, D.H. Context of maternal lineages in the Greater Southwest. American journal of physical anthropology 113:85-101, CAVALLI-SFORZA, L.L.; MENOZZI, P.; PIAZZA, A. The history and geography of human genes. Princeton University Press, Princeton, New Jersey, CHEVERUD, J.M.; WAGNER, G.P.; DOW, M.M. Methods for the comparative analysis of variation patterns. Systematic Biology 38(3): , CLARK, P.U.; MIX, A.C. Ice Sheets and Sea Level of the Last Glacial Maximum. Quaternary Science Reviews 21: 8, COUTINHO, L.M. O Cenceito de Bioma. Acta Botanica Brasileira 20(1): 10, COX, C.B.; MOORE, P.D. Biogeography: An Ecological and Evolutionary Approach. 7 ed. Wiley-Blackwell. Hoboken, DARWIN, C. A Origem das Espécies. Madras Editora Ltda., São Paulo, 2004 DEMARCHI, D.A.; PANZETTA-DUTARI, G.M.; MOTRAN, C.C.; BASUALDO, M.A.L.; MARCELINO, A.J. Mitochondrial DNA haplogroups in Amerindian populations from the Gran Chaco. American journal of physical anthropology 115: , DERBENEVA, O.A.; SUKERNIK, R.I.; VOLODKO, N.V.; HOSEINI, S.H.; LOTT, M.T.; WALLACE, D.C. Analysis of mitochondrial DNA diversity in the aleuts of the
126 120 commander islands and its implications for the genetic history of beringia. American Journal of Human Genetics vol. 71 (2) pp , DIAMOND, J.D. Armas, germes e aço: os destinos das sociedades humanas. 1 ed. São Paulo, 2005 DILLEHAY, T.D. Monte Verde: a late Pleistocene settlement in Chile 2; the archaeological context. Washington: Smithsonian Institution Press, DILLEHAY, T.D. Probing deeper into first American studies. Proceedings of the National Academy of Science 106(4): , DIXON, E.J. Human colonization of the Americas: timming, technology and process. Quaternary Science Reviews 20: 22, DOBZHANSKY, T. Genetics and the Origin of Species. Columbia University Press. Nova York, DOW, M.M.; CHEVERUD, J.M. Comparison of distance matrices in studies of population structure and genetic microdifferentiation: quadratic assignment. American Journal of Physical Anthropology. 68: , DOWLING, T.E.; MORITZ, C.; PALMER, J.D.; RIESEBERG, L.H. In:Hillis, D. M., Moritz, C., Mable, B. K. Molecular Systematics Chapter 8 Nucleic Acids III: Anaçysis of Fragments and Restriction Sites. 2 ed. Sinauer Associates, Inc. Stamford, DUPANLOUP, I.; SCHNEIDER, S.; EXCOFFIER, L. A simulated annealing approach to define the genetic structure of populations. Mol Ecol 11(12): , EASTON, R.D.; MERRIWETHER, D.A.; CREWS, D.E.; FERREL, R.E. mtdna variation in the Yanomami: evidence for additional New World founding lineages. American Journal of Human Genetics 59: , ESHLEMAN, J.A.; MALHI, R.S.; JOHNSON, J.R.; KAESTLE, F.A.; LORENZ, J.; SMITH, D.G. Mitochondrial DNA and Prehistoric Settlements: Native Migrations on the Western Edge of North America. Human Biology 76(1):55-75, 2004.
127 121 EXCOFFIER, L.L.; LAVAL, G.; SCHNEIDER, S. Arlequin 3.1 An Integrated Software Package for Population Genetics Data Analysis. Evolutionary Bioinformatics Online, 47-50, FAGUNDES, N.J.R.; BONATTO, S.L.; CALLEGARI-JACQUES, S.M.; SALZANO, F.M. Genetic, geographic, and linguistic variation among South American Indians: possible sex influence. American journal of physical anthropology vol. 117 (1) pp , FAGUNDES, N.J.R.; KANITZ, R.; BONATTO, S.L. A reevaluation of the Native American mtdna genome diversity and its bearing on the models of early colonization of Beringia. PLoS One 3(9): e3157, 2008a. FAGUNDES, N.J.R; KANITZ, R.; ECKERT, R.; VALLS, A.C.S.; BOGO, M.R.; SALZANO, F.M.; SMITH, D.G.; SILVA JR, W.A.; ZAGO, M.A.; RIBEIRO-DOS-SANTOS, A.K., SANTOS, S.E.B.; PETZL-ERLER, M.L.; BONATTO, S. Mitochondrial population genomics supports a single pre-clovis origin with a coastal route for the peopling of the Americas. Am J Hum Genet 82(3): , 2008b. FALCONER, D.S.; MACKAY, T.F.C. Introduction to Quantitative Genetics. 4 ed. Pearson England, FIELD, A. Descobrindo a estatística utilizando o SPSS. 1 ed. Artmed, Porto Alegre, FIX, A.G. Rapid deployment of the five founding amerind mtdna haplogroups via coastal and riverine colonization. American Journal of Physical Anthropology 128: 6, FOLEY, R. Apenas mais uma espécie única. 1 ed. EDUSP São Paulo, Brasil, FORSTER, P.; HARDING, R.; TORRONI, A.; BANDELT, H. Origin and evolution of Native American mtdna variation: a reappraisal. American Journal of Human Genetics vol. 59 (4) pp. 935, FORSTER, P.; MATSUMURA,S. Evolution. Did early humans go north or south? Science 308(5724): 965-6, 2005.
128 122 FUSELLI, S.; TARAZONA-SANTOS, E.; DUPANLOUP, I.; SOTO, A.; LUISELLI, D.; PETTENER, D. Mitochondrial DNA diversity in South America and the genetic history of Andean highlanders. Molecular biology and Evolution. 20(10): , 2003 GILBERT, M.T.P.; KIVISILD, T.; GRØNNOW,B.; ANDERSEN, P.K.; METSPALU, E.; REIDLA, M.; TAMM, E.; AXELSSON, E.; GÖTHERSTRÖM, A.; CAMPOS, P.F., RASMUSSEN, M.; METSPALU, M.; HIGHAM, T.F.G.; SCWENNINGER, J.; NATHAN, R.; HOOG, C.; KOCH, A.; MØLLER, L.N.; ANDREASEN, C.; MELDGAARD, M.; VILLEMS, R.; BENDIXEN, C.; WILLERSLEV, E. Paleo- mtdna genome reveals matrilineal discontinuity in Greenland. Science 320(5884): , GILLESPIE, J.R. Population genetics: a concise guide. 2 ed. The John Hopkins University Press, Baltimore, Maryland, GOICOECHEA, A.S.; CARNESE, F.R.; CARATINI, A.L.; AVENA, S.; SALABERRY, M.; SALZANO, F.M. Demography, genetic diversity, and population relationships among Argentinean Mapuche Indians Genetics and Molecular Biology 23(3): , GOICOECHEA, A.S.; CARNESE, F.R.; DEJEAN, C.; AVENA, S.A.; WEIMER, T.A.; FRANCO, M.H.L.P.; CALLEGARI-JACQUES, S.M.; ESTALOTE, A.C.; SIMÕES, M.L.M.S.; PALATNIK, M.; SALZANO, F.M. Genetic relationships between Amerindian populations of Argentina. American journal of physical anthropology 115: , GONZÁLEZ-JOSÉ, R.; BORTOLINI, M.C.; SANTOS, F.R.; BONATTO, S.L. The peopling of America: craniofacial shape variation on a continental scale and its interpretation from an interdisciplinary view. Am J Phys Anthropol 137(2): , 2008.
129 123 GONZÁLEZ-JOSÉ, R.; DAHINTEN, S.L.; LUIS, M.A.; HERNÁNDEZ, M.; PUCCIARELLI, H.M. Craniometric variation and the settlement of the Americas: testing hypotheses by means of R-matrix and matrix correlation analyses. Am J Phys Anthropol 116(2): , GONZÁLEZ-JOSÉ, R.; GONZÁLEZ-MARTÍN, A.; HERNÁNDEZ, M.; PUCCIARELLI, H.M.; SARDI, M.; ROSALES, A.; MOLEN, S.V. Craniometric evidence for Palaeoamerican survival in Baja California. Nature 425(6953): 62-5, GONZÁLEZ-OLIVIER, A.; MÁRQUEZ-MORFÍN, L.; JIMÉNEZ, J.C.; TORRE-BLANCO, A. Founding amerindian mitochondrial DNA lineages in ancient Maya from Xcaret, Quintana Roo. American journal of physical anthropology 116: , Google Earth 5.0 < acessado em 20/08/2010. GOULD, S.J.; ELDREDGE, N. Punctuated equilibrium comes of age. Nature 366(6452): 223-7, GRIFFITHS, A.J.F.; WESSLER, S.R.; LEWONTIN, R.C.; GELBART, W.M.; SUZUKI, D.T.; MILLER, J.H. Introdução à Genética. 8 ed Guanabara-Koogan. Rio de Janeiro, GUIDON, N. Primeiros habitantes do Brasil: as descobertas de São Raimundo Nonato. Revista FAPESP Edição Online, GUTHRIE, R.D. Origin and causes of the mammoth steppe: a story of cloud cover, woolly mammal tooth pits, buckles, and inside-out Beringia. Quaternary Science Reviews 20: 26, GUTHRIE, R.D. New carbon dates link climatic change with human colonization and Pleistocene extinctions. Nature 441(7090): 207-9, HAFFER, J. Speciation in Amazonian Forest Birds. Science 165(3889): , HALL, R. Pleistocene migration routes into the Americas: Human biological adaptations and environmental constraints. Evolutionary Anthropology 13(4): 13, 2004.
130 124 HANDLEY, L.J.L.; MANICA, A.; GOUDET, J.; BALLOUX, F. Going the distance: human population genetics in a clinal world. TRENDS in Genetics 23(9): , HARTL, D.L. Princípios de genética de populações. 3 ed. FUNPEC Lagoa Nova. Brasil, HEDRICK, P.W. Genetics of populations. 2 ed. Jones and Barlett Publishers Massachusetts, HETHERINGTON, R.; BARRIE, J.W.; REID, R.G.B.; MACLEOD, R.; SMITH, D.J.; JAMES, T.S.; KUNG, R. Late Pleistocene Coastal Paleogeography of the Queen Charlotte Islands, British Columbia, Canada, and its Implications for Terrestrial Biogeography and Early Postglacial Human Occupation. Can. J. Earth Sci. 40: 12, HILLIS, D.M.; MABLE, B.K.; LARSON, A.; DAVIS, S.K.; ZIMMER, E.A. In:Hillis, D. M., Moritz, C., Mable, B. K. Molecular Systematics Chapter 9 Nucleic Acids IV: Sequencing and Cloning. 2 ed. Sinauer Associates, Inc. Stamford, HILLIS, D.M., MORITZ, C.; MABLE, B.K. Molecular Systematics, Second Edition (Paperback). 2 ed. Sinauer Associates, Inc. Stamford, HORAI, S.; KONDO, R.; NAKAGAWA-HATTORI, Y.; HAYASHI, S.; SONODA, S.; TAJIMA, K. Peopling of the Americas, founded by four major lineages of mitochondrial DNA. Molecular biology and evolution 10 (1): 23-47, HUBBE, M.; NEVES, W.A.; HARVATI, K. Testing Evolutionary and Dispersion Scenarios for the Settlement of the New World. PLoS One 5(6): 1-9, HUNLEY, K.; LONG, J.C. Gene flow across linguistic boundaries in Native North American populations. Proc Natl Acad Sci U S A 102(5): , HUNLEY, K.L.; SPENCE, J.E.; MERRIWETHER, D.A. The impact of group fissions on genetic structure in Native South America and implications for human evolution. Am J Phys Anthropol 135(2): , 2008.
131 125 HUTSEMEKERS, V.; HARDY, O.J.; MARDULYN, P.; SHAW, A.J.; VANDERPOORTEN, A. Macroecological patterns of genetic structure and diversity in the aquatic moss Platyhypnidium riparioides. The New phytologist 185 (3): , JIN, L.; BASKETT, M.L.; CAVALLI-SFORZA, L.L.; ZHIVOTOVSKY, L.A.; FELDMAN, M.W.; ROSENBERG, N.A. Microsatellite evolution in modern humans: a comparison of two data sets from the same populations. Ann Hum Genet 64(Pt 2): , KAESTLE, F.A.; SMITH, D.G. Ancient mitochondrial DNA evidence for prehistoric population movement: the Numic Expansion. American journal of physical anthropology 115:1-12, KARAFET, T.M.; ZEGURA, S.L.; POSUKH, O.; OSIPOVA, L.; BERGEN, A.; LONG, J.; GOLDMAN, D.; KLITZ, W.; HARIHARA, S.; KNIJFF, P.; GRIFFITHS, R.C.; TEMPLETON, A.R.; HAMMER, M.F. Ancestral Asian source(s) of new world Y- chromosome founder haplotypes. Am J Hum Genet 64(3): , KATINAS, L.; POSADAS, P.; CRISCI, J.V. Historical Biogeography: an Introduction. Harvard University Press. Cambridge, KAYSER, M. The human genetic history of Oceania: near and remote views of dispersal. Current Biology 20: , KEMP, B.M.; GONZÁLEZ-OLIVER, A.; MALHI, R.S.; MONROE, C.; SCHROEDER, K.B.; MCDONOUGH, J.; RHETT, G.; RESENDÉZ, A.; PEÑALOZA-ESPINOSA, R.I.; BUENTELLO-MALO, L.; GORODESKY, C.; SMITH, D.G. Evaluating the Farming/ Language Dispersal Hypothesis with genetic variation exhibited by populations in the Southwest and Mesoamerica. Proceedings of the National Academy of Sciences of the United States of America 107(15): , 2010 KEYEUX, G.; RODAS, C.; GELVEZ, N.; CARTER, D. Possible migration routes into South America deduced from mitochondrial DNA Studies in Colombian Amerindian Populations. Human Biology 74(2): , 2002.
132 126 KITCHEN, A.; MIYAMOTO, M.M.; MULLIGAN, C.J. A three-stage colonization model for the peopling of the Americas. PLoS ONE 3(2): e1596, KLEIN, R.; EDGAR, B. O Despertar da Cultura. 1 ed. Jorge Zahar. Rio de Janeiro, KOLMAN, C.J.; BERMINGHAM, E.; COOKE, R.; WARD, R.H.; ARIAS, T.D.; GUIONNEAU- SINCLAIR, F. Reduced mtdna diversity in the Ngobe Amerinds of Panama. Genetics Society of America 140: , KOLMAN, C.J.; BERMINGHAM, E. Mitochondrial and nuclear DNA diversity in the Choco and Chibcha Amerinds of Panama. Genetics Society of America 147: , KORMINDY, E.J.; BROWN, D.E. Ecologia Humana. 1 ed. Atheneu Editora. São Paulo, KOTTEK, M.; GRIESER, J.; BECK, C.; RUDOLF, B.; RUBEL, F. World Map of the Köppen- Geiger Climate Classification Updated. Meteorologische Zeitschrift 15(3): 5, LALUEZA-FOX, C.; GILBERT, M.T.P.; MARTINEZ-FUENTES, A.J.; CALAFELL, F.; BERTRANPETIT, J. Mitochondrial DNA from pre-columbian Ciboneys from Cuba and the prehistoric Colonization of the Caribbean. American journal of physical anthropology 121:97-108, LECLERC, G.L. In: Lomolino, M. V., Sax, D. F., Brown, J. H. Foundations of Biogeography. Natural History, General and Particular. 1 ed. University of Chicago Press. Chicago, LEGENDRE, P. Comparison of permutation methods for the partial correlation and partial Mantel tests. Journal of Statistical Computation and Simulation 67(1):37-73, LESSA, E.P.; COOK, J.A.; PATTON, J.L. Genetic footprints of demographic expansion in North America, but not Amazonia, during the Late Quaternary. Proc Natl Acad Sci U S A 100(18): , 2003.
133 127 LÉVI-STRAUSS, C. O Pensamento Selvagem. 4 ed. M. R. Cornacchia e Editora Ltda. São Paulo, LÉVI-STRAUSS, C. Tristes Trópicos. 5 reimpressão Editora Schwarcz Ltda. São Paulo, LEWIN, R. Young Americans. New Scientist 160(2156): 24-28, LEWIS, C.M.; TITO, R.Y.; LIZÁRRAGA, B.; STONE, A.C Land, language, and loci: mtdna in Native Americans and the genetic history of Peru. American Journal of Physical Anthropology 127: , LI, Y.; KOROL, A.B.; FAHIMA, T.; BEILES, A.; NEVO, E. Microsatellites: genomic distribution, putative functions and mutational mechanisms: a review. Mol Ecol 11 (12): , LINNAEUS, C. In: Lomolino, M. V., Sax, D. F., Brown, J. H. Foundations of Biogeography: Classic Papers with Commentaries from Dissertation II. On the Increase of the Habitable Earth. 1 ed. University of Chicago Press. Chicago, LIU, Z.; REN, B.; WU, R.; ZHAO, L.; HAO, Y.; WANG, B.; WEI, F.; LONG, Y.; LI, M. The effect of landscape features on population genetic structure in Yunnan snub-nosed monkeys (Rhinopithecus bieti) implies an anthropogenic genetic discontinuity. Molecular Ecology 18(18): , LOMOLINO, M.V.; RIDDLE, B.R.; BROWN, J.H. Biogeography. 3 ed. Sinauer Associates, Inc. Stamford, LORENZ, J.G.; SMITH, D.G. Distribution of four founding mtdna haplogroups among Native North Americans. American journal of physical anthropology 101: , MAJUMDER, P.P. The human genetic history of South Asia. Current Biology 20: , 2010
134 128 MALHI, R.S.; SCHULTZ, B.A.; SMITH, D.G. Distribution of Mitchondrial DNA Lineages among Native American Tribes of Northeastern North America. Human Biology 73 (1):17-55, MALHI, R.S., MORTENSEN, H.M.; ESHLEMAN, J.A.; KEMP, B.M.; LORENZ, J.G.; KAESTLE, F.A.; JOHNSON, J.R.; GORODEZKY, C.; SMITH, D.G. Native American mtdna prehistory in the American Southwest. American journal of physical anthropology 120: , MALHI, R.S.; BREECE, K.E.; SHOOK, B.A.S.; KAESTLE, F.A.; CHATTERS, J.C.; HACKENBERGER, S.; SMITH, D.G. Patterns of mtdna diversity in northwestern North America. Human Biology 76(1):33-54, 2004 MALHI, R.S.; KEMP, B.M.; ESHLEMAN, J.A.; CYBULSKI, J.; SMITH, D.G.; COUSINS, S.; HARRY, H. Mitochondrial haplogroup M discovered in prehistoric North Americans. Journal of Archaeological Science 34: , MALHI, R.S.; CYBULSKI, J.S.; TITO, R.Y.; JOHNSON, J.; HARRY, H.; DAN, C. Brief communication: Mitochondrial haplotype C4c confirmed as a founding genome in the Americas. American Journal of Physical Anthropology 141: , MANEL, S.; SCHWARTZ, M.K.; LUIKART, G.; TABERLET, P. Landscape genetics: combining landscape ecology and population genetics. Trends Ecol Evol 18(4), MANICA, A.; PROGNOLLE, F.; BALLOUX, F. Geography is a better determinant of human genetic differentiation than ethnicity. Hum Genet 118(3-4): , MANTEL, N. The detection of disease clustering and a generalized regression approach. Cancer Res 27(2): , MARROIG, G.; CHEVERUD, J.M. A comparison of phenotypic variation and covariation patterns and the role of phylogeny, ecology, and ontogeny during cranial evolution of new world monkeys. Evolution 55(12): , 2001.
135 129 MARTIN, P.S. The Discovery of America: The first Americans may have swept the Western Hemisphere and decimated its fauna within 1000 years. Science 179(4077): , MARTINÓN-TORRES, M.; CASTRO, J.M.B.; GÓMEZ-ROBLES, A.; ARSUAGA, J.L.; CARBONELL, E.; LORDKIPANIDZE, D.; MANZI, G.; MARGVELASHVILI, A. Dental evidence on the hominin dispersals during the Pleistocene. Proceedings of the National Academy of Science 104(33): , MAYR, E. Populations, Species and Evolution. Harvard University Press. Cambridge, McDOUGALL, I.; BROWN, F.H.; FLEAGLE, J.G. McDougall, I. Stratigraphic placement and age of modern humans from Kibish, Ethiopia. Nature 433(7027): 733-6, MENDES, C.T. DNA mitocondrial na Amazônia Brasileira: Estrutura Genética Regional e Inferências Continentais. Tese de doutorado da Faculdade de Medicina de Ribeirão Preto, MERRIWETHER, D.A.; ROTHHAMMER, D.A.; FERREL, R.E. Distribution of the four founding lineage haplotypes in native americans suggests a single wave of migration for the New World. American Journal of Physical Anthropology 98: 19, MERRIWETHER, D.A.; HALL, W.W.; VAHLNE, A.; FERREL, R.E. mtdna variation indicates Mongolia may have been the source for the founding population for the New World. American Journal of Human Genetics 59: , MERRIWETHER, D.A.; FERREL, R.E. The four founding lineage hypothesis for the New World: a critical reevaluation. Molecular Phylogenetics and Evolution 5(1): , MESA, N.R.; MONDRAGÓN, M.C.; SOTO, I.D.; PARRA, M.V.; DUQUE, C.; ORTIZ- BARRIENTOS, D.; GARCÍA, L.F.; VELEZ, I.D.; BRAVO, M.L.; MÚNERA, J.G.;
136 130 BEDOYA, G.; BORTOLINI, M.C.; RUIZ-LINARES, A. Autosomal, mtdna, and Y- chromosome diversity in Amerinds: pre-and post-columbian Patterns of Gene Flow in South America. The American Journal of Human Genetics 67: , MONSALVE, M.V.; HELGASON, A.; DEVINE, D.V. Languages, geography and HLA haplotypes in native American and Asian populations. Proc Biol Sci 266(1434): , MORAGA, M.L.; ROCCO, P.; MIQUEL, J.F.; NERVI, F.; LLOP, E.; CHAKRABORTY, R.; ROTHHAMMER, F.; CARVALLO, P. Mitochondrial DNA polymorphisms in Chilean aboriginal populations: implications for the Peopling of the Southern Cone of the Continent. American journal of physical anthropology 113:19-29, MORRONE, J.J.; CRISCI, J.V. Historical Biogeography: Introduction to Methods. Annu. Rev. Ecol. Syst. 26: 29, MURPHY, R.W.; SITES, J.W.; BUTH, D.G.; HAUFLER, C.H.; In:Hillis, D. M., Moritz, C., Mable, B. K. Molecular Systematics, Chapter 4 Proteins: Isozyme Electrophoresis. 2 ed. Sinauer Associates, Inc. Stamford, NEEL, J.V.; SALZANO, F.M. Further studies on the Xavante Indians. X. Some hypothesesgeneralizations resulting from these studies. American Journal of Human Genetics 19(4): , NEI, M. Analysis of gene diversity in subdivided populations. Proc Natl Acad Sci U S A 70 (12): , NELSON, G. (1978). From Candolle to Croizat: comments on the history of biogeography. J Hist Biol 11(2): , NEVES, W.A.; POWELL, J.F.; PROUS, A.; OZOLINS, E.G.; BLUM, M. Lapa Vermelha IV hominid 1: morphological affinities of the earliest known American. Genetics and Molecular Biology 22(4): , 1999.
137 131 NEVES, W.A.; HUBBE, M. Cranial morphology of early Americans from Lagoa Santa, Brazil: implications for the settlement of the New World. Proc Natl Acad Sci U S A 102(51): , NEVES, W.A.; HUBBE, M.; CORREAL, G. Human skeletal remains from Sabana de Bogota, Colombia: a case of Paleoamerican morphology late survival in South America? Am J Phys Anthropol 133(4): , 2007a. NEVES, W.A.; HUBBE, M.; PILÓ, L.B. Early Holocene human skeletal remains from Sumidouro Cave, Lagoa Santa, Brazil: history of discoveries, geological and chronological context, and comparative cranial morphology. J Hum Evol 52(1): 16-30, 2007b. NEVES. W.A.; PROUS, A.; GONZÁLEZ-JOSÉ, R.; KIPNIS, R.; POWELL, J. Early Holocene human skeletal remains from Santana do Riacho, Brazil: implications for the settlement of the New World. J Hum Evol 45(1): 19-42, NEVES, W.A.,; PUCCIARELLI, H.M. Extracontinental biological relationships of early South American human remains: a multivariate analysis. Ciência e Cultura 41: 9, NOVICK, G.E.; NOVICK, C.C.; YUNIS, J.; YUNIS, E.; ANTUNEZ DE MAYOLO, P.; SCHEER, W.D.; DEININGER, P.L.; STONEKING, M.; YORK, D.S. BATZER, M.A.; HERRERA, R.J. Polymorphic Alu insertions and the Asian origin of Native American populations. Hum Biol 70(1): 23-39, OKSANEN, J.F.; BLANCHET, G.; KINDT, R.; LEGENDRE, P.; O HARA, R.B.; SIMPSON, G.L.; SOLYMOS, P.; STEVENS, M.H.H..; WAGNER, H. vegan: Community Ecology Package. R package version OLSON, D.M.; DINERSTEIN, E.; WIKRAMANAYAKE, E.D.; BURGESS, N.D.; POWELL, G.N.V.; UNDERWOOD, E.C.; D AMICO, J.A.; ITOUA, I.; STRAND, H.E.;
138 132 MORRISON, J.C.; LOUCKS, C.J.; ALLNUTT, T.F.; RICKETTS, T.H.; KURA, Y.; LAMOREUX, J.F.; WETTENGEL, W.W.; HEDAO, P.; KASSEM, K.R. Terrestrial Ecoregions of the World: A New Map of Life on Earth. BioScience 51: , O ROURKE, D.H.; MOBARRY, A.; SUAREZ, B.K. Patterns of Genetic Variation in Native America. Human Biology 64(3): 18, O ROURKE, D.H.; HAYES, M.G.; CARLYLE, S.W. Spatial and temporal stability of mtdna haplogroup frequencies in native North America. Human biology 72(1):15-34, PALUMBI, S.R. In:Hillis, D. M., Moritz, C., Mable, B. K. Molecular Systematics, Chapter 7 Nucleic Acids II: The Polymerase Chain Reaction. 2 ed. Sinauer Associates, Inc. Stamford, PAVLOV, P.; SVENDSEN, J.I.; INRELID, S. Human presence in the European Arctic nearly 40,000 years ago. Nature 413(6851): 64-7, PEREGO, U.A.; ACHILLI, A.; ANGERHOFER, N.; ACCETTURO, M.; PALA, M.; OLIVIERI, A.; KASHANI, B.H.; RITCHIE, K.H.; SCOZZARI, R.; KONG, Q.; MYRES, N.M.; SALAS, A.; SEMINO, O.; BANDELT, H.; WOODWARD, S.R.; TORRONI, A. Distinctive Paleo-Indian migration routes from Beringia marked by two rare mtdna haplogroups. Curr Biol 19(1): 1-8, PEREGO, U.A.; ANGERHOFER, N.; PALA, M.; OLIVIERI, A.; LANCIONI, H.; KASHANI, B.H.; CAROSSA, V.; EKINS, J.E.; GÓMEZ-CARBALLA, A.; HUBER, G.; ZIMMERMANN, B.; CORACH, D.; BABUDRI, N.; PANARA, F.; MYRES, N.M.; PARSON, W.; SEMINO, O.; SALAS, A.; WOODWARD, S.R.; ACHILLI, A.; TORRONI, A. The initial peopling of the Americas: A growing number of founding mitochondrial genomes from Beringia. Genome Researche Publicado online Junho, 2010.
139 133 PITULKO, V.V.; NIKOLSKY, P.A.; GIRYA, E.Y.; BASILYAN, A.E.; TUMSKOY, V.E.; KOULAKOV, S.A.; ASTAKHOV, S.N.; PAVLOVA, E.Y.; ANISIMOV, M.A. The Yana RHS site: humans in the Arctic before the last glacial maximum. Science 303(5654): 52-6, POWELL, J.F.; NEVES, W.A. Craniofacial morphology of the first Americans: Pattern and process in the peopling of the New World. Am J Phys Anthropol Suppl 29: , PRITCHARD, J.K., STEPHENS, M.; DONNELLY, P. Inference of population structure using multilocus genotype data. Genetics 155(2): , PUCCIARELLI, H.M.; GONZÁLEZ-JOSÉ, R.; NEVES, W.A.; SARDI, M.L.; ROZZI, F.R. East-West cranial differentiation in pre-columbian populations from Central and North America. J Hum Evol 54(3): , PUCCIARELLI, H.N.; NEVES, W.A.; GONZÁLEZ-JOSÉ, G.; SARDI, M.L.; ROZZI, F.R.; STRUCK, A.; BONILLA, M.Y. East-West cranial differentiation in pre-columbian human populations of South America. Homo 57(2): , PULLIAM, H.R.; DANIELSON, B.J. Sources, Links and Habitat Selection: a Landscape Perspective on Population Dynamics. American Naturalist 137: 17, R DEVELOPMENT CORE TEAM R: A language and environment for statistical computing. R Foundation for Statistical Computing, Vienna, Austria. ISBN , URL RAMACHANDRAN, S.; DESHPANDE, O.; ROSEMAN, C.C.; ROSENBERG, N.A.; FELDMAN, M.W.; CAVALLI-SFORZA, L.L. Support from the relationship of genetic and geographic distance in human populations for a serial founder effect originating in Africa. Proc Natl Acad Sci U S A 102(44): , RAY, N. PATHMATRIX: a geographical information system tool to compute effective distances among samples. Molecular Ecology Notes 5: , 2005.
140 134 RAY, N.; WEGMANN, D.; FAGUNDES, N.J.R.; WANG, S.; RUIZ-LINARES, A.; EXCOFFIER, L. A statistical evaluation of models for the initial settlement of the American continent emphasizes the importance of gene flow with Asia. Molecular biology and evolution 27(2): , RAYBOULD, A.F.; MOGG, R.J.; CLARKE, R.T. Genetic structure of a linear population of Beta vulgaris ssp. maritima (sea beet) revealed by isozyme and RFLP analysis. Heredity 77: , RIBEIRO, D. Os índios e a civilização. A integração das populações indigenas no Brasil Moderno. 7ed. Companhia das Letras, RICKARDS, O.; MARTINEZ-LABARGA, C.; LUM, J.K.; DE STEFANO, G.F.; CANN, R.L. mtdna history of the Cayapa Amerinds of Ecuador: detection of additional founding lineages for the Native American Populations. The American Journal of Human Genetics 65: , RICKLEFS, R.E. A economia da natureza." 5ed. Editora Guanabara-Koogan S.A. Rio de Janeiro, RJ, RIDLEY, M. Evolução. 3 ed. Artmed Editora S.A. São Paulo, ROSEMAN, C.C.; WEAVER, T.D. Multivariate apportionment of global human craniometric diversity. Am J Phys Anthropol 125(3): , ROSENBERG, N.A.; PRITCHARD, J.K.; WEBER, J.L.; CANN, H.M.; KIDD, K.K.; ZHIVOTOVSKY, L.A.; FELDMAN, M.W. Genetic structure of human populations. Science 298(5602): , ROOSEVELT. A.C.; DOUGLAS, J.; BROWN, L. The Migrations and Adaptations of the First Americans: Clovis and Pre Clovis Viewed from South America. The First Americans: The Pleistocene Colonization of the New World Editado por Nina Jablonski 1ed, 2002.
141 135 SAILLARD, J.; FORSTER, P.; LYNNERUP, N.; BANDELT, H.; NØRBY, S. Saillard et al. (2000) mtdna variation among Greenland s: the edge of the Beringian expansion. The American Journal of Human Genetics 67: , SALAS, A.; RICHARDS, M.; DE LA FE, T.; LAREU, M.; SOBRINO, B.; SANCHÉZ-DIZ, P.; MACAULAY, V.; CARRACEDO, A. The making of the African mtdna landscape. Am J Hum Genet 71(5): , SALZANO, F.M.; CALLEGARI-JACQUES, S.M. South American Indians: a case study in evolution. 1 ed. Clarendon Press, Oxford, SCHROEDER, K.B.; JAKOBSSON, M.; CRAWFORD, M.H.; SCHURR, T.G.; BOCA, S.M.; CONRAD, D.F.; TITO, R.Y.; OSIPOVA, L.P.; TARSKAIA, L.A.; ZHADANOV, S/I.; WALL, J.D..; PRITCHARD, J.K.; MALHI, R.S.; SMITH, D.G.; ROSENBERG, N.A. Haplotypic background of a private allele at high frequency in the Americas. Mol Biol Evol 26(5): , SCHROEDER, K.B.; SCHURR, T.G.; LONG, J.C.; ROSENBERG, N.A.; CRAWFORD, M.H.; TARSKAIA, L.A.; OSIPOVA, L.P.; ZHADANOV, S.I.; SMITH, D.G. A private allele ubiquitous in the Americas. Biol Lett 3(2): , SCHURR, T.G.; BALLINGER, S.W.; GAN, Y.; HODGE, J.A.; MERRIWETHER, D.A.; LAWRENCE, D.N.; KNOWLER, W.C.; WEISS, K.M.; WALLACE, D.C. Amerindian mitochondrial DNAs have rare Asian mutations at high frequencies, suggesting they derived from four primary maternal lineages. American Journal of Human Genetics 46: , SCHURR, T.G. The Peopling of the New World: Perspectives from Molecular Anthropology. Annual Review of Anthropology 33: 33, SCHURR, T.G.; SHERRY, S.T. Mitochondrial DNA and Y chromosome diversity and the peopling of the Americas: evolutionary and demographic evidence. Am J Hum Biol 16(4): , 2004.
142 136 SCHMITT, R.; BONATTO, S.L.; FREITAS, L.B.; MUSCHNER, V.C.; HILL, K.; HURTADO, A.M.; SALZANO, F.M. Extremely limited mitochondrial DNA variability among the Aché natives of Paraguay. Ann Hum Biol 31(1):87-94, SCOZZARI, R.; CRUCIANI, F.; SANTOLAMAZZA, P.; SELLITO, D.; COLE, D.E.C.; RUBIN, L.A.; LABUDA, D.; MARINI, E.; SUCCA, V.; VONA, G.; TORRONI, A. mtdna and Y chromosome-specific polymorphisms in modern Ojibwa: implications about the origin of their gene pool. American Journal of Human Genetics vol. 60 (1) pp , SHEA, J.J. Neandertals, Competition, and the Origin of Modern Human Behavior in the Levant. Evolutionary Anthropology 12: 15, SHIELDS, G.; SCHMIECHEN, A.M.; FRAZIER, B.L.; REDD, A.; VOEVODA, M.I.; REED, J.K.; WARD, R.H. mtdna sequences suggest a recent evolutionary divergence for Beringian and northern North American Populations. American Journal of Human Genetics 53: , SHIN, S.I.; LIU, Z.; OTTO-BLIESNER, B.; BRADY, E.C.; KUTZBACH, J.E.; HARRISON, S.P. A Simulation of the Last Glacial Maximum Climate Using the NCAR-CCSM. Climate Dynamics 20: 25, SHINODA, K.; ADACHI, N.; GUILLEN, S.; SHIMADA, I. Mitochondrial DNA analysis of ancient Peruvian highlanders. American journal of physical anthropology vol. 131 (1) pp. 98, SHOOK, B.A.; Smith, D.G. Using ancient mtdna to reconstruct the population history of northeastern North America. Am J Phys Anthropol 137(1): 14-29, SLATKIN, M. A measure of population subdivision based on microsatellite allele frequencies. Genetics 139(1): , 2005.
143 137 SMITH, D.G.; MALHI, R.S.; ESHLEMAN, J.; LORENZ, J.G.; KAESTLE, F.A. Distribution of mtdna haplogroup X among native North Americans. American journal of physical anthropology 110: , SMOUSE, P.E.; LONG, J.C.; SOKAL, R.P. Multiple Regression and Correlation Extensions of the Mantel Test of Matrix Correspondence. Systematic Zoology 35(4): 6, SMOUSE, P.E.; LONG, J.C. Matrix correlation analysis in anthropology and genetics. Yearbook of Physical Anthropology 35: , SOARES,P.; ACHILLI, A.; SEMINO, O.; DAVIES, W.; MACAULAY, V.; BANDELT, H.; TORRONI, A.; RICHARDS, M.B. The archaeogenetics of Europe. Current Biology 20: , STARIKOVSKAYA, Y.B.; SUKERNIK, R.; SCHURR, T.G.; KOGELNIK, A.M.; WALLACE, D.C. mtdna diversity in Chukchi and Siberian s: implications for the genetic history of Ancient Beringia and the Peopling of the New World. The American Journal of Human Genetics 63: , STONE, A.C.; STONEKING, M. mtdna analysis of a prehistoric Oneota population: implications for the peopling of the New World. The American Journal of Human Genetics 62: , STONEKING, M.; DELFIN, F. The human genetic history of East Asia: weaving a complex tapestry. Current Biology 20: , STRINGER, C. Human evolution: Out of Ethiopia. Nature 423(6941): 692-3, 695, TAMM, E.; KIVISILD, T.; REIDLA,.; METSPALU, M.; SMITH, D.; MULLIGAN, C.J.; BRAVI, C.M.; RICKARDS, O.; MARTINEZ-LABARGA, C.; KHUSNUTDINOVA, E.K.; FEDOROVA, S.A.; GOLUBENKO, M.V.; STEPANOV, V.A.; GUBINA, M.A.; ZHADANOV, S.I.; OSSIPOVA, L.P.; DAMBA, L.; VOEVODA, M.I.; DIPIERRI, J.E.; VILLEMS, R.; MALHI, R.S. Beringian standstill and spread of native american founders. PLoS One 9 e829(6): 1, 2007
144 138 TARAZONA-SANTOS, E.; CARVALHO-SILVA, D.; PETTENER, D.; LUISELLI, D.; STEFANO, G.F.; LABARGA, C.M.; RICKARDS, O.; TYLER-SMITH, C.; PENA, S.D.J.; SANTOS, F.R. Genetic differentiation in South Amerindians is related to environmental and cultural diversity: evidence from the Y chromosome. Am J Hum Genet 68(6): , TEMPLETON, A.R. Population Genetics and Microevolutionary Theory. 1 ed. John Wiley & Sons, Inc. Hoboken. New Jersey, THOMAS, M.; GILBERT, P.; JENKINS, D.; GÖTHERSTROM, A.; NAVERAN, N.; SANCHEZ, J.J.; HOFREITER,M.; THOSEN, P.F.; BINLADEN, J.; HIGHAM, T.G.F.; YOHE II, R.M.; PARR, P.; CUMMINGS, L.S.; WILLERSLEV, E. DNA from pre-clovis human coprolites in Oregon, North America. Science 320(786): , THORPE, S.K.S.; HOLDER, R.L.; CROMPTON, R.H. Origin of human bipedalism as an adaptation for locomotion on flexible branches. Science 316: , TORRONI, A.; SCHURR, T.G.; YANG, C.C.; SZATHMARY, E.J.E.; WILLIAMS, R.C.; SCHANFIELD, M.S.; TROUP, G.A.; KNOWLER, W.C.; LAWRENCE, D.N.; WEISS, K.M.; WALLACE, D.C. Native American mitochondrial DNA analysis indicates that the Amerind and the Nadene populations were founded by two independent migrations. Genetics 130 (1): , TORRONI, A.; SCHURR, T.G.; CABELL, M.F.; BROWN, M.D.; NEEL, J.V.; LARSEN, M.; SMITH, D.; VULLO, C.M.; WALLACE, D.C. Asian affinities and continental radiation of the four founding Native American mtdnas. American Journal of Human Genetics 53: , TORRONI, A.; NEEL, J.V.; BARRANTES, R.; SCHURR, T.G.; WALLACE, D.C. Mitochondrial DNA "clock" for the Amerinds and its implications for timing their entry into North America. Proceedings of the National Academy of Sciences of the United States of America 91(3): , 1994a.
145 139 TORRONI, A.; CHEN, Y.S.; SEMINO, O.; SANTACHIARA-BENECERETTI, A.; SCOTT, C.R.; LOTT, M.T.; WINTER, M.; WALLACE, D.C. mtdna and Y-chromosome polymorphisms in four Native American populations from Southern Mexico. American Journal of Human Genetics 54: , 1994b. TRIGGER, G. História do pensamento arqueológico. 1 ed. Odysseus. São Paulo, VANZOLINI, P.E. Paleoclimas e Especiação em Animais da América do Sul Tropical. Estudos Avançados 6(15): 25, VERZANI, J. Using R for Introductory Statistics. 1 ed. Chapman & Hall/CRC Press Boca Raton, VOLODKO, N.V.; STARIKOVSKAYA, E.B.; MAZUNIN, I.O.; ELTSOV, N.P.; NAIDENKO, P.V.; WALLACE, D.C.; SUKERNIK, R.I. Mitochondrial genome diversity in arctic Siberians, with particular reference to the evolutionary history of Beringia and Pleistocenic peopling of the Americas. Am J Hum Genet 82(5): , VONA, G.; FALCHI, A.; MORAL, P.; CALO, C.M.; VARESI, L. Mitochondrial sequence variation in the Guahibo Amerindian population from Venezuela. Am J Phys Anthropol 127: , WAGUESPACK, N.M. Why we re still arguing about the pleistocene occupation of the Americas? Evolutionary Anthropology 16:63-74, WALLACE, A.R. In: Lomolino, M. V., Sax, D. F., Brown, J. H. Foundations of Biogeography.The Geographical Distribution of Animals. 1 ed. University of Chicago Press. Chicago, WANG, S.; LEWIS, C.M.; JAKOBSSON, M.; RAMACHANDRAN, S.; RAY, N.; BEDOYA, G.; ROJAS, W.; PARRA, M.V.; MOLINA, J.A.; GALLO, G.; MAZZOTTI, G.; POLETTI, G.; HILL, K.; HURTADO, A.M.; LABUDA, D.; KLITZ, W.; BARRANTES, R.; BORTOLINI, M.C.; SALZANO, F.M.; PETZL-ERLER, M.L.; TSUNETO, L.T.; LLOP, E.; ROTHHAMMER, F.; EXCOFFIER, L.; FELDMAN, M.W.; ROSENBERG,
146 140 N.A.; RUIZ-LINARES, A. Genetic variation and population structure in native Americans. PLoS Genet 3(11): e185, WARD, R.H.; FRAZIER, B.; DEW-JAGER, K.; PÄÄBO, S. Extensive mitochondrial diversity within a single Amerindian tribe. Proceedings of the National Academy of Sciences of USA 88: , WARD, R.H.; REDD, A.; VALENCIA, D.; FRAZIER, B.L.; PÄÄBO, S. Genetic and linguistic differentiation in the Americas. Proceedings of the National Academy of Sciences of USA 90: , WATERS, M.R.; STAFFORD JR, T.W. Redefining the age of Clovis: implications for the peopling of the Americas. Science 315(5815): , WHITMORE, T.C.; PRANCE, G.T. Biogeography and Quaternary History in Tropical America. Oxford University Press, WIENS, J.J.; DONOGHUE, M.J. Historical biogeography, ecology and species richness. Trends Ecol Evol 19(12): , WILLIAMS, S.R.; CHAGNON, N.A.; SPIELMAN, R.S. Nuclear and mitochondrial genetic variation in the Yanomamö: A test case for ancient DNA Studies of Prehistoric Populations. American journal of physical anthropology 117: , WILSON, J.A.P. A New Perspective on Later Migration(s). Critique of Anthropology 28(3): 12, WOLPOFF, M.H.; HAWKS, J.; CASPARI, R. Multiregional, not multiple origins. Am J Phys Anthropol 112(1): , WRIGHT, S. The interpretation of population structure by F-statistics with sepcial regard to systems of mating. Evolution 19(3): 25, ZIMMER, C. O Livro de Ouro da Evolução. Ediouro. Rio de Janeiro, 2001.
147 8. APÊNDICES Apêndice I: Frequências de haplogrupos nas populações originalmente colhidas com suas respectivas referências originais. As cores agrupam as populações por referência População N Haplogrupo A Haplogrupo B Haplogrupo C Haplogrupo D Referências Ache Akimal O odham Aleuts Aleuts ancient Aleuts St. Paul Anasazi ancient Ancash Aónikenk Apache Apache Apache Arequipa Peru Atacameno Atacameno Atacamenos Atacamana San Pedro Aymara Aymara Aymara groups Baníwa Bella Coola Bella Coola Bella Coola Bella Coola Bella Coola Boruca Boruca Bribi-Cabecar Butalelbun Pehuenche Butalelbum Pehuenche Caquena Aymara Cayapa Cayapa Ecuador Cherokee Cherokee Oklahoma Red Cross Cherokee Stillwell Cheyenne/Arapahoe Cheyenne/Araphoe Chickasaw Chickasaw/Choctaw Schmitt et al Malhi et al Derbeneva et al O'Rourke et al Merriwether et al Carlyle et al Shinoda et al Demarchi et al Lorenz and Smith Merriwether et al O Rourke et al Lewis et al Merriwether et al Bailliet et al Shinoda et al Easton et al Bert et al Shinoda et al Easton et al Mendes, O'Rourke et al Merriwether et al merriwether et al Forster et al Eshleman et al Torroni Easton et al Torroni Merriwether et al Merriwether et al Merriwether et al Rickards et al Lewis et al Lorenz and Smith Malhi et al Malhi et al Lorenz and Smith O Rourke et al Bolnick and Smith O Rourke et al 2000
148 149 População N Haplogrupo A Haplogrupo B Haplogrupo C Haplogrupo D Referências Chickasaw/Choctaw Chimane Chimila Chipewa Chippewa Turtle Muntain Chippewa Wisconsin Chippewa/Kickapoo Choctaw Choroti Choroti Chukchi Chukchi Chukchi Chumash Ciboneys Cochimi Codpa Aymara Columbia Coreguaje Creek Creek (Muskoke) Creek/Seminole Curripaco Dogrib Dogrib Dogrib Dogrib Dogrib Embera Embera Embera s Savoonga Ammassalik (Angmagssalik) Ilulissat (Jakobshavn) Ittoqqortoormiitt (Scoresbysund) Nanortalik Nuuk (Godthab) Upernarik Uummannaq (Umanak) s St. Lawrence Siberian s Gambell s Lorenz and Smith Bert et al Keyeux et al Merriwether et al Malhi et al Malhi et al O Rourke et al Bolnick and Smith Bianchi et al Goicoechea et al Merriwether et al Merriwether et al Starikovskaya et al Eshleman et al Lalueza-Fox et al Lorenz and Smith Merriwether et al Merriwether et al Keyeux et al Bolnick and smith Bolnick and smith O Rourke et al Keyeux et al Merriwether et al Easton et al Lorenz and Smith Starikovskaya et al O Rourke et al Mesa et al Keyeux et al Kolman and Bermingham Merriwether et al Saillard et al Saillard et al Saillard et al Saillard et al Saillard et al Saillard et al Saillard et al Starikovskaya et al Merriwether et al Merriwether et al Merriwether et al 1996
149 150 População N Haplogrupo A Haplogrupo B Haplogrupo C Haplogrupo D Referências s Ouzinkie Siberian Siberian s Old Harbor Yupik Fremont ancient Fueguian Argentina Gambel Gaviao Great Salt lake fremont ancient Guahibo Guahibo-Sikuani Guallatiri Aymara Guambiano Guanacagua Aymara Guane-Butaregua Guatuso Guayabero Guaymi Haida Haida Haida Haida Havasupai/Hualapai/ Yavapai/Mojave Hokan Northern California hokan (central coast) Hokan Northern Hopi Huetar Huetar Huilliche Chile Huilliche Chile Huilliche Huilliche Huitoto Hupa Ignaciano Ijka-Arhuaco Illapata Aymara Ingano Ingano Colombia Inuit eskimo Jemez/Taos/San Idelfonso Merriwether et al Starikovskaya et al O Rourke et al Merriwether et al Merriwether et al Kaestle & Smith Lewis et al Merriwether et al Demarchi et al Stone & Stonecking Vona et al Keyeux et al Merriwether et al Keyeux et al Merriwether et al Keyeux et al Torroni et al. 1994b Keyeux et al Torroni O'Rourke et al Merriwether et al Eshleman et al Merriwether et al Lorenz and Smith Lorenz and Smith Kaestle & Smith Kaestle & Smith Lorenz and Smith Forster et al Bianchi et al Merriwether et al Bailliet et al Shinoda et al Easton et al Keyeux et al Lorenz and Smith Bert et al Keyeux et al Merriwether et al Keyeux et al Mesa et al Lorenz and Smith O Rourke et al 2000
150 População N Haplogrupo A Haplogrupo B Haplogrupo C Haplogrupo D Referências Jemez Kanamarí Katukina Katukina Kaxináwa Kaxináwa Kawéskar Kiliwa Kiliwa/Paipai Kiowa Kogui Kraho Kumiai Kuna Kuna Kuna Macushi Makiritare Makiritare Makiritare Makiritare Venezuela Mapuche (Chile) Mapuche Mapuche Aguada Guzmán Mapuche Anecón Grande Mapuche Cerro policia Mapuche Chile Mapuche Mapuche Mapuche Mapuche Argentina Mapuche Mapuche Marúbo Brasil Marúbo Mataco (Chaco) Mataco (Salta) Mataco (Wichí) Argentina Mataco (Wichí) Formosa Maya ancient copán Maya ancient Xcaret Maya Mexico Maya Malhi et al Mendes, Mendes, Mendes, Mendes, Mendes, Demarchi et al Malhi et al Lorenz and Smith Lorenz and Smith Keyeux et al Torroni Lorenz and Smith Torroni Forster et al Stone & Stonecking Torroni Torroni Bailliet et al Easton et al Lewis et al Demarchi et al Bianchi et al Bailliet et al Goicoechea et al Bailliet et al Moraga et al Forster et al Shinoda et al Shinoda et al Goicoechea et al Bailliet et al Torroni Mendes, Demarchi et al Demarchi et al Goicoechea et al Demarchi et al Gonzalez-Oliver et al Gonzalez-Oliver et al Torroni et al Easton et al
151 152 População N Haplogrupo A Haplogrupo B Haplogrupo C Haplogrupo D Referências Maya Contenporary Maya Maya Micmac/Narragansett Micmac Mixe Mixtec (Alta) Mixtec (Baja) Mixtec Mohawk Mohawk Mohawk Mohawk Mosetén Bolivia Movima Bolívia Murui-Muinane Muskoke Oklahoma Muskoke Oklahoma Nahua/Cora Nahua Navajo Navajo Navajo Ngöbé Nukak Nuu-Chah-Nulth Nuu-Chah-Nulth Nuu-Chah-Nulth Nuu-Chah-Nulth Ojibwa Manitoulin Island Ojibwa Manitoulin Island Ojibwa N Ojibwa North Ontario Ojibwa/Chippewa Old Harbor Oneota ancient Ouzinkie Paez Paiute/Shoshone Paiute Northern Paiute N./Shoshoni Parinacota Aymara Pasto Pawnee Gonzalez-Oliver et al bianchi et al bailliet et al Lorenz and Smith bolnick and smith Torroni et al. 1994a Torroni et al. 1994a Torroni et al. 1994a Lorenz and Smith Stone & Stonecking Merriwether et al Malhi et al Easton et al Bert et al Bert et al Keyeux et al Easton et al Stone & Stonecking Lorenz and Smith Malhi et al Lorenz and Smith Merriwether et al O Rourke et al Kolman et al Keyeux et al Brown et al Brown et al Forster et al Eshleman et al Scozzari et al Bolnick & Smith Scozzari et al Bolnick & Smith O Rourke et al Merriwether et al Stone & Stoneking Merriwether et al Keyeux et al Lorenz and Smith Eshleman et al Malhi et al Merriwether et al Keyeux et al Malhi et al. 2001
152 153 População N Haplogrupo A Haplogrupo B Haplogrupo C Haplogrupo D Referências Pawnee Pehuenche Trapa Trapa Peneunche Chile Peneunche Chile 2 Pehuenche Trapa Trapa Penutian California Penutian California Pewenche Piaroa Colombia Piaroa Pilagá Formosa Pima Pima Pima Pyramid Lake ancient Quebrada de Humahuaca Argentina Quechan/Cocopa Bolivia (Arequipa) (Tayacaja) /Nmatsiquenga (San Martin) River Yuman Sahaptian Plateau Saint Paul Aleut Salinan/Chumash Salish Plateau Savoonga Sel kuns Selknam Selkman ancient Seminole San Salvador de Jujuy Argentina Seri Siona Siouan Sioux Sisseton/ Wapheton Sioux Stillwater Marsh ancient Takik Taono O odham (Papago) Tayacaja Peru Lorenz & Smith Moraga et al Lewis et al Lewis et al Easton et al Kaestle & Smith Lorenz and Smith Demarchi et al Torroni Keyeux et al Demarchi et al Lorenz and Smith Merriwether et al Bailliet et al Kaestle and Smith Lewis et al Lorenz and Smith Merriwether et al Easton et al Bert et al Fuselli et al Fuselli et al Fuselli et al Shinoda et al Malhi et al Malhi et al Merriwether et al Lorenz and Smith Malhi et al Merriwether et al Merriwether et al Demarchi et al Stone & Stonecking Bolnick & Smith Lewis et al Malhi et al Keyeux et al Lorenz & Smith Malhi et al Kaestle & Smith Eshleman et al Malhi et al Lewis et al 2004
153 154 População N Haplogrupo A Haplogrupo B Haplogrupo C Haplogrupo D Referências Tehuelche Argentina Teribe Teribe Tikúna Colombia Tikúna Tikúna Tikúna Tikúna Tikúna Tikúna Tikúna Tikúna Tikúna Brasil Ticuna Toba Toba Argentina Toba Toba (chaco) Toba (Formosa) Trapa Trapa Pehuenche Trapa Trapa Pehuenche Trinitario Bolivia Tucano Tule-Cuna Udegeys Uto-Aztecan Northern Uto-Aztecan Central Uto-Aztecan California Vantage Visviri Aymara Wapishana Brasil Washo Waunana Wayuu Wayuu Colombia Wishram Wiwa Wounan Xavante Yaghan Chile Yakama Yakima Yámana (Yaghan) Yamana Yaminawa Goicoechea et al Torroni et al. 1994b Easton et al Mesa et al Mendes, Mendes, Mendes, Mendes, Mendes, Mendes, Mendes, Mendes, Schurr et al Easton et al Bianchi et al Lewis et al Bianchi et al Demarchi et al Demarchi et al Merriwether et al Merriwether et al Bert et al Keyeux et al Keyeux et al Merriwether et al Kaestle and Smith Kaestle & Smith Lorenz and Smith Malhi et al Merriwether et al Torroni Lorenz and Smith Keyeux et al Keyeux et al Mesa et al Malhi et al Keyeux et al Kolman & Bermingham Demarchi et al Lewis et al Brown et al Forster et al Moraga et al Demarchi et al Mendes, 2005
154 155 População N Haplogrupo A Haplogrupo B Haplogrupo C Haplogrupo D Referências Yanomama Brasil Torroni 1993 Yanomami Easton et al Yanomami Easton et al Yanomamö Williams et al Yok-Utian Eshleman et al Yuko-Yukpa Keyeux et al Yuman Delta Malhi et al Yuman Pai Malhi et al Yuman (Hokan) Kaestle & Smith 2001 Yuman Eshleman et al 2004 Yuracaré Bert et al 2001 Zapotec Torroni et al. 1994a Zenu Mesa et al Zenu Keyeux et al Zoro Demarchi et al 2001 Zuni Lorenz & Smith 1996 Zuni O Rourke et al 2000 Zuni (penutian) Kaestle & Smith 2001 Arara Santos 1996 Arara Santos 1996 Assurini Santos 1996 Assurini Santos 1996 Awá-Guajá Santos 1996 Katuena Santos 1996 Kayapó Santos 1996 Parakanã Santos 1996 Poturujara (Zoé) Santos 1996 Tiryó Santos 1996 Waiãpi Santos 1996 Yamamadi Santos 1996 Yanomama Santos 1996
155 Apêndice II: Banco de dados de DNA mitocondrial final. (N) número de indivíduos amostrados em cada população. (Haplogrupo A-D) frequencia absoluta dos haplogrupos. População N Haplogrupo A Haplogrupo B Haplogrupo C Haplogrupo D Latitude Longitude Ache Akimal O odham Aleuts Aleuts ancient Aleuts St. Paul Anasazi ancient Ancash Aónikenk Apache Arara Arequipa Peru Assurini Assurini Atacameno Atacameno Atacamenos Awá-Guajá Aymara Aymara Baníwa Bella Coola Bella Coola Bella Coola Bella Coola Boruca Bribi-Cabecar Cayapa Cayapa Ecuador Cherokee Cherokee Oklahoma Red Cross Cherokee Stillwell Cheyenne/Arapahoe Cheyenne/Araphoe Chickasaw Chickasaw/Choctaw Chimane Chimila Chippewa Chippewa Turtle Muntain º30 S 55º50 W 0 33º22 N 111º54 W 30 55º00 N 166º14 L 11 53º13 N 168º25 W 38 59º47 N 166º10 W 0 36º28 N 110º30 W 7 9º19 S 77º33 W 11 51º01 S 70º49 W 2 34º36 N 114º16 W 0 03º51 S 52º47 W 2 16º25 S 71º32 W 0 05º26 S 52º37 W 0 03º03 S 49º46 W 3 23º05S 68º0 W 0 22º0 S 68º37 W 3 23º11 S 69º12 W 0 02º18 S 46º18 W 1 18º26 S 67º31 W 24 17º22 S 68º9 W 0 01º33 N 68º44 W 9 52º21 N 128º11 W 2 52º21 N 126º42 W 10 53º43 N 127º38 W 12 50º26 N 124º11 W 1 8º58 N 83º19 W 0 10º55 N 84º01 W 0 0º55 N 79º39 W 9 1º47 S 79º21 W 0 35º35 N 83º12 W 1 36º43 N 98º24 W 0 35º27 N 83º18 W 5 40º07 N 102º04 W 4 38º50 N 102º10 W 0 34º32 N 89º22 W 0 31º39 N 86º01 W 0 16º31 S 66º27 W 2 9º45 N 73º56 W 0 45º54 N 91º15 W 0 46º22 N 84º21 W
156 População N Haplogrupo A Haplogrupo B Haplogrupo C Haplogrupo D Latitude Longitude Chippewa Wisconsin Chippewa/Kickapoo Choctaw Choroti Choroti Chumash Ciboneys Cochimi Coreguaje Creek (Muskoke) Creek/Seminole Curripaco Dogrib Dogrib Embera Embera Embera Ammassalik (Angmagssalik) Gambel Ilulissat (Jakobshavn) Ittoqqortoormiitt (Scoresbysund) Nanortalik Nuuk (Godthab) Savoonga Siberian Upernarik Uummannaq (Umanak) Yupik s s (Savoonga e Gambel) s Old Harbor s Ouzinkie Fueguian Argentina Gaviao Guahibo Guahibo-Sikuani Guambiano Guane-Butaregua Guatuso Guayabero Guaymi Haida Havasupai/Hualapai/ Yavapai/Mojave º0 N 91º29 W 0 40º40 N 89º39 W 1 31º33 N 89º6 W 3 24º31 S 60º24 W 3 22º11 S W 7 34º38 N 120º21 W 5 22º26 N 83º51 W 0 27º45 N 113º26 W 1 0º55 N 76º36 W 38 32º55 N 85º15 W 3 31º22 N 83º25 W 0 1º54 N 68º55 W 0 62º59 N 116º0 W 0 62º55 N 133º41 W 0 6º21 N 75º56 W 2 5º12 N 76º50 W 0 7º54 N 77º49 W 0 66º14 N 36º17 W 13 63º14 N 169º03 W 0 68º5 N 52º5 W 0 70º48 N 23º0 W 0 61º23 N 48º28 W 0 64º41 N 51º49 W 1 63º28 N 171º38 W 16 65º8 N 172º53 W 0 74º15 N 56º45 W 0 69º51 N 53º26 W º3 N 156º32 W 8 65º39 N 172º43 W 15 63º23 N 170º10 W 56 56º59 N 154º19 W 8 57º31 N 153º11 W 25 46º06 S 69º16 W 19 10º10 S 61º08 W 0 5º0 N 67º0 W 0 4º42 N 71º5 W 3 1º47 N 77º7 W 8 6º11 N 74º50 W 0 10º30 N 84º39 W 0 4º14 N 71º54 W 0 7º57 N 81º25 W 2 56º41 N 133º41 W 0 36º18 N 116º05 W 157
157 158 População N Haplogrupo A Haplogrupo B Haplogrupo C Haplogrupo D Latitude Longitude hokan (central coast) º45 N 119º56 W Hokan Northern º17 N 122º5 W Hopi º5 N 110º32 W Huetar º43 N 83º45 W Huetar º42 N 84º18 W Huilliche º16 S 73ºW Huilliche º16 S 73ºW Huilliche Chile º42 S 72º31 W Huitoto º11 S 74º25 W Hupa º12 N 123º39 W Ignaciano º50 S 66º47 W Ijka-Arhuaco º18 N 73º13 W Ingano º3 N 77º13 W Ingano Colombia º51 N 76º6 W Inuit eskimo º52 N 136º11 W Jemez º36 N 106º43 W Kanamarí º37 S 69º32 W Katuena º43 S 58º00 W Katukina º09 S 70º21 W Katukina º17 S 71º34 W Kawéskar º14 S 74º23 W Kaxináwa º07 S 70º19 W Kayapó Kiliwa Kiliwa/Paipai Kiowa Kogui Kraho Kumiai Kuna Kuna Kuna Macushi Makiritare Mapuche Mapuche Mapuche (Chile) Mapuche Anecón Grande Mapuche Argentina Mapuche Chile Marúbo Marúbo Brasil Mataco (Chaco) Mataco (Salta) º00 S 53º00 W 0 31º09 N 116º12 W 0 31º45 N 115º47 W 0 32º06 N 98º50 W 0 10º58 N 72º58 W 0 12º32 S 44º02 W 0 34º32 N 118º21 W 0 8º0 N 77º42 W 0 9º30 N 79º26 W 0 9º02 N 79º29 W 4 4º13 N 61º50 W 1 2º0 N 66º35 W 17 46º06 S 69º11 W 42 39º10 S 68º37 W 15 40º57 S 73º21 W S W 28 40º1 S 69º27 W 54 40º15 S 72º25 W 6 06º47 S 72º08 W 3 7º59 S 70º03 W 15 24º34 S 61º27 W 13 22º16 S 62º42 W
158 159 População N Haplogrupo A Haplogrupo B Haplogrupo C Haplogrupo D Latitude Longitude Mataco (Wichí) Argentina S W Mataco (Wichí) Formosa º22 S 61º58 W Maya º44 N 88º54 W Maya Contenporary º34 N 89º26 W Micmac º52 N 59º3 W Micmac/Narragansett º35 N 61º40 W Mixe º38 N 95º6 W Mixtec º20 N 96º18 W Mohawk º02 N 83º53 W Mohawk º14 N 78º46 W Mosetén Bolivia º31 S 66º27 W Movima Bolívia º48 S 65º33 W Murui-Muinane º10 N 73º30 W Muskoke Oklahoma º44 N 95º22 W Nahua º22 N 101º13 W Nahua/Cora º07 N 97º29 W Navajo º53 N 112º58 W Ngöbé º55 N 82º09 W Nukak º38 N 71º43 W Nuu-Chah-Nulth º24 N 125º05 W Ojibwa Manitoulin º46 N 82º01 W Island Ojibwa North Ontario º36 N 86º34 W Ojibwa/Chippewa º13 N 90º56 W Old Harbor º52 N 152º31 W Ouzinkie º59 N 154º15 W Paez º11 N 76º28 W Paiute N./Shoshoni º54 N 119º27 W Paiute/Shoshone º19 N 113º17 W Parakanã º21 S 52º07 W Pasto º50 N 77º37 W Pawnee º12 N 99º16 W Pehuenche Butalelbum º29 S 72º20 W Pehuenche Trapa Trapa º43 S 71º16 W Peneunche Chile º28 S 71º32 W Penutian California º54 N 121º23 W Penutian California º42 N 122º25 W Piaroa º35 N 68º36 W Piaroa Colombia º01 N 70º29 W Pilagá Formosa º09 S 60º42 W Pima º13 N 117º9 W Pima º30 N 110º46 W Pima º26 N 112º06 W Poturujara (Zoé) º00 S 56º00 W Pyramid Lake ancient º0 N 119º39 W
159 160 População N Haplogrupo A Haplogrupo B Haplogrupo C Haplogrupo D Latitude Longitude Quechan/Cocopa º45 N 118º33 W º20 S 71º32 W (Arequipa) º32 S 72º39 W (Tayacaja) º24 S 74º41 W Bolivia º02 S 64º39 W /Nmatsiquenga º9 S 72º59 W (San Martin) Sahaptian Plateau º21 N 118º59 W Aleut Saint Paul º09 N 170º17 W Salinan/Chumash º47 N 121º41 W Salish Plateau º56 N 121º57 W Selknam º0 S 68º41 W Seminole º16 N 81º2 W Seri º56 N 112º23 W Siona º24 N 76º36 W Siouan º23N 97º38 W Sioux Sisseton/ Wapheton Sioux º31 N 94º6 W Stillwater Marsh ancient º34 N 118º28 W Takik º44 N 116º24 W Taono O odham º20 N 115º41 W (Papago) Tayacaja Peru º53 S 73º16 W Tehuelche Argentina S 71 W Teribe º0 N 83º20 W Teribe º17 N 82º43 W Tikúna º20 S 69º42 W Tikúna Brasil º33 S 71º58 W Tikúna Colombia º37 S 70º10 W Toba º26 S 63º42 W Toba (chaco) º26 S 60º37 W Toba (Formosa) º10 S 58º09 W Trinitario Bolivia º52 S 64º53 W Tucano º45 N 69º42 W Tule-Cuna º19 N 77º12 W Uto-Aztecan California º24 N 119º27 W Uto-Aztecan Central º45 N 111º54 W Uto-Aztecan Northern º13 N 114º28 W Waiãpi º09 N 52º36 W Wapishana Brasil º34S 57º49 W Washo º50 N 120º38 W Waunana º0 N 76º53 W Wayuu º55 N 71º43 W Wayuu Colombia º36 N 72º16 W Wiwa º55 N 73º50 W Wounan º31 N 77º58 W Xavante º20 S 51º40 W
160 161 População N Haplogrupo A Haplogrupo B Haplogrupo C Haplogrupo D Latitude Longitude Yaghan Chile º48 S 68º19 W Yakama º28 N 120º34 W Yakima º45 N 120º44 W Yamamadi º15 S 66º41 W Yámana (Yaghan) º00 S 67º40 W Yaminawa º17 S 71º34 W Yanomama º47 N 62º12 W Yanomama Brasil º39 S 70º30 W Yanomami º51 N 61º28 W Yanomamö º51 N 65º18 W Yok-Utian º13 N 120º28 W Yuko-Yukpa º50 N 72º55 W Yuman º38 N 114º09 W Yuman (Hokan) º4 N 114º13 W Yuman Delta º53 N 114º51 W Yuman Pai º47 N 112º35 W Yuman River º24 N 113º49 W Yuracaré º23 S 65º47 W Zapotec º1 N 95º23 W Zenu º25 N 74º40 W Zoro º20 S 60º20 W Zuni º35 N 111º44 W Zuni (penutian) º52 N 109º4 W
161 Chipewyan Cree Ojibwa Pima Mixtec Zapotec Mixe Maya Kaqchikel Cabecar Guaymi Kogi Arhuaco Waunana Embera Zenu Inga Aymara Huilliche Kaingang Guarani Wayuu Piapoco TicunaT TicunaA Karitiana Surui Ache 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , Chipewyan Cree Ojibwa Pima Mixtec Zapotec Mixe Maya Kaqchikel Cabecar Guaymi Kogi Arhuaco Waunana Embera Zenu Inga Aymara Huilliche Kaingang Guarani Wayuu Piapoco TicunaT TicunaA Karitiana Surui Ache Apêndice III: Matriz de Fst entre populações da América com microssatélites 162
162 163 Chipewyan Cree Ojibwa Pima Mixtec Zapotec Mixe Maya Kaqchikel Cabecar Guaymi Kogi Arhuaco Waunana Embera Zenu Inga Aymara Huilliche Kaingang Guarani Wayuu Piapoco TicunaT TicunaA Karitiana Surui Ache Apêndice III: Matriz de Fst para populações de microssatélite da América Apêndice III: Matriz de Fst para populações de microssatélite da América Chipewyan Cree Ojibwa Pima Mixtec Zapotec Mixe Maya Kaqchikel Cabecar Guaymi Kogi Arhuaco Waunana Embera Zenu Inga Aymara Huilliche Kaingang Guarani Wayuu Piapoco TicunaTarapaca TIcunaArara Karitiana Surui Ache 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,00000 Apêndice IV: Matriz de Rst entre populações da América com microssatélites
163 164 Chipewyan Cree Ojibwa Chipewyan Cree Ojibwa 0, , , , , ,00000 Apêndice V: Matriz de Fst entre populações da América do Norte com microssatélites Chipewyan Cree Ojibwa Chipewyan Cree Ojibwa 0, , , , , ,00000 Apêndice VI: Matriz de Rst entre populações da América do Norte com microssatélites Pima Mixtec Zapotec Mixe Maya Kaqchikel Cabecar Guaymi Pima Mixtec Zapotec Mixe Maya Kaqchikel Cabecar Guaymi 0 0, , , , ,0316 0, , , , , , , , , , , , , , , , , , , , ,0575 0, , Apêndice VII: Matriz de Fst entre populações da América Central com microssatélite Pima Mixtec Zapotec Mixe Maya Kaqchikel Cabecar Guaymi Pima Mixtec Zapotec Mixe Maya Kaqchikel Cabecar Guaymi 0 0, , , , , , , , , , , , ,0456 0, , ,0238 0, , , , , , , , , , , , Apêndice VIII: Matriz de Rst entre populações da América Central com microssatélites
164 165 Kogi Arhuaco Waunana Embera Zenu Inga Aymara Huilliche Kaingang Guarani Wayuu Piapoco TicunaT TicunaA Karitiana Surui Ache Kogi Arhuaco Waunana Embera Zenu Inga Aymara Huilliche Kaingang Guarani Wayuu Piapoco TicunaT TicunaA Karitiana Surui Ache 0 0, , , , , , , , , , , , , , , , , , , , , , , , , ,033 0, , , , , , , , ,0167 0, , , , , , , , , , , , , , , , , , , , , ,0594 0, , , ,0335 0, , , , , , , , , , , , , , , , , ,1307 0, , , , , , , , , , ,0718 0, , , , , , , , , , , , ,0626 0, , , , , , ,0933 0, , , , , , , , , , , , , , , ,1403 0, , , , , , ,1617 0, , , , , , , , ,1305 0,112 0, , , , ,1258 0, , , , , Apêndice IX: Matriz de Fst entre populações da América do Sul com microssatélites
165 166 Kogi Arhuaco Waunana Embera Zenu Inga Aymara Huilliche Kaingang Guarani Wayuu Piapoco TicunaTarapaca TIcunaArara Karitiana Surui Ache Kogi Arhuaco Waunana Embera Zenu Inga Aymara Huilliche Kaingang Guarani Wayuu Piapoco TicunaT TicunaA Karitiana Surui Ache 0 0, ,0506 0, , , , , , , , , ,0964 0, , , , , , , , , , , ,06 0, , , , , , ,0589 0, , , , , , , , ,0976 0, , , , , , ,0814 0, ,0391 0, , , , , , , ,0898 0, , ,0537 0, , , ,0278 0, , , , , , ,0325 0,0268 0, ,024 0, , , , , ,1445 0, , , , , , , , ,0866 0, , , , , , , , , , , , , , , , ,057 0, , ,0507 0, , , , , , , , , , , , , ,0764 0, , ,0635 0, , , , , , , , , , , , , , , , , , , , , , , , ,1325 0, , Apêndice X: Matriz de Rst entre populações da América do Sul com microssatélites
166 167 Salish Plateau Yakama Yakima Nuu- Chah- Nulth Bella Coola Sahaptian Plateau Paiute N./ Shoshoni Uto- Aztecan Northern Salish Plateau Yakama Yakima Nuu- Chah- Nulth 0 0, , , , , , Bella Coola 0, ,3591 0,3445-0, Sahaptian Plateau -0, , , , , Paiute N./ Shoshoni 0, , , , , , Uto- Aztecan Northern 0, , , , , , , Apêndice XI: Matriz de Fst entre populações do conjunto 1 com quatro haplogrupos de DNA mitocondrial Salish Plateau Yakama Yakima Nuu- Chah- Nulth Bella Coola4 Sahaptian Plateau Paiute N./ Shoshoni Uto- Aztecan Northern Salish Plateau Yakama Yakima Nuu- Chah- Nulth 0 0, , , , , , Bella Coola4 0, , , , Sahaptian Plateau -0, , , , , Paiute N./ Shoshoni 0, , , , , , Uto- Aztecan Northern 0, , , , , , , Apêndice XII: Matriz de Fst entre populações do conjunto 1 com cinco haplogrupos de DNA mitocondrial
167 168 Chippewa Chippewa/ Kickapoo Chickasaw Chippewa Chippewa /Kickapoo 0 Chickasaw 0, , , Apêndice XIII: Matriz de Fst entre populações do conjunto 2 com quatro haplogrupos de DNA mitocondrial Chippewa Chippewa/ Kickapoo Chickasaw Chippewa Chippewa /Kickapoo 0 Chickasaw 0, , , Apêndice XIV: Matriz de Fst entre populações do conjunto 2 com cinco haplogrupos de DNA mitocondrial Kayapó Xavante Kraho Kayapó Xavante Kraho 0 0, , , Apêndice XV: Matriz de Fst entre populações do conjunto 3 com quatro haplogrupos de DNA mitocondrial Kayapó Xavante Kraho Kayapó Xavante Kraho 0 0, , , Apêndice XVI: Matriz de Fst entre populações do conjunto 3 com cinco haplogrupos de DNA mitocondrial Fueguian Argentina Mapuche Mapuche Chile Fueguian Argentina Mapuche Mapuche Chile 0 0, , , Apêndice XVII: Matriz de Fst entre populações do conjunto 4 com quatro haplogrupos de DNA mitocondrial
168 169 Fueguian Argentina Mapuche Mapuche Chile Fueguian Argentina Mapuche Mapuche Chile 0 0, , , Apêndice XVIII: Matriz de Fst entre populações do conjunto 4 com cinco haplogrupos de DNA mitocondrial Atacameno Atacamenos Aymara Atacameno Atacamenos Aymara 0-0, , , Apêndice XIX: Matriz de Fst entre populações do conjunto 5 com quatro haplogrupos de DNA mitocondrial Atacameno Atacamenos Aymara Atacameno Atacamenos Aymara 0-0, , , Apêndice XX: Matriz de Fst entre populações do conjunto 5 com cinco haplogrupos de DNA mitocondrial Movima Bolívia Murui- Muinane Pilagá Formosa Zoro Movima Bolívia 0 Murui- Muinane 0, Pilagá Formosa Zoro 0, , , ,0985 0, Apêndice XXI: Matriz de Fst entre populações do conjunto 6 com quatro haplogrupos de DNA mitocondrial Movima Bolívia Murui- Muinane Pilagá Formosa Zoro Movima Bolívia 0 Murui- Muinane 0, Pilagá Formosa Zoro 0, , , , , Apêndice XXII: Matriz de Fst entre populações do conjunto 6 com cinco haplogrupos de DNA mitocondrial
169 Figura esquemática da localização original dos apêndices XXIII e XXIV A-AC da matriz de DNA mitocondrial de populações da América (n10). 170
170 171 Ache Akimal O odham Aleuts St. Paul Ancash Aónikenk Apache Arara Arequipa Peru Assurini Atacameno Atacameno Atacamenos Awá-Guajá Aymara Aymara Ache Akimal O odham Aleuts St. Paul Ancash Aónikenk Apache Arara Arequipa Peru Assurini Atacameno Atacameno Atacamenos Awá-Guajá Aymara Aymara Baníwa Bella Coola Bella Coola Bella Coola Bella Coola Boruca Bribi-Cabecar Cayapa Cayapa Ecuador Cherokee Cherokee Oklahoma Red Cross Cherokee Stillwell Cheyenne/ Arapahoe Cheyenne/ Araphoe Chickasaw/ Choctaw Chimane Chimila Chippewa Chippewa Turtle Muntain Chippewa Wisconsin Chippewa/ Kickapoo Choctaw Choroti Choroti Chumash Ciboneys Cochimi Coreguaje Creek (Muskoke) Creek/Seminole Curripaco Dogrib Dogrib Embera Embera Embera Gambel Ilulissat (Jakobshavn) Ittoqqortoormiitt (Scoresbysund) Nuuk (Godthab) Savoonga Siberian Upernarik Uummannaq (Umanak) Yupik s s (Savoonga e Gambel) s Old Harbor s Ouzinkie Fueguian Argentina Gaviao Guahibo Guahibo-Sikuani Guambiano 0 0, , , , , , , , , , ,587 0, , ,2603 0, ,6995 0, , , ,482 0, , , , , , , , ,5424 0, , , , , , , , , , , , ,3692 0, , , , , , , , , , , , , , , ,5174 0, , , , , , , , , ,3146 0,714 0, , , , , ,5605 0, , , , ,2784 0, , , , , , , , , , , , , ,4588 0, , , , , , , , , , , , , , , , , , ,1981 0, ,2315 0, , , , , , , , , , , , , , , , , , , , ,7067 0,4704 0,4377 0, , , , , , , , , , ,7324 0, , , , , , ,0376 0,4206 0, , ,3873 0, , , , ,3749 0, ,3791 0, , , , , , , , , , , ,5712 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0778 0, ,144 0, , , , , , , ,2482 0, , , , ,0784 0, , ,261 0, , , , , , , , , , ,5372 0, ,4469 0, , , , , , , , , , , , , , , , ,0184 0, , , , , , ,5571 0, , , , , , , , , , , , , , , , , , , , , ,0578 0, , , , , , , ,5181 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,8106 0, , , , , , , , , , , , , , , , , , , , ,1065 0, , , ,2155 0, , ,6263 0, , , , ,2338 0, ,0271 0, ,3044 0, , , , ,6164 0, , , , , , ,3599 0, , , , , , , , , , , , , , , , , , , , , , ,5889 0, , , , , , ,558 0, , , , , , , , , , , , , , , , , , ,0104 0, , ,0301 0, , , , , ,3481-0, , , , , , , , , , , , , , , , , , , , , , , , ,6824 0, , ,7205 0,2662 0, ,2364 0, , , , , , , , , , , , , , , , , , , ,0788 0, , , , , , , , , , , , , , , , , , , , , , , , , , ,0919 0, ,2179 0, , , , ,4389 0, , , , , , , , , , , , ,2104 0, , , , , , , , , , , , , , ,0669 0, , , ,2024 0, , , , , , , ,8598 0, , , , , , , , ,7128 0,6717 0, ,8823 0, , , , ,7199 0, , , , , , ,4731 0, , ,0275 0, , , ,417 0, , , , , , , , ,0324 0, , , , ,103 0, , , , , ,079 0,2274 0, ,4414 0, , , , , , , , , , , , , , , , , , , , , , , , , , , ,8505 0, , , , , , , , , ,6934 0, , , ,8505 0, , , , , , , , , ,6934 0, , , ,8505 0, , , , , , , , , ,6934 0, , , ,8705 0, , , , , , , , , ,7899 0, , , , , , , , , ,0734 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,6491 0, , , , , , , , , , , , , , , , ,4397 0, , , ,6814 0, , , , , , , , , , , , , , ,6134 0, , , , , , , , , , , , , ,1542 0, , , , , , ,6073 0, , , , , , , , , , , , ,4825 0, , , , , , , , , , , , , ,4778 0,4814 0, , , , ,6661 0, , , , , ,5305 0,3912 0, , , , , , , , , , ,2955 0, , , , , , , , ,6274 0, , , , , , ,6294 0, , , ,3784 0, , , , , , , , , , , , ,0162 0, , , , , , , ,45001 Apêndice XXIII A: Matriz de Fst para populações de DNA mitocondrial com quatro haplogrupos de n10
171 172 Guane- Butaregua Guatuso Guayabero Guaymi Haida Havasupai/ Hualapai/ Yavapai/Mojave hokan (central coast) Huetar Huetar Huilliche Huilliche Huilliche Chile Huitoto Ignaciano Ijka-Arhuaco Ingano Ingano Colombia Inuit eskimo Jemez Kanamarí Katuena Katukina Katukina Kawéskar Kaxináwa Kayapó Kiliwa/Paipai Kogui Kraho Kumiai Kuna Kuna Kuna Mapuche Mapuche Mapuche (Chile) Mapuche Anecón Grande Mapuche Argentina Mapuche Chile Marúbo Mataco (Chaco) Mataco (Salta) Mataco (Wichí) Argentina Mataco (Wichí) Formosa Maya Maya Contenporary Mixe Mixtec Mohawk Mohawk Mosetén Bolivia Movima Bolívia Murui-Muinane Muskoke Oklahoma Nahua Nahua/Cora Navajo Ngöbé Nukak Nuu-Chah-Nulth Ojibwa Manitoulin Island Ojibwa North Ontario Ojibwa/ Chippewa Old Harbor Ouzinkie Paez Paiute N./ Shoshoni Parakanã Pehuenche Butalelbum Pehuenche Trapa Trapa Peneunche Chile 2 Ache Akimal O odham Aleuts St. Paul Ancash Aónikenk Apache Arara Arequipa Peru Assurini Atacameno Atacameno Atacamenos Awá-Guajá Aymara Aymara 0, , ,4009 0, , , , , , , , , , , , , , , ,4179 0, , , , , , , , , , , , , , , , , ,3949 0, , , , , , , , , , , , , , , , , , , , , , , ,7608 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,2388 0, , , , ,7107 0, , , , ,3444 0,4584 0, , , , , , , , , , , , , , , , ,282 0, , , , ,242 0, , , , , , ,0799 0, ,293 0, , , , , , , , , , ,2115 0, , , , ,2857 0, , , , ,2497 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,5243 0, , , , , , , ,5642 0,5397 0, , , , , , , , ,3991 0, , , , , , , , , , , , , , , , , , , ,1045 0, , , ,3377 0, , , , , , , , ,7045 0, , , , , , , , , , , , , , , , , ,2385 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,6205 0, , , , , , , , ,163 0, , , , , , , , , , , , , , , , , , ,3584 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,1985 0, , , ,4729 0, ,4685 0, , , , ,0602 0, , , , , , , , ,0577 0, , , ,037 0, , , , , , , , , , , , , , , , ,6252 0, ,5533 0, , , , , , , , , , , , , , , , , ,3813 0, , ,2988 0, , , , , , , , , , , , , , , , , , , ,313 0,0901 0,2575-0, , , , , , , , , , , , ,3176 0, , , , , , ,1067-0, ,1566 0, , , , , , , , , , , , ,1895-0, ,2563 0,2378 0, , , , , , , , , , , ,0551-0, , , , , , , , , , , , ,2273 0, ,0806-0, , , , ,3081 0, , , , , , , , , , , , ,4033 0, , , , ,618 0, , , , , , , , , , , ,6261 0, , , , , , , , , , , , , , , ,4644 0, , ,1459 0, , , , , , ,1647 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0256 0, , , , , , , , , , ,0035 0, , ,1522 0,2666 0, ,2585 0, ,4523 0, , , , , ,39-0, , , , , ,1717 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,325 0, , , , , ,336 0, , , , , , , , , , , , , , ,2946 0, ,3187 0, ,3106 0,5078 0, , , , , , , , , , ,2145 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , ,1649-0, , , , , , , , , , , , , , , , , , , , , ,2658 0, , , , , , , , , , , ,2664 0,5542 0,5359 0, , , , , , , , , , , , , , , , , , , ,1689 0, , , , ,217 0,1958 0, , , , , , , , , , , ,5251 0,3279 0, , , , , , , , ,2024 0, , , , , , , , ,4897 0, , , , , , , , , , ,3269 0, , , , , , , ,327 0, , , , , ,0211 0,2036 0, , , ,245 0, , , , , , , , , , , , , , , , , , , , , , , , , , ,4036 0, ,4286 0, ,4175 0, , , , , , , , , , , , , , , , , , , , , , ,5627 0, , , , , , , , , , , , ,4204 0,2817 0, , , , , ,4082 0, ,4008 0, , , , , , , , , , , , , , , , , , , , , , , ,2194 0, , , , , , , , , ,7122 0, , , , , , , , , , , , , , , , , ,1707 0, , , ,2987-0, , , , , , , , , ,1869 0, , , , , ,0036 0, , , , , ,31402 Apêndice XXIII B: Matriz de Fst para populações de DNA mitocondrial com quatro haplogrupos de n10
172 173 Penutian California Penutian California Piaroa Pilagá Formosa Pima Pima Pima Poturujara (Zoé) Quechan/ Cocopa (Arequipa) (Tayacaja) Bolivia / Nmatsiquenga (San Martin) Aleut Saint Paul Salinan/ Chumash Salish Plateau Selknam Seminole Siona Siouan Sioux Sisseton/ Wapheton Sioux Takik Taono O odham (Papago) Tayacaja Peru Tehuelche Argentina Teribe Teribe Tikúna Tikúna Brasil Tikúna Colombia Toba (chaco) Toba (Formosa) Trinitario Bolivia Tucano Tule-Cuna Uto-Aztecan California Uto-Aztecan Central Uto-Aztecan Northern Waiãpi Wapishana Brasil Washo Waunana Wayuu Wayuu Colombia Wounan Xavante Yaghan Chile Yakama Yakima Yámana (Yaghan) Yaminawa Yanomama Yanomama Brasil Yanomami Yanomamö Yok-Utian Yuko-Yukpa Yuman Yuman (Hokan) Yuman Delta Yuman Pai Yuman River Yuracaré Zapotec Zenu Zoro Zuni Zuni (penutian) Ache Akimal O odham Aleuts St. Paul Ancash Aónikenk Apache Arara Arequipa Peru Assurini Atacameno Atacameno Atacamenos Awá-Guajá Aymara Aymara 0, , , , , , , ,1073 0, , , , , , , ,4145 0, , , , , , , , , , , , , , , ,1643 0, , , , , , ,0486 0, ,1875 0, , , , , , , , , , , , , , , , , ,3262 0, , , ,5127 0, , ,3253 0, , , ,0788 0, , , , ,0717 0, , , , , , , , ,1171 0, , , , , ,1085 0, , , , , , , , , , , , , , , , ,3113 0, , , ,0549 0, , ,0383 0, , , , , , , , , , , , , ,0052 0,2057 0, , , , , , , , , ,022 0, , , ,0047 0, , ,0325 0, , , , , , , , , , , , , , , , , , , , , , , ,1854 0, , , , , , ,1405 0,335 0, , , , , , , , , , , , , ,0198 0, , , , , ,3502-0,0187 0, , , , , , , , , , , , , ,0156 0, , , , , , , , , , , , , , , , , , , , , , , , , , , ,2362 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,4678-0, , , , , , , , , ,3209 0, , , , ,5618-0, , , ,2619 0, , , , , , , , , , ,4132-0, , , , , , , , , , , , , , , , , , ,1484 0, , , , , , ,6156 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,1854 0, , , , , , ,1405 0,335 0, , , , , ,1229 0, , ,3093 0, , , , , , , , , ,459 0, , , , , , , , , , , , ,44 0, , ,6077 0, , , , ,7014 0,6001 0, , , , , , , , , , , , , ,0115 0, , ,3122 0, , , , , , , , , ,287 0, , , , , , , , , , , , , , , , , , ,2508 0, ,4803 0, , , , , , , , , , , , , , , , , , , , , , , , , ,0092 0, , , , , ,141 0, , , , , , , , , , , , , , , , , ,3076 0, ,1193 0, , , , ,3249 0, , , , , , , , , , , ,3025 0, , , , , , , , , , , ,0411 0,1723 0,2854 0, , , , , , , , , , , ,5127 0, , ,3253 0, , , ,0788 0, , , , ,0717 0, , , , , , , , , , , , , ,2921 0, , , , , , , , , , , , , , , , , , , , , , ,4632 0, , , , , , , , , , , , , , , , , , , ,0888 0, ,2732 0, , , , , , , , , , ,0359 0, , , , , , , , , , , , , , , , , , ,628 0, , , , , , ,141 0, , , , , , , , , , ,117 0, , , , , ,2265 0, , , ,283 0, , , , , , , , , , , , ,0067-0, ,0052 0, , ,0484 0, ,3341 0, , , , , , ,0393 0, , , ,7423 0, , , , , , , , , , , , , , , ,0925-0, , , , , , , , ,0232 0, , , , , , ,0122 0, , , , , , , , , , , , ,733 0, , , , , , , , , ,3947 0, , , , , , , , ,0621 0, ,1219 0, , , , , , , , , , , , , , , , , , , , ,3439 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,2567 0, , , ,2282 0, , , , , ,0998 0, , ,4661 0, , , , , , , , , ,1703 0, , , , , , , , ,1848 0, ,0878 0, , ,2162 0, , , , , , , , , , , , , , , , , , , , , , , , ,1539 0, , , , , , , , , , , , , , , , , ,0114 0,2149 0, , , , , , , , , , , ,0636 0, , , , , , , , , , , , , , , , , ,437 0, , , , , , , , , , , , , , ,5102 0, , , , , , , , , , , , , , , , , , , , , , ,284 0, , , , , , , , , , ,1232 0, ,0734 0, ,3844 0,4668-0, , ,0209-0, , , , , , , , , , , , , , , , , , , ,01055 Apêndice XXIII C: Matriz de Fst para populações de DNA mitocondrial com quatro haplogrupos de n10
173 174 Baníwa Bella Coola Bella Coola Bella Coola Bella Coola Boruca Bribi-Cabecar Cayapa Cayapa Ecuador Cherokee Cherokee Oklahoma Red Cross Cherokee Stillwell Cheyenne/ Arapahoe Cheyenne/ Araphoe Chickasaw/ Choctaw Chimane Chimila Chippewa Chippewa Turtle Muntain Chippewa Wisconsin Chippewa/ Kickapoo Choctaw Choroti Choroti Chumash Ciboneys Cochimi Coreguaje Creek (Muskoke) Creek/ Seminole Curripaco Dogrib Dogrib Embera Embera Embera Gambel Ilulissat (Jakobshavn) Ittoqqortoorm iitt (Scoresbysun d) Nuuk (Godthab) Savoonga Siberian Upernarik Uummannaq (Umanak) Yupik s s (Savoonga e Gambel) s Old Harbor s Ouzinkie Fueguian Argentina Gaviao Guahibo Guahibo- Sikuani Guambiano Baníwa Bella Coola Bella Coola Bella Coola Bella Coola Boruca Bribi-Cabecar Cayapa Cayapa Ecuador 0 0, , , , , , ,0449 0,0106 0, , ,1684 0, , , , , , , , , , , , , , , , , Cherokee 0,0554 0, , , , , , , , ,3203 0, , , , , , , Cherokee Oklahoma Red Cross 0,1162 0, , , , , , , , , Cherokee Stillwell 0, , , , , , , , , , , Cheyenne/ Arapahoe 0, ,0357 0, , , , , , , , , , Cheyenne/ Araphoe 0, , , , , , , , , , , , , Chickasaw/ Choctaw -0, , , , , , , ,1194 0, , , , , , , , , ,2191 0, , , ,0121 0, , , , , , , , , , , , , ,3449 0, , , , , , ,3603 0, ,0235 0, , ,1366 0, , , , , , , , , , , , , , , , , ,06 0, ,0836 0, , , , , , , , , , , , , , , , , , , , , , , , , , , ,1073 0, , , , , , , , , , , , , , ,0781 0, , , , , , , , ,1032 0, ,3734 0, , , , , ,0365 0, , , , , , , , , , , , , , , ,0054 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,3444 0, , , ,4225 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0052 0,2352 0, ,1098-0, ,2187 0,0774 0, , , , , , , , , , , ,0005 0,1101 0, , , , , ,0398 0, , , , , , , , , , , , , , ,3845 0,4326 0, , , , , , , , , , ,6752 0, ,5362 0, , ,111 0,0175 0, , , , , , , , , , , , , , , , ,1627-0, , ,0181 0,0559 0, , , , ,1186 0, , , ,3304 0, , , , , , , , , , ,1134 0, , , ,0547-0, , , , , , , , ,3278 0, , , , , , , ,119 0, , , , , , , , , , , , , , ,119 0, , , , , , , , , , , , , , ,119 0, , , , , , , , , , , , , , , , , , , , , , ,4507 0, , , , , , , , , , ,2028 0, ,4172 0, , ,2542 0,0954 0, ,2066 0, , , , , , , , , , , , , , ,2066 0, , , , , , , , , , , , , , , ,4299 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,3308 0, , , , ,1619 0, , , , ,4937 0, , , , , , , , , ,3966 0, , , , , , , , , , , , , , , ,2241 0, , , , , , , , , , , ,258 0, , , , , , , , , ,1476 0, ,054 0, , , , , , , , , , , , , , , , ,188 0, , , , , , , , , , , , , , , ,1572 0,51825 Apêndice XXIII D: Matriz de Fst para populações de DNA mitocondrial com quatro haplogrupos de n10
174 175 Guane- Butaregua Guatuso Guayabero Guaymi Haida Havasupai/ Hualapai/ Yavapai/ Mojave hokan (central coast) Huetar Huetar Huilliche Huilliche Huilliche Chile Huitoto Ignaciano Ijka-Arhuaco Ingano Ingano Colombia Inuit eskimo Jemez Kanamarí Katuena Katukina Katukina Kawéskar Kaxináwa Kayapó Kiliwa/Paipai Kogui Kraho Kumiai Kuna Kuna Kuna Mapuche Mapuche Mapuche (Chile) Mapuche Anecón Grande Mapuche Argentina Mapuche Chile Marúbo Mataco (Chaco) Mataco (Salta) Mataco (Wichí) Argentina Mataco (Wichí) Formosa Maya Maya Contenporary Mixe Mixtec Mohawk Mohawk Mosetén Bolivia Movima Bolívia Murui- Muinane Muskoke Oklahoma Nahua Nahua/Cora Navajo Ngöbé Nukak Nuu-Chah- Nulth Ojibwa Manitoulin Island Ojibwa North Ontario Ojibwa/ Chippewa Old Harbor Ouzinkie Paez Paiute N./ Shoshoni Parakanã Pehuenche Butalelbum Pehuenche Trapa Trapa Peneunche Chile 2 Baníwa Bella Coola Bella Coola Bella Coola Bella Coola Boruca Bribi-Cabecar Cayapa Cayapa Ecuador Cherokee Cherokee Oklahoma Red Cross Cherokee Stillwell Cheyenne/ Arapahoe Cheyenne/ Araphoe Chickasaw/ Choctaw 0, , , , , , , , , , , , , ,2475 0, , ,1383-0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0311 0, , , , , , , , ,174-0, , , , ,1296 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0411 0,0045 0, , , , , , , ,0089 0, ,0525 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,2349 0, , , , , , , , , , , , , , , , , , , , , , , , , ,2125 0, ,2277 0, , ,0683 0, ,1851 0, , , , , , , , , , , , , , , ,2385 0, , , , , , , , , , , , , , , , ,0368 0, , , , , , , , ,3085 0, ,5902 0, , , , , , ,1298 0, , , , , , , ,1598-0, , , , , , , , , , , , , , ,0396 0, , , , , , , , , , , , , , , , , , , , , ,6218 0,7642 0, , , , , , , , , , , , , , , , , , , ,0661-0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,2 0, , , , , , ,0995-0, , , , , ,1667 0, ,4972 0, , , , , , , , , , ,6101 0, , , , , , , , , , , , , , , ,2379 0, , , , , , , , ,0136 0, , , ,1604 0, , ,0253 0,1107 0, , , , , , , , , , , , , , , , , , , , , , , , , , ,482 0, , , , , , , , , , , ,3388 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,122 0, , , , ,7787 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,3606 0, , , ,2016 0, , , , , ,1091 0, ,2798 0, , , , , , , , , , , ,0774 0, , , , , , , , , , , , , , , , , , , , ,3071 0, , , , , , , , , , , , , , , ,184 0, , ,1962 0, , , , , , , ,2621 0, , , , , , , , , , , , , , , , , , , , , , , , ,0932 0, , , , , , , , ,2099 0, , , , , , , ,2707 0, , , , , , ,3407 0, , , , , , , , , , , ,2656 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0519 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0717 0, , , , , , , , , , ,1059 0,1184-0, , , , , , , , , , , , , , , , , , , , ,0209 0, , , , , , , , , , , , , , , , , , , ,2773 0, , , , , , , , , , , , , , , , , , , ,136 0, , , , , , , , , , ,0024 0, , ,0514 0, , , , ,1663 0, , , , , , , , , , , , , , ,0182 0, , , , , ,261 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0043 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,524 0, , , , ,1453 0, , , , , , , , , ,1484 0, , , , , , , , , , , , , , , ,1371 0, , ,0216 0, , , , , , , , , , , , , , , , ,2067 0, , , , , , , , , , , , , , , ,1026 0, ,1646 0, , ,0051 0, , , ,2746 0, , ,3354 0, , , , , , , , , , , ,2968 0, , , , , , , ,0405 0,0618 0, , , , , , , ,3009 0, , , , , , , , , , , , , , ,2221 0, , ,2335 0, , , , , , ,2662 0, , , , , , , ,0167-0, , , , , , ,4105 0, , , ,1644 0, , , , , , , ,2115 0, ,2403 0, ,3591 0, , , , ,1276 0, , , , , , , , , , , , , , ,1482 0, , , ,38791 Apêndice XXIII E: Matriz de Fst para populações de DNA mitocondrial com quatro haplogrupos de n10
175 176 Penutian California Penutian California Piaroa Pilagá Formosa Pima Pima Pima Poturujara (Zoé) Quechan/ Cocopa (Arequipa) (Tayacaja) Bolivia / Nmatsiqueng a (San Martin) Aleut Saint Paul Salinan/ Chumash Salish Plateau Selknam Seminole Siona Siouan Sioux Sisseton/ Wapheton Sioux Takik Taono O odham (Papago) Tayacaja Peru Tehuelche Argentina Teribe Teribe Tikúna Tikúna Brasil Tikúna Colombia Toba (chaco) Toba (Formosa) Trinitario Bolivia Tucano Tule-Cuna Uto-Aztecan California Uto-Aztecan Central Uto-Aztecan Northern Waiãpi Wapishana Brasil Washo Waunana Wayuu Wayuu Colombia Wounan Xavante Yaghan Chile Yakama Yakima Yámana (Yaghan) Yaminawa Yanomama Yanomama Brasil Yanomami Yanomamö Yok-Utian Yuko-Yukpa Yuman Yuman (Hokan) Yuman Delta Yuman Pai Yuman River Yuracaré Zapotec Zenu Zoro Zuni Zuni (penutian) Baníwa Bella Coola Bella Coola Bella Coola Bella Coola Boruca Bribi-Cabecar Cayapa Cayapa Ecuador Cherokee Cherokee Oklahoma Red Cross Cherokee Stillwell Cheyenne/ Arapahoe Cheyenne/ Araphoe Chickasaw/ Choctaw 0, , , , , , , , , , , , , ,1681 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,032 0, ,171 0, , , , , , , , , , , , ,0786 0, , , , ,3614 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,3603 0, , , , , ,0571-0, , , , , ,0238 0, , , , , , , , , ,2417 0, , , , , , , , , , , ,2441-0, ,1895 0, ,2569 0, , , , , , , , , , , , ,1522 0, , , , ,1981 0, , , , , , , , , , , , , , , , , ,1058 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0411 0,0045 0, , , , , , , ,0089 0, ,0525 0, , , , , , , , , , , , , , , , , , , , ,4739 0,4708 0,3864 0, , , , , , , , , , , , , ,0204 0, , ,3389 0, , , , , , , , , , , , , , ,435 0, , ,1215 0, , , , , ,0309-0, , , , , , , , , , , ,0221 0, , ,0468 0, , , , , , ,1366 0, , , ,0048 0, , , , , , , , ,108 0, , , , , , , , , , , , , ,1415 0, , , , , , , , , , ,1058 0, , , , , , , , , , , , , , , , , , ,3044 0, , ,2007 0, , , , , , , ,1148 0, , , ,1911 0, , , , , , , , , , , , , , , , , , , , , , ,156 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,2637 0, ,0836 0, , , , , , , , , ,1725 0, ,0884 0, , , , , , , , , , , , ,0249 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,2419 0, , , , , , ,0707 0, , , , , ,3614 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,1322 0, , , , ,1652 0, , , , , , , , , , , , , , , , , , , , , , , , , , ,0864-0, , , , , , , , , , , , , , ,0129-0,0042 0, ,0364 0, , , , , , , , , ,1507 0, , ,0371 0, , , , , , , , , , , , , , , , , ,3993 0, , , , , , , , , , , , , , , , , ,3591 0, , , , , , , , , , , , , , ,3445 0, , , , ,2254 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,1714 0, ,0017-0,0403 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,262 0, , , , , , , , , , , ,1349 0, , , , ,1944 0, , , , , , , , , , , , , , , , , , , ,5094 0,7284 0, , , , , , , , , , , , , , , , ,2384-0, , , , , , , , , , ,4081 0, , , , ,0135 0, , , , , , ,3533 0,5201 0,3915 0, , ,3036 0, , , , , , , , , , , , , , , ,0848 0, , , , , , , ,2475 0, , , , , , , , , , , ,2294 0, , , , , , , , , , , , , ,0784 0, , , , , , , , , ,0821 0, , , , , , , , , , ,3085 0, , , , , , ,002 0, , ,0605 0, , , , , , , , , , ,0568 0, ,2523 0,3233 0,1545 0, , ,2401 0, , , , , , , ,2593 0, , , , , , , , , , , , , , , , , , , , ,30378 Apêndice XXIII F: Matriz de Fst para populações de DNA mitocondrial com quatro haplogrupos de n10
176 177 Chimane Chimila Chippewa Chippewa Turtle Muntain Chippewa Wisconsin Chippewa/ Kickapoo Choctaw Choroti Choroti Chumash Ciboneys Cochimi Coreguaje Creek (Muskoke) Creek/ Seminole Curripaco Dogrib Dogrib Embera Embera Embera Gambel Ilulissat (Jakobshavn) Ittoqqortoorm iitt (Scoresbysun d) Nuuk (Godthab) Savoonga Siberian Upernarik Uummannaq (Umanak) Yupik s s (Savoonga e Gambel) s Old Harbor s Ouzinkie Fueguian Argentina Gaviao Guahibo Guahibo- Sikuani Guambiano Chimane Chimila Chippewa Chippewa Turtle Muntain 0 0, , , , ,1615 0, Chippewa Wisconsin 0, , , , Chippewa/ Kickapoo Choctaw Choroti Choroti Chumash Ciboneys Cochimi Coreguaje Creek (Muskoke) 0, , , , , ,1797 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,1093 0, , , , , , , , , , , , , ,0223 0,3063 0, , , , , , , , , , , , , , , , , , , , , , , , , , Creek/ Seminole 0, ,2563 0, ,0003 0, , ,0656 0, , , , , , , , ,3766 0, , , ,1063 0, , , , , , , , , , , ,7786 0, , , , ,8367 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,013 0, , , ,121 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0099 0, , ,51 0, , , ,1186 0, , , , , , , ,3125 0, , ,51 0, , , ,1186 0, , , , , , , ,3125 0, , ,51 0, , , ,1186 0, , , , , , , ,3125 0, , , , , , , , , , , , , , , , , , , , , , ,4287 0, , , ,5272 0, , , , , , , , , , , , , , , , ,2776 0, , , , , , , , , , , , , , ,2776 0, , , , , , ,4126 0,4317 0, , , , , , , , ,3833 0, , , , , , , , , , ,5854 0, , , , , , , , , , , , , , , , , , , , , ,1897 0,2766 0, , , , , , , , , , , , , , , , , , , , , ,498 0, , , , , , , , , , , , , , , , , , , , , , , , , , , ,2023 0,3178 0, , ,1086 0,2286 0, , , , ,0102 0, , , ,1798 0, , , , , ,0696 0, ,0139 0, , , , , , , ,15 0, , ,5258 0, , , , , , , , , , , ,3904 0, , ,0261 0, ,31339 Apêndice XXIII G: Matriz de Fst para populações de DNA mitocondrial com quatro haplogrupos de n10
177 178 Guane- Butaregua Guatuso Guayabero Guaymi Haida Havasupai/ Hualapai/ Yavapai/ Mojave hokan (central coast) Huetar Huetar Huilliche Huilliche Huilliche Chile Huitoto Ignaciano Ijka-Arhuaco Ingano Ingano Colombia Inuit eskimo Jemez Kanamarí Katuena Katukina Katukina Kawéskar Kaxináwa Kayapó Kiliwa/Paipai Kogui Kraho Kumiai Kuna Kuna Kuna Mapuche Mapuche Mapuche (Chile) Mapuche Anecón Grande Mapuche Argentina Mapuche Chile Marúbo Mataco (Chaco) Mataco (Salta) Mataco (Wichí) Argentina Mataco (Wichí) Formosa Maya Maya Contenporary Mixe Mixtec Mohawk Mohawk Mosetén Bolivia Movima Bolívia Murui- Muinane Muskoke Oklahoma Nahua Nahua/Cora Navajo Ngöbé Nukak Nuu-Chah- Nulth Ojibwa Manitoulin Island Ojibwa North Ontario Ojibwa/ Chippewa Old Harbor Ouzinkie Paez Paiute N./ Shoshoni Parakanã Pehuenche Butalelbum Pehuenche Trapa Trapa Peneunche Chile 2 Chimane Chimila Chippewa Chippewa Turtle Muntain Chippewa Wisconsin Chippewa/ Kickapoo Choctaw Choroti Choroti Chumash Ciboneys Cochimi Coreguaje Creek (Muskoke) Creek/ Seminole 0, , , , , , , , , , , , , , , , , ,3178 0, , , ,022 0, , , , , , ,2064 0, , , , , ,1136-0, , , , , , , , , , , , , , , , , , , , , , , , , ,3633-0, , , , , , , , , , , ,5868 0, , , , , ,2196 0, , , , , ,2977 0,2199-0, , , , , , , , ,1149 0, ,0668 0,056 0, , ,2131 0, , , , , , , , , , , , , ,0077 0, , , , , , , ,2489 0, , , , , , , , , , , ,0449 0, ,4835 0, , , , ,361 0, , , , , , ,0874 0, , , , , , , ,3624 0, , , , , , , ,142 0, , , , , ,3173 0, , , ,1916 0, , , , , , , , , , ,1961 0, , , , , , , , , , ,5286-0, , , , , , , , , , , , , , , , , , , , , , , ,5321 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,5747 0, , , , , , , , ,6829 0, , , , , , , ,7613 0, , , , , , , , , , ,1434 0, , , , , , , , , ,4498 0, , , ,1492 0, , , ,0557 0, , , , , , , , , , , , , , , , , , , , , ,0886 0,2311 0, , , ,0497 0, , , , , , , , , , ,5133 0, , , , , , , , , , , , , ,3851-0, , , , , , , , , , , , ,0182-0, , , , , , ,1411 0, , , , , , , ,1055 0, , ,45 0, , , , , , , , , , , , , , , ,197-0, ,1615 0, , , , , , , , , , , , , , , , , ,008 0, , , , , , , , ,1762 0, , , , , ,0229 0, , , , , , , , , , ,3976 0, , ,6028 0, , ,7067 0, , , , , , , , , , , , , , , ,4049 0, , ,117 0, , , ,0489 0, , , , ,3676 0, , , , , , , , , , , ,2526 0, , , ,1922 0, , , , , , , , ,2131 0, , , , , , , , , , , ,2594 0, , , , , , , , , , , , , , , , , , , , , , , , , , ,1945 0, , , , , , , , , , , ,1727 0, , , , , ,4026 0,5514 0, , , , , , , , , , , , , , , , , , ,3044 0, , ,0523 0, , , , , , , , , , , , , ,0576 0, ,2043 0, , , , , , , , , , , , , , , , , , , , , ,5917 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,3171 0, , , , , , , , ,016 0, , , , , , , , , , ,1088-0, , , , , , , , , , , , , , , , , , , ,4185 0, , , , , , , , , , , , , , , , , , , , , , , , , , ,0774-0, , , , , , , , ,1059 0, , , , , , , , , , , , , , , ,1455 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0542 0, , , , , , , , , ,008 0, , ,3107-0, , , ,2052 0, , , , ,0038 0, , , , , , , , , , , , , , , , ,1362 0, , , , , , , , , , , , , , , , , , , , , , , , , , , ,0012 0,2456 0, , ,4871 0, , , , , , , ,4297 0, ,3974 0, , , , , , , , , , , , , , ,0617 0,0907 0, , , , , , , , , , ,0539 0, , , , , , , , , , , , , , , , , , , , ,3793 0, , , , , , , , ,0875-0, , , , , , , , , , , , , ,1546 0, , , , ,1042 0,2952 0, , , , , , , , , , , , , , , ,3776 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,1894 0, , , ,118 0, , , , , , , ,4933 0,194 0, , , , , , , , , , , ,1736 Apêndice XXIII H: Matriz de Fst para populações de DNA mitocondrial com quatro haplogrupos de n10
178 179 Penutian California Penutian California Piaroa Pilagá Formosa Pima Pima Pima Poturujara (Zoé) Quechan/ Cocopa (Arequipa) (Tayacaja) Bolivia / Nmatsiqueng a (San Martin) Aleut Saint Paul Salinan/ Chumash Salish Plateau Selknam Seminole Siona Siouan Sioux Sisseton/ Wapheton Sioux Takik Taono O odham (Papago) Tayacaja Peru Tehuelche Argentina Teribe Teribe Tikúna Tikúna Brasil Tikúna Colombia Toba (chaco) Toba (Formosa) Trinitario Bolivia Tucano Tule-Cuna Uto-Aztecan California Uto-Aztecan Central Uto-Aztecan Northern Waiãpi Wapishana Brasil Washo Waunana Wayuu Wayuu Colombia Wounan Xavante Yaghan Chile Yakama Yakima Yámana (Yaghan) Yaminawa Yanomama Yanomama Brasil Yanomami Yanomamö Yok-Utian Yuko-Yukpa Yuman Yuman (Hokan) Yuman Delta Yuman Pai Yuman River Yuracaré Zapotec Zenu Zoro Zuni Zuni (penutian) Chimane Chimila Chippewa Chippewa Turtle Muntain Chippewa Wisconsin Chippewa/ Kickapoo Choctaw Choroti Choroti Chumash Ciboneys Cochimi Coreguaje Creek (Muskoke) Creek/ Seminole 0, , , , , , , , , ,1982 0, , , , , , , , , , , , , , , ,2384 0, ,3351 0, , , , , , , , , , , , , , , ,0102-0, , , , , , , , , , , , , ,1681 0, , , ,6135 0, , , ,3046 0, , , , , , , , , , , , , , , , , , , , , , , , , , , ,2727 0, , , , , , , , ,092 0, , , , , , , , , , , , , , , , , , , , , , , , , , ,4362 0, , , , ,3102 0, , , , ,2469 0, , , , , , , , , , , , , ,3044 0, , , , , , , , , ,2179 0, , , , , , , ,2099-0, , , , , , , , ,0699 0, , , , ,3637 0, , , , ,4912 0,1552 0, ,2757 0, , , , , , , , ,0449 0, , , , , ,1352 0, , , , , , , ,4516 0, , , , , , , , , , , , ,1149 0, ,0668 0,056 0, , ,2131 0, , , , , ,5414 0, , ,215 0, , , , , ,3161 0, , , , , , ,2015 0, , ,3707 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0138 0, , , ,0491 0, , , , ,022 0, ,1608 0,0363-0, , ,0116 0,0168 0, , , , ,2099 0, , , , ,6461 0,0731 0, , , , , , , , , , , , ,1813 0, , , , , , , , , , , , , , , , , , , , ,2099-0, , , , , , , , , , , , ,2721 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,6364 0, , , ,7237 0, , , , , , ,1658 0, , , , , , , , , , , , , , ,271 0, , , ,1815 0, , , , , , , , , , , , , , ,0863 0, , , , , , , , , , , , , , , , , ,2726 0,1133 0, , , , , , , , , ,209-0, , ,0927 0, , , , , , , , , , , ,3071 0, , , , , , , , , , , , , , , , , , ,0203 0, , , , , ,2401 0, , , ,0034 0, ,1143 0, , , , , ,0867-0,0035 0, , ,0743 0, , , , , , , , , , , , , ,6135 0, , , ,3046 0, , , , , , , , , ,2259 0,5238 0, , , , , , , , , , ,2919 0, , , , , , , , , , , , , , , , , , , , , , , , ,1332 0, , ,2487 0, , , , , , , , , ,3137 0, , , , , , , , , , , , , , , ,4509 0, , , , , ,2268 0, , , , , , , , , , , , , , , , , , , ,0236 0, , , , , , , , , , , , , , , , , , , , , ,1601 0, ,0355 0, , , , , , , , , , , , , , , , , , , , ,2346 0,3956 0, , , ,2494 0, , ,0065 0, , , , , , , , , , , , , , , , , , , ,1222 0, , , , , ,4311-0, , , ,4077 0, , , , , , , , , , , , , , , , , , ,2214 0, , , , , ,0143 0, , , ,0361 0, , , ,0936-0,0119 0, , , , , , , , , , , , , , , , , ,0985 0, , , , , ,0648 0, , , , , , , , , , , , , , , , , , , , , , ,5473 0, , , , , , , , , , , , , , , , , , , , , ,0456 0, , , , , , ,5137 0, , , , , , , , , , , , , , , , , , , ,3396 0, , , , , ,0196 0, , , , ,6055 0, , , , , , , , , , , ,2755 0, , , , , , , , , , , , , , , , , , , , , , , , ,0387 0, , , , , , ,1785 0, , , , , , , , , , , , , , , , , , , , , , , ,1087 0, , , , , , ,4345-0,0542 0, , , , , , , , , ,1357 0, , , , , , , , , ,0054-0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,1119 0, , ,24041 Apêndice XXIII I: Matriz de Fst para populações de DNA mitocondrial com quatro haplogrupos de n10
179 180 Curripaco Dogrib Dogrib Embera Embera Embera Gambel Ilulissat (Jakobshavn) Ittoqqortoorm iitt (Scoresbysun d) Nuuk (Godthab) Savoonga Siberian Upernarik Uummannaq (Umanak) Yupik s s (Savoonga e Gambel) s Old Harbor s Ouzinkie Fueguian Argentina Gaviao Guahibo Guahibo- Sikuani Guambiano Curripaco Dogrib Dogrib Embera Embera Embera Gambel 0 0, , , , , , , , , , , , , , , , , , , , , , , , , , , Ilulissat (Jakobshavn) Ittoqqortoorm iitt (Scoresbysun d) Nuuk (Godthab) 0, , , , , , , , , , , , Savoonga 0,5495-0, , , , , , , , , Siberian 0, , , , , , , , , , , Upernarik 0,3769-0, , , , , , , Yupik Uummannaq (Umanak) 0,3769-0, , , , , , , , ,664 0, , , , , ,549 0,549 0,549 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,4295 0, , , , , , , , , ,1074 0, , ,1089 0, , ,2022 0,0877 0, , , , , , , , , , , , , , ,5457 0, ,3672 0, , , , , , ,6423 0,6423 0, , , , , , , , ,6605 0,6605 0,6605 0, , , , , , , , , , , , , , , , ,2752 0, , , , , , , , , , , , , , ,0495 0, , , ,5298 0, , , , , , , , , , , , , ,52285 Apêndice XXIII J: Matriz de Fst para populações de DNA mitocondrial com quatro haplogrupos de n10
180 181 Guane- Butaregua Guatuso Guayabero Guaymi Haida Havasupai/ Hualapai/ Yavapai/ Mojave hokan (central coast) Huetar Huetar Huilliche Huilliche Huilliche Chile Huitoto Ignaciano Ijka-Arhuaco Ingano Ingano Colombia Inuit eskimo Jemez Kanamarí Katuena Katukina Katukina Kawéskar Kaxináwa Kayapó Kiliwa/Paipai Kogui Kraho Kumiai Kuna Kuna Kuna Mapuche Mapuche Mapuche (Chile) Mapuche Anecón Grande Mapuche Argentina Mapuche Chile Marúbo Mataco (Chaco) Mataco (Salta) Mataco (Wichí) Argentina Mataco (Wichí) Formosa Maya Maya Contenporary Mixe Mixtec Mohawk Mohawk Mosetén Bolivia Movima Bolívia Murui- Muinane Muskoke Oklahoma Nahua Nahua/Cora Navajo Ngöbé Nukak Nuu-Chah- Nulth Ojibwa Manitoulin Island Ojibwa North Ontario Ojibwa/ Chippewa Old Harbor Ouzinkie Paez Paiute N./ Shoshoni Parakanã Pehuenche Butalelbum Pehuenche Trapa Trapa Peneunche Chile 2 Curripaco Dogrib Dogrib Embera Embera Embera Gambel Ilulissat (Jakobshavn) Ittoqqortoorm iitt (Scoresbysun d) Nuuk (Godthab) Savoonga Siberian Upernarik Yupik Uummannaq (Umanak) 0,1609 0,8183 0, , , , , , , , , ,4939 0,5914 0,5914 0, ,174 0, , , , , ,1307 0, , , , , , , , , , , , , , , , , , , , , , , , , , ,0435 0, , , ,2578 0,2578 0,2578 0, , , , ,4503 0, , , , , , , , , , , , ,0112 0,0112 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,1955 0, , , ,2237 0, , , , , , , , , , , , , ,082 0,2306 0, ,0037 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,2852 0, , , ,1725 0, , , , , , , , , , ,2604 0, , , , , , , , , , , , , , , , ,689 0, , , , , , , , , , , , , , , , , , ,1054 0, , , , , , , , , , , , , ,1613 0, , , , , , , , , , , , , , ,0091 0, , , , , , , , ,471 0, , , , , , , , , , , , , , , , ,9548 0, ,7499 0, , ,6489 0, , , , , , , , , , , ,1676 0, , , ,3795 0,3795 0,3795 0, , , , , , , , , , , , , , , , , , , , , , , , ,2821 0, , , , , , , , , ,2539 0, , , , , , , , , , , , ,125 0,125 0, , , ,9098 0, , , , , , , ,8827 0, , , , ,2311 0, , , , , , , , , , , , , ,4032-0, , ,6995 0, , , , , , , , , , , ,4651 0, , , , , , ,5424 0, , , , , , , , , , , , , , ,2367 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,5436 0,4285-0, , , , , , , , , , , , ,1458 0, , , , , , , , , , , , , , , , , , , , , , , , , , , ,5887 0, , , , , , , , , , , ,2736 0,203 0, , , , , , , , , , , , , , ,2841 0, , , , , , , , , , , , , , , , , , , , , ,4603 0,4603 0,4603 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,5706 0,5706 0, , , , , , , , , , , , , , , , , ,8183 0, , , , , , , , , , ,5711 0,5711 0, , , , , , , , ,6141 0,6141 0,6141 0, ,5136 0, , , , , , , , , , , , , , , , , ,4081 0, , , , , , , , , , , , , , , , , , , , , ,0645 0, , , , , , , , , , , ,0308 0, , , , , , , , , , , , , ,4418-0, , , , , , , , ,1432 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0538 0, , , , , , , , , , , , , , ,1745-0,0319 0, , , , , , , ,4849 0,4849 0, , , , , , , , , , , , ,5006 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0147 0, , ,0045 0, , , , , , , , , , ,4305-0,0319 0, , ,0199 0, , , , , , , , , , , , , , , , ,095 0,1761 0,288 0,288 0,288 0, , , , , ,0041 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0771 0, , , , , , , , ,2388 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,22 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,074 0, , , , , , , , , , , , , , , , , , , , , , , , , , , ,1263 0, , , , , , , , , ,3138 0, ,8768 0, , , ,3832 0, , , , , ,5256 0, , ,1275 0, , , , , , , , , , , , , , ,2136 0, , , , , , , , , , , , , , ,18636 Apêndice XXIII K: Matriz de Fst para populações de DNA mitocondrial com quatro haplogrupos de n10
181 182 Penutian California Penutian California Piaroa Pilagá Formosa Pima Pima Pima Poturujara (Zoé) Quechan/ Cocopa (Arequipa) (Tayacaja) Bolivia / Nmatsiqueng a (San Martin) Aleut Saint Paul Salinan/ Chumash Salish Plateau Selknam Seminole Siona Siouan Sioux Sisseton/ Wapheton Sioux Takik Taono O odham (Papago) Tayacaja Peru Tehuelche Argentina Teribe Teribe Tikúna Tikúna Brasil Tikúna Colombia Toba (chaco) Toba (Formosa) Trinitario Bolivia Tucano Tule-Cuna Uto-Aztecan California Uto-Aztecan Central Uto-Aztecan Northern Waiãpi Wapishana Brasil Washo Waunana Wayuu Wayuu Colombia Wounan Xavante Yaghan Chile Yakama Yakima Yámana (Yaghan) Yaminawa Yanomama Yanomama Brasil Yanomami Yanomamö Yok-Utian Yuko-Yukpa Yuman Yuman (Hokan) Yuman Delta Yuman Pai Yuman River Yuracaré Zapotec Zenu Zoro Zuni Zuni (penutian) Curripaco Dogrib Dogrib Embera Embera Embera Gambel Ilulissat (Jakobshavn) Ittoqqortoorm iitt (Scoresbysun d) Nuuk (Godthab) Savoonga Siberian Upernarik Yupik Uummannaq (Umanak) 0, , , , , , ,259 0, , , , , , , , , , ,7422 0, , , , , , , , , , , , , , , , , ,1033 0, , , , , , , , ,2037 0, , , , , , , , , , , , , , , , , , ,4236 0,1244 0, , , , , , , , , ,5157 0, , ,7304 0, , ,0209 0, , , , , , , , ,5049 0, , , , , , , , , , , , , , , , , , , , , , , , , ,4688 0, , , , , , , , , , , , , , , , , , ,5559 0, , , , , , , , , , , , , , , , ,8598 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,6858 0,6858 0,6858 0, , , , , , , , , , , ,3181 0,6236 0,6236 0,6236 0, , , , ,3572 0, , , , ,3918 0, , ,5951 0,5951 0,5951 0, , ,5802 0,5802-0, , , , , , , , , , , , , , , , , , , , , ,0292 0, , , , ,7185 0,394 0,5375 0,5375 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,362 0, , ,2835 0, , , , , , , , , , , ,3708 0, ,8737 0, , , , , , , , , , , , , ,2645 0, , , , ,0357 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,4006 0, , , , , , , , , , , , , , , ,1746 0, , ,4687 0,4687 0,4687 0, , , , ,2289 0, ,7843 0,666 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , ,7628 0, , ,081 0, , , , , , , , , , , , , , ,2328 0, , , , , , , , , , , , , , , , , ,2673 0,2673 0,2673 0, , , , , , , , , , , , , , , , , ,6173 0,6173 0, , , , ,4236 0,1244 0, , , , , , , , , ,5157 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,4318 0, , ,3683 0,6387 0,6387 0,6387 0, , , , , ,2517 0, , , , , , , , , , , , , , , , , , , ,1504 0,2477 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,2366 0,9005 0, , , ,1193 0, , , , , , ,778 0,778 0, , , ,8226 0, , , , , , , , , , , ,2448 0, , , , , , , , , , , , , , ,4748 0, , , , , , , , , , ,7313 0, , , , , , , , , , ,3057 0, , , ,7908 0, , , , ,2008 0, , , , , ,0226 0, , , , ,1233 0, , , ,2521 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,109 0, , ,5831 0,5831 0,5831 0, , , , , , , , , , , , , , ,266 0, , , ,1561 0, , , , , , , , , , , , , , ,1405 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,489 0, , , , , , , , , , , , , , , , , , , , , , , ,3079 0,3079 0, , ,7523 0, , , , , , , , , , , , , , , , , , , ,2338 0, , , , , , , , ,3422 0, , , , , , , , , ,6486 0, , , , ,1774 0, , , ,0712 0,055 0,4481 0, , , , , , , , ,1032 0, , , , ,0104 0, , , , , , ,6332 0,6332 0,5221 Apêndice XXIII L: Matriz de Fst para populações de DNA mitocondrial com quatro haplogrupos de n10
182 183 s s (Savoonga e Gambel) s Old Harbor s Ouzinkie Fueguian Argentina Gaviao Guahibo Guahibo- Sikuani Guambiano s 0 s (Savoonga e Gambel) -0, s Old Harbor 0, , s Ouzinkie -0,0122-0,0162 0, Fueguian Argentina Gaviao Guahibo Guahibo- Sikuani 0, , ,3093 0, , , , ,4223 0, , , , ,2528 0, , Guambiano 0, , , , , , , , , , , , , , , Guane- Butaregua Apêndice XXIII M: Matriz de Fst para populações de DNA mitocondrial com quatro haplogrupos de n10 Guatuso Guayabero Guaymi Haida Havasupai/ Hualapai/ Yavapai/ Mojave
183 184 Guane- Butaregua Guatuso Guayabero Guaymi Haida Havasupai/ Hualapai/ Yavapai/ Mojave hokan (central coast) Huetar Huetar Huilliche Huilliche Huilliche Chile Huitoto Ignaciano Ijka-Arhuaco Ingano Ingano Colombia Inuit eskimo Jemez Kanamarí Katuena Katukina Katukina Kawéskar Kaxináwa Kayapó Kiliwa/Paipai Kogui Kraho Kumiai Kuna Kuna Kuna Mapuche Mapuche Mapuche (Chile) Mapuche Anecón Grande Mapuche Argentina Mapuche Chile Marúbo Mataco (Chaco) Mataco (Salta) Mataco (Wichí) Argentina Mataco (Wichí) Formosa Maya Maya Contenporary Mixe Mixtec Mohawk Mohawk Mosetén Bolivia Movima Bolívia Murui- Muinane Muskoke Oklahoma Nahua Nahua/Cora Navajo Ngöbé Nukak Nuu-Chah- Nulth Ojibwa Manitoulin Island Ojibwa North Ontario Ojibwa/ Chippewa Old Harbor Ouzinkie Paez Paiute N./ Shoshoni Parakanã Pehuenche Butalelbum Pehuenche Trapa Trapa Peneunche Chile 2 s s (Savoonga e Gambel) s Old Harbor s Ouzinkie Fueguian Argentina Gaviao Guahibo Guahibo- Sikuani Guambiano Guane- Butaregua Guatuso Guayabero Guaymi Haida Havasupai/ Hualapai/ Yavapai/ Mojave 0, , , , , , , , , , , ,2294 0, , , , ,0028 0, , , , , , , , , , , , , , , , , , , , , , ,2988 0, , , , , ,031 0, ,5681 0, , , , , , , , , , , , , , , , , , , ,2943 0, , , , , ,2579 0,1649 0, , , , , ,0222 0, ,2088 0, ,0161 0, , , , , , , , , , , , , ,4071 0, , , , , , , , , , , ,0827 0,0973 0, , ,4307 0, , , ,0981 0, , , , , ,4302 0, ,3367 0, , ,4359 0, , , , , , , , , , , , , , , ,4264 0,226 0, , , , , , , , , , , , , , , ,2361 0, , , , , , , , , , ,2311 0, , , , , , , , ,182 0, , , , , , , , , , , , , , , , , , , , , , ,3318 0, , , , ,0205 0, , , ,3414 0, , , , , , , , , , , , ,2352 0,1454 0, , , , , , , , , , , , , , ,6456 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,1516 0, , , , , , , , , ,351 0, , , , ,2051 0,0742 0,062 0, , , , , , , , , , , , , , , , ,5816 0, , ,5138 0, ,5628 0, , , , , , , ,2718 0, , , , , , , , , , , , ,1607 0, , , , ,4852 0, ,2918 0, , , , , , , , , , , , , , , , , , , , , , , , ,4034 0, , , , ,0064 0, , , , , , , , , , , , , , , , , , , , , , , , ,5926 0, ,5695 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,2671 0, , , , , , , , , , , ,1019 0, , , , , , , , ,0187 0, , , , , , , , , , , , , , , , , ,3838 0, , , , , , , , , , , , , , , , , ,3863 0, , , , , , , , , ,4664 0, , , , , , , , , , , , , , , , ,3974 0, ,2355 0, , , , , , , , ,2166 0, , , , , , , , , ,2876 0, , ,3349 0, , , , , ,5608 0,5079 0,3288 0,4655 0, , , , , , , , , , , , , , ,402 0, , , , , , , , , , , , , , , , , , , , , ,4939 0, , , ,1322 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,374 0, , ,0193 0, ,1072 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0373 0, , , , , , , , , , , , , ,1028 0, , , , , , , , , , , ,0235 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,5018 0, , , , , , , , , ,3077 0, ,5451 0, , , , , , , , , , , , , , , , , , , , , , , ,2166 0, , , ,1206 0, , , , , , , , , , ,0969 0, , , , , , ,2307 0, , , , , , , ,1349 0, , ,0964-0, , , , , , , , , , , , , , , , ,0036 0, , , , , , , , , , , , , ,0133-0, , , , , , , ,3402 0, , , , , , , , , , , , , , ,2345 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0457 0, , , , , ,5071-0, , , , , , , , , , , , , , ,4025 0, , , ,0468 0, , , , , , , , , , , , , , , , ,0163-0, , , , , , , ,5918 0, , , , , , , , , , , ,4297 0, , , , , , , , , , , , , , , , , , , , ,3554 0, , , , , , , , ,2252 0, , , , , , , , , , , , ,5005 0, , , , ,3759 0, , , , , , , , , , , , , , , , , , , , , , , ,2537 0, , , , , , , , , , , ,23021 Apêndice XXIII N: Matriz de Fst para populações de DNA mitocondrial com quatro haplogrupos de n10
184 185 Penutian California Penutian California Piaroa Pilagá Formosa Pima Pima Pima Poturujara (Zoé) Quechan/ Cocopa (Arequipa) (Tayacaja) Bolivia / Nmatsiqueng a (San Martin) Aleut Saint Paul Salinan/ Chumash Salish Plateau Selknam Seminole Siona Siouan Sioux Sisseton/ Wapheton Sioux Takik Taono O odham (Papago) Tayacaja Peru Tehuelche Argentina Teribe Teribe Tikúna Tikúna Brasil Tikúna Colombia Toba (chaco) Toba (Formosa) Trinitario Bolivia Tucano Tule-Cuna Uto-Aztecan California Uto-Aztecan Central Uto-Aztecan Northern Waiãpi Wapishana Brasil Washo Waunana Wayuu Wayuu Colombia Wounan Xavante Yaghan Chile Yakama Yakima Yámana (Yaghan) Yaminawa Yanomama Yanomama Brasil Yanomami Yanomamö Yok-Utian Yuko-Yukpa Yuman Yuman (Hokan) Yuman Delta Yuman Pai Yuman River Yuracaré Zapotec Zenu Zoro Zuni Zuni (penutian) s s (Savoonga e Gambel) s Old Harbor s Ouzinkie Fueguian Argentina Gaviao Guahibo Guahibo- Sikuani Guambiano Guane- Butaregua Guatuso Guayabero Guaymi Haida Havasupai/ Hualapai/ Yavapai/ Mojave 0, ,4521 0, , , , , , , , , , , , , , , , , , , ,3378 0, , , , , , ,505 0, , , ,1216 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,3509 0, , , , , , ,3396 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,2329 0, ,1717 0, , , , , ,584 0, , , , , , , , , , , ,4482 0, , , , , , , , , , ,2338 0, , , , , , , , ,3912 0, , , , , , , , , , , ,1554 0, , , , , , , , , , , , ,6069 0, , , , , , , , , , , , , , , , , ,4461 0, , , , ,4492-0, ,4705 0,2809 0, , , , , , ,3728 0, , , ,5091 0, , , , , , , , , , , ,2579 0,1649 0, , , , , ,0222 0, ,2088 0, , , , , , , , , , , , , , , , , , , , , , , , , , ,6177 0,3947 0, , , , , , ,0835 0, , , , , , , , , , , , , , , , , , , , , , , ,0387 0,0008 0, , , , , , , , , , , , ,0326 0, , ,2039 0, , , , , ,3522 0, , , , , , , , , , , , , , , , , , , ,5455 0,3092 0, , , , ,5559 0, , , , , , , , , , , , , , , ,1554 0, , , , , , , , , , , , , , , , , ,0578 0, , , , ,5244 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , ,6417 0, , , , , , ,253 0, , , , , , , , , , , , , ,4288 0, , ,3297 0, , , , , ,3161 0, , , , , , , , , , , , , ,1253 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,1524 0, , , , , , , , , , , , , , ,1837 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0909 0, , , , , , ,2804 0, , , , , , , , , , , , , , , ,3509 0, , , , ,2829 0, , , , , , ,0725 0, , ,3507 0, , , , , , , , , , , , ,0503 0, , , , , , , , , , , , , , ,609 0, , , ,3122 0, ,5399 0, , , , , , , , , , , , , , , , , , ,4172 0, , , , , , , , , , ,4344 0, , , , , , , , , , , , , , , , , , , , , , ,1794 0, , ,1868 0, , ,3939 0, ,2988 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,3131 0, , , , , ,1921 0, , , , , , , , , , , , ,4377 0, , , , , , , , , , , , , , ,4265 0, , , ,5235 0, , , , , , , ,4961-0, , , , , , , , , , , , , , , , ,4047 0,0007 0, , , , , , , ,2629 0, , , , , , , , , , ,4248 0, , , , , ,5131 0, , , , , , , , , , , , , , , , , , , , , , , , ,5273 0, , , , , ,4096 0, , , , , , ,0911 0, , , , , , , , , , , , , , , , , , , , , , , , , ,8489 0, , , , , , , , , ,5679 0, , , , , , , , , , , , , , , , ,468 0,5292 0, , ,3465 0, , , , ,3359 0, ,5617-0, , ,5813 0, , , , , , ,2937 0, , , , , , , , , ,5343 0, , , , , , , , , , , ,6413 0, , , ,413 0,4683 0,352 0, ,3695 0, , , , , , ,279 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0006 0, , , , , , ,1664 0, , , , , ,1925 0, , , ,3792 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,4827 0, , , , , , , , , ,06271 Apêndice XXIII O: Matriz de Fst para populações de DNA mitocondrial com quatro haplogrupos de n10
185 186 hokan (central coast) Huetar Huetar Huilliche Huilliche Huilliche Chile Huitoto Ignaciano Ijka-Arhuaco Ingano Ingano Colombia Inuit eskimo Jemez Kanamarí Katuena Katukina Katukina Kawéskar Kaxináwa Kayapó Kiliwa/Paipai Kogui Kraho Kumiai Kuna Kuna Kuna Mapuche Mapuche Mapuche (Chile) Mapuche Anecón Grande Mapuche Argentina Mapuche Chile Marúbo Mataco (Chaco) Mataco (Salta) Mataco (Wichí) Argentina Mataco (Wichí) Formosa Maya Maya Contenporary Mixe Mixtec Mohawk Mohawk Mosetén Bolivia Movima Bolívia Murui- Muinane Muskoke Oklahoma Nahua Nahua/Cora Navajo Ngöbé Nukak Nuu-Chah- Nulth Ojibwa Manitoulin Island Ojibwa North Ontario Ojibwa/ Chippewa Old Harbor Ouzinkie Paez Paiute N./ Shoshoni Parakanã Pehuenche Butalelbum Pehuenche Trapa Trapa Peneunche Chile 2 hokan (central coast) Huetar Huetar Huilliche Huilliche Huilliche Chile 0 0, , , , , , , , , , Huitoto Ignaciano Ijka-Arhuaco Ingano Ingano Colombia 0, , , , , , , ,1694 0, , , ,1271 0, , , , , , , , , , , , , , , , , , , ,2671 0, ,05 0, Inuit eskimo Jemez Kanamarí Katuena 0, , , ,1795 0, , , , , , , , , , ,5268 0, , ,5928 0, ,5153 0, , ,7469 0, , , , , , , , ,38 0, ,0219 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0848 0, , , , , , , , , , ,0446 0, , , , ,3851 0, , ,314 0, , , , , , , , , , , , , , , , ,5148 0, , , , ,265 0, , , , , , , , , , , , , , , , , , , , , , ,1421 0, , , , ,4984 0, , ,439 0, , , , , , , , , , , , , , ,2373 0, , , , , , , ,347-0, , , , , , , , , , , , , , , , , , , , ,2037 0, , , , ,2152 0,2138 0, ,0271 0, , , , , , , , , , , , , , , , ,4784 0,5644-0, , , , , , , , , , , , , , , ,1779 0, , , , ,1106 0, , , , , , , , , ,1232 0, , , , , , , , , , , , , , , , , , , , , , ,0184 0, , , , , , , , , , , , , ,0377 0, , , , , , , , , , , ,038 0,2461 0, , , , , , , , , , , , , , , , , , ,0186 0, , , , , ,4768 0, , , ,22 0, , , , , ,0709 0, , , , , , , , , , ,4041 0, , , , ,0903 0, , , , , , , , , , , ,024 0, ,0928 0,254 0, ,3542 0, , , , , , , , , , , , , , , , , , , , , , , , , , , ,2063 0, , , , , , , , , , ,1057 0,1135 0, , , ,468 0, ,0225 0, , , , , , ,0775 0, , , , , , , , ,1912 0, , , , , , , , , , , , , , , , , , , , , , , , , , ,1398 0, , , , , , , ,0857 0, , , , , , , ,3009 0, , , , , , , , , ,3056 0, , , , , , , , , , ,0344 0, , , , , , ,142 0, , ,1783 0, , , , ,1154 0,2886 0,2938 0, , , , , , , , , , , , , , , , , , , , , ,0605 0, , , , , , ,2873 0, , , , ,0282 0, ,4268 0, , , ,5556 0, , , , , , , , , , , , , , , , , , , , , , , , , ,1477 0, , , , , , ,0368 0, , , ,2869 0, , , , , , , ,5287 0,0635 0,2192 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,5964 0, , , , , ,6374 0,1649 0,1774 0, ,7818 0, , ,0383 0, , , , , , , , , , , , ,0292 0, , , , , , , , , , , ,1071 0, , , , , , , , ,4323 0, ,3378 0,388-0, , , ,0464 0, , , , , , , , , , ,2284 0,0107 0, , , , , , , , , , , , , , , , , , , ,166 0, , , , , , , , , , , , , , , , , , , , , , , , , , , ,2934 0, , , , , , , , , , , ,5186 0, , , ,3611 0, , , , , ,0362 0, , , , , , , , , , , , , , , , , , , , ,3691 0, , , , , , ,3452 0, , , , , , , , , , , , , , , , , , , , , , , , , , , ,03782 Apêndice XXIII P: Matriz de Fst para populações de DNA mitocondrial com quatro haplogrupos de n10
186 187 Penutian California Penutian California Piaroa Pilagá Formosa Pima Pima Pima Poturujara (Zoé) Quechan/ Cocopa (Arequipa) (Tayacaja) Bolivia / Nmatsiqueng a (San Martin) Aleut Saint Paul Salinan/ Chumash Salish Plateau Selknam Seminole Siona Siouan Sioux Sisseton/ Wapheton Sioux Takik Taono O odham (Papago) Tayacaja Peru Tehuelche Argentina Teribe Teribe Tikúna Tikúna Brasil Tikúna Colombia Toba (chaco) Toba (Formosa) Trinitario Bolivia Tucano Tule-Cuna Uto-Aztecan California Uto-Aztecan Central Uto-Aztecan Northern Waiãpi Wapishana Brasil Washo Waunana Wayuu Wayuu Colombia Wounan Xavante Yaghan Chile Yakama Yakima Yámana (Yaghan) Yaminawa Yanomama Yanomama Brasil Yanomami Yanomamö Yok-Utian Yuko-Yukpa Yuman Yuman (Hokan) Yuman Delta Yuman Pai Yuman River Yuracaré Zapotec Zenu Zoro Zuni Zuni (penutian) hokan (central coast) Huetar Huetar Huilliche Huilliche Huilliche Chile Huitoto Ignaciano Ijka-Arhuaco Ingano Ingano Colombia Inuit eskimo Jemez Kanamarí Katuena 0,0711 0, , , , , , ,1913 0, , , , , , , , , , , , , , , , , , , , , , , ,15 0,1595 0, , , , , , , , ,4788 0, , , , , ,3239 0, , , , , , , , , , , , , ,4587 0, , , , , , , , ,0148 0,658 0, ,119 0, , , , , , , , , ,5105 0, , , , , , , , , , , , , , , , , , , , , , , , ,1591 0, , , , , ,0836 0, ,3775 0, , , ,3165 0, , , , , , , , , ,0263 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,7045 0, , , , , , , , , , , , , , , , , , , , , , ,2854 0, , , , , ,1131 0, , , ,375 0, , ,371 0, , , , , , , , , , , , ,1927 0, , , , , , , , , , , , , , ,1 0, , , , , , ,1271 0, , , , , ,0219 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0995 0, , , , , , , , ,0417 0, , , , , , , , , , , , , , , , , , , , , , ,1739 0, ,5884 0, , , , , , , , , , , , , , , ,0221 0, , , , ,0892 0, , , , , , ,0839 0, ,6512 0, , , , , , , , , , , , , , , ,1316 0, , , , , , , , , , , , , , , , , , , , , ,012 0, , , , , , , , , , , , , , , , , , , , ,1355 0, , , , ,2158 0, , , , , , , , , , , , ,5193 0, , , , , , , , ,2731 0, , , , , , , , , , ,0479 0, , , , , , , , , , , , , , , ,0501 0, , , , , , , , , , , , , , , , , ,4657 0, ,0922 0, , , , ,0169 0, , , , , ,0527 0, , , ,1366 0, ,4453 0, , ,1383 0, , , , , , , , , , , , , , , , , ,0494 0, , , , , , , , ,6391 0, , , , , , , , , , , , , , , , , , , , , , , , ,0326 0, , , , , , , , ,4587 0, , , , , , , , ,0148 0,658 0, ,119 0, , ,3532 0, ,0103 0, , , , , , ,1605 0, , , , ,1057-0, , , , , , , , , , , , , , , , , , , , ,0722 0, , ,3889 0, , , , , , , , ,1378 0, , , , , , , , , , , , , , , , , , , ,5789 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,042-0,0164 0, , , , , , , , , , , , , , , , , , , ,3966 0, , , ,3419 0, , , , , , ,8047 0, , , , , , , , , , , , , , , , , , , ,4671 0, , , , , , , , ,1278 0, , , ,3255 0, , , , , , , , , , , , , , , , , , , , , , , , , ,2849 0, , , , , , , , , , , , , , , , , , , , , , , , , , ,0211 0, , , , , , , , ,4092 0, , , , , , ,5196 0, , , , , ,0258 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0276 0, , , , , ,6263 0,5503 0, , , , , ,5068 0, , , , , , , , , ,7326 0, , ,4822 0, , , , , , , , , , , , , ,2993 0, , , , , , , , , ,0141 0, ,21 0,17 0,2863 0, , , , , , ,3094 0, , , , , , , , , , , ,2209 0, , , , , , , , ,204 0, , ,2986 0, , , , , , , , , , , , , , , , , ,1877 0, , , , ,222 0, , , , , , ,0372 0, , , , , , , , , ,0143 0, , , ,0638 0,0889 0,2945 0, , , , , , ,3723 0, , ,5106 0, , , ,0783 0, , , , , , , , , , , , ,1446 0, , , , , , , , , ,6065 0, ,1264 0, , , , , , ,4391 0, , , , , , , , , , , ,31128 Apêndice XXIII Q: Matriz de Fst para populações de DNA mitocondrial com quatro haplogrupos de n10
187 188 Katukina Katukina Kawéskar Kaxináwa Kayapó Kiliwa/Paipai Kogui Kraho Kumiai Kuna Kuna Kuna Mapuche Mapuche Mapuche (Chile) Mapuche Anecón Grande Mapuche Argentina Mapuche Chile Marúbo Mataco (Chaco) Mataco (Salta) Mataco (Wichí) Argentina Mataco (Wichí) Formosa Maya Maya Contenporary Mixe Mixtec Mohawk Mohawk Mosetén Bolivia Movima Bolívia Murui- Muinane Muskoke Oklahoma Nahua Nahua/Cora Navajo Ngöbé Nukak Nuu-Chah- Nulth Ojibwa Manitoulin Island Ojibwa North Ontario Ojibwa/ Chippewa Old Harbor Ouzinkie Paez Paiute N./ Shoshoni Parakanã Pehuenche Butalelbum Pehuenche Trapa Trapa Peneunche Chile 2 Katukina Katukina Kawéskar Kaxináwa Kayapó Kiliwa/Paipai Kogui Kraho Kumiai Kuna Kuna Kuna Mapuche Mapuche Mapuche (Chile) 0 0, ,2452 0, , ,2382 0, ,3838 0,1386 0, , , , , , , , ,3935 0, , ,3934 0, , ,2512 0, , , , , , , , ,2844 0, , , , , , , , , , ,5493 0, ,75 0 0, , , , , ,5311 0, , , , , , , , , , , , , ,1198-0, , , , ,1916 0, ,1984 0, , , ,5036 0, , , , , , , ,2366 0, , ,1016 0, , , , ,0124 0, , , , , , , ,1467 0, , , , , , , , , , ,1965 0, , , , , , , , , ,087 0, , , , , , , , ,4669 0, , ,0137-0, , , , , , , , , ,3475 0, , , , , , , , ,466 0, , , , , , , ,6623 0, , , , , , , , , , , , , , ,6054 0,404 0, , , , ,3615 0,4229 0, , , , , , , , , , , , , , , , , , , , ,0847 0, , , , , , , ,2209 0, , ,2333 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,1537 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,1582 0, , , , , , , , , ,4817 0, , ,4317 0, , , ,1784 0, , , , , , ,1889 0, ,2803-0,0657 0, , , , , , ,2228 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,1637 0,2839 0, , , ,0926 0, , , , , , , , , , , , , ,0244 0, , , , , , , , , , , ,2859 0, , , , , , , , , , , , , , , , ,038 0,087 0, , , , , , ,2475 0, , , , , , , , ,2925 0, , , , , , ,5244 0, , , , , , , , ,2062 0, , ,1147 0, , , , , , , , , , , , , , , , , , , ,0432 0, , , ,1764 0, , , , ,5101 0, ,7157 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,6915 0, , , , , ,0846 0, , , , , , , , ,5371 0, , , , , , , ,2214 0, ,1996 0, , , , , , , , , , , , , , , , , , ,2183 0, , , , , , , , , , , , ,0081 0, ,487 0, , , , ,7205 0, , , , , , , , , , , ,2341 0,294 0, , ,4442 0, ,0911 0, ,0071-0, , , , , ,4396 0, , , , , , , , ,01701 Apêndice XXIII R: Matriz de Fst para populações de DNA mitocondrial com quatro haplogrupos de n10
188 189 Penutian California Penutian California Piaroa Pilagá Formosa Pima Pima Pima Poturujara (Zoé) Quechan/ Cocopa (Arequipa) (Tayacaja) Bolivia / Nmatsiqueng a (San Martin) Aleut Saint Paul Salinan/ Chumash Salish Plateau Selknam Seminole Siona Siouan Sioux Sisseton/ Wapheton Sioux Takik Taono O odham (Papago) Tayacaja Peru Tehuelche Argentina Teribe Teribe Tikúna Tikúna Brasil Tikúna Colombia Toba (chaco) Toba (Formosa) Trinitario Bolivia Tucano Tule-Cuna Uto-Aztecan California Uto-Aztecan Central Uto-Aztecan Northern Waiãpi Wapishana Brasil Washo Waunana Wayuu Wayuu Colombia Wounan Xavante Yaghan Chile Yakama Yakima Yámana (Yaghan) Yaminawa Yanomama Yanomama Brasil Yanomami Yanomamö Yok-Utian Yuko-Yukpa Yuman Yuman (Hokan) Yuman Delta Yuman Pai Yuman River Yuracaré Zapotec Zenu Zoro Zuni Zuni (penutian) Katukina Katukina Kawéskar Kaxináwa Kayapó Kiliwa/Paipai Kogui Kraho Kumiai Kuna Kuna Kuna Mapuche Mapuche Mapuche (Chile) 0, , , , , , , , , , , ,4367 0, , , , , , , , , , , , , , , , , , , , ,2661 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0827 0, , ,4086 0, , , , , , , , , , , , , , ,1816 0, , , , ,15 0, , , , , , , , , , , , , ,0678 0, ,0586-0, , , , , , ,1902 0, , ,3535 0, , , , ,0223 0, , , ,3751 0, , , , , , , , , , , , , ,3813 0, , ,1067 0,1895 0,1139 0, , , ,0925 0, ,2542 0,0581 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,6309 0, , , , , , , , ,41 0,477 0, ,5018 0, , ,5985 0, , , , ,2615 0, , , , , ,439 0,2373 0, , , , , , , , , , , , , , , , , , , , , ,0674 0, , , , , , ,6129 0, , , ,7606 0, , ,1355 0, ,0142 0, , , , , , , , , , , , , , , , ,0686 0, , , , , , , , , , , , , , , , , , , , , , , , ,0539 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,1139 0, , , ,0925 0, ,2542 0,0581 0, , , , , , , ,0452 0, , , , , , , , ,6302 0,4725 0, ,047 0, , , , , , , , , , , , , ,03 0, , , , , , ,4672 0, , , , , , , , , , , ,272 0, , , , , , , , , , , , , , , , ,1532 0, , , , , , , ,4622 0, , , , , , , , , , , , , , ,3996 0, , , , , , , , , ,3659 0, , , , , , , , , , , , , , , , , , , , , ,0427 0, , , , , , , ,1773 0,0283 0, , , , , , , , , , , , , , , , , , , , , ,0348 0, , , , , , ,1791 0, , , , , , , , , , , , , , , ,1498 0,0917 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0065 0,0281 0, , , , , , , , , , , , , , , , , ,5327 0, , , , , ,3205 0, , ,5156 0, , , , , , , , , , , , , , , , , , , ,3073 0, , , , , ,3813 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,2003 0, , ,1292 0, , , , , , , ,5842 0, , , , , , , ,1936 0, , , ,2132 0,2456 0, ,0557 0, , , , , , ,3913 0, , ,5472 0, , , ,0343 0, , ,5209 0, , , , , , , , , , , , , , , , ,1468 0,0404 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,1092 0, ,0612-0, , , , , , , , ,187 0, , , , , , , , , , , , , ,5202 0, , , ,6599 0, , , , , , , , , ,3269 0, , , , , , ,0678 0, , , , , , , , , , , , , , ,0047 0, , , , , , , , , , , ,7791 0, , , , , , , , , , ,0704-0, , ,4221 0, , , , , , , ,3061 0, , , , , , , ,4828 0, , , , , , ,2569 0,2454 0, ,3151 0, , , , , , , , , , , , ,1459 0, , , , , , , , , , , , , , , , , , , , , , , , , , ,1097 0, , , , , , , ,3463 0,1081 0, , , , , ,1894 0, , , , , , , , , , , , , , , ,4265 0, , , , ,0051 0, , , , , , , , , , , , , , , , , , , , , , ,4859 0, , , , , , , , , , , , , , ,4084 0,3814 0,6096 0, , ,0383 0, ,0382 0, , ,3264 0, , , ,24465 Apêndice XXIII S: Matriz de Fst para populações de DNA mitocondrial com quatro haplogrupos de n10
189 190 Mapuche Anecón Grande Mapuche Argentina Mapuche Chile Marúbo Mataco (Chaco) Mataco (Salta) Mataco (Wichí) Argentina Mataco (Wichí) Formosa Maya Maya Contenporary Mixe Mixtec Mohawk Mohawk Mosetén Bolivia Movima Bolívia Murui- Muinane Muskoke Oklahoma Nahua Nahua/Cora Navajo Ngöbé Nukak Nuu-Chah- Nulth Ojibwa Manitoulin Island Ojibwa North Ontario Ojibwa/ Chippewa Old Harbor Ouzinkie Paez Paiute N./ Shoshoni Parakanã Pehuenche Butalelbum Pehuenche Trapa Trapa Peneunche Chile 2 Mapuche Anecón Grande 0 Mapuche Argentina -0, Mapuche Chile Marúbo 0, , , , , Mataco (Chaco) 0, , , , Mataco (Salta) 0, , , , , Mataco (Wichí) Argentina 0, , , , , , Mataco (Wichí) Formosa 0, , , , ,1279 0, , , , , ,2766 0, , , , Maya Maya Contenporary Mixe Mixtec Mohawk Mohawk Mosetén Bolivia 0, , , , , , , ,1725-0, , , , , , , , ,2126-0, , , , , , ,4837 0, , , , , , , ,2273 0, , ,3387 0, , ,2594-0, , , , , , , , , , , , ,0023-0, , , , ,1099 0, , , ,2548 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,2155 0, ,1631 0,0931 0, , , , , ,1817 0, , , , , , , , , , , , , , , , , , , , , , , , , , , ,0043 0, , , , , , , , , , , , , , , , , , , , , , , ,3543 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,041 0, ,2011 0, , , , ,2913 0, , , , , , , , , , , , ,4985 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0408 0, , , , , , , , , , , , , , , , , , ,3723 0, , , , , , , , ,1868 0, , , , ,4476 0, , , , , , , , , ,36 0, , , , , , , ,4941 0, ,0773-0, , , , ,2833 0, , , , , , ,3018 0, ,1142 0, , , , , , , , , , , , , ,37766 Apêndice XXIII T: Matriz de Fst para populações de DNA mitocondrial com quatro haplogrupos de n10
190 191 Penutian California Penutian California Piaroa Pilagá Formosa Pima Pima Pima Poturujara (Zoé) Quechan/ Cocopa (Arequipa) (Tayacaja) Bolivia / Nmatsiqueng a (San Martin) Aleut Saint Paul Salinan/ Chumash Salish Plateau Selknam Seminole Siona Siouan Sioux Sisseton/ Wapheton Sioux Takik Taono O odham (Papago) Tayacaja Peru Tehuelche Argentina Teribe Teribe Tikúna Tikúna Brasil Tikúna Colombia Toba (chaco) Toba (Formosa) Trinitario Bolivia Tucano Tule-Cuna Uto-Aztecan California Uto-Aztecan Central Uto-Aztecan Northern Waiãpi Wapishana Brasil Washo Waunana Wayuu Wayuu Colombia Wounan Xavante Yaghan Chile Yakama Yakima Yámana (Yaghan) Yaminawa Yanomama Yanomama Brasil Yanomami Yanomamö Yok-Utian Yuko-Yukpa Yuman Yuman (Hokan) Yuman Delta Yuman Pai Yuman River Yuracaré Zapotec Zenu Zoro Zuni Zuni (penutian) Mapuche Anecón Grande Mapuche Argentina Mapuche Chile Marúbo Mataco (Chaco) Mataco (Salta) Mataco (Wichí) Argentina Mataco (Wichí) Formosa Maya Maya Contenporary Mixe Mixtec Mohawk Mohawk Mosetén Bolivia 0, ,0174 0, , , , , , , , , , , , , , , , , , , , , ,2 0, , , , , , ,0049 0, , , , , , , , ,0417 0, , , ,0901 0, ,0133-0, , , , , , , , , , , ,2516 0,2363 0, , ,0914 0, , , , , , , , , , , ,272 0, , , , , , ,1628 0, , , , , , , , , , , , , , , , , , , , , , , , , ,1355 0,173 0, , , , , , , , , , , , , ,1155 0, , ,302 0, , , , , , ,5856 0, , , ,0016 0,0152 0, , , , , , , , , , , , , ,0551 0,0806 0, , , , , , , , , , ,336 0,2946 0, , , , , , , , , , , ,1295 0, , , , , ,1588 0, , , ,0507 0, , , , , , , ,3165 0, , , , ,2009 0, , , , , , , , , , , , , , ,3247 0, , , , , , , , , ,3813 0, ,038 0, ,22 0, , , , , , , , , , ,0344 0,1154-0, , , , , , , , , ,0631 0, , , , , , , , , , , , , ,3416 0, , , , , , , , ,3921 0, , ,3114 0, , , ,0196-0, , , , , , , , , , , , , , , , , , , , , , ,3357 0, , , , , , ,0338-0, , , , , , , , ,2693 0, ,3008 0, , , , ,0167 0, , , ,1156 0, , , , , , , , , , , , ,3175 0, , , , , , , , , , ,2738 0, , , , , , , , , , , , , , , , ,1295 0, , , , ,0694 0, , ,0454 0, , , , , , , , , , ,3528 0, , , , , , , , , , , ,0195-0, , , , , , , , , ,6252 0, , , , , , , , , ,0795 0, , , , , , , , ,2097 0, , , , , , , , , , , , , , , , , , , ,0425 0, , , , , , , , , ,244 0, , ,1979 0,2684-0, , , , , , , , , , , , , , , , , , ,1557 0, , ,0872 0,0652 0, , , , , , , , , , , , , , , , , , , , , , , , ,0085-0,0595 0, , , , , , , , , , , , , , ,2466 0, , , , , , , , , , , , , , , , , , ,0576 0, , , , , , , , ,0914 0, , , , , , , , , , , ,272 0, , , , ,1137 0, , , , , , , , ,3428 0, , , , , , , , , , , , , ,0397 0, , , , , , , , , , , , , , , , , , , ,0427 0, , ,2077 0, , , , , , , , , , , , , , , ,0692 0, , , , , , , , , , , , , , , , , , ,1613 0, , , , , ,049 0, , , ,2456 0, , , , , , , , ,1035 0, , , , , , ,293 0, , , , , , , , , ,1986 0, , , , , , , , , , , , , ,1172 0, , , , ,2163 0, , , ,3533 0, , , , , , , , , , , ,014-0, , , , , , , , , , , , , , , , , , , , , , , ,1268 0,1676 0, , , , , , , , , , , , , , , , , , , , , , , , , , ,0172-0, , , , ,1794 0, , , , , , , , , , , , , , , , , , , , , , , , , ,2856 0, , , , , , , , , ,4508 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,8439 0, , , , , , , ,203 0, , , , ,2849 0, , , ,361 0, , , , , ,2293 0, , , , , , , , , , , , , , , , ,1593 0, , , , , , , , , , , , , , , ,0901 0, , , , , , , ,1288 0, , , , , , , , , , , , , , , , , , , ,2093 0, , , , , , , , , , ,0326 0, , , , , , , , , , , , , , , , , , , , , , , , , , ,1531 0, , , , , , ,0582 0, , , , , , , , ,2896 0, , , , , , ,0497 0, , , ,2813 0, , , , , , , , , , , , , , , , , , , ,02186 Apêndice XXIII U: Matriz de Fst para populações de DNA mitocondrial com quatro haplogrupos de n10
191 192 Movima Bolívia Murui- Muinane Muskoke Oklahoma Nahua Nahua/Cora Navajo Ngöbé Nukak Nuu-Chah- Nulth Ojibwa Manitoulin Island Ojibwa North Ontario Ojibwa/ Chippewa Old Harbor Ouzinkie Paez Paiute N./ Shoshoni Parakanã Pehuenche Butalelbum Pehuenche Trapa Trapa Peneunche Chile 2 Movima Bolívia 0 Murui- Muinane 0, Muskoke Oklahoma Nahua Nahua/Cora Navajo Ngöbé Nukak Nuu-Chah- Nulth 0, , , , , , , , , ,369 0, , , , , , , , , , , ,1599 0, , , , , , , , , , , ,1339 0, Ojibwa Manitoulin Island 0, ,0779 0, ,0909 0, ,1263 0, , , Ojibwa North Ontario 0, , , , , , , , , , Ojibwa/ Chippewa 0, , ,158 0, , , , , , , , Old Harbor 0, ,2792 0,0613 0, ,1744 0, , , , , , , , , ,1662 0, , , , , ,0827 0, , , , , , , ,0974 0, , , , , , , , , , , , , , ,3132 0, , , , ,3091 0, , ,3375 0, , ,0028-0, , , , , , , , ,0768 0, , , , , , , , , ,4625 0, , , , , , , , , , , , , , , , , ,227 0, , , ,3583 0, , , , , , , ,3568 0,3661 0, , , , , , , , ,29834 Apêndice XXIII V: Matriz de Fst para populações de DNA mitocondrial com quatro haplogrupos de n10 Ouzinkie Paez
192 Penutian California Penutian California Piaroa Pilagá Formosa Pima Pima Pima Poturujara (Zoé) Quechan/ Cocopa (Arequipa) (Tayacaja) Bolivia / Nmatsiqueng a (San Martin) Aleut Saint Paul Salinan/ Chumash Salish Plateau Selknam Seminole Siona Siouan Sioux Sisseton/ Wapheton Sioux Takik Taono O odham (Papago) Tayacaja Peru Tehuelche Argentina Teribe Teribe Tikúna Tikúna Brasil Tikúna Colombia Toba (chaco) Toba (Formosa) Trinitario Bolivia Tucano Tule-Cuna Uto-Aztecan California Uto-Aztecan Central Uto-Aztecan Northern Waiãpi Wapishana Brasil Washo Waunana Wayuu Wayuu Colombia Wounan Xavante Yaghan Chile Yakama Yakima Yámana (Yaghan) Yaminawa Yanomama Yanomama Brasil Yanomami Yanomamö Yok-Utian Yuko-Yukpa Yuman Yuman (Hokan) Yuman Delta Yuman Pai Yuman River Yuracaré Zapotec Zenu Zoro Zuni Zuni (penutian) Movima Bolívia Murui- Muinane Muskoke Oklahoma Nahua Nahua/Cora Navajo Ngöbé Nukak Nuu-Chah- Nulth Ojibwa Manitoulin Island Ojibwa North Ontario Ojibwa/ Chippewa Old Harbor 0, , , , , , , , , , ,4853 0, , , , , , , , , , , , , , , , , , , , ,0282 0, , , , , , , ,0208 0, , , , , , , , ,2482 0, , , , , , , , , , , , , ,2642 0,2887 0,2495 0, , , , , , , , , , , , , , , , , , , , , , , ,5017 0, , , , , , , , , , , , , , , ,2759 0,1622 0, , , , , , , ,0202 0,0268 0, , , , ,0572 0, , ,3063 0, , , , , , ,2977 0, ,4556 0, , , , , , , , ,1633 0, , , , , , , , , , ,1649 0, , , , ,3279 0, , , , ,4036 0, , , , , , ,1412 0, , , , , ,1157 0, , , , , , , , , , , , , , , , , , , , , , , , , ,1876 0, , , , , , ,342 0, , , , , , , , , , , , , ,4487 0, , , , , , , ,0368 0, , , ,0383 0, , , , , , , ,0932 0,1103 0, , , , , , , , , , , , , ,0221 0, ,4441 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,01 0, , ,0784-0, , , , , , , , ,0051 0, ,0361 0, , , , , , , , , , , , , , , , , , , ,0682-0, , ,0881 0, , , , , , , , , , , , , , , ,2564 0, , , , , , ,3918 0, , , , ,4014 0, , , , , , ,1412 0, , , , , ,1157 0, , , , , , , , , , , , , ,1896 0, , ,4079 0, , , , , ,1972 0, , , , , , , , , , , ,1413 0, ,5921 0, , , , ,2312 0, , , , , , , , , , , , , , , , , , , , , , , , ,0324 0, ,3281 0, , , , , , , , ,2197 0,3588 0, , , , , ,2418 0, , , , ,0984 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,1462 0, , ,1526 0, ,2198 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , ,4794 0, ,2404 0, , , , , , , , ,0054 0, , , , , , , , , , , , , , , ,5028 0, , , , , , ,2642 0,2887 0,2495 0, , , , , , , , , , ,2402 0,0686 0, , , , , , , ,2732 0, ,3875 0, ,4377 0, , , , , , , , , , , , , ,0367-0, , , , ,1058 0, , , , , , , , , , , , , , , , , , ,3763 0, , , ,5174 0, , , , , ,1291 0, , , , , , , , , , , , , , , , , , , ,3537 0, ,1784 0, , ,2213 0, , , ,1054 0, ,1549 0,1376 0, , , , ,1475 0, ,31 0, , ,3344 0, , , ,1512 0, , , , , , ,0031 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,3413 0, , , ,5459 0, , , , , , , , , , ,3272 0, , , , , , ,0492 0, , , , ,5214 0, , ,2582 0, , , , , , , , ,105 0, ,167 0, , , , , , , , , , , , , ,226 0, , , , , , , , , ,1482-0,0144-0, , , , , , , , , , , , ,4888 0, , , , , , , , , , , , , , ,6484 0, , , , , , , , , , , ,4839 0, , , , , , , , , , , , , , , , , , , , , , , , , , , ,6328 0, , ,8396 0, , , , , , , , ,2644 0, , , , , , , , , , , , , , , , ,2812 0, , , , , , , ,1927 0, , , , , , ,2077 0, , , , , , , , , , ,2837 0, , , , ,3131 0, , ,4055 0, , , , , , , , , , , , , ,5867 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0985 0, , , , , , , , ,4412 0, , , , ,4478 0, ,3023 0, , , , , , , , , , , , , , , , , , , , , , , , , , ,35213 Ouzinkie 193 Paez Apêndice XXIII W: Matriz de Fst para populações de DNA mitocondrial com quatro haplogrupos de n10 Paiute N./ Shoshoni Parakanã Pehuenche Butalelbum Pehuenche Trapa Trapa Peneunche Chile 2 Paiute N./ Shoshoni Parakanã 0 0, Pehuenche Butalelbum 0, , Pehuenche Trapa Trapa 0,1364 0, , Peneunche Chile 2 0, , , , Penutian California Penutian California Piaroa Pilagá Formosa Pima Pima Pima Poturujara (Zoé) Apêndice XXIII X: Matriz de Fst para populações de DNA mitocondrial com quatro haplogrupos de n10 Quechan/ Cocopa
193 194 Penutian California Penutian California Piaroa Pilagá Formosa Pima Pima Pima Poturujara (Zoé) Quechan/ Cocopa (Arequipa) (Tayacaja) Bolivia / Nmatsiqueng a (San Martin) Aleut Saint Paul Salinan/ Chumash Salish Plateau Selknam Seminole Siona Siouan Sioux Sisseton/ Wapheton Sioux Takik Taono O odham (Papago) Tayacaja Peru Tehuelche Argentina Teribe Teribe Tikúna Tikúna Brasil Tikúna Colombia Toba (chaco) Toba (Formosa) Trinitario Bolivia Tucano Tule-Cuna Uto-Aztecan California Uto-Aztecan Central Uto-Aztecan Northern Waiãpi Wapishana Brasil Washo Waunana Wayuu Wayuu Colombia Wounan Xavante Yaghan Chile Yakama Yakima Yámana (Yaghan) Yaminawa Yanomama Yanomama Brasil Yanomami Yanomamö Yok-Utian Yuko-Yukpa Yuman Yuman (Hokan) Yuman Delta Yuman Pai Yuman River Yuracaré Zapotec Zenu Zoro Zuni Zuni (penutian) Paiute N./ Shoshoni Parakanã Pehuenche Butalelbum Pehuenche Trapa Trapa Peneunche Chile 2 Penutian California -0, , , , , Penutian California Piaroa -0, , , , , , , , , , , ,074 0, Pilagá Formosa Pima Pima Pima Poturujara (Zoé) 0,0363-0,0276 0,1499 0, , , , , , , , , , , ,1748 0, , , , , , , , , , , , , , , , , , , , ,0828-0, , , ,0858 0, ,1384 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0751 0, , , , , , ,2987 0, ,1073 0, , , , , , , ,0052 0,0047 0, , ,1652 0,1154 0, , , ,0002 0, , ,1258 0, , , ,0224 0, ,2644 0,5286 0, , , , , , , , , , , , , , , , , , , , ,0312 0, , , , , , , ,3143 0, ,2046 0, ,216 0,2109 0, , ,5166 0, , , , , , , , , , ,0711 0, , , , , , , ,3165 0, , , , , , , , , , , ,0734 0, , , , , , , ,0294-0,0252 0, , , , , , , , , , , , , , , , , , , ,3095 0,2955 0, , , , , , , , , , , , , , ,3716 0, , , , , , , , , , , , , , ,2498 0, ,0485 0, , , , , ,2896 0, , , , ,2048 0,259 0, , , , , , , , , , , , , , , , , , , , ,1835 0, , , , , , , , , , , , , , , , ,1652 0,1154 0, , , ,0002 0, , ,1258 0, , , ,0224 0, , , , , , , , ,0397 0, , , , , , , , , , , , , , , , , , , , , , ,6648 0, , , , , , , , , , , ,7638 0, , , , , ,0431 0, , , , , , , , , , , , , , , ,1574 0,1256 0, , , , , , , , , , , , , , , , ,028 0, , , , , , , , , , ,1893-0, , , , , ,124 0, , , , , , , ,1563 0, , , , ,0417 0, , , , , , , , , ,1795 0, , , , , , ,0198-0,0082 0, , , , , , , , , , ,025 0, , , , , , , , , , , , , , , , , , , , , , , , , , , ,104 0, , , , , , , , , , , , , , , ,1748 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0137 0,1151 0, , , , , , , , , , , , , , , , ,0177-0, , , , , , , , , , ,1682 0, , , , , , , , , , , , , , ,3228 0, , , , , , , , , , , , , , , , , , , ,0163 0, , , , ,2761 0, , , , , , , , , ,0791 0, , , , , , , , , , , , ,2473 0, ,2401 0,2313 0, , , , , ,0164-0, , , , , , ,3448 0, , , , , , , , , , , ,0834 0, ,0993 0, , , , , , , , , , , , , , , ,0944 0,1313 0, , , , , , , , , ,2476 0,205 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0281 0, , , , , , , , , , , , ,0506 0,1702 0, , , , , , , , , , ,5514 0, , , , , , , , , , , , , ,2251 0, , , , , , , , , , , , , , , , ,0459 0, , , ,1986 0, , , , , , , , , , , , , , , , , , ,3503 0, , , , , , , ,2093 0,1012-0, ,0101 0, , , , , , , , ,3035 0, , , , , , , , , , ,225 0, , , , , , , , , , , , ,0167 0, , , , , , , ,1627 0,2029 0, , , , , , , , , , , ,2932 0, , , , , , , , , , , , , ,2297 0, , , , , , , , , , , , , , , , , , , , , , , , , , , ,0096 0, , ,1963 0, , , , ,0109 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , ,1806 0, , , , , ,07 0, , , ,3536 0, , , , , , , , , , ,04145 Apêndice XXIII Y: Matriz de Fst para populações de DNA mitocondrial com quatro haplogrupos de n10 Quechan/ Cocopa
194 195 (Arequipa) (Tayacaja) Bolivia / Nmatsiqueng a (San Martin) Aleut Saint Paul Salinan/ Chumash Salish Plateau Selknam Seminole Siona Siouan Sioux Sisseton/ Wapheton Sioux Takik Taono O odham (Papago) Tayacaja Peru Tehuelche Argentina Teribe Teribe Tikúna Tikúna Brasil Tikúna Colombia Toba (chaco) Toba (Formosa) Trinitario Bolivia Tucano Tule-Cuna Uto-Aztecan California Uto-Aztecan Central Uto-Aztecan Northern Waiãpi Wapishana Brasil Washo Waunana Wayuu Wayuu Colombia Wounan Xavante Yaghan Chile Yakama Yakima Yámana (Yaghan) Yaminawa Yanomama Yanomama Brasil Yanomami Yanomamö Yok-Utian Yuko-Yukpa Yuman Yuman (Hokan) Yuman Delta Yuman Pai Yuman River Yuracaré Zapotec Zenu Zoro Zuni Zuni (penutian) (Arequipa) 0 (Tayacaja) 0, Bolivia -0, , / Nmatsiqueng a (San Martin) 0, ,0224 0, Aleut Saint Paul 0, , , , Salinan/ Chumash 0, , , , , Salish Plateau Selknam Seminole Siona Siouan Sioux Sisseton/ Wapheton Sioux -0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,242 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , Takik Taono O odham (Papago) Tayacaja Peru 0, , , , , , , , , , , , , , , , ,0224 0, , , , , , , , , , , , , , , , ,2147 0, , , , ,3081 0,0761 0, , , , , , , , , , , , , , , , , ,7014 0, , ,6572 0, , ,6359 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0209 0, , , , , , , , , , , , , ,0169 0, , , , , , ,1649 0, , , ,0783 0, , , ,1383 0, , , , , , , , ,0783 0, , , , , , , ,0282 0, , , , , , , , , , ,2045 0, , , , , , , , , , , , , , , , , ,1164 0, , , , , , , , , , , , ,5166 0, , , ,3095 0, , ,259 0, , , , , , ,0217 0, , , , , , , , ,1258 0, , , ,2639 0, , , ,1057 0, , , , , , , ,5418 0,2639 0, , , , , , , , , , , , , , , , , , , , , ,052 0, , , , , , , , , , , , , , ,0482 0, , , , , , , , , ,1758 0, , , , , , , ,2959 0, , , , ,1758 0, , , , , , , , , ,1962 0, , , , , ,2265 0, , , , , ,1304 0, , , , , , , , , , , ,1248 0, ,3966 0, , , , , , , , , , , , , , , , , ,4226 0, , , , , , , , , , , , , , , , ,3276 0, , , , ,0232 0,096 0, , , , , , ,3244 0, , , , , ,096 0, , , , , , , , ,424 0,4914 0, , , , , ,3947 0, ,4563 0, ,4013 0, , ,3074 0, , , , , ,3648 0, , , , , ,4491 0, , , , , , , , ,0838 0, , , , , , , , , , , , , , , , , , , , , , , , , ,6253 0, , , , , , , , ,0622 0,437 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,8463 0, , , , , , ,5224 0, , , ,3432 0, , , , , , , , ,0668 0, , ,2993 0, , , , , ,3011 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0788 0, ,5545 0,2986 0, , , , , , , , , , ,0493 0, , , , , , , , , , , , ,0493 0, ,054 0, , , ,0372 0,0042 0,2399 0, , , , , , ,054 0, , , , , ,0889-0, , , , ,1226 0,1158 0, , , , , , , , ,0783 0, , , , , , , , , , , , , , , ,0119 0, , ,4506 0, , , , , , , , ,0505 0, , , , , , ,2433 0, , , ,11758 Apêndice XXIII Z: Matriz de Fst para populações de DNA mitocondrial com quatro haplogrupos de n10
195 196 Tehuelche Argentina Teribe Teribe Tikúna Tikúna Brasil Tikúna Colombia Toba (chaco) Toba (Formosa) Trinitario Bolivia Tucano Tule-Cuna Uto-Aztecan California Uto-Aztecan Central Uto-Aztecan Northern Waiãpi Wapishana Brasil Washo Waunana Wayuu Wayuu Colombia Wounan Xavante Yaghan Chile Yakama Yakima Yámana (Yaghan) Yaminawa Yanomama Yanomama Brasil Yanomami Yanomamö Yok-Utian Yuko-Yukpa Yuman Yuman (Hokan) Yuman Delta Yuman Pai Yuman River Yuracaré Zapotec Zenu Zoro Zuni Zuni (penutian) Tehuelche Argentina Teribe Teribe Tikúna Tikúna Brasil Tikúna Colombia 0 0, , , , , , , , , , Toba (chaco) 0, , , , , ,1116 0, , ,155 0,1936 0, Toba (Formosa) 0, ,2542 0, , , , , Trinitario Bolivia Tucano Tule-Cuna Uto-Aztecan California 0, , , , , , ,09 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , Uto-Aztecan Central 0, , , , , , , , , , , , Uto-Aztecan Northern 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,7928 0, , ,1065 0, ,0748 0, , , , , , , , ,4652 0, , ,2588 0, , , , , , , ,0177 0, , , , , , , , , , , ,1894 0, , , , , , , , , , , , , , , , , , , , , , , ,1232 0, , , , , , , , , , ,3431 0, , , , , , , ,1653 0, , , , , , , , , , , , , , , , , , , , , , , , ,7423 0, , , , ,219 0, , , , ,3448 0,1749 0, , , ,6836 0, , , ,0236 0, , , , , , , , , , , , , , , , , , , , ,0944 0, , , , ,733 0,0522 0, , , , , , , , , , , , , , , , , , , ,2048 0, , , , , , , , , , , , , , , , , , , ,2112 0, , , , , , , , , , , ,2544-0,0371 0,1702 0, , ,4014 0, , , , ,2029 0, , , , , , , , , , , , , , , , , ,0286 0, , , , , , , , , , ,0501 0, , , , , , , , , , , , , , , , , , , , , , , , , ,4708 0, , , , , , , , , , , , , , , ,6354 0, ,3723 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,2047 0, ,1113-0, , , , , , , , , , , ,0408 0, , , , , , , , ,5102 0,1011 0, , , , ,0148 0, , , , , , , , , , , , , , ,023 0, , , ,0109 0, ,361 0, , , , , , ,1654 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,3127 0, , , , ,51933 Waiãpi Apêndice XXIII AA: Matriz de Fst para populações de DNA mitocondrial com quatro haplogrupos de n10 Wapishana Brasil Washo Waunana Wayuu Wayuu Colombia Wounan Xavante Yaghan Chile Yakama Yakima Yámana (Yaghan) Yaminawa Yanomama Yanomama Brasil Yanomami Yanomamö Yok-Utian Yuko-Yukpa Yuman Yuman (Hokan) Yuman Delta Yuman Pai Yuman River Yuracaré Zapotec Zenu Zoro Zuni Zuni (penutian) Wapishana Brasil Washo Waunana Wayuu Wayuu Colombia 0 0, , , , , , Wounan Xavante Yaghan Chile Yakama Yakima Yámana (Yaghan) 0, , , , , , , , , ,5212 0, , ,4933 0, , , ,2729 0, , , , , , , , , , , , , , , , , , , ,0552 0, , Yaminawa Yanomama Yanomama Brasil 0, , , , , , , , , , , , ,3985 0, , , , , , , , ,3158 0, , ,0209 0, , , , , , , , ,1937 0, , , ,1141 0, , , , , , , , , , , , , , ,68 0, , , , , , , , , , ,2041 0, , , , , , , , ,1115 0, , , , , , , ,1831 0,3526 0, , , , , , , , ,8944 0, , , , ,7385 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0237-0, ,1619 0,0525 0, , , , , , , ,0663 0, ,3507 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , ,2065 0, ,2826 0, , , , ,0386 0, , , , , , , , , , , , , , , ,2851 0, , , , , , , , , , , ,0798-0, ,0364 0, , , , , , , , , , , , , , , , ,0928 0, , , , , , , ,4401 0, , , , , , , , , , , , ,3921 0, , , , , , , , , , , , , , , ,57713 Yanomami Apêndice XXIII AB: Matriz de Fst para populações de DNA mitocondrial com quatro haplogrupos de n10 Yanomamö Yok-Utian Yuko-Yukpa Yuman Yuman (Hokan) Yuman Delta Yuman Pai Yuman River Yuracaré Zapotec Zenu Zoro Zuni Zuni (penutian) Yanomamö Yok-Utian Yuko-Yukpa Yuman Yuman (Hokan) 0 0, , , , , , Yuman Delta Yuman Pai Yuman River Yuracaré Zapotec Zenu Zoro Zuni Zuni (penutian) 0, , , , , , , , , , , ,4358-0, , , , , ,5788-0, , , , , , , , , ,1502 0, , , , , , , , , , , , , , , , , , , , , , ,0131 0, , , , , , , , , , , , , , , , , , , , , , , , , , ,1099-0, , , , , , , Apêndice XXIII AC: Matriz de Fst para populações de DNA mitocondrial com quatro haplogrupos de n10
196 197 Ache Akimal Aleuts St. Ancash Aónikenk Apache Arara Arequipa Assurini Atacameno Atacameno2 Atacamenos O odham Paul Peru Awá- Guajá Aymara Aymara2 Ache Akimal O odham Aleuts St. Paul Ancash Aónikenk Apache Arara Arequipa Peru Assurini Atacameno Atacameno2 Atacamenos Awá-Guajá Aymara Aymara2 Baníwa Bella Coola Bella Coola2 Bella Coola3 Bella Coola4 Boruca Bribi-Cabecar Cayapa Cayapa Ecuador Cherokee Cherokee Oklahoma Red Cross Cherokee Stillwell Cheyenne/ Arapahoe Cheyenne/ Araphoe2 Chickasaw/ Choctaw Chimane Chimila Chippewa Chippewa Turtle Muntain Chippewa Wisconsin Chippewa/ Kickapoo Choctaw Choroti Choroti2 Chumash Ciboneys Cochimi Coreguaje Creek (Muskoke) Creek/Seminole Curripaco Dogrib Dogrib0 Embera Embera0 Embera01 Gambel Ilulissat (Jakobshavn) Ittoqqortoormiitt (Scoresbysund) Nuuk (Godthab) Savoonga Siberian Upernarik Uummannaq (Umanak) Yupik s s (Savoonga e Gambel) s Old Harbor s Ouzinkie Fueguian Argentina Gaviao Guahibo Guahibo-Sikuani Guambiano 0 0, , , , , , , , , , , , , ,2603 0, , ,1248 0,5398 0, ,482 0, , , , , , , , , ,1354 0, , , , , , , , , , , ,3692 0, , , , , , , , , , , , , , , ,4896 0, , , , , , , , , , , , , , , , ,5605 0, , , , , ,6588 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,1981 0, ,2315 0, , , , , , , ,1637 0, , , , , , , , , , ,5646 0, , , , , , , , , , , , , , ,7324 0, , , , , , ,0376 0,4206 0, , ,3873 0, , , , ,3749 0, , , , , , , , , , , , , ,5712 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,1422 0, , , , ,0536 0, , , , , , , ,0784 0, , ,261 0, , , , , , , , , , , , , , , , ,1186 0, , , , , , ,5471 0, , , , , ,0184 0, , , , , , ,5571 0, , , , , , , , , , , , , , , , ,5002 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0081 0, , , , , , , ,6125 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,497 0, , , , , ,558 0, , , , , , , , , , , , , , , , , , ,0104 0, , ,0301 0, , , , , , , , , , , , , , , , , , ,643 0, , , , , , , , , , , ,6824 0, , , , , ,2364 0, , , , , , , , , , , , , , , , , , , ,0788 0, , , , , , , , , ,207 0,3882 0, , , , , , , , , , , , , , , ,0919 0, ,2179 0, , , , ,4389 0, , , , , , , , , , , , , ,2874 0,5667 0, , , , , , , , , , , , ,0669 0, , , ,2024 0, , , , , , , ,7307 0, , , , , , , , , , , ,8823 0, , , , ,7199 0, , , , , , , , , , , ,3831 0, , , , ,7067 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,547 0, , , , , , , ,6934 0, , , , , ,547 0, , , , , , , ,6934 0, , , , , ,547 0, , , , , , , ,6934 0, , , , , , , ,7947 0,1766 0, , , , , , , , , ,6874 0, , , , ,0734 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,5514 0, , , , , , , ,3297 0, ,5032 0, , ,4885 0, , , , , , , ,5629 0, , , , , , , , , , , , , , , , , , , , , , ,2625 0,4738 0,4122 0, , , , , , ,2 0, , , , , , , , , , , , , , , , , , ,5305 0,3912 0, , , , , , , , , , ,2955 0, , , , , , , , ,6274 0, , , , , , , ,0459 0, , , , , , , , , , , , , , , ,0162 0, , , , , , , ,45001 Apêndice XXIV A: Matriz de Fst entre populações da América (n10) com cinco haplogrupos de DNA mitocondrial
197 198 Ache Akimal Aleuts St. Ancash Aónikenk Apache Arara Arequipa Assurini Atacameno Atacameno2 Atacamenos O odham Paul Peru Awá- Guajá Aymara Aymara2 Guane-Butaregua Guatuso Guayabero Guaymi Haida Havasupai/ Hualapai/Yavapai/ Mojave hokan (central coast) Huetar Huetar2 Huilliche Huilliche2 Huilliche Chile Huitoto Ignaciano Ijka-Arhuaco Ingano Ingano Colombia Inuit eskimo Jemez Kanamarí Katuena Katukina Katukina2 Kawéskar Kaxináwa Kayapó Kiliwa/Paipai Kogui Kraho Kumiai Kuna Kuna2 Kuna3 Mapuche Mapuche0 Mapuche (Chile) Mapuche Anecón Grande Mapuche Argentina Mapuche Chile Marúbo 110: Mataco (Chaco) Mataco (Salta) Mataco (Wichí) Argentina Mataco (Wichí) Formosa Maya Maya Contenporary Mixe Mixtec Mohawk Mohawk2 Mosetén Bolivia Movima Bolívia Murui-Muinane Muskoke Oklahoma Nahua Nahua/Cora Navajo Ngöbé Nukak Nuu-Chah-Nulth Ojibwa Manitoulin Island Ojibwa North Ontario Ojibwa/Chippewa Old Harbor Ouzinkie Paez Paiute N./Shoshoni Parakanã Pehuenche Butalelbum Pehuenche Trapa Trapa Peneunche Chile2 0, ,1538 0,3695 0, , , , , , , , , , , , ,7187 0, , ,4179 0, , , , , , , , , , , , , , , , , , ,2589 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0355 0, , , , , , , , , , , , , , ,2446 0, , , , , , , , , , , , , , , , , , , , , ,2388 0, , , , ,7107 0, , , , ,3444 0,4584 0, , , , , , , , , , , , ,1406 0, , , ,282 0, , , , ,242 0, , , , ,2034 0,1369 0,0799 0, ,293 0, , , , , , , , , , , , , , , ,2857 0, , , , ,2497 0, , , , , ,0587 0, , , , , , , , , , , , , , ,3995 0, , , , , , , , , , , , , , , , , , , , , ,5642 0,5397 0, , , , , , , , ,3991 0, , , , , , , , , , , ,0144 0, , , ,2153 0, , , , , , , , , , , , , , , , ,7045 0, , , , , , , , , , , , , , , , , , ,061 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,6205 0, , ,6721 0, , , , , , , , , , , , , , , , , , , , , , , , ,3584 0, , , , , , , , , , , , , , , , , , , , , ,3633 0, , , , , , , , , , , , , , ,4235 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , ,2808 0, , , ,1985 0, , , ,4729 0, ,4685 0, , , ,1998 0, , , , , , , , , ,0577 0, , , ,037 0, , , , , , , , , , , , , , , , , , ,5533 0, , , , , , , , , , , , , , , , , ,3813 0, , ,2988 0, , , , , , , , , , , , , , , , , , , , , , ,0096 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,1895-0, ,2563 0,2378 0, , , , , , , , , , , ,0551-0, , , , , , , , , , , , , , , , , , , , , , ,5586 0, , , , , , , , , ,4033 0, , , , ,5962 0,1532 0, , , , , , , , , , ,6261 0, , , , , , , , , , , , , , , ,4644 0, , , ,3745 0, , , , , , , , , ,1689 0,1332 0, , , ,3632 0, , , , , , , ,0524 0, , , , , , ,3574-0, , , , , , ,0256 0, , , , , , , , , , , , , , , , , ,4529 0, , , , , , , , , ,221 0, ,2611 0, , , , ,2706 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,2946 0, ,3187 0, ,3106 0,5078 0, , , ,1752 0, , , , ,4245 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,45 0, , , , , , , , , , , , , ,2658 0, , , , , , , , , , , ,2664 0,5542 0,5359 0, , , , , , , , , , ,2197 0, , , , ,2419 0, ,2269 0, , , , , , , , , , , , , , , , , , , ,5251 0,3279 0, , , , , , , ,705 0, , , , , , , , , ,4897 0, , , , ,4634 0, , , , , ,3057 0, , , , , , , , , , ,3054 0, , , , , ,0926 0, ,2315 0, , , ,3142 0, , , , , ,036 0, , , , , , , , , , , , , , , , , , , , , , ,5598 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,2817 0, , , , , ,4082 0, ,4008 0, , , , , , , , , , , , , , , , , , , ,0627 0, , , ,2194 0, , , , , , , , , , , , , , , , , , , , , , , , , , , ,1707 0, , , ,2987-0, , , , , , , , , , , , , , , , , , , , , ,31763 Apêndice XXIV B: Matriz de Fst entre populações da América (n10) com cinco haplogrupos de DNA mitocondrial
198 Ache Akimal Aleuts St. Ancash Aónikenk Apache Arara Arequipa Assurini Atacameno Atacameno2 Atacamenos O odham Paul Peru Awá- Guajá Aymara Aymara2 Penutian California Penutian California2 Piaroa Pilagá Formosa Pima Pima2 Pima3 Poturujara (Zoé) Quechan/Cocopa (Arequipa) (Tayacaja) Bolivia / Nmatsiquenga (San Martin) Aleut Saint Paul Salinan/Chumash Salish Plateau Selknam Seminole Siona Siouan Sioux Sisseton/ Wapheton Sioux Takik Taono O odham (Papago) Tayacaja Peru Tehuelche Argentina Teribe Teribe2 Tikúna Tikúna Brasil Tikúna Colombia Toba (chaco) Toba (Formosa) Trinitario Bolivia Tucano Tule-Cuna Uto-Aztecan California Uto-Aztecan Central Uto-Aztecan Northern Waiãpi Wapishana Brasil Washo Waunana Wayuu Wayuu Colombia Wounan Xavante Yaghan Chile Yakama Yakima Yámana (Yaghan) Yaminawa Yanomama Yanomama Brasil Yanomami Yanomamö Yok-Utian Yuko-Yukpa Yuman Yuman (Hokan) Yuman Delta Yuman Pai Yuman River Yuracaré Zapotec Zenu Zoro Zuni Zuni (penutian) 0, , , , , , , ,1073 0, , , , , , , , , ,1772 0, , , , , , , , , , , , , , , , , , , , ,0486 0, ,1875 0, , , , , , , , , , , ,0805-0, , , , , , , , , , , , ,3253 0, , , ,0788 0, , , , ,0717 0, , , , , , , , ,1171 0, , , , , ,1085 0, , , , , , , , , , ,1188 0, , , ,122 0, , ,1531 0, , ,0549 0, , ,0383 0, , , , , , , , , , , , , , , , ,0458 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0181 0, , , , , , , ,1403 0, , , , , , , , , , , , , , ,0198 0, , , , , , , , ,2931 0, , , , , , , , , , , , , , , , ,4473 0, , , , , , , , ,2446 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0419 0, , , , , , , , , , , , , , ,4678-0, , , , , , , , , ,3209 0, , , , ,5618-0, , , ,2619 0, , , , , , , , , , , , , , , , , , , , , , ,2467 0, ,1932 0, , , , , , , ,283 0, , , ,5919 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0181 0, , , , , , , ,1403 0, , , , , , ,1229 0, , ,3093 0, , , , , , , , , , , , , , , , , , , , , , ,44 0, , , , , , , ,7014 0,6001 0, , , , , , , , , , , , , ,0115 0, , ,3122 0, , , , , , , , , ,287 0, , , , , , , , , , , , , , , , , , ,2508 0, ,4803 0, , , , , , ,2622 0, , , , , , , ,2883 0, , , , , , , , , , ,0092 0, , , , , ,141 0, , , , , , , , , , , , , , ,1151 0, , , , ,1193 0, , , , ,3249 0, , , , , , , , , , , , , , , , , , , , , , ,2982 0,0411 0,1723 0,2854 0, , , , , , , , , , , , , , ,3253 0, , , ,0788 0, , , , ,0717 0, , , , , , , , , , , , , ,2921 0, , ,512 0, , , , , , , , , , , , , , , , , , , ,4632 0, , , , , , , , , , , , , , , , , , , ,0888 0, ,2732 0, , , ,4778 0, , , , , , ,0359 0, , , , , , , , , , , , , , , , , , ,628 0, , , , , , , , , , , , , ,3889 0, , , , , , , , , ,2265 0, , , ,283 0, , , , , , , , , , , , ,0067-0, ,0052 0, , ,0484 0, ,2922 0, , , , , , , , , , , ,6655 0, ,0995 0, ,4216 0, , , ,3774-0, , , , , , , , , , , , , , , ,0232 0, , , , , , ,0122 0, , , , , , , , , , , , ,733 0, , , , , , , , , ,3947 0, , , , , , , , , , ,1219 0, , , , , , , , , , , , , , , , , , , , ,3439 0, , , , , , , , ,3328 0, , , , , , , , , , , , , , , , , , , , , , , , , , , ,2049 0,107 0, , ,2567 0, , , ,2282 0, , , , , , , , ,4661 0, , , , , , , , , ,1703 0, , ,4966 0, , , , , ,1848 0, ,0878 0, , ,2162 0, , , , , , , , , , , , , , , , , ,0295 0, , , , , , ,1539 0, , , , , , , ,0041 0, , , , , , , , , ,0114 0,2149 0, , , , , , , , , , , ,0636 0, , , , , , , , , , , , , , , , , , ,409 0, , , , , , , , , , , , , ,5102 0, , , , , , , , , , , , , , , , ,0921 0, , , , , ,284 0, , , , , , , , , , , , ,0734 0, ,3844 0,4668-0, , ,0209-0, , , , , , ,0836 0, , , , , , , , , , , , ,01055 Apêndice XXIV C: Matriz de Fst entre populações da América (n10) com cinco haplogrupos de DNA mitocondrial
199 200 Baníwa Bella Coola Bella Coola2 Bella Coola3 Bella Coola4 Boruca Bribi- Cayapa Cayapa Cherokee Cherokee Cherokee Cheyenne/ Cheyenne/ Chickasaw Cabecar Ecuador Oklahoma Red Cross Stillwell Arapahoe Araphoe2 /Choctaw Baníwa 0 Bella Coola 0, Bella Coola2 0, , Bella Coola3 0, , , Bella Coola4 0,0449 0, , , Boruca 0,1684 0, , , , Bribi-Cabecar 0, ,1432 0, , , , Cayapa 0, , , , , , , Cayapa Ecuador 0,0554 0, , , , , , , Cherokee 0, ,2455 0, ,3177 0, , , , , Cherokee Oklahoma Red Cross 0,1162 0,1537 0, , , , , , , , Cherokee Stillwell 0, , , , , , , ,1143 0, ,0935 0, Cheyenne/ Arapahoe 0, , , , , , , , , , , , Cheyenne/ Araphoe2 0, , , , , , , , ,0081 0, , , , Chickasaw/ Choctaw -0, ,0635 0, , ,0089 0, , , , , , , , , Chimane 0, , ,2671 0, , , , , , ,2195 0, , , , ,11447 Chimila 0, , , ,1523 0, , , , , , , , , , ,10318 Chippewa 0, , , ,1568 0, , , , , , , , , , ,15537 Chippewa Turtle Muntain -0, , , , , , , , , , , , , , ,01653 Chippewa Wisconsin 0, , , , , , , , , , , , , , ,18129 Chippewa/ Kickapoo 0, , , , , , , , , , , , , , ,04214 Choctaw 0, , , , , , ,0781 0,1703 0, , , , , , ,02614 Choroti 0,1032 0, ,3734 0, , , , , ,0365 0, , , , , ,22565 Choroti2 0, , , , , , , , , , , , , , ,20707 Chumash 0, , , , , , , , , ,3052 0, , , , ,10982 Ciboneys 0, , , , , , , , , , , , , , ,41044 Cochimi 0, , , , , , , , , , , , , , ,31026 Coreguaje 0, , , , , , , , , , , , , , ,38705 Creek (Muskoke) 0, , , , , , , , , , , , , , ,12328 Creek/Seminole 0, , , , , , , , , ,1143 0, , , , ,06292 Curripaco -0, , , , , , , , ,1101 0, , , , , ,03444 Dogrib 0, , , , , , , , , , , , , , ,18936 Dogrib0 0,4326 0, , , , , , , , , , ,6752 0, , ,2782 Embera 0, , , , , , , , , ,383 0, , , , ,03268 Embera0 0, ,12 0, , , , , , , , ,1571 0, ,1025 0, ,10322 Embera01 0, , ,3304 0, , , , , , , , , , , ,1932 Gambel 0, , , , , , ,1894 0, , , , , ,0669 0, ,0792 Ilulissat (Jakobshavn) 0, , , , ,119 0, , , , , , , , , ,16417 Ittoqqortoormiitt (Scoresbysund) 0, , , , ,119 0, , , , , , , , , ,16417 Nuuk (Godthab) 0, , , , ,119 0, , , , , , , , , ,16417 Savoonga 0, ,2604 0, , , , , , , , , , , , ,18072 Siberian 0, , , , , , , , ,2028 0, ,4172 0, , , ,0952 Upernarik 0, , , , , , , , , ,5992 0, , , ,3409 0,13754 Uummannaq (Umanak) 0, , , , , , , , , ,5992 0, , , ,3409 0,13754 Yupik 0, , , , , , , , , , , , , , ,3712 s 0, , , , , , , , , , , , , , ,08576 s (Savoonga e Gambel) 0, , , ,0375 0, , ,2187 0, , ,4543 0, , , ,2031 0,06127 s Old Harbor 0, , , , , , , , , , , , ,1587 0, ,17845 s Ouzinkie 0, , , , , , , , , , , , , , ,05806 Fueguian Argentina 0, , , , , , , , , , , , , , ,46095 Gaviao 0, , , , , , , , , , , , , , ,42581 Guahibo 0, , , , , , , , ,1476 0, ,054 0, , , ,17022 Guahibo-Sikuani 0,0847 0, , , , , , ,1357 0,1455 0, , , , ,1154 0,04452 Guambiano 0, , , , , , , , , , , , , , ,49746 Apêndice XXIV D: Matriz de Fst entre populações da América (n10) com cinco haplogrupos de DNA mitocondrial
200 Guane-Butaregua 0, , , , , , , , , ,2339 0, , , , ,32042 Guatuso 0, , , , , , , , , , , , ,2349 0, ,01166 Guayabero 0, , , , , , , , , , ,1418 0, , , ,01474 Guaymi -0, , , , ,0311 0, , , , , , , ,1533 0,1691-0,03543 Haida 0, , , ,1296 0, , , , , , , , , , ,06992 Havasupai/ Hualapai/Yavapai/ Mojave 0, , , , , , , , , , , , ,1067 0, ,27988 hokan (central coast) 0, ,044 0, ,0411 0,0045 0, , , , ,2067 0, , , , ,03984 Huetar 0, , , , , , , ,2262 0, , , , , , ,07223 Huetar2 0, , , , , , , , , , , , , , ,07056 Huilliche 0, , , , , , , , , ,2077 0, , , , ,32784 Huilliche2 0, , , , , , , ,2091 0, , , , , , ,32775 Huilliche Chile 0, , , ,2125 0, ,2277 0, ,2069 0,0683 0, ,1851 0, , ,1339 0,3344 Huitoto 0, , , , , , , , , , , ,2385 0, , ,23548 Ignaciano 0, , , , , , ,1746 0, , , ,018-0, , , ,20999 Ijka-Arhuaco 0, , , , , , , , , , , , , ,2428 0,07463 Ingano 0, ,1216 0, , , , , , , ,1916-0, , , , ,1868 Ingano Colombia 0,1013 0, , , , , ,1712 0, , , , , , ,0683 0,23404 Inuit eskimo 0, , , , , , , , , , , , , , ,18546 Jemez 0, , , ,5722 0, , , , , , , , , ,4709 0,56138 Kanamarí 0, , , , , , , , , , , , , , ,09066 Katuena 0, , , , , , , , , , , , , , ,22379 Katukina 0, , , ,1887 0, , , , , , , , , , ,33957 Katukina2 0, , , , , , , , , , , , , , ,03266 Kawéskar 0, ,3407 0, , , ,6101 0, , , , , , , , ,58212 Kaxináwa 0, , , , , , ,2379 0, , , , , , , ,15256 Kayapó 0,0136 0, , , ,1604 0, , , ,1107 0, , , , , ,07827 Kiliwa/Paipai 0, , , , , , , ,1965 0, , , , ,3805 0, ,50395 Kogui 0, ,1999 0, , , ,482 0, , , , , , , , ,29565 Kraho 0, , ,3388 0, , , , , , , , , ,1256 0, ,1623 Kumiai 0, , , , , , , , , , , , , , ,38756 Kuna 0, , , , ,122 0, , , , , , , ,3412 0, ,16992 Kuna2 0, , , , , , , , , , , , , , ,01182 Kuna3 0, , , , , , , ,2113 0, , , , , , ,00216 Mapuche 0, , , , , , , , , , , , , , ,24109 Mapuche0 0, , , , , ,1514 0, , , , ,0706 0, ,0694 0, ,24245 Mapuche (Chile) 0, , , , , , , , , , , , , , ,3116 Mapuche Anecón Grande 0, , ,3071 0, , , , , , , , , , ,053 0,20394 Mapuche Argentina 0, , , , , , , , , , ,0889 0, , , ,23354 Mapuche Chile 0, , , , , , , , , , , , , ,1376 0,42147 Marúbo 0, ,2057 0, , , , , , , , , , , , , : Mataco (Chaco) 0, , , ,2099 0, , , , , , , ,2707 0, , ,3439 Mataco (Salta) 0, ,2941 0, ,3239 0, , , , , , , ,159 0, , ,33596 Mataco (Wichí) Argentina 0, , , , , ,0242 0, , , , , , , , ,35111 Mataco (Wichí) Formosa 0, , , , , , , , , ,1225 0, , , , ,27169 Maya -0, , , , , , , , , , , ,1931 0, , ,0042 Maya Contenporary -0, , , , , , , , , , , , , ,0323-0,00962 Mixe -0, , , , , , , , , , , , , ,1162-0,04054 Mixtec 0, , , , , , , ,2389 0, , , , , , ,01445 Mohawk 0, , , , , , , , , , , , , , ,07495 Mohawk2-0, , , , , , , , , , , , , , ,01074 Mosetén Bolivia 0, , , , , , , , , , ,2319 0, , , ,11338 Movima Bolívia 0, , , , , , , , , , ,0024 0, ,0712 0, ,37645 Murui-Muinane 0, , ,3431 0, , , , ,1226 0, , , , , , ,24957 Muskoke Oklahoma 0, ,0146 0, ,0182 0, , , , , ,2378 0, , , , ,13825 Nahua -0, , , , , , , , , , , , , , ,0214 Nahua/Cora -0, , , , , , , , , , , , , , ,00437 Navajo -0, , , , , , , , , , , , , ,1421 0,0245 Ngöbé 0, , , , , , , , , ,3871 0, , , , ,01086 Nukak 0, , , , , , ,524 0, , , , ,1453 0, , ,51408 Nuu-Chah-Nulth 0, , , , , , , , ,0049 0, ,1341 0, , , ,07734 Ojibwa Manitoulin Island 0, , , , , , , , , , , , , , ,12227 Ojibwa North Ontario 0, , , , , , , , , , , , ,1202 0, ,02939 Ojibwa/Chippewa 0, , , , , , , , , , , , , , ,05227 Old Harbor 0,1646 0, , ,0051 0, , , , , , ,3354 0, , , ,12369 Ouzinkie 0, , , , , , , , , , , , , , ,04794 Paez 0,0405 0, , , , , , , , , , , , , ,06571 Paiute N./ Shoshoni 0, , , , , , , , , , , , , , ,37611 Parakanã 0, , , , ,2662 0, , ,132 0, , , , , , ,2768 Pehuenche Butalelbum 0, , , , ,4105 0, , ,3528 0,1644 0,3541 0, , , ,2123 0,48676 Pehuenche Trapa Trapa 0, ,2018 0, ,2403 0, ,3591 0, , , , ,1276 0, , , ,37463 Peneunche Chile2 0,3106 0, , ,2467 0, , , , , , , , , , ,38498 Apêndice XXIV E: Matriz de Fst entre populações da América (n10) com cinco haplogrupos de DNA mitocondrial 201
201 202 Baníwa Bella Coola Bella Coola2 Bella Coola3 Bella Coola4 Boruca Bribi- Cayapa Cayapa Cherokee Cherokee Cherokee Cheyenne/ Cheyenne/ Chickasaw Cabecar Ecuador Oklahoma Red Cross Stillwell Arapahoe Araphoe2 /Choctaw Penutian California 0, , , , , , , , , ,2181 0, , , , ,2995 Penutian California2 0, , , , , , , , , , , , , , ,28022 Piaroa 0, ,0174 0, , , , , , , , , , , , ,14798 Pilagá Formosa 0, , , , , , , , , , , , , , ,27461 Pima 0, ,3099 0, ,3614 0, , , , , , , , , , ,34133 Pima2 0, , , , , , , , , , , , , , ,32497 Pima3 0, ,2938 0, , , , , , , , ,0547-0, , , ,32297 Poturujara (Zoé) 0, , , , , , , , , ,2482 0, ,2417 0,0029 0, ,12508 Quechan/Cocopa 0, , , , , , , ,123 0, , , , , , , , ,1951 0, ,2276 0, , , ,073 0, , , , , , ,24629 (Arequipa) 0,1981 0, , , , , , , , , , , , , ,33007 (Tayacaja) 0, , ,2504 0,1038 0, , ,1214 0, ,0029 0, , , , , ,16544 Bolivia 0, , , , , , , , , , , , , , ,35454 / Nmatsiquenga (San Martin) 0, , , , , , , , , , , , , ,1627 0,28739 Aleut Saint Paul 0, , , , , , , , , , , , , , ,39209 Salinan/Chumash 0, ,044 0, ,0411 0,0045 0, , , , ,2067 0, , , , ,03984 Salish Plateau 0, , , , , , , , , , , , , , ,14977 Selknam 0, , , , , , , , , ,1977 0,1236 0, , , ,42022 Seminole -0, ,0527 0, , , , ,0204 0, , ,3126 0, , , ,1166-0,02575 Siona 0, , , , , , , , , , , , , ,1383-0,05106 Siouan -0, , , , , , , ,0687 0, , , , , , ,0066 Sioux Sisseton/ Wapheton Sioux -0, , , , , , ,0506 0, , , , ,197 0, , ,00781 Takik 0, , , , , , , , ,108 0, , , , , ,37272 Taono O odham (Papago) 0, , , , , , , , , , , , , , ,36932 Tayacaja Peru 0, , ,2504 0,1038 0, , ,1214 0, ,0029 0, , , , , ,16544 Tehuelche Argentina 0, , , , , , , , , , ,2007 0, , , ,39333 Teribe 0, , , ,1148 0, , , , , , , , , , ,01531 Teribe2 0, , , , , , , , , , , , , , ,1908 Tikúna 0,156 0, , , , , , , , , , , ,0248 0, ,21616 Tikúna Brasil 0, , , , , , , , , , , , , , ,32513 Tikúna Colombia 0, , , , , , , , , , , , , , ,2658 Toba (chaco) 0, , , ,1924 0, , , , , ,1239 0, , , , ,23156 Toba (Formosa) 0, ,0759 0, , , , , , , , , ,1725 0, , ,16875 Trinitario Bolivia 0, , ,342 0, , , , , , , , ,0185 0,0611 0, ,22257 Tucano 0, , , , , , , , , , , , ,078 0, ,37056 Tule-Cuna -0, , , ,0781 0, , , , , ,1897 0,0958 0, , , ,00327 Uto-Aztecan California 0, , , , , ,2419 0, , , , , , , , ,3387 Uto-Aztecan Central 0, ,3099 0, ,3614 0, , , , , , , , , , ,34133 Uto-Aztecan Northern 0, , , , , , , , , , , , , , ,3508 Waiãpi 0, , , , , , , , , , , , , , ,08073 Wapishana Brasil 0, , , , , , , , , , , , , , ,44238 Washo 0, , , , , , , ,1286 0, , , , , , ,34796 Waunana 0, , , , , , , , , , , , , , ,3785 Wayuu 0, , , , , , , , , , , , , , ,27251 Wayuu Colombia 0,0504 0, , , , , , , , , , , , , ,16288 Wounan 0, , , , , , , ,1342 0, ,1312-0,0371 0, , , ,1853 Xavante 0, , , , , , , ,1567 0, , , , , , ,43181 Yaghan Chile 0, , , ,2597 0, , , , , , , , , , ,41823 Yakama 0, , , , , , , , , , ,2652 0, , , ,35405 Yakima 0, , , , , , ,2254 0, , , , , ,2659 0, ,36836 Yámana (Yaghan) 0, ,2418 0, , , , , , , , , , , , ,45996 Yaminawa 0, , , , , , , , , , , , ,0157 0, ,06279 Yanomama 0, , , , , , , ,1518 0, , ,0403 0, , , ,26761 Yanomama Brasil 0, , , , , , , , , , , , , , ,38483 Yanomami 0, ,3638 0, , , , , , , , , ,2056 0, , ,48453 Yanomamö 0, , , , , , , , , , ,1349 0, , , ,35021 Yok-Utian 0,1944 0, , , , , , ,1572 0, , , , ,1091 0, ,28695 Yuko-Yukpa 0, , , , , ,5094 0,7284 0, , , , , ,7107 0, ,8149 Yuman 0, , , , , , , , ,2384 0,1462 0, , , , ,37335 Yuman (Hokan) 0, , , , ,4081 0, , , , , , , , , ,37424 Yuman Delta 0, , ,5201 0,3915 0, , ,3036 0, , , , , , , ,387 Yuman Pai 0, , , , , , , ,1087 0, , , , , , ,34892 Yuman River 0,2475 0, , , , , , , , , , , , , ,3958 Yuracaré -0,0247 0,0605 0, , , , , , ,0268 0, , , , , ,04651 Zapotec -0, , , , , , ,0821 0, , , , , , , ,10049 Zenu 0, , , , , , , , , ,0564 0, ,0018 0, , ,17648 Zoro 0, , , , , , , , ,0568 0, ,2523 0,3233 0, , ,33616 Zuni 0,2401 0, , , , , , , ,2593 0, , , ,3023 0,3036 0,36641 Zuni (penutian) 0, ,3002 0, , , , , , , , , , , , ,28622 Apêndice XXIV F: Matriz de Fst entre populações da América (n10) com cinco haplogrupos de DNA mitocondrial
202 Chimane Chimila Chippewa Chippewa Turtle Muntain Chippewa Chippewa Choctaw Choroti Choroti2 Chumash Ciboneys Cochimi Coreguaje Wisconsin /Kickapoo 203 Creek (Muskoke) Chimane 0 Chimila 0, Chippewa 0, , Chippewa Turtle Muntain 0, , , Chippewa Wisconsin 0, ,3275-0, , Chippewa/ Kickapoo 0, , , ,0024 0, Choctaw 0, , , , , , Choroti 0, , , , , , , Choroti2 0, , , , , , , , Chumash 0, , , , , , , , , Ciboneys 0, , , , , , , , , , Cochimi 0, ,615 0, , , , , , ,0223 0,3063 0, Coreguaje 0,3301 0, , , , , , , , , , , Creek (Muskoke) 0, , , , , , , , , , , , , Creek/Seminole 0, , , , , , , , , , , , , ,01119 Curripaco -0, , , , , , , , , , , , , ,13686 Dogrib 0, , , , , , , , , , , , , ,36451 Dogrib0 0, , , , , , , , , , , , , ,34681 Embera 0, , , , , , ,0346 0, ,2661 0, , , , ,15287 Embera0-0, , , , , , ,1635-0, ,0267 0, , , , ,08827 Embera01 0, , , , , , , , , , , , , ,16119 Gambel 0, , , ,0551 0, , , , , , , , , ,04182 Ilulissat (Jakobshavn) 0, , , , , , ,1186 0, , , , , , ,29128 Ittoqqortoormiitt (Scoresbysund) 0, , , , , , ,1186 0, , , , , , ,29128 Nuuk (Godthab) 0, , , , , , ,1186 0, , , , , , ,29128 Savoonga 0, , , , , , , , , , , , , ,30448 Siberian 0, , , , , , , ,4287 0, , , ,5272 0, ,15669 Upernarik 0,4005-0, , , , , , , , , , , , ,2776 Uummannaq (Umanak) 0,4005-0, , , , , , , , , , , , ,2776 Yupik 0, , , , , , , , , , , , , ,1012 s 0, , , , , ,1489 0, , , , , , , ,17405 s (Savoonga e Gambel) 0, , , , , , , , , , , , , ,14192 s Old Harbor 0, , , , , , , , ,2667 0, , , , ,04404 s Ouzinkie 0, , , , , , , , , , , , , ,10635 Fueguian Argentina 0, , , , , , , , , , , ,3043 0, ,16589 Gaviao 0, , , , , , , , , ,2023 0,3178 0, , ,1086 Guahibo 0, , , , , , , , ,1798 0, , , , ,16355 Guahibo-Sikuani 0, , , , , , , , , , , , , ,14797 Guambiano 0, , , , , , , , , ,3904 0, , , ,29279 Apêndice XXIV G: Matriz de Fst entre populações da América (n10) com cinco haplogrupos de DNA mitocondrial
203 Chimane Chimila Chippewa Chippewa Turtle Muntain Chippewa Chippewa Choctaw Choroti Choroti2 Chumash Ciboneys Cochimi Coreguaje Wisconsin /Kickapoo Creek (Muskoke) Guane-Butaregua 0, , , , , , , , , , , , , ,17912 Guatuso 0, , , , , , ,022 0, , , , , , ,2064 Guayabero 0, , , ,0167 0, , , , , , , , , ,09478 Guaymi 0, , , , ,2293 0, , , , , , , , ,13838 Haida 0, , , , ,3056 0, , , , , , , , ,21711 Havasupai/ Hualapai/Yavapai/ Mojave 0, , , , , , , , , ,2977 0,2199-0, , ,18648 hokan (central coast) 0, , , , , , ,0668 0,056 0, , ,2131 0, , ,04011 Huetar 0,2647 0, , , , , , , , ,0077 0, , , ,07896 Huetar2 0, , , , , , , , , , , , , ,08616 Huilliche 0, , , , , ,2685 0,361 0, , , , , , ,0874 Huilliche2 0, , , , , , ,3624 0, , , , , , ,08212 Huilliche Chile 0, , , , , , , , , ,1916 0, , , ,08769 Huitoto 0, , , , , , , , , , , , , ,00113 Ignaciano 0,1157 0, , , , , ,2863 0, , , , , , ,1414 Ijka-Arhuaco 0,3516 0, , , , , , , , , ,5321 0, , ,21849 Ingano 0, , , , , , , , , , , , , ,14401 Ingano Colombia 0, , ,0609 0,157 0,1244 0, , , , , ,1944-0, , ,16272 Inuit eskimo 0, , ,5249 0, , , , , ,5747 0, , , , ,29346 Jemez 0, ,787 0,4968 0, , , , , , , , , ,5112 0,40043 Kanamarí 0, , , , , , ,1434 0, , , , , , ,02063 Katuena 0, , , , , , , , , ,0557 0, , , ,03596 Katukina 0, , , ,2307 0, , , , , , , , , ,08768 Katukina2 0, , ,246 0, , , ,0497 0, , , , , , ,16421 Kawéskar 0, , , ,5132 0, , , , , , , , ,498 0,21798 Kaxináwa 0, , , , , , , , , , , , ,1207 0,10826 Kayapó -0, , , , , , ,1411 0, , , , , , ,14465 Kiliwa/Paipai 0, , , , ,4108 0, , , , , , , , ,36149 Kogui 0, , , , , , , , , , , , , ,21431 Kraho -0, , , , , , , , ,008 0, , , , ,14738 Kumiai 0,1658 0, , , , , , , ,0229 0, , , , ,25925 Kuna 0, , ,4775 0, , , , ,6028 0, , ,7067 0, , ,29415 Kuna2 0, , , , , , , , , , , ,4049 0, ,17867 Kuna3 0, , , , , , , , ,3676 0, , , , ,20896 Mapuche 0, , ,0825 0, , , , ,0102 0, , , , , ,08437 Mapuche0 0, , , ,1853 0,1344 0, , , , , , , , ,07318 Mapuche (Chile) 0, , , , , , , , , , , , , ,09823 Mapuche Anecón Grande 0,095 0, , , , , , , , , , , , ,05554 Mapuche Argentina 0,1378 0, , ,1797 0, , , , , , , , , ,07256 Mapuche Chile 0, , ,2278 0, , , , , , , , , ,19 0,15811 Marúbo 0, , , , , , , , ,0523 0, , , , , : Mataco (Chaco) 0, , , , , , , ,0576 0, ,2043 0, , , ,08664 Mataco (Salta) 0, , ,2652 0, ,3063 0, , , , , , , , ,19636 Mataco (Wichí) Argentina 0, , , , , , , , , , , , , ,19896 Mataco (Wichí) Formosa 0, , , , , , , , , , , , , ,15008 Maya 0, , , , , , ,0224 0,1347 0, , , , , ,0738 Maya Contenporary 0, ,1707 0, , , , , , ,0881 0, , , , ,048 Mixe 0, ,1834 0, , , , , , , , , , ,3695 0,10858 Mixtec 0, , , ,0564 0, , , ,4185 0, , , , , ,19957 Mohawk 0, , , , , , , , , , , , , ,1197 Mohawk2 0, , , , , , , , , , , , , ,1059 Mosetén Bolivia -0,0355 0, , , , , , , , , , , , ,15712 Movima Bolívia 0, , ,1059 0, ,1158 0, , , , , , , , ,18716 Murui-Muinane 0, , ,0343 0, , , , ,031-0,0132 0,13 0,0184 0, , ,05792 Muskoke Oklahoma 0, , , , , , , , , ,008 0, , , ,00981 Nahua 0, , , , , , ,0038 0, , , , , , ,12187 Nahua/Cora 0,0216 0, , , ,1591 0, , , ,1185 0,1343 0, , , ,10667 Navajo 0, , , ,0356 0, , , , , , , , , ,13032 Ngöbé 0, ,1655 0, , , ,1077 0,0012 0,2456 0, , ,4871 0, , ,16107 Nukak 0, , , ,3995 0, , , , , , , , , ,33285 Nuu-Chah-Nulth 0,1728 0,1642 0, , , , , , , , , , , ,01044 Ojibwa Manitoulin Island 0,1762 0, , , , , , , , , ,1702 0, , ,1047 Ojibwa North Ontario 0, ,1004 0, , , , , ,3011 0, , , ,367 0, ,15104 Ojibwa/Chippewa 0, ,1741 0, , , , , , ,1783 0, , , , ,12108 Old Harbor 0,2832 0, , , ,2633 0, ,1042 0,2952 0, , , , , ,06685 Ouzinkie 0, , , , , , ,0274 0, , , , ,4486 0, ,12941 Paez 0, , , , , , , , , , , , ,2496 0,12539 Paiute N./Shoshoni 0, , , , ,3007 0, , , , , , , , ,13945 Parakanã 0, , , , , , , , , , , , , ,07076 Pehuenche Butalelbum 0, ,6521 0,3197 0, , , , , , , , , , ,15461 Pehuenche Trapa Trapa 0, , ,188 0, , , , , ,118 0, , , , ,12451 Peneunche Chile2 0, , , , ,2147 0, , , , , , , , ,13153 Apêndice XXIV H: Matriz de Fst entre populações da América (n10) com cinco haplogrupos de DNA mitocondrial 204
204 205 Penutian California Chimane Chimila Chippewa Chippewa Turtle Muntain Chippewa Chippewa Choctaw Choroti Choroti2 Chumash Ciboneys Cochimi Coreguaje Wisconsin /Kickapoo Creek (Muskoke) 0,1591 0, , , , , , , , ,1982 0, , , ,07472 Penutian California2 0, , ,1819 0, , , , , , , ,2384 0, ,2963 0,05904 Piaroa 0, ,369 0, , , , , , , , , , , ,0102 Pilagá Formosa 0, ,4843 0, , , , ,3281-0,0139-0, , , , , ,0895 Pima 0,1481 0, , , , , , , , , , , , ,23834 Pima2 0, , , , , , , , , , , , , ,22009 Pima3 0, , ,1379 0, , , , , , , , , , ,23306 Poturujara (Zoé) 0, , , , , ,0721 0, , , , , , , ,00318 Quechan/Cocopa 0, , , , ,2591 0, , , , , , , , , , , , , , , , , , , , , , ,12029 (Arequipa) 0, , , , , , , , , , , , , ,2179 (Tayacaja) 0,0939 0, ,0955 0, , , , , , ,1001 0, ,1017 0, ,02533 Bolivia 0, , , , , , , , , , ,4912 0,1552 0, ,2757 / Nmatsiquenga (San Martin) 0,0934 0, , , , , , , , , , , , ,12723 Aleut Saint Paul 0, , , , , , , , , , , , ,4604 0,10396 Salinan/Chumash 0, , , , , , ,0668 0,056 0, , ,2131 0, , ,04011 Salish Plateau -0, , , , , , , , , , ,3161 0, , ,09969 Selknam 0, , , , , , , ,2032 0, , , , , ,13576 Seminole 0, , , , , , , , , , , , , ,09799 Siona 0, , , , , , , , , , , , ,4201 0,13144 Siouan 0, , , , ,0867-0, , , , , , , , ,06067 Sioux Sisseton/ Wapheton Sioux 0, , ,079-0, , , , , , , , , , ,074 Takik 0, , , , , , , , , , , , , ,14786 Taono O odham (Papago) 0, , , , ,234 0,2812 0, , , , , , , ,23919 Tayacaja Peru 0,0939 0, ,0955 0, , , , , , ,1001 0, ,1017 0, ,02533 Tehuelche Argentina 0, , ,2264 0,3246 0,2388 0,2974 0, , , , , , , ,10259 Teribe 0, , , , , , , , , , , , ,4975 0,18169 Teribe2 0, , ,5134 0, ,3897 0, , ,6364 0, , , ,7237 0, ,30438 Tikúna 0, , ,0969 0, , , , , , , , , , ,03782 Tikúna Brasil 0, , , , ,1671 0, , ,1815 0, , , , , ,05744 Tikúna Colombia 0, , , , , ,1752 0, ,0863 0, , , , ,1046 0,06501 Toba (chaco) 0, , , , , , ,2876-0, , , ,2539 0, ,2583 0,08711 Toba (Formosa) 0, , , , , , ,209-0, , ,0927 0, , , ,01474 Trinitario Bolivia 0, , , , , , , , ,0236 0, , , , ,14541 Tucano 0, , , , , , , , , , ,0203 0, , ,12219 Tule-Cuna 0, , , ,0236 0, , , , , , , , , ,08237 Uto-Aztecan California 0, ,5977 0, , , , , , , , , , , ,11291 Uto-Aztecan Central 0,1481 0, , , , , , , , , , , , ,23834 Uto-Aztecan Northern 0,2194 0, , , , , , , , , , , , ,13055 Waiãpi 0, , ,3157 0, , , , , , , , , , ,14897 Wapishana Brasil 0, , , , , , , ,1332 0, , ,2487 0, , ,11625 Washo 0, , , , , , , , , , , , , ,20351 Waunana 0, , , , , , ,4509 0, , , , , , ,24545 Wayuu 0, ,5115 0, , , ,1439 0, , , , , , , ,18089 Wayuu Colombia 0, , , , , ,0984 0, , , , , , ,0776 0,12608 Wounan 0, , , , , , , , , ,1601 0, ,0355 0, ,12288 Xavante 0, , , , , , , , , , , , , ,32758 Yaghan Chile 0, , , ,325 0, , , , , , , , ,1859 0,14075 Yakama 0, , , , ,3044 0, , , , , ,3957 0, , ,22224 Yakima 0, , , , ,3228 0,3196 0,4311-0, , , ,4077 0, , ,22523 Yámana (Yaghan) 0,4359 0, , , ,2178 0, , , , , , , , ,16227 Yaminawa 0, , , , , , , , , ,0361 0, , , ,0936 Yanomama 0, , , ,1706 0, , , , , , , , , ,16577 Yanomama Brasil 0, , , , , , , , ,0648 0, , , , ,15948 Yanomami 0, , , , , , , , , , , , ,0228 0,31312 Yanomamö 0, , , , , , , , , , , , , ,22659 Yok-Utian 0, , , , , , , , ,0456 0, , , , ,03241 Yuko-Yukpa 0, , , , , , , , , , , , , ,55213 Yuman 0, , , ,3107 0, , , , , , , ,0196 0, ,27494 Yuman (Hokan) 0, , , , , , , , , , , , ,1944 0,2755 Yuman Delta 0, , ,1799 0, , ,2827 0, , , , , , ,1146 0,25854 Yuman Pai 0, ,6259 0, , , , , , ,0387 0, , , , ,25071 Yuman River 0, , , , , , , , , , , , , ,26949 Yuracaré 0, , , , , , , , , , , , , ,06546 Zapotec 0, ,3981 0,0105 0,0313 0, , , , , , , , , ,08771 Zenu 0, , , , , , ,2425-0, , , , , , ,11241 Zoro 0, ,4911 0,2396 0,2798 0, , , , , , , , , ,04845 Zuni 0, , , , , ,3204 0, , , , , , , ,28229 Zuni (penutian) 0, , , ,2469 0,287 0, , , , , , ,1119 0, ,22978 Apêndice XXIV I: Matriz de Fst entre populações da América (n10) com cinco haplogrupos de DNA mitocondrial
205 206 Creek/ Curripaco Dogrib Dogrib0 Embera Embera0 Embera01 Seminole Gambel Ilulissat (Jakobshavn) Ittoqqortoormiitt (Scoresbysund) Nuuk (Godthab) Savoonga Siberian Upernarik Uummannaq (Umanak) Creek/Seminole 0 Curripaco 0, Dogrib 0, , Dogrib0 0, , , Embera 0, , , , Embera0 0, ,0109 0, , , Embera01 0, , ,5939 0, , , Gambel 0, , ,2556 0, , , , Ilulissat (Jakobshavn) 0, , , , , , , Ittoqqortoormiitt (Scoresbysund) 0, , , , , , , Nuuk (Godthab) 0, , , , , , , Savoonga 0, , , , , , , , , , , Siberian 0, , , , , , , , , , , , Upernarik 0, ,3769-0, , , , , , , Uummannaq (Umanak) 0, ,3769-0, , , , , , , Yupik 0, , , , , , , , , , , ,4956 0, , , s 0, , , ,1378 0, , , , , , , , , , , ,35052 s (Savoonga e Gambel) 0, , , , , , , , , , , , , , , ,31344 s Old Harbor 0, , , , , , ,3346 0, , , , , , , , ,08921 s Ouzinkie 0, , , , , , , , , , , , , , , ,26813 Fueguian Argentina 0, , ,7223 0,731 0, , , ,3008 0, , , , , , , ,17999 Gaviao 0, , , , , , , , ,6605 0,6605 0,6605 0, , , , ,0219 Guahibo 0, , , , , , , , , , , , ,2752 0, , ,39343 Guahibo-Sikuani 0, , , , , ,1874 0, , , , , , , , , ,34684 Guambiano 0, ,5298 0, , , , , , , , , , , , , ,46708 Apêndice XXIV J: Matriz de Fst entre populações da América (n10) com cinco haplogrupos de DNA mitocondrial Yupik
206 207 Creek/ Curripaco Dogrib Dogrib0 Embera Embera0 Embera01 Seminole Gambel Ilulissat (Jakobshavn) Ittoqqortoormiitt (Scoresbysund) Nuuk (Godthab) Savoonga Siberian Upernarik Uummannaq (Umanak) Guane-Butaregua 0, ,1609 0,6989 0, , , , ,3483 0, , , , ,4939 0,5914 0,5914 0,35505 Guatuso 0, ,174 0, , , ,2616 0, , , , , , , , , ,43718 Guayabero -0, , , , , , , , , , , , , ,2102 0,2102 0,31767 Guaymi 0, , , , , , , , ,2578 0,2578 0,2578 0, , , , ,3938 Haida 0, , , , ,0631 0, , , , , , , , ,0112 0,0112 0,42796 Havasupai/ Hualapai/Yavapai/ Mojave 0, , , , , , , , , , , , , , , ,43091 hokan (central coast) -0, , ,4266 0, , , , , , , , , , , , ,13667 Huetar 0,082 0,1955 0,2173 0, , , , , , , , , , , , ,23394 Huetar2 0, , , , , , , , , , , , , , , ,24496 Huilliche 0, , , , , , , , , , , , , , , ,13723 Huilliche2 0, , , , , , , , , , , , , , , ,13269 Huilliche Chile 0, ,2852 0, , , , , , , , , , , , , ,13908 Huitoto 0, ,2604 0,5825 0, , , , , , , , , , , , ,05279 Ignaciano 0, , , , , , , , , , , , ,425 0, , ,387 Ijka-Arhuaco 0, , , , , ,3528 0, ,1059 0, , , , , , , ,44096 Ingano 0,0751 0, , , , , ,1613 0, , , , , , , , ,38947 Ingano Colombia 0, , , , ,2955 0, , , , , , , , , , ,40337 Inuit eskimo 0, , , , , , , , , , , , , , , ,48903 Jemez 0, , , , , ,2575 0, , , , , , , , , ,57611 Kanamarí -0,0122 0, , , , , , , ,3795 0,3795 0,3795 0, , , , ,24898 Katuena 0, , , , , ,1708 0, , , , , , , , , ,18366 Katukina 0, , , , ,4088 0, , , , , , , , ,61 0,61 0,18919 Katukina2 0, , , , , , , , , , , , , ,125 0,125 0,41012 Kawéskar 0, , , ,9098 0, , , , , , , , , , , ,09596 Kaxináwa 0, ,2311 0, , , , , , , , , ,4822 0, , , ,34669 Kayapó 0, , ,5501 0,6995 0, , , , , , , , , , , ,40573 Kiliwa/Paipai 0, , , , , , , , , , , , , , , ,56115 Kogui 0, , , , , , , ,2302 0, , , , , , , ,43568 Kraho 0, , , , , , , , , , , ,6267 0, , , ,40794 Kumiai 0, , , , , , , , , , , , , , , ,48505 Kuna 0, ,4285 0, , , , , ,0094 0, ,49683 Kuna2 0, , ,1996 0, , , , , , , , , , , , ,41617 Kuna3 0, , , , , , , , , , , , , , , ,43803 Mapuche 0, , , , , , , ,2165 0, , , , , , , ,21468 Mapuche0 0, , , , , , ,0892 0,1983 0, , , , , , , ,19625 Mapuche (Chile) 0, ,2841 0, , , , , , , , , , , , , ,2286 Mapuche Anecón Grande 0, , , , , , , , ,4603 0,4603 0,4603 0, , , , ,22917 Mapuche Argentina 0, , , , , , ,078 0, , , , , , , , ,20052 Mapuche Chile 0, , , , , , , , , , , ,5985 0, ,5706 0,5706 0,20367 Marúbo 0, , , , , , , , , , , ,7067 0, , , , : Mataco (Chaco) 0, , , , , , , , , , , , , ,5711 0,5711 0,12237 Mataco (Salta) 0, , , , ,3718 0, , ,3637 0, , , ,6273 0, , , ,36235 Mataco (Wichí) Argentina 0, , , , , , , ,3701 0, , , , , , , ,35155 Mataco (Wichí) Formosa 0, , , , , , , , , , , , , , , ,35058 Maya 0,0173 0, , , , ,055 0, , , , , , , , , ,30173 Maya Contenporary -0, , , , , , , , , , , , , , , ,28753 Mixe 0, , , ,4418-0, ,0424 0, , , , , , ,1432 0, , ,37275 Mixtec 0, , , , , , , , , , , , , , , ,43024 Mohawk -0, , , , , , , ,1086 0, , , , , , , ,31869 Mohawk2 0, , , , , , , , , , , , , , , ,34185 Mosetén Bolivia 0, , ,5641 0, , , , , , , , , , , , ,40416 Movima Bolívia 0, , , , , , , , , , , , ,5006 0, , ,36894 Murui-Muinane 0,0415 0, , , , , , , , , , , , , , ,21898 Muskoke Oklahoma 0, , , , , , , , , , , , , , , ,08435 Nahua 0, ,0147 0, , , , , , , , , , , , , ,37762 Nahua/Cora 0, , ,3424 0, , , , ,1217 0, , , , , , , ,35577 Navajo 0, , , , ,041 0, , , , , , , , , , ,37336 Ngöbé 0, ,0041 0, , , , , , , , , , , , , ,40486 Nukak 0, , ,7966 0, , , , , , , , , , , , ,52742 Nuu-Chah-Nulth -0, , , , , , , , , , , , , , , ,14609 Ojibwa Manitoulin Island -0, , , , , , , , , , , , , , , ,29776 Ojibwa North Ontario 0, , , , , , , , , , , , , , , ,35579 Ojibwa/Chippewa -0, , , , , , , , , , , , , , , ,3218 Old Harbor 0, ,22 0, , , , , , , , , , , , , ,17368 Ouzinkie 0,0899 0, , , ,0486 0, , , , , , , , , , ,31111 Paez 0, , , , , , , ,0715 0, , , , , , , ,37248 Paiute N./Shoshoni 0, , , , ,41 0, , , , , , ,5976 0, , , ,2048 Parakanã 0, , , , , , , , , , , , , , , ,25596 Pehuenche Butalelbum 0, , , , , , ,3832 0, , , , , ,5256 0, , ,10677 Pehuenche Trapa Trapa 0,1678 0, , , , , , , , , , ,5631 0, , , ,18364 Peneunche Chile2 0, , , , , , , , , , , , , ,5302 0,5302 0,16796 Apêndice XXIV K: Matriz de Fst entre populações da América (n10) com cinco haplogrupos de DNA mitocondrial Yupik
207 208 Creek/ Curripaco Dogrib Dogrib0 Embera Embera0 Embera01 Seminole Gambel Ilulissat (Jakobshavn) Ittoqqortoormiitt (Scoresbysund) Nuuk (Godthab) Savoonga Siberian Upernarik Uummannaq (Umanak) Penutian California 0,1316 0, , , , , , , , , , ,6745 0, , , ,17427 Penutian California2 0, , , ,7422 0, ,0558 0, , , , , , , , , ,16944 Piaroa -0, , , , , , ,1033 0, , , , , , , , ,15682 Pilagá Formosa 0, , ,6412 0, , , , , , , , , , , , ,239 Pima 0, , , , ,4009 0, , , , , , , , , , ,46216 Pima2 0, , , ,7304 0, , ,0209 0, , , , , , , , ,44968 Pima3 0, , , , , , , ,3549 0, , , ,5857 0, , , ,44401 Poturujara (Zoé) -0, , , , , , , , , , , , , , , ,14668 Quechan/Cocopa 0, , , , , , ,0287 0, , , , , , ,681 0,681 0, , , , , , ,0258 0, , , , , , , , , ,29128 (Arequipa) 0, , ,7307 0, ,3831 0, , , , , , , , , , ,43361 (Tayacaja) 0, , , , , , ,0746 0, , , , , , , , ,16688 Bolivia 0, , , , , , , , ,6858 0,6858 0,6858 0, , , , ,49464 / Nmatsiquenga (San Martin) 0, , , , , ,0246 0, , ,5996 0,5996 0,5996 0, , , , ,29192 Aleut Saint Paul 0, , , , , , , , , , , , , , , ,00927 Salinan/Chumash -0, , ,4266 0, , , , , , , , , , , , ,13667 Salish Plateau 0, , , , , , ,0292 0, , , , , ,394 0,5375 0,5375 0,34882 Selknam 0,1475 0, , , , , , , , , , ,744 0, , , ,19535 Seminole 0, , , , , , , , , , , , , , , ,34025 Siona 0, , , , , , , , , , , , , , , ,38676 Siouan -0, , , , ,021 0, , , , , , , , , , ,28912 Sioux Sisseton/ Wapheton Sioux 0, , , , , , , , , , , , ,1145 0,1718 0,1718 0,31543 Takik 0, , , , , , , , , , , , , , , ,31427 Taono O odham (Papago) 0, ,2645 0, , , ,1368 0,0357 0, , , , , , , , ,45047 Tayacaja Peru 0, , , , , , ,0746 0, , , , , , , , ,16688 Tehuelche Argentina 0, , , , , , , , , , , , , , , ,12636 Teribe 0, , , , , , , , , , , ,1185 0, , , ,42067 Teribe2 0, , , , , , , , , ,50409 Tikúna 0, , ,4223 0, , , , ,0987 0, , , , , , , ,13527 Tikúna Brasil 0, , , , , ,2499 0, , , , , , , , , ,08801 Tikúna Colombia 0, , , , , , , , ,4687 0,4687 0,4687 0, , , , ,18993 Toba (chaco) 0, , , , , , , , , , , , , , , ,24636 Toba (Formosa) 0, , , , , , , , , , , , , , , ,14921 Trinitario Bolivia 0, , , , , , , , ,5004 0,5004 0,5004 0, , , , ,37515 Tucano 0, , , , , , , , , , , , , , , ,2536 Tule-Cuna 0, , , , , ,0361 0, , , , , , , , , ,34049 Uto-Aztecan California 0, , , , , , , , , , , , , ,6173 0,6173 0,2792 Uto-Aztecan Central 0, , , , ,4009 0, , , , , , , , , , ,46216 Uto-Aztecan Northern 0, , , , , , , , , , , , , , , ,21471 Waiãpi 0, , , , ,0742 0, , , , , , , , , , ,32902 Wapishana Brasil 0, , , , , , , , , , , , , , , ,08437 Washo 0, , , , , , , , ,6387 0,6387 0,6387 0, , , , ,41257 Waunana 0, ,2517 0, , , , , , , , , , , , , ,4558 Wayuu 0, , , , , , ,1504 0, , , , , , , , ,4178 Wayuu Colombia 0, , , , , , , , , , , ,4899 0, ,4094 0,4094 0,37105 Wounan 0, , ,5581 0, , ,1434 0, , , , , , , , , ,36256 Xavante 0, ,2366 0, , , , ,1193 0, , , , , , ,778 0,778 0,5338 Yaghan Chile 0, , , , , , , ,2666 0, , , , , ,6265 0,6265 0,18319 Yakama 0, ,198 0,7113 0, , , , , , , , , , , , ,40432 Yakima 0, , , , , ,0749 0, , , , , , , , , ,41036 Yámana (Yaghan) 0, , , , , , , , , , , , , , , ,20725 Yaminawa 0, ,2008 0, , , , , , , , , , ,1233 0, , ,33112 Yanomama 0, ,2521 0, , ,3361 0, , , , , , , , , , ,39638 Yanomama Brasil 0, , , , , , , , , , , , , , , ,3094 Yanomami 0, , , , , , , , , , , , , , , ,46419 Yanomamö 0, , , , , , , , , , , , , , , ,4053 Yok-Utian 0, , , , , , , , ,5831 0,5831 0,5831 0, , , , ,09969 Yuko-Yukpa 0, , , , , , , , , ,6872 Yuman 0,2764 0,266 0, , ,4248 0, , , , , , ,6305 0, , , ,47822 Yuman (Hokan) 0, , , , , , , , , , , , , , , ,48228 Yuman Delta 0, , , , , , , , , , , , , , , ,48058 Yuman Pai 0, , , , , , , , , , , , , , , ,47609 Yuman River 0, , ,7643 0, , , , , , , , ,7531 0, , , ,49001 Yuracaré 0,0082 0, , , , , , , , , , , , , , ,31734 Zapotec 0, , , , ,166 0, , , , , , , , , , ,35922 Zenu 0, , , , ,229 0, , , , , , , , , , ,34213 Zoro 0, ,3422 0, , , , , , , , , , , , , ,00805 Zuni 0, ,1774 0, , , , ,055 0, , , , , , , , ,5009 Zuni (penutian) 0, ,1032 0, , ,3382 0, ,0104 0,373 0, , , , , ,6332 0,6332 0,46482 Apêndice XXIV L: Matriz de Fst entre populações da América (n10) com cinco haplogrupos de DNA mitocondrial Yupik
208 209 s s (Savoonga e Gambel) s Old Harbor s Ouzinkie Fueguian Argentina Gaviao Guahibo Guahibo- Sikuani Guambiano s 0 s (Savoonga e Gambel) -0, s Old Harbor 0, , s Ouzinkie -0, , , Fueguian Argentina 0, , , , Gaviao 0, , , , , Guahibo 0, , , , , , Guahibo-Sikuani 0, ,0612 0, ,0422 0, , , Guambiano 0,6229 0, , , ,229 0, , , Apêndice XXIV M: Matriz de Fst entre populações da América (n10) com cinco haplogrupos de DNA mitocondrial
209 210 s s (Savoonga e Gambel) s Old Harbor s Fueguian Gaviao Guahibo Guahibo- Guambiano Ouzinkie Argentina Sikuani Guane- Butaregua Guatuso Guayabero Guaymi Haida Havasupai/ Hualapai/ Yavapai/ Mojave Guane-Butaregua 0,5051 0, ,3353 0, ,3868 0, , , , Guatuso 0, , , , , , , , , , Guayabero 0, , , , , , , , , , , Guaymi 0, , ,1994 0, , , , , , ,2988 0, , Haida 0, , , , , ,5681 0, , , , , , , Havasupai/ Hualapai/Yavapai/ Mojave 0, ,4467 0, , , , , , , , , , ,2943 0, hokan (central coast) 0, , , , , ,1649 0, , , , , , , ,2088 0,17585 Huetar 0, , ,0324-0, , , , , , , , , , , ,4071 Huetar2 0, , , , , , , , , , , , ,0973 0, ,41466 Huilliche 0, , , , , , , , , , ,4302 0, ,3367 0, ,17878 Huilliche2 0, , , , , , , , , , , , , , ,17818 Huilliche Chile 0, , , , , , , , , , , ,2659 0, , ,18066 Huitoto 0, , , , , , , , , , , ,162 0, , ,2311 Ignaciano 0, , , , , , , , , , , , ,2331 0, ,03183 Ijka-Arhuaco 0, , , , , , , , , , , , , , ,49491 Ingano 0, , , , , , ,0205 0, , , ,3414 0, , , ,13602 Ingano Colombia 0, , , , ,3009 0, ,183 0, , , ,4035 0, , , ,04334 Inuit eskimo 0, , , , , , , , , ,6456 0, ,2703 0, , ,67155 Jemez 0, , , , , , , , ,7007 0, , , , , ,28421 Kanamarí 0, , , , ,2081 0, , , , , , , , , ,05205 Katuena 0, ,2675 0, , , , , , , , , , , , ,1516 Katukina 0, , , , , , , , , , , , , , ,18641 Katukina2 0, , , , , , , , , , , , , , ,37977 Kawéskar 0,6312 0, , ,5129 0, , ,5138 0, ,5628 0, , ,4801 0, , ,52267 Kaxináwa 0, , , , , , , , , , , ,0834 0, , ,1607 Kayapó 0, , , , , , ,2918 0, , , , , , , ,14422 Kiliwa/Paipai 0, , , , ,5642 0, , , , , , , , , ,18013 Kogui 0, , , , , , ,0064 0, , , , , , , ,23537 Kraho 0, , , , , , , , , , , , , , ,01401 Kumiai 0, , , , , , , , , , , , , , ,03527 Kuna 0, , , , , , , , , , , , , , ,63594 Kuna2 0,1184 0, , , , , ,2671 0, , , , , , , ,36517 Kuna3 0, ,0869 0, ,1005 0, , , , , , , , ,0187 0, ,44013 Mapuche 0, , , , , , , ,2511 0, , , , , , ,06471 Mapuche0 0, , ,2152 0, , ,1455 0, , , , , , , , ,0634 Mapuche (Chile) 0, , , ,3392 0, , , , , , , , , ,4664 0,09454 Mapuche Anecón Grande 0, , , , , , , , , , , ,1413 0, , ,03935 Mapuche Argentina 0, , , , , , , , , , , , , , ,06197 Mapuche Chile 0, , , , , , ,2876 0, , ,3349 0, , , , ,25685 Marúbo 0, , , , ,0348 0, , , , , , , , , , : Mataco (Chaco) 0, , , , , , , , , , , , , , ,24999 Mataco (Salta) 0,5042 0, ,3538 0, , , , , , , ,4733 0, , , ,12077 Mataco (Wichí) Argentina 0, , , , , , , , , , , ,2955 0,3333 0, ,13946 Mataco (Wichí) Formosa 0, ,4175 0, ,3725 0, , , , , , , , , , ,00935 Maya 0, , , ,0722 0, , ,1121 0, , , , , , , ,17895 Maya Contenporary 0, , , , , , , , , , , , , , ,15869 Mixe 0, , , ,0948 0,4548 0, , , , , , , , , ,22391 Mixtec 0, , , , , , , ,0914 0, , , , , , ,46197 Mohawk 0, , , , , , , ,0112 0, , , , , , ,22123 Mohawk2 0, , , , , , , , , , , , , , ,20878 Mosetén Bolivia 0, , , , , , , , , , , , , , ,13059 Movima Bolívia 0, ,4472 0, ,4093 0, , , , , , , , , ,5451 0,14548 Murui-Muinane 0, , , , , , , , , , , , , ,4495 0,05417 Muskoke Oklahoma 0, , ,0323 0,1119 0, , , , , , , , , , ,21123 Nahua 0, , , , , , , , , , , , , , ,2307 Nahua/Cora 0, , , , ,4197 0, , , , , , , , , ,17353 Navajo 0, ,186 0,2275 0, , , , , , , , , ,0015 0, ,17541 Ngöbé 0, , , , , , , , , , , ,0715-0, , ,31659 Nukak 0, , , , , , , , , , , , , , ,19048 Nuu-Chah-Nulth 0, , , , , , , , , , , , , , ,21524 Ojibwa Manitoulin Island 0, , , , , , , , , , , , , , ,14744 Ojibwa North Ontario 0, , ,1774 0, , , , , , , , , , , ,34733 Ojibwa/Chippewa 0, , , , , ,3695 0, , , , , , , , ,24129 Old Harbor 0, , , , , , , ,146 0, , , , , , ,40792 Ouzinkie -0, ,0175 0, , , , , , , , , , ,0907 0, ,42532 Paez 0, , , , , ,4297 0, , , , , , , , ,23844 Paiute N./ Shoshoni 0, ,442 0, , ,1955 0, , , , , , , , , ,19879 Parakanã 0, ,3843 0, ,3308 0,0828 0,2252 0, , , , , , , , ,02377 Pehuenche Butalelbum 0, , , , ,0061 0, , , ,3759 0, , , , , ,37615 Pehuenche Trapa Trapa 0,4452 0, , , , , , , , , , , , , ,21187 Peneunche Chile2 0, , , , , ,1403 0, , , , , , , , ,23493 Apêndice XXIV N: Matriz de Fst entre populações da América (n10) com cinco haplogrupos de DNA mitocondrial
210 211 s s (Savoonga e Gambel) s Old Harbor s Fueguian Gaviao Guahibo Guahibo- Guambiano Ouzinkie Argentina Sikuani Guane- Butaregua Guatuso Guayabero Guaymi Haida Havasupai/ Hualapai/ Yavapai/ Mojave Penutian California 0, ,3941 0, , , , , , , , , , , , ,17755 Penutian California2 0, ,3794 0, , , , ,3378 0, , , , , , ,505 0,13984 Piaroa 0, , ,1114 0, , , , , , , , , , , ,09664 Pilagá Formosa 0, , , , , , , , , , ,4107 0, , , ,04633 Pima 0, , , , , , , , , , , , ,3509 0, ,03556 Pima2 0, , , , , , , , , , , , , , ,04288 Pima3 0, , , , , , , , , , , , , , ,02984 Poturujara (Zoé) 0, , , , , , , , , , ,2329 0, ,1717 0, ,23727 Quechan/Cocopa 0, , , , , ,4578 0, , , , , ,2886 0, , , , , , , , , ,2913 0, , , , , , , ,04401 (Arequipa) 0, ,4885 0, , , ,3912 0, , , , , ,2589 0, , ,04076 (Tayacaja) 0, , , , , , , , , , , , , , ,0895 Bolivia 0, , , , , , , , , , , , , , ,10033 / Nmatsiquenga (San Martin) 0,4771 0, , , , , ,3632 0, , , , , , , ,09477 Aleut Saint Paul 0, , , , , ,0138 0, , , , , , , , ,44215 Salinan/Chumash 0, , , , , ,1649 0, , , , , , , ,2088 0,17585 Salish Plateau 0, , , , ,3655 0, , , , , , , , , ,02401 Selknam 0, , , , , , , , , , , , , , ,24643 Seminole 0,0967 0, , , , , , , , , , , , , ,24961 Siona 0, ,0209 0, , , , , , , , , , ,0387 0,0008 0,32792 Siouan 0, , , , , , , , , , , , ,0166 0, ,18261 Sioux Sisseton/ Wapheton Sioux 0,1156 0, , , , , , , , , , , , , ,18521 Takik 0, , , ,4125 0, , , , , , ,5455 0, , , ,08457 Taono O odham (Papago) 0, , , ,473 0,3397 0, , , , , , , , , ,02439 Tayacaja Peru 0, , , , , , , , , , , , , , ,0895 Tehuelche Argentina 0, ,4394 0, , , ,0578 0, , , , ,5244 0, , , ,22989 Teribe 0,0603 0, , , , , , , , , , , , , ,41422 Teribe2 0, , , , , , , , , ,6417 0, , , , ,66429 Tikúna 0, ,227 0, , , , , , , , , , , , ,17699 Tikúna Brasil 0, , , , , , , , , ,3161 0, , , , ,26345 Tikúna Colombia 0, , , , , , , , ,1253 0, , , , , ,11774 Toba (chaco) 0, , , , , , ,3074 0, , , , , , , ,08211 Toba (Formosa) 0, , , , , , , , , , , , , , ,14411 Trinitario Bolivia 0,4267 0, , , , , , , , , , , , , ,03158 Tucano 0, , , , , , , , , , , , , , ,12014 Tule-Cuna 0, , , , , , , ,0676 0, , , , , , ,12541 Uto-Aztecan California 0, , , ,3892 0, , , , , , , , , , ,04236 Uto-Aztecan Central 0, , , , , , , , , , , , ,3509 0, ,03556 Uto-Aztecan Northern 0,4563 0, , , , , , , , ,0725 0, , ,3507 0, ,15253 Waiãpi -0, , , , , , , , , , ,0503 0, , , ,48809 Wapishana Brasil 0, , , , , , , ,4389 0, , ,609 0,3417 0, , ,3122 Washo 0, , , , , , , ,3882 0, , , , , , ,02466 Waunana 0, , , , , ,4172 0, , , , , , , , ,00646 Wayuu 0, , , , , , , , , , , , , , ,09266 Wayuu Colombia 0, , , ,278 0, , ,1038 0, ,1948 0, , ,0951 0, , ,00854 Wounan 0, , , , ,2406 0, , , , , , , , , ,05748 Xavante 0, , , ,55 0, , , , , , , , , , ,21365 Yaghan Chile 0,5181 0, , , , , , , , , ,5584 0, , , ,26192 Yakama 0, , , , , , , , , , , , , , ,09605 Yakima 0,5537 0,5013 0, , , , ,4265 0, , , ,5235 0, , , ,10246 Yámana (Yaghan) 0, , , , , , , , , , , , , , ,29934 Yaminawa 0, , , , , ,4047 0,0007 0, , , , , , , ,2629 Yanomama 0,4546 0, , , , , , , , , ,4248 0, , , ,01823 Yanomama Brasil 0, , , , , , , , , , , , , , ,12153 Yanomami 0, , , , , , , ,4442-0, , , , ,5349 0, ,24165 Yanomamö 0, , , , , , , , , ,0911 0, , , , ,00048 Yok-Utian 0, , , , ,1188 0, , , , , , , , , ,16503 Yuko-Yukpa 0, , , , , ,8489 0, , , , , , , , ,66711 Yuman 0, , , , , , , , , , , , , , ,01915 Yuman (Hokan) 0, , , , , , ,3465 0, , , , , , ,5617-0,01146 Yuman Delta 0,5938 0, , , ,3634 0, , , ,2937 0, , , , , ,03395 Yuman Pai 0, ,5075 0, , , , , ,4113 0, , , , , , ,00444 Yuman River 0, , , , , ,4683 0,352 0, ,3695 0, , , , , ,02205 Yuracaré 0,2426 0,1932 0, , , , , , , , , , , , ,05682 Zapotec 0, ,2678 0,2576 0, , , , , , , , , , , ,0006 Zenu 0,3789 0,3316 0, , , , , , , , , , , ,3878-0,02054 Zoro 0, , , , , , , , , , , ,2624 0, , ,31865 Zuni 0, , , , , , , , , , , , , , ,11478 Zuni (penutian) 0, , , ,4334 0, , , , , , , , , , ,06271 Apêndice XXIV O: Matriz de Fst entre populações da América (n10) com cinco haplogrupos de DNA mitocondrial
211 212 hokan (central coast) Huetar Huetar2 Huilliche Huilliche2 Huilliche Chile Huitoto Ignaciano Ijka- Arhuaco Ingano Ingano Colombia Inuit eskimo Jemez Kanamarí Katuena hokan (central coast) 0 Huetar 0, Huetar2 0, , Huilliche 0, , , Huilliche2 0, , , , Huilliche Chile 0, , , , , Huitoto 0, , ,1694 0, , , Ignaciano 0, , , , , , , Ijka-Arhuaco 0, , , , , , , , Ingano 0, , , , , ,2671 0, , , Ingano Colombia 0, , , , , , , , , , Inuit eskimo 0, , , , ,5268 0, , , , ,5153 0, Jemez 0, , , , , , , , , , , , Kanamarí -0,0219 0, , , , , , ,0057 0, , , , , Katuena 0, , , , , , , , , , , , , , Katukina 0, , , , , , , , , , , , , , ,0421 Katukina2 0, , , , ,3851 0, , , , , , , , , ,30037 Kawéskar 0, , , , , , , , , ,5148 0, , , , ,265 Kaxináwa 0, , , , , , , ,0686 0, , , , , , ,01936 Kayapó 0, , , , , , , , , , , , , , ,25737 Kiliwa/Paipai 0,439 0, , , , , , , , , ,1898 0, , , ,45973 Kogui 0,2373 0, , , , , , , ,347-0, , , , , ,08907 Kraho 0, , , , , , , , , , , , , , ,2037 Kumiai 0, , , , ,2152 0,2138 0, , , , ,0036 0, , , ,24906 Kuna 0, , , , , , , , , ,4784 0, , , , ,48851 Kuna2 0, , , , , , , , , , , ,1779 0, , ,33446 Kuna3 0, ,1106 0, , , , , , , , , ,1232 0, , ,38137 Mapuche 0, , , , , , , , , , , , , , ,08283 Mapuche0 0, , , , , , , , , ,1488 0, , , , ,04642 Mapuche (Chile) 0, , , ,0377 0, , , ,0704 0, , , , ,4153 0, ,01569 Mapuche Anecón Grande 0,038 0,2461 0, , , , , , , , , , , , ,06717 Mapuche Argentina 0, ,2585 0, , , , , , , , , , , , ,06662 Mapuche Chile 0,22 0, , , , , ,0709 0, , , , , , , ,05184 Marúbo 0, , ,4041 0, , , , , , , ,1127 0, , , , : Mataco (Chaco) 0, , , , ,024 0, ,0928 0, , ,3542 0, , , , ,17514 Mataco (Salta) 0, , ,4123 0, , , ,2698 0, , , , , , , ,28335 Mataco (Wichí) Argentina 0, , , ,1246 0, ,1271 0, ,1867 0, , , , , , ,2891 Mataco (Wichí) Formosa 0, , , ,1057 0,1135 0, , , ,468 0, ,0216 0, ,1575 0, ,17391 Maya -0, , , , , , , , , , , , , , ,13459 Maya Contenporary -0, , , , , , , , , ,0907 0, , , , ,10133 Mixe 0, , , , , , , ,1667 0,1398 0, , , ,533 0, ,20998 Mixtec 0, ,0857 0, , , , , , , ,3009 0, , , , ,34227 Mohawk 0, , , , , , , , , , , , , , ,14939 Mohawk2 0,0344 0, , , , , , , , , , , , , ,13823 Mosetén Bolivia 0, , ,2764 0, , , , , , , , , , , ,26406 Movima Bolívia 0, , , , , , , , , ,0605 0, , , , ,02688 Murui-Muinane 0, ,2779 0, , , , , , , , , , , , ,01698 Muskoke Oklahoma -0, , , , , , , , , , , , , , ,05183 Nahua 0, , , , , , , , ,1477 0, ,1929 0, , , ,22833 Nahua/Cora 0, , ,1307 0, , , , , , , , , , ,0505 0,19695 Navajo 0, , , , , , , ,1464 0,2167 0, , , ,3522 0, ,23461 Ngöbé 0, , , , , , , , , , , , , , ,30626 Nukak 0, , ,5964 0, , , , , ,6374 0,1649 0, , , , ,21053 Nuu-Chah-Nulth -0, , , , , , , ,1508 0, , , , , , ,04349 Ojibwa Manitoulin Island 0, , , , , , , , , , , , , , ,07043 Ojibwa North Ontario 0, , ,0853 0, , , , , , ,2006 0, , , , ,22507 Ojibwa/Chippewa 0,0586 0, , , , , , , , , , , ,514 0, ,14577 Old Harbor 0, , , , , , , , , , , , , ,166 0,20002 Ouzinkie 0, , , , , ,37 0, , , , , ,0935 0, , ,24499 Paez 0, , , , , , , , , , , ,2934 0, , ,10898 Paiute N./ Shoshoni 0, , , , , , , , ,5186 0, , , , , ,19273 Parakanã 0, , , ,0362 0, , , , , , , , , , ,01899 Pehuenche Butalelbum 0, , , , , , , , , ,3691 0, , , , ,12614 Pehuenche Trapa Trapa 0, ,3452 0, , , , , , , , , , , , ,02995 Peneunche Chile2 0, , , ,0533 0, , , , , , , , , , ,04289 Apêndice XXIV P: Matriz de Fst entre populações da América (n10) com cinco haplogrupos de DNA mitocondrial
212 213 hokan (central coast) Huetar Huetar2 Huilliche Huilliche2 Huilliche Chile Huitoto Ignaciano Ijka- Arhuaco Ingano Ingano Colombia Inuit eskimo Jemez Kanamarí Katuena Penutian California 0,0711 0, , , , , , , , , , , , , ,16316 Penutian California2 0, , , , , , , , , , , , , , ,12474 Piaroa -0, ,15 0,1595 0, , , , , , , , ,4788 0, , ,03405 Pilagá Formosa 0, ,308 0, , , , , ,0491 0, , , , , , ,06744 Pima 0, ,4587 0, , , , , , , , , ,658 0, ,119 0,22383 Pima2 0, , , , , , , , ,5105 0, , , , , ,17789 Pima3 0, , , , , , ,2746-0, , , , , , , ,1874 Poturujara (Zoé) -0, , , ,1591 0, , , , , ,0836 0, ,3775 0, , ,00358 Quechan/Cocopa 0,2899 0, , ,2181 0, , , , ,5974 0, , , , , , ,1053 0, , , , , , , ,4254 0, , , , , ,15643 (Arequipa) 0, , , , , , , , , , , ,7045 0, , ,27937 (Tayacaja) 0, , , , , , , , , , , , , , ,06422 Bolivia 0, , , , ,2854 0, , , , , , , , , ,375 / Nmatsiquenga (San Martin) 0,1151 0,3453 0,3553 0, , ,0715 0, , , , , , , , ,19703 Aleut Saint Paul 0,1501 0, , , , , , , , , , , , , ,19097 Salinan/ Chumash -0,1 0, , , , , , , , , , , , ,0219 0,03487 Salish Plateau 0, , , , , , , , , , , , , , ,16875 Selknam 0,1846 0, , , , , , , , ,1993 0, , , , ,01053 Seminole 0, , , , , , , , ,0995 0, , , , , ,19963 Siona 0, , ,0417 0, , , , , , , , , , , ,23118 Siouan -0, , , , , , , ,1155 0, , , , , ,015 0,10881 Sioux Sisseton/ Wapheton Sioux -0,0024 0, , , , , ,1503 0, , , , , ,4707 0, ,12231 Takik 0, , , ,0892 0, , , , , , ,0784 0, , , ,01668 Taono O odham (Papago) 0, , , , , , , , , , , , , ,1316 0,22114 Tayacaja Peru 0, , , , , , , , , , , , , , ,06422 Tehuelche Argentina 0, , , , , ,012 0, , , , , , , , ,08354 Teribe 0, , , , , , , , , , , ,1355 0, , ,33045 Teribe2 0, ,2158 0, , , , , , , , , , , , ,5193 Tikúna 0, , , , , , , , ,2731 0, , , , , ,02378 Tikúna Brasil 0, , , , ,0479 0, , , , , , , , , ,00286 Tikúna Colombia 0, , , , , ,0501 0, , , , , , , , ,01679 Toba (chaco) 0, , , , , , , , , , ,078 0, , , ,14631 Toba (Formosa) -0,0169 0, , , , , ,0527 0, , , ,1286 0, ,3576 0, ,09897 Trinitario Bolivia 0, , , , , , , , , , ,0314 0, , , ,09965 Tucano 0, , , ,0494 0, , , , , , ,111 0, ,5346 0, ,00769 Tule-Cuna 0, , , ,2418 0, , , , , , , ,297 0, , ,1183 Uto-Aztecan California 0, , , , , , , , , , , , ,4584 0, ,02219 Uto-Aztecan Central 0, ,4587 0, , , , , , , , , ,658 0, ,119 0,22383 Uto-Aztecan Northern 0, ,3532 0, ,0103 0, , , , , , , , , , ,16216 Waiãpi 0,1057-0, , , , , , ,3991 0, , , , , , ,29697 Wapishana Brasil 0, , , , , , ,0722 0, , ,3889 0, , , , ,16128 Washo 0, , , ,1378 0, , , , , , , , , , ,17887 Waunana 0, , , , , , , , ,5789 0, , , , , ,25774 Wayuu 0, , , , , , , , , , , , ,5738 0, ,05416 Wayuu Colombia 0, , , ,1818 0, , , , , ,0413-0, , , ,0025 0,08409 Wounan 0, , , , , , , , , , , , , , ,03139 Xavante 0,3966 0, , , ,3419 0, , , , , , ,8047 0, , ,46917 Yaghan Chile 0, , , , , , , , , , , , , ,15 0,03004 Yakama 0,2334 0, , ,1613 0, , , , , , , , , , ,29959 Yakima 0, , , , , , , , , , , , , , ,30823 Yámana (Yaghan) 0, , , , , , , , , , , , , , ,04644 Yaminawa 0, , , , , , , , , , , , , , ,07609 Yanomama 0, , , , , , , , , , , , ,4484 0, ,06091 Yanomama Brasil 0, ,4092 0, , , , , , ,5196 0, , , , , ,0258 Yanomami 0, , , , , , , , , , , , , , ,15925 Yanomamö 0, , , , , , , ,0541 0, , , , , , ,21813 Yok-Utian 0, , , , , ,0276 0, , , , , ,6263 0, , ,07044 Yuko-Yukpa 0, , , ,5068 0, , , , , , ,6008 0, , ,7326 0,82362 Yuman 0, ,4822 0, , , , , , , , , , , , ,26716 Yuman (Hokan) 0,2993 0, , , , , , , , , , , , ,17 0,2863 Yuman Delta 0, , , , , , ,3094 0, , , , , , , ,22894 Yuman Pai 0, , , ,2209 0, , , , , , , , , , ,28313 Yuman River 0,2986 0, , , , , , , , , , , , , ,26956 Yuracaré 0, , , , , , ,1288 0, , , , , ,3613-0, ,08741 Zapotec 0,0372 0, , , , , , , , , , , , , ,0638 Zenu 0, , , , , , , , , , , , ,254 0, ,09288 Zoro 0,0783 0, , , , , , , , , , , , ,1446 0,07841 Zuni 0, , , , , , , ,1796 0,6065 0, , , , , ,38626 Zuni (penutian) 0, , ,4391 0, , , , , , , , , , , ,31128 Apêndice XXIV Q: Matriz de Fst entre populações da América (n10) com cinco haplogrupos de DNA mitocondrial
213 214 Katukina Katukina2 Kawéskar Kaxináwa Kayapó Kiliwa/ Paipai Kogui Kraho Kumiai Kuna Kuna2 Kuna3 Mapuche Mapuche0 Mapuche (Chile) Katukina 0 Katukina2 0, Kawéskar 0, , Kaxináwa 0, ,2382 0, Kayapó 0,3526 0,1386 0, , Kiliwa/Paipai 0, , , , , Kogui 0,1441 0,3935 0, , ,3934 0, Kraho 0, ,2512 0, , , , , Kumiai 0, , , ,2844 0, , , , Kuna 0, , , , , , ,5493 0, ,75 0 Kuna2 0, , , , , ,5311 0, , , , Kuna3 0, , , , , , , , , ,1198-0, Mapuche 0,0749 0, , , ,1482 0, , , , ,4676 0,3124 0, Mapuche0 0, , , , , , , , , , , , , Mapuche (Chile) -0, , , , , , , , ,1467 0, , , , , Mapuche Anecón Grande 0,0919 0, , , , ,1965 0, , , , , , , , ,03252 Mapuche Argentina 0, , , , , , , , , ,4464 0, ,3474-0, , ,02168 Mapuche Chile -0, , , , , , , ,3475 0, , , , , ,0805 0,02489 Marúbo -0, ,466 0, , , , , , , ,6623 0, , , , , : Mataco (Chaco) 0, , , , , , , , , ,6054 0,404 0, , , ,12368 Mataco (Salta) 0, , , , , , , , , ,596 0, , , , ,19076 Mataco (Wichí) Argentina 0, , , , , , , , , , , , ,0675 0, ,19179 Mataco (Wichí) Formosa 0, , , ,2333 0, , , , , , , , , , ,09808 Maya 0, ,029 0, , , , , , , , , , ,1681 0, ,21835 Maya Contenporary 0, ,0322 0, , , , ,1929 0, , , , , ,1363 0, ,18691 Mixe 0, , , , , , , , , , , , , ,2105 0,28226 Mixtec 0, , , , , , , , , , , , , , ,42888 Mohawk 0, , , , ,1485 0,4597 0, , , , ,1679 0, , ,1815 0,23328 Mohawk2 0, , ,4817 0, , ,4317 0, , , ,1784 0, , , , ,24332 Mosetén Bolivia 0, ,1758 0, , , , , , , , , , , , ,26035 Movima Bolívia -0, , , , , , , , , , , , , , ,03966 Murui-Muinane -0, , , , , , , , , , , ,4176 0,0038-0, ,02955 Muskoke Oklahoma 0, , , , , , , ,1637 0,2839 0, , , , , ,11221 Nahua 0, , , , , , , , , , , ,0244 0, ,2184 0,29377 Nahua/Cora 0, , , , ,0114 0,3498 0, , , , , , , , ,25259 Navajo 0, ,0578 0, , , , , , , ,283 0, , , , ,27336 Ngöbé 0, , , ,2475 0, , , , , , , , , , ,36433 Nukak 0, , , , ,5244 0, , , , , , , , , ,16493 Nuu-Chah-Nulth 0, , , , , , , , , , , , , ,1208 0,14428 Ojibwa Manitoulin Island 0, , , , , , , ,1504 0, , , , , , ,14584 Ojibwa North Ontario 0, , , , , , , , , , , , , , ,33355 Ojibwa/Chippewa 0, , , , ,166 0, , , , ,2329 0, , , , ,24726 Old Harbor 0, , , , , , , , , , , , , , ,31353 Ouzinkie 0, , , , , ,6309 0, ,3452 0, , , , , , ,36393 Paez 0,2142 0, , , , ,5371 0, , , , , , , , ,23531 Paiute N./ Shoshoni 0, , , , , , , , , , , , , , ,09911 Parakanã -0, , , , ,2183 0, , , , , , , , , ,04827 Pehuenche Butalelbum 0, , ,0081 0, ,487 0, , , , ,7205 0, , , , ,09629 Pehuenche Trapa Trapa -0, , , , , , ,2341 0,294 0, , ,4442 0, , , ,0071 Peneunche Chile2-0, , ,1195 0, , , , ,3115 0, , , , , , ,01836 Apêndice XXIV R: Matriz de Fst entre populações da América (n10) com cinco haplogrupos de DNA mitocondrial
214 215 Katukina Katukina2 Kawéskar Kaxináwa Kayapó Kiliwa/ Paipai Kogui Kraho Kumiai Kuna Kuna2 Kuna3 Mapuche Mapuche0 Mapuche (Chile) Penutian California 0, , , , , , , , , , , ,4367 0, , ,10884 Penutian California2 0, , , , , , , , , , , , , , ,07245 Piaroa -0,0008 0, ,2661 0, , , , , , , , , , , ,01351 Pilagá Formosa 0, , , , , , , , , , , , , , ,00511 Pima 0, , , , , , , , , , , , , , ,13757 Pima2 0, , , , , , , , , , , , , , ,10633 Pima3 0,2103 0, , , , , , , , ,5618 0, , , , ,11607 Poturujara (Zoé) 0, ,1816 0, , , , ,15 0, , , , , , , ,11994 Quechan/Cocopa 0, , , ,3165 0, , , , , , , , , , , , , , , , , , , , , , , ,0102 0, ,09099 (Arequipa) 0, , , , , , , , , , ,3813 0, , , ,1895 (Tayacaja) 0, , , , , , ,2479 0, , , , ,2804 0, , ,05595 Bolivia 0, , , , , , , , , , , , , , ,28649 / Nmatsiquenga (San Martin) 0, , , , , , , , , , , , , , ,12425 Aleut Saint Paul 0, , , ,3616 0, , , ,422 0, , , ,4745 0, , ,22746 Salinan/Chumash 0, , , , , ,439 0,2373 0, , , , , , , ,11884 Salish Plateau 0, , , , , , , , , , , , , , ,14456 Selknam -0, , , , , , , , , , , , , ,0657 0,00766 Seminole 0, , , , , , , , , , , , , , ,28266 Siona 0, ,0686 0, , , , , , , , , , , , ,33412 Siouan 0, , ,4532 0, , , , , , ,2072 0, , , , ,20291 Sioux Sisseton/ Wapheton Sioux 0, , , , , , , , , , , , , , ,21637 Takik -0, , , , , , , , , , , , , , ,02705 Taono O odham (Papago) 0, , , , , , , , , , , , , , ,11864 Tayacaja Peru 0, , , , , , ,2479 0, , , , ,2804 0, , ,05595 Tehuelche Argentina 0, , , , , , , , , ,6302 0,4725 0, , , ,02749 Teribe 0, , , , , , , , , , , ,03 0, , ,40592 Teribe2 0, , , ,4672 0, , , , , , , , , ,55325 Tikúna -0,0136 0,272 0, , , , , , , , , , , , ,03584 Tikúna Brasil -0,0241 0, , ,1532 0, , , , , , , ,4622 0, ,0753 0,04438 Tikúna Colombia -0, , , , , , , , , , , ,3996 0, , ,01135 Toba (chaco) 0, , , , ,0837 0, , , , , , , ,0007 0, ,09622 Toba (Formosa) 0, , , , , , , , , , , , , , ,09918 Trinitario Bolivia 0, , , , , , , , , , , , , , ,06897 Tucano -0, , , , , , , , , , , , , , ,0348 Tule-Cuna 0, , , , , , , ,0552 0, , ,0602 0, ,143 0, ,19244 Uto-Aztecan California -0,0069 0, , , , , , ,1498 0,0917 0, , , , ,0169-0,04198 Uto-Aztecan Central 0, , , , , , , , , , , , , , ,13757 Uto-Aztecan Northern 0, , , , , , , , , , , , , , ,06984 Waiãpi 0,4358 0, , , , , , , , , , , ,3471 0, ,40146 Wapishana Brasil 0, ,5327 0, , , , , ,3205 0, , ,5156 0, , , ,09857 Washo 0, , , , , , , , , , , , , , ,08078 Waunana 0, , , , , , ,3813 0, , , , , , , ,14815 Wayuu 0, , , , , , , , , , , , , ,1286 0,10257 Wayuu Colombia 0,1314 0, , , , , , ,0289 0, , , , , ,0695 0,09082 Wounan 0, , , , ,2003 0, , ,1292 0, , , , , , ,08206 Xavante 0, , , , , , , , ,1936 0, , , , ,2401 0,36611 Yaghan Chile -0, , , , , , , , , , , , , , ,02563 Yakama 0, , , , , , , , , , ,3968 0, , , ,19994 Yakima 0, , , , ,1468 0,0404 0, , , , , , , , ,20343 Yámana (Yaghan) -0, , , , , , , , , , , , , , ,03668 Yaminawa 0, , , , , , , , , , , , , , ,22081 Yanomama 0, , , , , , , , , , , , , , ,0612 Yanomama Brasil -0, , , , , , , , ,187 0, , , , , ,01197 Yanomami 0, , , , , , , , , , , , , , ,1464 Yanomamö 0,2323 0, , , , , ,3269 0, , , , , , , ,11186 Yok-Utian 0, , , , , , , , , , , , , , ,03643 Yuko-Yukpa 0, , , , , , , , , , ,7791 0, , ,65151 Yuman 0, , , , , , , ,0704-0, , ,4221 0, ,1216 0, ,16771 Yuman (Hokan) 0, , , ,3061 0, , , , , , , ,4828 0, , ,18448 Yuman Delta 0, , , ,2569 0,2454 0, ,3151 0, , , , , , , ,13066 Yuman Pai 0, , , , ,1459 0, , , , , , , , , ,19041 Yuman River 0, , , , , , , , , , , , , , ,16293 Yuracaré 0,1595 0, ,4427 0, , , , , , , , , , , ,13254 Zapotec 0, ,1894 0, , , , , , , , , , , ,0676 0,09661 Zenu 0, , , , , , , , , , , , , ,0379 0,07282 Zoro 0, , , , , , , , , , , , , , ,10531 Zuni 0, , , , , , , , , , , , , , ,29585 Zuni (penutian) 0, ,3814 0,6096 0, , ,0383 0, ,0382 0, , ,3264 0, , , ,24465 Apêndice XXIV S: Matriz de Fst entre populações da América (n10) com cinco haplogrupos de DNA mitocondrial
215 216 Mapuche Anecón Grande Mapuche Mapuche Argentina Chile Marúbo Mataco (Chaco) Mataco (Salta) Mataco (Wichí) Argentina Mataco (Wichí) Formosa Maya Maya Contenporary Mixe Mixtec Mohawk Mohawk2 Mosetén Bolivia Mapuche Anecón Grande 0 Mapuche Argentina -0, Mapuche Chile 0, , Marúbo 0, , , : Mataco (Chaco) 0, , , , Mataco (Salta) 0, , , , , Mataco (Wichí) Argentina 0, , , , , , Mataco (Wichí) Formosa 0, , , , ,1279 0, , Maya 0, ,1607 0, , ,2526 0, ,2652 0, Maya Contenporary 0, , , , , ,2396 0, ,1655-0, Mixe 0, , , , , ,2764 0, ,2126-0, , Mixtec 0, , , , ,4837 0, , , , , , Mohawk 0, , , , , , ,322 0, , , , , Mohawk2 0, , , , , , , , , , , , , Mosetén Bolivia 0, , , , , ,106 0, , , , , , , , Movima Bolívia 0, , , , , , , , , , , , , , ,37898 Murui-Muinane 0, , ,0412-0, , ,1701 0, , , , , ,3979 0, , ,2023 Muskoke Oklahoma 0, , , , , , ,205 0,1631 0, , , , , , ,16859 Nahua 0, , , , , , , , , , , , , , ,05127 Nahua/Cora 0,1253 0, ,3829 0, , , , , ,0028-0, , , , , ,01304 Navajo 0, ,1747 0, , , , , , , , , , , , ,00085 Ngöbé 0, , , , , , , , , ,0295-0, , , , ,08781 Nukak 0, , , , , , , , , , , , , , ,49438 Nuu-Chah-Nulth 0,1063 0, , , , , , , , , , ,1349 0, , ,17203 Ojibwa Manitoulin Island 0, , , , , , , , , , , , , , ,17281 Ojibwa North Ontario 0, , , , ,3841 0, , , , ,0429 0, , , , ,22479 Ojibwa/Chippewa 0, , , ,2805 0,3172 0, , , , , , , , , ,17485 Old Harbor 0, , , , , , , , , , , , , , ,2849 Ouzinkie 0, , , , , , , , , ,0678 0, , , , ,29286 Paez 0, , , , , , , , , , , , , , ,24013 Paiute N./Shoshoni 0, , , , , , ,0662 0, , , , , , , ,24634 Parakanã -0, , , , , , , , , , , ,4476 0,1786 0, ,21343 Pehuenche Butalelbum 0, , , , , , , , , , , , , , ,47105 Pehuenche Trapa Trapa 0, ,0783-0, , , , , , , , , , ,3005 0,3018 0,35797 Peneunche Chile2 0, , , , , , , , , ,2757 0,373 0, , ,3153 0,37059 Apêndice XXIV T: Matriz de Fst entre populações da América (n10) com cinco haplogrupos de DNA mitocondrial
216 217 Mapuche Anecón Grande Mapuche Mapuche Argentina Chile Marúbo Mataco (Chaco) Mataco (Salta) Mataco (Wichí) Argentina Mataco (Wichí) Formosa Maya Maya Contenporary Mixe Mixtec Mohawk Mohawk2 Mosetén Bolivia Penutian California 0, , , , , , , , , , , , , , ,15238 Penutian California2-0, , , , , ,0347 0, , , , , , , , ,14342 Piaroa 0,0049 0, , , , , , , , , , , , ,0901 0,15677 Pilagá Formosa -0, , , ,0291 0, , , , , , ,2311 0,41 0, , ,16508 Pima 0, , , , , , , , , , , , ,2924 0,272 0,16805 Pima2 0, , , , , , , , , , , , , , ,18313 Pima3 0, , , , , , , , , , , , , , ,19404 Poturujara (Zoé) 0, , ,173 0, , , , , , , , , , , ,24262 Quechan/Cocopa 0,0891 0, , ,2265 0, , , , ,2745 0, ,3305 0, , , , ,0031 0, , , , , , , , , , , , , ,08169 (Arequipa) 0,0551 0, , , , , , , , ,221 0, , , ,2946 0,07698 (Tayacaja) -0, ,004 0, , , ,0735 0, , , , , , , , ,09102 Bolivia 0, , , , , , , , , , , , , ,3165 0,07103 / Nmatsiquenga (San Martin) 0, ,0212 0,2587 0, , , , , , , , , , , ,08607 Aleut Saint Paul 0, , , , , , , ,3534 0, , , , , ,3522 0,41995 Salinan/Chumash 0,038 0, ,22 0, , , , , , , , , , ,0344 0,09763 Salish Plateau -0, , , , , , , , , , , , , , ,04289 Selknam 0, , , ,0262 0, , , , , , , , , , ,41132 Seminole 0, , ,3921 0, , , , , , , , , , , ,08967 Siona 0, , , , , ,3895 0, , , , , , , , ,1685 Siouan 0, , , , , , , , , , , , , , ,0945 Sioux Sisseton/ Wapheton Sioux 0, , , , , , , , , ,0274-0, ,0637 0, , ,1008 Takik 0, , , , , , , , , , , , , , ,32266 Taono O odham (Papago) 0, , , , , ,1086 0, , , ,2515 0, , , , ,19429 Tayacaja Peru -0, ,004 0, , , ,0735 0, , , , , , , , ,09102 Tehuelche Argentina 0,0694 0, , ,0454 0, , , , , ,2631 0, , , , ,33265 Teribe 0, , , , , , , , , , , ,0195 0, , ,21053 Teribe2 0, ,4574 0, , , , , , , , , , , , ,5307 Tikúna 0, , , , , , , , , , ,2097 0, ,1712 0, ,25578 Tikúna Brasil 0, , , , , , , , , , , , , , ,35063 Tikúna Colombia 0,0425 0, , , , ,2185 0, , , , ,244 0, , ,1979 0,252 Toba (chaco) -0, ,0062 0,2222 0, ,036 0, , , , , , , , , ,07114 Toba (Formosa) 0, , , ,1557 0, , ,0787 0,0652 0, , , , ,1523 0, ,08791 Trinitario Bolivia 0, , , , ,2161 0, ,1489 0,0276 0, , ,1775 0, , , ,11961 Tucano 0, , ,0085-0,0595 0, , , , ,2573 0,2248 0, , , , ,32355 Tule-Cuna 0, , , , , , , , , , , , , , ,05959 Uto-Aztecan California 0, , , , , , , ,0576 0, , , , , , ,25404 Uto-Aztecan Central 0, , , , , , , , , , , , ,2924 0,272 0,16805 Uto-Aztecan Northern 0, , , ,1137 0, , , , , , , , , , ,22256 Waiãpi 0, , , , , , ,4861 0, ,1022 0, , ,0397 0, , ,346 Wapishana Brasil 0, ,0719 0, , , , , , , ,3025 0, , ,3496 0, ,36384 Washo 0, , , , ,2077 0, ,0952-0, , , , , , , ,17788 Waunana 0, , , , , , , , , , , , ,3254 0, ,16909 Wayuu 0, , , , , , , , , , , , , , ,29825 Wayuu Colombia 0, , , ,1108 0, , , , , , , ,3005 0, , ,1067 Wounan 0, ,1077 0, , , , ,2918 0, , , , , , , ,20646 Xavante 0,1986 0, , , , , , , , , , , , , ,11737 Yaghan Chile 0, , , , , , , , , , , ,5409 0, , ,41057 Yakama 0, , ,3415 0, , , ,0063 0, , , , , ,3132 0, ,1078 Yakima 0, , , , , , , , , , , , , , ,11915 Yámana (Yaghan) 0,1676 0, , , , , , , , , , , , , ,45677 Yaminawa 0, , , , , , , , , , , , , , ,25978 Yanomama 0, , ,1794 0, , ,2564 0, , , , , , , , ,23443 Yanomama Brasil 0, , , , , ,2822 0, , , , , , , ,2856 0,34776 Yanomami 0, , , , , , , , , , , ,572 0, , ,48808 Yanomamö 0, , , , , , , , , , , , , , ,17641 Yok-Utian 0, , , , , , , , , , , , , , ,20021 Yuko-Yukpa 0, , , , , ,3043 0, , , ,7415 0,8439 0, , , ,63993 Yuman 0, ,1176 0, ,203 0, , , , , , , , , , ,19641 Yuman (Hokan) 0, , , ,2293 0, , , , , , , , , , ,17764 Yuman Delta 0, , , , , , , , , , , , , , ,21935 Yuman Pai 0, , , , , , , , , , , , , , ,11984 Yuman River 0, , , , , , , , ,2793 0,2732 0, , , , ,19465 Yuracaré 0, , , , , , , , , , ,0114 0, , , ,03218 Zapotec 0,0326 0, , , , , , , , , , , , , ,0682 Zenu 0, , , ,1008 0,1782 0, , , , , , , , , ,07282 Zoro 0, , , , ,0582 0, , , , , , , , ,2896 0,3413 Zuni 0, , , , , , ,0709 0, , , , , , , ,07504 Zuni (penutian) 0, ,1256 0, , , ,0386 0, , , ,194 0, , , , ,02115 Apêndice XXIV U: Matriz de Fst entre populações da América (n10) com cinco haplogrupos de DNA mitocondrial
217 218 Movima Bolívia Murui- Muinane Muskoke Oklahoma Nahua Nahua/ Cora Navajo Ngöbé Nukak Nuu- Chah- Nulth Ojibwa Manitoulin Island Ojibwa North Ontario Ojibwa/ Chippewa Old Harbor Ouzinkie Paez Movima Bolívia 0 Murui-Muinane 0, Muskoke Oklahoma 0, , Nahua 0, , , Nahua/Cora 0, ,1853 0, , Navajo 0, , ,1438-0,0082-0, Ngöbé 0, , , , , , Nukak 0, , , , , , , Nuu-Chah-Nulth 0, , , , , , , , Ojibwa Manitoulin Island 0, , , ,1396 0, , , , , Ojibwa North Ontario 0, , , , ,0774 0, , ,525 0, , Ojibwa/Chippewa 0, , , , , , , , , , , Old Harbor 0, , ,0613 0, , , , , , , , , Ouzinkie 0, , ,1397 0, , , , , , , , , , Paez 0, , , ,0974 0, , , , , , , , , , Paiute N./ Shoshoni 0, , , , , , , , , , , , ,3375 0, ,38998 Parakanã -0,0028-0, , , , , , , ,1093 0, , , , , ,18655 Pehuenche Butalelbum 0, , , , ,4345 0, , , ,2196 0, , , , ,4818 0,42597 Pehuenche Trapa Trapa 0, , , , , , , ,227 0, , , , , , ,29045 Peneunche Chile2 0, , , , ,3482 0, , , , , , ,3201 0, , ,30684 Apêndice XXIV V: Matriz de Fst entre populações da América (n10) com cinco haplogrupos de DNA mitocondrial
218 219 Movima Bolívia Murui- Muinane Muskoke Oklahoma Nahua Nahua/ Cora Navajo Ngöbé Nukak Nuu- Chah- Nulth Ojibwa Manitoulin Island Ojibwa North Ontario Ojibwa/ Chippewa Old Harbor Ouzinkie Paez Penutian California 0, , , , , , , , , , , , , , ,34391 Penutian California2 0, , , , , , , , , , , , , ,3605 0,31056 Piaroa 0, , , , ,1091 0,1398 0, , , , , , , , ,09252 Pilagá Formosa 0, , , , , , , , , , , , , ,355 0,24907 Pima 0, , ,2642 0,2887 0, , , , , , , , , , ,31245 Pima2 0, , , , , , , , , , , ,2804 0, ,4553 0,27289 Pima3 0, , , , , ,2321 0, , , , , , , ,4385 0,27115 Poturujara (Zoé) 0,1622 0, , , , ,1796 0, , , , , , , , ,0572 Quechan/Cocopa 0, , , , , , , , , , ,4502 0, , , , , , , , , , , , , , , , , , ,27358 (Arequipa) 0, , , , , , ,3279 0, , , , , , , ,37045 (Tayacaja) 0,1734 0,0198 0, , , , , , , , , , ,1785 0, ,18388 Bolivia 0, ,2643 0, , , , , , , , , , , , ,41586 / Nmatsiquenga (San Martin) 0, , , ,234 0, , , , , , , , , , ,33636 Aleut Saint Paul 0, , , , , , , , , ,3042 0,3777 0, , , ,39052 Salinan/ Chumash 0, , , , , , , , , , , ,0586 0, , ,07693 Salish Plateau 0, , ,1103 0, , , , , , , , , , , ,22244 Selknam 0,0481 0, , , , , , , , , ,4011 0, , , ,30923 Seminole 0, , , , , , , , , ,1279 0, , , ,0608 0,07199 Siona 0, , , , , , ,01 0, , , , , , , ,04811 Siouan 0,246 0, , , , , ,0424 0, , , , , , , ,025 Sioux Sisseton/ Wapheton Sioux 0, , , , , ,0329 0,036 0,3779 0, , , , , , ,01659 Takik -0, , , , , , , , , , , ,2739 0, , ,2564 Taono O odham (Papago) 0, , , , , , ,3918 0, , , , , , , ,33849 Tayacaja Peru 0,1734 0,0198 0, , , , , , , , , , ,1785 0, ,18388 Tehuelche Argentina 0, , , , , , , , , , , , , , ,35327 Teribe 0, , ,1972 0, , , , , , , , , , , ,1413 Teribe2 0, , , , , , ,2312 0, , ,3737 0, , , , ,29213 Tikúna 0, , , , , , , , , , , , , , ,13488 Tikúna Brasil 0, , , ,3281 0, , , , , ,1682 0,3178 0, ,2197 0, ,24145 Tikúna Colombia 0, , , , ,2231 0, , , , , , , , , ,18128 Toba (chaco) 0, , , , , , ,2472 0, , , , , , , ,27619 Toba (Formosa) 0, , , , , , , , , , , , ,1526 0, ,2198 Trinitario Bolivia 0, , , , , , , , , , , , , , ,17697 Tucano 0, , , , , , , , , , ,3827 0, , , ,26915 Tule-Cuna 0, , , , ,0114 0, , , , , , ,0384 0, ,1153 0,02644 Uto-Aztecan California 0, , , , , , , , , , , ,2574 0, , ,25629 Uto-Aztecan Central 0, , ,2642 0,2887 0, , , , , , , , , , ,31245 Uto-Aztecan Northern 0,2402 0, , , , , , , , , , , , ,4075 0,35614 Waiãpi 0, , , , , , , , , , , , ,0367-0, ,16521 Wapishana Brasil 0, , ,1058 0, , , , , , , , , , , ,43127 Washo 0, , , , , , ,3763 0, , , , , , , ,31745 Waunana 0, , , , , , , , , , , ,3451 0, , ,37106 Wayuu 0, ,0561 0, , , , ,3537 0, , , , ,1835 0, , ,10367 Wayuu Colombia 0, , , , , , , , , , , , , , ,11005 Wounan 0, , ,1512 0, , ,1772 0, , , , , ,13 0, , ,06136 Xavante 0, ,3626 0, , , , , , , ,3893 0, , , , ,50362 Yaghan Chile 0,0785 0, , , , ,3978 0, , , , , ,2979 0, , ,31853 Yakama 0, , , , , , , , , , , , , , ,40266 Yakima 0, , , , , , , , ,2895 0, , ,3629 0, , ,41468 Yámana (Yaghan) 0, , , , , , ,5214 0, , , , , , , ,35231 Yaminawa 0, , , ,105 0, , , , , , , , , , ,04269 Yanomama 0, , , , , , , , , , , , , , ,1482 Yanomama Brasil -0,0144-0, , , , , , , , , , ,2891 0, , ,26781 Yanomami 0,0113 0, , , , ,4546 0, , , ,2285 0, , , , ,33064 Yanomamö 0, , , , , ,2416 0, , , , , , , , ,33662 Yok-Utian 0, , , , , , , , , , , , , , ,28827 Yuko-Yukpa 0, , , , , , , , , , , , , , ,82512 Yuman 0, , , , , , , , , ,2618 0, , , , ,34885 Yuman (Hokan) 0, , , , , , , ,2812 0, , , , , , ,36318 Yuman Delta 0,1927 0, , , ,2742 0, , ,2077 0, , , , , , ,34102 Yuman Pai 0, , , ,2837 0, , , , , , , , , , ,35925 Yuman River 0, , , , , , , , ,3108 0, , , , , ,37327 Yuracaré 0, ,0661 0, , , , , , , , , , , , ,06015 Zapotec 0, , , , , , , , ,0848 0, ,1706 0, , , ,05805 Zenu 0, , , , , , , , , , , , ,3013 0, ,15687 Zoro 0, , , , , , , , , , , ,2782 0, , ,31034 Zuni 0,4478 0, ,3023 0, , , , , , ,3216 0, ,3698 0, , ,42964 Zuni (penutian) 0, , , , , ,1216 0, , , , , , , , ,35213 Apêndice XXIV W: Matriz de Fst entre populações da América (n10) com cinco haplogrupos de DNA mitocondrial
219 220 Paiute N./ Shoshoni Parakanã Pehuenche Butalelbum Pehuenche Trapa Trapa Peneunche Chile2 Paiute N./ Shoshoni 0 Parakanã 0, Pehuenche Butalelbum 0, , Pehuenche Trapa Trapa 0,1364 0, , Peneunche Chile2 0, , , , Apêndice XXIV X: Matriz de Fst entre populações da América (n10) com cinco haplogrupos de DNA mitocondrial
220 221 Paiute N./ Parakanã Shoshoni Pehuenche Butalelbum Pehuenche Peneunche Penutian Penutian Trapa Trapa Chile2 California California 2 Piaroa Pilagá Formosa Pima Pima2 Pima3 Poturujara (Zoé) Quechan/ Cocopa Penutian California -0, , , , , Penutian California2-0, , , , , , Piaroa 0, , , , , ,074 0, Pilagá Formosa 0,0375-0,0279 0, ,0679 0, , , , Pima 0, , , , , , ,1748 0, , Pima2 0, , , , ,241 0, , , , , Pima3 0, , ,3735 0, , , , ,1462 0, , , Poturujara (Zoé) 0, ,0858 0, ,1384 0, , , , , , , , Quechan/ Cocopa 0, , ,4497 0, , ,2052 0, , , , , ,0189 0, , ,0503 0, , , , , ,0774 0, , , , , , (Arequipa) 0, , , ,2987 0, ,1073 0, , ,0805 0, , , , , ,00901 (Tayacaja) 0, , , , , , , , , , , ,1402 0, , ,01869 Bolivia 0, ,2644 0,5286 0, , , , , , , , , , , ,06874 / Nmatsiquenga (San Martin) 0, , , , , , , , , , , , ,2358 0, ,01904 Aleut Saint Paul 0, , , , , , , , , , , , , ,4989 0,28936 Salinan/ Chumash 0, , , , , ,0711 0, , , , , , , ,2899 0,1053 Salish Plateau 0, , , , , , , , , , ,0734 0, , , ,01486 Selknam 0, , , ,0274-0, , , , , , , , , , ,21252 Seminole 0, , , , , , , , , ,3095 0,2955 0, , , ,21441 Siona 0, , , , , , , , , , ,3716 0, , , ,2869 Siouan 0, , , , , ,2052 0, , , , , , , , ,17364 Sioux Sisseton/ Wapheton Sioux 0, , , , , ,2281 0, , ,1929 0,2522 0, , , , ,18666 Takik 0, , , , , , , , , , , , , , ,1302 Taono O odham (Papago) 0,1835 0, , , , , , , , , , , , , ,05314 Tayacaja Peru 0, , , , , , , , , , , ,1402 0, , ,01869 Tehuelche Argentina 0, , , , , , , , , , , , , , ,12482 Teribe 0, , , , , , , , , , , , , ,5138 0,33406 Teribe2 0, ,6648 0, , , , , , ,542 0, , , , , ,50577 Tikúna 0, , , , , , , , , , , , , ,2725 0,15945 Tikúna Brasil 0, , , , , ,1574 0,1256 0, , , , , , , ,20166 Tikúna Colombia 0, , , , , , , , , , , , , , ,11379 Toba (chaco) 0, , , , , , , , , , , , , , ,01583 Toba (Formosa) 0, , , ,1563 0, , , , , , , , , , ,03227 Trinitario Bolivia 0, , , , , , , , , , , , , , ,04805 Tucano 0, , , , , , , ,025 0, , , , , , ,12495 Tule-Cuna 0, , ,3872 0, , , , , , , ,1718 0, , , ,1464 Uto-Aztecan California 0, , , , , ,104 0, , , , , , , , ,05892 Uto-Aztecan Central 0, , , , , , ,1748 0, , , , , , , ,06178 Uto-Aztecan Northern -0, , , , , , , , , , , , , , ,03752 Waiãpi 0, , , , ,4199 0, , , , , , , , , ,38067 Wapishana Brasil 0, , , , , , ,0137 0,1151 0, , , , , , ,14161 Washo 0, , , , ,207 0, , , , ,0177-0, , , , ,02784 Waunana 0, , , , , ,1682 0, , , , , , , , ,03491 Wayuu 0, , , , , ,3228 0, , ,1465 0, , , , , ,2138 Wayuu Colombia 0, , , , , , , , , , , , ,1365 0, ,07944 Wounan 0,2761 0, , , , , , , , , ,0791 0, , , ,15593 Xavante 0, , , , , , , , , , ,2401 0, , , ,11891 Yaghan Chile 0, , , , ,0091 0, , , ,0994 0,314 0, , , , ,21837 Yakama 0, , , , , , , , , , , , , , ,01114 Yakima 0, , , , , , , , , ,0944 0,1313 0, , , ,01423 Yámana (Yaghan) 0, , , , , ,2476 0,205 0, , , , , , , ,24913 Yaminawa 0, , , , , , , , , , , , , , ,27683 Yanomama 0, , , , , , , , , , ,0281 0, , , ,13807 Yanomama Brasil 0, , , , , , , , , ,1702 0, , , , ,15839 Yanomami 0, , , , , , , , , , , , , , ,32782 Yanomamö 0, , , , , , , , , , , , , , ,033 Yok-Utian -0, , , , , , ,0459 0, , , ,1986 0, , , ,04236 Yuko-Yukpa 0, , , , , , , , , , , , , , ,34703 Yuman 0, , , , , , , ,2093 0,1017-0, ,0101 0, , , ,08127 Yuman (Hokan) 0, , , , , , , , , , , ,017 0, ,0234 0,07109 Yuman Delta 0,225 0, , , , , , , , , , , , , ,07915 Yuman Pai 0, , , , , , ,1627 0,2029 0, , , ,0341 0, , ,04335 Yuman River 0, , , , , , , , , , , , , ,0403 0,06413 Yuracaré 0, ,0653 0, , , , , , , , , , ,0779 0, ,07981 Zapotec 0, , , , , , , , , , , , , , ,07451 Zenu 0, , , , ,1955 0, , , , , , , , , ,0264 Zoro 0, , , , , , , , , , , , , , ,19452 Zuni 0, , , , , , , , , , , ,1537 0, , ,0732 Zuni (penutian) 0, , , ,3536 0, , , , , , , , , , ,04378 Apêndice XXIV Y: Matriz de Fst entre populações da América (n10) com cinco haplogrupos de DNA mitocondrial
221 222 (Arequipa) (Tayacaja) Bolivia / Nmatsiquenga (San Martin) Aleut Saint Paul Salinan/ Chumash Salish Plateau Selknam Seminole Siona Siouan Sioux Sisseton/ Wapheton Sioux Takik Taono Tayacaja O odham Peru (Papago) (Arequipa) 0 (Tayacaja) 0, Bolivia -0, , / Nmatsiquenga (San Martin) 0, , , Aleut Saint Paul 0,4473 0, , , Salinan/ Chumash 0, , , ,1151 0, Salish Plateau -0, ,0104 0, , , , Selknam 0, , , , , ,1846 0, Seminole 0, , , , , , , , Siona 0, , , , , , , , , Siouan 0, , , , , , , , , , Sioux Sisseton/ Wapheton Sioux 0, , , , , ,0024 0, ,3022-0, , , Takik 0, , , , , , , , , , , , Taono O odham (Papago) 0, , , , ,4611 0, , ,2818 0, , , , , Tayacaja Peru 0, , , , , , ,0104 0, , , , , , , Tehuelche Argentina 0, , , , , , ,2147 0, , , , , ,0761 0, ,06763 Teribe 0, , , , , , , , , , , , , , ,24432 Teribe2 0,7014 0, , ,6334 0,5583 0, ,6359 0, , , , , , , ,40939 Tikúna 0, ,0699 0, , , , , , , , , , , , ,0699 Tikúna Brasil 0, ,0949 0, , , , , , , , , , , , ,0949 Tikúna Colombia 0, , , , ,189 0, , , , , , , , , ,05324 Toba (chaco) 0, ,0034 0, , , , , , ,1972 0, , , , , ,0034 Toba (Formosa) 0, ,0171 0, , , ,0169 0, ,1861 0, , , , ,1649 0, ,0171 Trinitario Bolivia 0, , , , , , , , ,2017 0, , , , ,0143 0,06768 Tucano 0, , , , , , , , , , , , , , ,08285 Tule-Cuna 0, , , , , , , , ,0002 0, , , , ,2218 0,08932 Uto-Aztecan California 0, , , , , , ,1164 0, , , ,2093 0, , , ,04572 Uto-Aztecan Central 0, , , , , , , , ,3095 0, , ,2522 0, , ,13031 Uto-Aztecan Northern 0, ,0414 0, , , , , ,1575 0, , , , ,1258 0, ,0414 Waiãpi 0, , , , ,361 0,1057 0, , , , , , , ,5418 0,25861 Wapishana Brasil 0, , , , ,0793 0, , , , , , ,3448 0, , ,07767 Washo 0, , , , ,4208 0, ,052 0, , , , , , , ,08997 Waunana -0, , , , , , ,0482 0, , , , , , , ,12098 Wayuu 0, ,1707 0, , ,4323 0, , , , ,2959 0, , , , ,1707 Wayuu Colombia 0, ,075 0, , , , , , , , , , , ,0612 0,075 Wounan 0,2265 0, , , , , ,1304 0, , , , , , , ,11235 Xavante 0, , , , , ,3966 0, , , , , , , , ,21335 Yaghan Chile 0, , , , ,1871 0, ,306-0, , , , , , , ,13916 Yakama -0, , , , , ,2334 0, , , , , , , , ,09283 Yakima -0,0232 0,0966 0, , , , , , ,3244 0, , , , , ,0966 Yámana (Yaghan) 0, , , , , , , , ,424 0,4914 0, , , , ,16774 Yaminawa 0,3947 0, ,4563 0, , , , , , , , , , ,3648 0,16888 Yanomama 0, , , , , , , , , , , , , ,0838 0,12345 Yanomama Brasil 0, ,1221 0, , , , , , , , , , , , ,1221 Yanomami 0, , , ,4184 0, , ,4131 0, , , ,3689 0, , , ,29969 Yanomamö 0, ,1077 0, , , , , , , , , , , , ,1077 Yok-Utian 0, , , , , , , ,0913 0, , , , , , ,00926 Yuko-Yukpa 0, , , , , , , , , , , , ,8463 0, ,49107 Yuman 0, , , , , , , , ,3432 0, , , , , ,16385 Yuman (Hokan) 0, , ,0668 0, , ,2993 0, , , , , , , , ,15995 Yuman Delta 0, , , , , , , ,3008 0, , , , , , ,14864 Yuman Pai -0, , , , , , , , , , , , , , ,12996 Yuman River 0, , ,0788 0, , ,2986 0, , , , , , , , ,14944 Yuracaré 0, , , , , , ,0038 0, , , , , , , ,04362 Zapotec 0, , , ,1224 0, ,0372 0,0042 0, , , , , , , ,05176 Zenu 0, , , , , , , , , , , , , , ,03908 Zoro 0, , , , ,0054 0,0783 0, , , , , , , , ,08312 Zuni -0, , , ,0803 0, , ,0119 0, , ,4506 0, , , , ,16376 Zuni (penutian) -0, , , , , , , , , , , , , , ,11665 Apêndice XXIV Z: Matriz de Fst entre populações da América (n10) com cinco haplogrupos de DNA mitocondrial
222 223 Tehuelche Argentina Teribe Teribe2 Tikúna Tikúna Brasil Tikúna Colombia Toba (chaco) Toba (Formosa) Trinitario Bolivia Tucano Tule- Cuna Uto- Aztecan California Uto- Aztecan Central Uto- Waiãpi Aztecan Northern Tehuelche Argentina 0 Teribe 0, Teribe2 0, , Tikúna 0, , , Tikúna Brasil 0, , , , Tikúna Colombia 0, , , , , Toba (chaco) 0, , , , , , Toba (Formosa) 0, ,2542 0, , , , , Trinitario Bolivia 0, , , , , , , , Tucano 0, , , , , , ,1336 0, , Tule-Cuna 0,2885 0, , , , , , ,1031 0, , Uto-Aztecan California 0, , , , , , , , , , , Uto-Aztecan Central 0, , , , , , , , , , , , Uto-Aztecan Northern 0, , , , , , , , , , ,2585 0, , Waiãpi 0, , , , , , , , , , , , , , Wapishana Brasil -0, , ,7928 0, , ,1065 0, ,0748 0, , , , , , ,53111 Washo 0, ,4652 0, , ,2588 0, , , , , , , ,0177 0, ,51938 Waunana 0, , , , , , , , , ,1894 0, , , , ,56077 Wayuu 0, , , , , , ,2412 0, , , , , , , ,43283 Wayuu Colombia 0, , , , , , , , , , ,0428 0, , , ,33545 Wounan 0, , , , , , , ,1653 0, , , , , , ,33155 Xavante 0, , , , , , , , , , , , , , ,65474 Yaghan Chile 0, , , , , , ,211 0, , , , , ,314 0, ,48678 Yakama 0, , , , , , , , ,115 0, , , , , ,51921 Yakima 0, , , , , , , , ,1244 0, , , ,0944 0, ,53582 Yámana (Yaghan) 0, , ,733 0,0522 0, , , , , , ,3405 0, , , ,52922 Yaminawa 0, , , , , , , , ,1919 0, ,0321 0, , , ,12645 Yanomama 0, , , , , , , , , , , , , ,2112 0,42494 Yanomama Brasil 0, , , , , , ,1773 0, , , , ,0371 0,1702 0, ,49468 Yanomami 0, , , , , , , , , , , , , ,3441 0,57847 Yanomamö 0, , , , , , , , , , , , , , ,48958 Yok-Utian -0, , , , ,0501 0, , , ,1324 0, , , , , ,37995 Yuko-Yukpa 0, , , , , , , , , , , , , ,93309 Yuman 0, ,4708 0, , , , , , , , , , , , ,53589 Yuman (Hokan) 0, , ,6354 0, ,3723 0, , , ,0378 0, , , , , ,54405 Yuman Delta 0, , , , , , , , , , ,2265 0, , , ,57839 Yuman Pai 0, , , , , , , , , , , , , , ,55378 Yuman River 0, , , , , , , , , ,2047 0, ,1113-0, , ,59443 Yuracaré 0, , , , , , , , , , , , , , ,21513 Zapotec 0, , ,5102 0,1011 0, , , , , , , , , , ,30726 Zenu 0, , , , , , , , , , ,0586 0,0356 0, , ,34599 Zoro 0, , , , , , , , , , , , , , ,34949 Zuni 0, , , , , , , , , , , , , , ,58736 Zuni (penutian) 0, , , , , , , , , ,3127 0, , , , ,51933 Apêndice XXIV AA: Matriz de Fst entre populações da América (n10) com cinco haplogrupos de DNA mitocondrial
223 224 Wapishana Washo Waunana Wayuu Wayuu Wounan Xavante Yaghan Yakama Yakima Yámana Yaminawa Yanomama Yanomama Yanomami Brasil Colombia Chile (Yaghan) Brasil Wapishana Brasil 0 Washo 0, Waunana 0, , Wayuu 0, , , Wayuu Colombia 0, , , , Wounan 0, , , , , Xavante 0,5212 0, , ,4933 0, , Yaghan Chile 0, , , , ,2187 0, , Yakama 0, , , , ,16 0, , , Yakima 0, , , , , , ,0552 0, , Yámana (Yaghan) 0, , , , , , , , ,3993 0, Yaminawa 0, , ,3985 0, , , , , , , , Yanomama 0,3158 0, , ,0209 0,0013-0, , , , , , , Yanomama Brasil 0,1937 0, , , , , , , , , , , , Yanomami 0, , , ,0767 0, ,1317 0, , , , , , , , Yanomamö 0, , , ,2041 0, , , , , , , , ,1115 0, ,28368 Yok-Utian -0, , , , , ,1831 0,3526 0, , , , , , , ,35827 Yuko-Yukpa 0,8944 0, , , , ,7385 0, , , , , , , , ,76317 Yuman 0, , , , , , , , , , , , , , ,29769 Yuman (Hokan) 0, , , , , , , , ,0761 0, , , , , ,33495 Yuman Delta 0, ,0237-0, ,1619 0, , , , , , , , ,0663 0, ,26413 Yuman Pai 0, , , , , , , ,3785 0, , , , , , ,38701 Yuman River 0, , , , ,0759 0, , , , , , , , ,2065 0,33178 Yuracaré 0, , , , ,0041 0, , , , , ,2872 0, , ,1822 0,30962 Zapotec 0, , , , , , ,2851 0, , , , , , , ,24634 Zenu 0, , , , , ,0359 0, , , , ,2375 0, , , ,22999 Zoro 0, , , , ,2626 0, , , , , , , , , ,39131 Zuni 0,4401 0, , , , , , , , , , , , ,3921 0,51723 Zuni (penutian) 0, , , , , , , , ,0163 0, , , , , ,47455 Apêndice XXIV AB: Matriz de Fst entre populações da América (n10) com cinco haplogrupos de DNA mitocondrial Yanomamö Yok-Utian Yuko- Yuman Yuman Yuman Yuman Yuman Yuracaré Zapotec Zenu Zoro Zuni Zuni Yukpa (Hokan) Delta Pai River (penutian) Yanomamö 0 Yok-Utian 0, Yuko-Yukpa 0, , Yuman 0, , , Yuman (Hokan) 0, , , , Yuman Delta 0, , , , , Yuman Pai 0, , ,4358-0, , , Yuman River -0, , ,5788-0, , , , Yuracaré 0, , , , ,1584 0, , , Zapotec 0, , , , , , , , , Zenu 0, , , , , , , , , , Zoro 0, ,0131 0, , , , , , , , , Zuni 0, , , , , , , , , , ,1125 0, Zuni (penutian) 0, , , , , ,1099-0, , ,1029 0, , , , Apêndice XXIV AC: Matriz de Fst entre populações da América (n10) com cinco haplogrupos de DNA mitocondrial
224 Apêndice XXV: Matriz de Fst entre populações da Patagônia (biom31) com quatro haplogrupos de DNA mitocondrial 225
225 226 Aónikenk Fueguian Argentina Huilliche Huilliche2 Huilliche Chile Kawéskar Mapuche Mapuche (Chile) Mapuche Anecón Grande Mapuche Argentina Pehuenche Butalelbum Pehuenche Trapa Trapa Peneunche Chile2 Selknam Tehuelche Argentina Yaghan Chile Yámana (Yaghan) Aónikenk Fueguian Argentina 0 Huilliche Huilliche2 Huilliche Chile 0, , , , , , Kawéskar Mapuche Mapuche (Chile) 0, , , , , , , , , , , , , , , , , ,0377 0, , , , Mapuche Anecón Grande 0, , , , , , , , Mapuche Argentina 0, , , , , , , , , Pehuenche Butalelbum -0, ,0061 0, , , ,0081 0, , , , Pehuenche Trapa Trapa 0, , , , , , , ,0071 0, ,0783 0, Peneunche Chile2 0, , ,0533 0, , ,1195 0, , , , , , ,0419-0, , , , , ,0657 0, , , , ,0274-0, Selknam Tehuelche Argentina 0, , , , ,012 0, , , ,0694 0, , , , , , , , , , , , , , , , , ,0091-0, , , , , , , , , , ,1676 0, , , , , , , Apêndice XXVI: Matriz de Fst entre populações da Patagônia (biom31) com cinco haplogrupos de DNA mitocondrial Yaghan Chile Yámana (Yaghan)
226 Figura esquemática da localização original dos apêndices XXVII e XXVIII A-G da matriz de DNA mitocondrial do centro norte da América do Sul (biom 42). 227
227 228 Ache Ancash Arara Arequipa Peru Assurini Assurini2 Atacameno Atacameno2 Atacamenos Awá-Guajá Aymara Aymara2 Baniwa Boruca Bribi-Cabecar Cayapa Cayapa Ecuador Chimane Chimila Choroti Choroti2 Coreguaje Curripaco Embera Embera2 Embera3 Gaviao Guahibo Guahibo- Sikuani Guambiano Guane- Butaregua Guatuso Guayabero Guaymi Huetar Huetar2 Huitoto Ignaciano Ijka-Arhuaco Ingano Colombia Kanamari Katuena Katukina Katukina2 Kaxináwa Kayapo Kogui Kraho Kuna Kuna2 Kuna3 Macushi Makiritare Marúbo Marúbo Brasil Mataco (Chaco) Mataco (Salta) Mataco (Wichí) Argentina Mataco (Wichí) Formosa Mosetén Bolivia Movima Bolívia Murui- Muinane Ngobe Nukak Paez Parakana Pasto Piaroa Piaroa Colombia Pilaga Formosa Poturujara (Zoe) (Arequipa) (Tayacaja) Bolivia / Nmatsiqueng a (San Martin) Siona Tayacaja Peru Teribe Teribe2 Tikuna Tikuna Brasil Tikuna Colombia Toba Toba (chaco) Toba (Formosa) Tucano Tule-Cuna Waiãpi Wapishana Brasil Waunana Wayuu Wayuu Colombia Wiwa Wounan Xavante Yamamadi Yaminawa Yanomama Yanomama Brasil Yanomami Yanomamö Yuko-Yukpa Yuracaré Zenu Zoro Ache Ancash Arara Arequipa Peru Assurini Assurini2 Atacameno Atacameno2 Atacamenos Awá-Guajá Aymara Aymara2 Baníwa Boruca Bribi-Cabecar 0 0, ,6995 0, , , , ,5424 0, , , , , ,7066 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,1981 0, , ,2315 0, , , , , , , , , , , , , , , , , , , , , , , ,4176 0, , , , , , , , , ,0778 0, , , , , , , , ,2482 0, , , , , ,0784 0,261 0, , , , , , , , , ,0554 0, , , , , , , , , , , , , , , , , ,8106 0, , , , , , , , , , , , , ,3449 0, , , , ,0104 0, , , ,0301 0, , , ,1032 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,4225 0, , , , , , , , ,0669 0, , , ,2024-0, , , , , , , , , ,417 0, , , , , , , , , ,0324 0, , ,103 0, , , , , , ,079 0, , , ,2274 0, , , , , , , , , , , , , , ,6661 0, ,5305 0,3912 0, , , , , , , , , , , , ,2955 0, , , , , , , ,6274 0, , , , , , , , , ,3784 0, , , , , , , , , , , , ,0162 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , ,4179 0, , , , , , , , , , , , , , , ,3949 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,2388 0, , , , , ,7107 0, , , , , ,3444 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,5642 0,5397 0, , , , , , , , , , , , , ,1045 0, , , ,3377 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , ,6205 0, , , , , , , ,163 0, , , , , , , , , , , , , , , , , , , ,3584 0, , , , , , , , , , , , , , , , , , , , , ,2379 0, , , , , ,3765 0, , , , , , ,0136 0, , , , , , , , ,4729 0, ,4685 0, , , , ,482 0, , , , , , , , ,0577 0, , , ,037 0, , , , ,5533 0, , , , , , , , , , , , , , ,3813 0, , , ,2988 0, , , , , , , , , , , , , , , , , , , , , , , , ,1899 0, , , , , , , , , , , , , , , , , , , , , , , , , ,4844 0,422 0,618 0, , , , , , , , ,6261 0, , , , , , , , , ,0199 0,7 0, , , , , , , , , , , , , , , , , , , ,4644 0, , , , , , , , ,1647 0, , , , , , , , , , , , , , , , , , , , , , ,2656 0, , , , , , , , ,0256 0, , , , , , , , , , , , ,2145 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , ,1649-0, , , , , , , , , , , , , ,5251 0,3279 0, , , , , , , , , , , , , , , , , , ,4897 0, , , , , , ,524 0, ,2817 0, , , ,6022 0,4082 0, ,4008 0, , , ,0405 0, , , , , , , , , , , , , , , , , , , , , , , , ,1875 0, , , , , , , , , , , ,0486 0, , ,1875 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,3262 0, ,171 0, , , , , , ,0383 0, , , , , , , , , , , ,022 0, ,0047 0, , , ,0325 0, , , , , , , , , , , , , , , , , , , ,1981-0, , , , , , , , , , ,1405 0,335 0, , , , , , , , , , , , , ,0198 0, , , , , , , ,0187 0, , , , , , , , , , , , , , , , , ,2619 0, , , , , , , , , , , , , , , , , , ,1405 0,335 0, , , , , , , , , , , , , , , , ,44 0, , , , , , ,7014 0, , , , , , , , , , , , , ,0115 0, , , ,3122 0, , , ,156 0, , , , ,287 0, , , , , , , , , , , , , , , , , , , ,2508 0, ,4803 0, , , , , , , , , , , , , , ,66 0, , ,2777 0,2408 0, , , , , , , , , , , , , , , , , , , , ,0092 0, , , , , , ,141 0, , , , , , , , , ,3249 0, , , , , , , , , , ,3025 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,4632 0, , , , , , , , , , , , , , , , , ,1808 0,0359 0, , , , , , , , , , , , , , , , , , ,628 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,2554 0,5197 0, , , , ,2265 0, , , , ,283 0, , , , , , , , , , , , ,0067-0, ,0052 0, , ,0484 0, , , , , , , , , , , , , , , , , , , ,3947 0, , , , , , , , , , , , ,1219 0, , , , , , , , , , , , , , , , , , , ,3439 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0998 0,4661 0, , , , , , , ,1703 0, ,5094 0,7284 0, , , , , , , , , ,437 0, , , , , , , , , , ,2661 0, , , , , , , , , , , , , , , , , , , , , , , ,34998 Apêndice XXVII A: Matriz de Fst entre populações da região centro norte da América do Sul (biom42) com quatro haplogrupos de DNA mitocondrial
228 229 Cayapa Cayapa Ecuador Chimane Chimila Choroti Choroti2 Coreguaje Curripaco Embera Embera2 Embera3 Gaviao Guahibo Guahibo- Sikuani Guambiano Cayapa 0 Cayapa 0, Ecuador Chimane -0,0121 0, Chimila 0, , , Choroti 0, ,0365 0, , Choroti2 0, , , , , Coreguaje 0, , , , , , Curripaco -0,0005 0,1101-0, ,3766 0, , , Embera 0, , , , , , , , Embera2-0,0181 0,0559-0, , ,013 0, , , , Embera3 0, , , , , , , , , , Gaviao 0, , , , , , , , , , , Guahibo 0, ,1476 0, , , ,1798 0, , , , , , Guahibo- 0, , , ,0139 0, , ,5258 0, , , , , , Sikuani Guambiano 0, , , , , , ,0261 0,5298 0, , , , , , Guane- Butaregua 0, , , , , , , ,1609 0, , , , , , ,49938 Guatuso 0, , , , , , , ,174-0, , , , , ,0028 0,65625 Guayabero 0, , , , , , , , , , , , , , ,43187 Guaymi 0, , , , , , , , ,0435 0, , , , , ,56452 Huetar 0, , , , , , , ,1955 0, ,2237 0, , , , ,54399 Huetar2 0, , , , , , , , ,082 0,2306 0, , , , ,55042 Huitoto 0, , , , , , , ,2604 0, , , , , , ,28047 Ignaciano 0, , , ,5286 0, , , , , , , , , , ,182 Ijka-Arhuaco 0,3085 0, , , , , , , , , , , , , ,59842 Ingano Colombia 0, , , , , , , , , , ,0091 0, , , ,2352 Kanamari 0,0661-0, , , , , , , ,1676 0, , , , , ,21901 Katuena 0, , , ,4498 0, , , , , , , , , , ,13421 Katukina 0, , , , , , , , , ,2821 0, , , , ,09082 Katukina2 0,1667 0, , ,0886 0, , , , , , , , , , ,60821 Kaxináwa 0, , , ,3851 0, , , ,2311 0, , , , , , ,16654 Kayapo -0,0253 0,1107-0, , , , , , , , , , ,2918 0, ,53211 Kogui 0, , , , , , , , , , , , ,0064 0, ,11318 Kraho -0, , , , , ,008 0, , , , , , , , ,43044 Kuna 0, , , , ,6028 0, , ,4285 0, , , , , , ,76951 Kuna2 0, , , , , , , , , ,1458 0, , ,2671 0, ,57808 Kuna3 0, , , , , ,3676 0, , , , , , , , ,61836 Macushi 0, , , , , , , , , , , , , , ,2082 Makiritare 0, , ,395 0, , , , , , , , , , , ,03214 Marúbo 0, , , , , ,0523 0, , , , , , , , ,08177 Marúbo Brasil 0, , , , , , , , , , , , , , ,00352 Mataco 0, , , , ,0576 0, , , , , , , , , ,45633 (Chaco) Mataco 0, , , , , , , , , , , , , , ,48771 (Salta) Mataco (Wichí) 0, , , ,5917-0, , , , , , , , , , ,48716 Argentina Mataco (Wichí) 0, , , , , , , , , , , , , , ,33264 Formosa Mosetén -0, , , , , , , , ,1745-0,0319 0, , , , ,53455 Bolivia Movima 0, , , , , , , , , , , , , , ,01466 Bolívia Murui- 0, , , , , , , , , , , , , , ,12779 Muinane Ngobe 0, , , , ,2456 0, , ,0041-0, , , , , , ,55781 Nukak 0, , , , , , , , , , , , , , ,01347 Paez 0, , , , , , , , , , , ,4297 0, , ,32051 Parakana 0, , , , , , , , , ,1263 0, ,2252 0, , ,11187 Pasto 0, , , , , , , , , , ,1476 0, ,2183 0, ,55593 Piaroa 0, , , , , , , , , , ,1033 0, , , ,20698 Piaroa 0, , , , , , , , , , ,2644 0, , ,2048 0,42652 Colombia Pilaga 0, , , , , , ,1681 0, , , , , , , ,23123 Formosa Poturujara 0, , , , , , , , , , , , , , ,29282 (Zoe) 0, , , , , , , , , , , , , , ,34183 (Arequipa) 0, , , , , , , , , , , ,3912 0, , ,46597 (Tayacaja) 0, , , , , , , , , , , , , , , , , ,0699 0, , , , , , , , , , , ,54686 Bolivia / Nmatsiqueng a (San Martin) 0, , , , ,0449 0, , , , , , , , , ,4492 Siona 0, , , , , , , , , , , , , , ,54658 Tayacaja Peru 0, , , , , , , , , , , , , , ,29239 Teribe 0,1911 0, , , , , , , , , , , , , ,62234 Teribe2 0, , , , ,6364 0, , , , , , , , , ,78775 Tikuna 0, , , , , , , , , , , , , , ,15945 Tikuna Brasil 0, , , , ,1815 0, , , ,4006 0, , , , , ,23854 Tikuna 0, , , , ,0863 0, , , , ,1746 0, , , , ,1253 Colombia Toba 0, , , , ,0287 0, , , , , , , ,3867 0, ,41768 Toba (chaco) 0, , , , , , , , , , , , , , ,39534 Toba 0, , , , , , , , , , , , , , ,38397 (Formosa) Tucano 0, , , , , , , , , ,2328 0, , , , ,10321 Tule-Cuna 0, , , ,2401 0,1143 0, , , , , , , , , ,35839 Waiãpi 0, , , , , , , , , , , , , , ,62282 Wapishana Brasil 0, , , , ,1332 0, , , , , , , , , ,46981 Waunana 0, , , , , , ,2268 0,2517 0, , , ,4172 0, , ,37363 Wayuu 0, , , , , , , , , , ,1504 0, , , ,05907 Wayuu Colombia 0, , , , , , , , , , , , , , ,19835 Wiwa 0, , , , , , , , , , , , ,0393 0,4924-0,00486 Wounan 0,1507 0, , , , , , , , , , , , , ,11444 Xavante 0, , , , , , , ,2366 0, , ,1193 0, , , ,65483 Yamamadi 0,5085 0, , , ,5369 0, , , ,7526 0, , , , , ,05062 Yaminawa 0, , , , , , , ,2008 0, , , ,4047 0,0007 0, ,34053 Yanomama 0,1714 0, , , , , , ,2521 0, , , , , , ,08344 Yanomama Brasil 0, , , , , ,0648 0, , , , , , , , ,05457 Yanomami 0, , , , , , , , , , , , , , ,02223 Yanomamö 0, , , ,5473 0, , , , , , , , , , ,29501 Yuko-Yukpa 0, , ,5137 0, , , , , , , , ,8489 0, , ,90879 Yuracaré 0, , , , , , , , , , , , , , ,30871 Zenu 0, , , , ,0054-0, , , , , , , ,1664 0, ,23746 Zoro 0, ,0568 0, , , , , ,3422 0, , , , , , ,38338 Apêndice XXVII B: Matriz de Fst entre populações da região centro norte da América do Sul (biom42) com quatro haplogrupos de DNA mitocondrial
229 230 Guane- Butaregua Guatuso Guayabero Guaymi Huetar Huetar2 Huitoto Ignaciano Ijka-Arhuaco Ingano Colombia Kanamarí Katuena Katukina Katukina2 Kaxináwa Guane- Butaregua 0 Guatuso 0, Guayabero 0, , Guaymi 0,2988 0, , Huetar 0, , , , Huetar2 0, , ,0827 0,0973-0, Huitoto 0, , , , , , Ignaciano 0, , , , , , , Ijka-Arhuaco 0, , , , , , , , Ingano Colombia 0,1454 0, , , , , , , , Kanamari 0, , , , , , , , , , Katuena 0, , , , , , , , , , , Katukina 0,351 0, , , , , , , , , , , Katukina2 0, , , , , , , ,314 0, , , , , Kaxináwa 0, , , , , , , , , , , , , , Kayapo 0, , , , , , , ,1421 0, , , , ,3838 0,1386 0,24732 Kogui 0, , , , , , , , ,347 0, , , , ,3935-0,01141 Kraho 0, , , , , , , , , , , ,2037 0, ,2512 0,20608 Kuna 0, , , , , , , , , ,5644 0, , , , ,43512 Kuna2 0, , , , , , , , , , , , , , ,26646 Kuna3 0, , , ,0187 0,1106 0, , , , , , , , , ,30347 Macushi 0, , , , , , , , , , , , , , ,11302 Makiritare 0, , , , , , , , , , , ,0275 0, , ,01295 Marúbo 0, , , , , ,4041 0, ,0903 0, , , , , ,466 0,13642 Marúbo Brasil 0, , , , ,3915 0, , ,1207 0, , , , , ,4911 0,06711 Mataco 0, , , , , , ,0928 0,254 0, , , , , , ,32888 (Chaco) Mataco -0, ,4939 0, , , , , , , , , , ,3615 0,4229 0,38055 (Salta) Mataco (Wichí) -0, , , , , , , ,2063 0, , , , , , ,39053 Argentina Mataco (Wichí) 0, , , , , , , , ,468 0,0225 0, , ,2209 0, ,2333 Formosa Mosetén 0, , , , ,2886 0,2938 0, , , , , , , ,1889 0,2803 Bolivia Movima 0, , ,3077 0, , , , , , , , , , , ,07384 Bolívia Murui- 0, , , , ,2873 0, ,0282 0, ,4268 0, , , , , ,07313 Muinane Ngobe 0, , ,0133-0, , , , , , , , , , , ,2475 Nukak 0, , , , , ,5964 0, , ,6374 0,1774 0, , , , ,21355 Paez 0, , , , , , , , , , , , , , ,00576 Parakana 0, , , , , , , , , , , , , , ,07103 Pasto 0, , ,0363-0, , , , , , , ,0754 0, , , ,18705 Piaroa 0, , , , ,15 0,1595-0, , , , , , , , ,03207 Piaroa 0, , ,1073 0, , , ,0196 0, , , , , , , ,1203 Colombia Pilaga 0, , , , , ,3239 0, , , , , , , , ,17482 Formosa Poturujara 0, ,2329 0, ,1717 0, , , , , , , , , ,1816 0,03531 (Zoe) 0, , ,2338 0, , , , , , , , , , , ,24121 (Arequipa) 0, , , , , , , , , , , , , , ,33071 (Tayacaja) 0, , , , , , , , , , , , ,1139 0, , , , , , , , , , , ,1131 0, ,375 0, , ,40131 Bolivia / Nmatsiqueng a (San Martin) -0, ,4705 0,2809 0, , ,371 0, , , , , , , , ,30795 Siona 0, , , ,0387 0, ,0417 0, , , , , , , ,0686 0,15323 Tayacaja Peru 0, , , , , , , , , , , , ,1139 0, ,14993 Teribe 0, , , , , , , , , , , , , , ,26457 Teribe2 0,6417 0, , , ,2158 0, , , , , , ,5193 0, , ,4672 Tikuna 0, , , , , , , , ,2731 0, , , , ,272 0,06618 Tikuna Brasil 0,3161 0, , , , , , , , , , , , , ,1532 Tikuna 0, , , , , , , , , , , , , , ,08663 Colombia Toba 0, , , , , , , , , , , ,114 0, , ,29904 Toba (chaco) -0, , , , , , , , ,4657 0,0922 0, , , , ,25116 Toba 0, , ,1524 0, , , ,0527 0, , ,1366 0, , , , ,19267 (Formosa) Tucano 0, , , , , , , , , , , , , , ,14111 Tule-Cuna 0, , , , , , , , , , , , , , ,05815 Waiãpi 0, ,0503 0, , , , , , , , , , , , ,27281 Wapishana Brasil 0, ,609 0, , , , ,0722 0, , , , , , ,5327 0,36079 Waunana 0, , , , , , , , ,5789 0, , , ,3073 0, ,30656 Wayuu 0, , , , , , , , , , , , , , ,00379 Wayuu Colombia 0,1794 0, , ,1868 0, , , , , ,0164-0, , , , ,05727 Wiwa 0, , , , , , , , , , ,1184 0, , , ,00852 Wounan 0, , , , , , , , , , , , , , ,01381 Xavante 0, , , , , , , , , , , , ,5842 0, ,50555 Yamamadi 0, , , , ,6879 0, , , , , , , , , ,32196 Yaminawa 0, , , , , , , , , , , , , , ,01411 Yanomama 0, ,4248 0, , , , , ,0211 0, , , , , , ,04533 Yanomama Brasil 0, , , , ,4092 0, , , ,5196 0, , ,0258-0, , ,13448 Yanomami 0, , ,5273 0, , , , , , , , , , , ,26998 Yanomamö 0,0911 0, , , , , , , , , , , , , ,27143 Yuko-Yukpa 0, , , , , , , , , , ,7326 0, , , ,85789 Yuracaré 0, , , , , , , , ,222 0, , , , ,1097 0,05814 Zenu 0, , , ,1925 0,2945 0, , , ,3723-0, , , , , ,10731 Zoro 0, , , , , , , , , , ,1446 0, , , ,25303 Apêndice XXVII C: Matriz de Fst entre populações da região centro norte da América do Sul (biom42) com quatro haplogrupos de DNA mitocondrial
230 231 Kayapó Kogui Kraho Kuna Kuna2 Kuna3 Macushi Makiritare Marúbo Marúbo Brasil Mataco (Chaco) Mataco (Salta) Mataco (Wichí) Argentina Mataco (Wichí) Formosa Mosetén Bolivia Kayapo 0 Kogui 0, Kraho -0, , Kuna 0, ,5493 0, Kuna2 0, , , , Kuna3 0, , , ,1198-0, Macushi 0, , ,1498 0, , , Makiritare 0, , , , , , , Marúbo 0, , , ,6623 0, , , , Marúbo Brasil 0, , , , , , , , , Mataco 0, , , ,6054 0,404 0, , , , , (Chaco) Mataco 0, , , , , , ,1764 0, , , , (Salta) Mataco (Wichí) 0, , ,0847 0, , , , ,4532 0, , , , Argentina Mataco (Wichí) 0, , , , , , ,0731 0, , , ,1279 0, , Formosa Mosetén -0,0657 0, , , , ,2228 0, , , , ,2548 0, , , Bolivia Movima 0, , , , , , , ,0469-0, , , , , , ,40157 Bolívia Murui- 0, , , , , , , ,0576-0, , , , , , ,23346 Muinane Ngobe 0, , , , , , , , , ,466 0, , ,3543 0, ,09046 Nukak 0,5244 0, , , , , , , , ,1217 0, , , , ,52006 Paez 0, , , , , , , , , , , , , , ,25643 Parakana 0,2183 0, , , , , , , , , , , ,1868 0, ,23648 Pasto -0, , , ,3557-0, , , , , , , ,3019 0, , ,03511 Piaroa 0, , , , , , , , , , , , , , ,17557 Piaroa 0,2347 0, , , , , , , , , , , , , ,27642 Colombia Pilaga 0, , , , , , , , , , , , , , ,18532 Formosa Poturujara 0, ,15 0, , , , , , , , , , , , ,26279 (Zoe) 0, , ,0223 0, , ,3751 0, ,293 0, , , , , , ,09678 (Arequipa) 0, , , , ,3813 0, , , , , , , , , ,08415 (Tayacaja) 0,0925 0,2542 0,0581 0, , , , , , ,1536 0, , , , , , , , , , , , , , ,4816 0, ,0507 0, , ,07187 Bolivia / Nmatsiqueng a (San Martin) 0, , , ,6309 0, , , ,3808 0,2009 0, , , , , ,10574 Siona 0, , , , , , , , , , , , , , ,18388 Tayacaja Peru 0,0925 0,2542 0,0581 0, , , , , , ,1536 0, , , , ,10559 Teribe 0, , , , , ,03 0, , , , , , , , ,22354 Teribe2 0, , , , , ,68 0, , , , , ,6252 0, ,56284 Tikuna 0, , , , , , , , , , , , , , ,26959 Tikuna Brasil 0, , , , , ,4622-0, , , , , , , , ,37141 Tikuna 0, , , , , ,3996-0, , , , , , , , ,2684 Colombia Toba 0, , , , , , , , , , , ,1209 0, , ,29742 Toba (chaco) 0, ,3659 0, , , , , ,3225 0, , , , , , ,08794 Toba 0, , , , , , , , ,1557 0, , , ,0872 0,0652 0,10065 (Formosa) Tucano 0, , , , , , , , ,0595-0, , , , , ,34689 Tule-Cuna 0, ,1791 0, , , , , , ,2466 0, , , , , ,07303 Waiãpi 0, , , , , , , , , , , , , , ,36727 Wapishana Brasil 0, , ,3205 0, ,5156 0, , , , , , , , , ,39016 Waunana 0, ,3813 0, , , , , , , , , ,0692 0, , ,18141 Wayuu 0, , , , , , , , , , , , , , ,31949 Wayuu Colombia 0, , , , , , , ,111 0, , ,2456 0, , , ,12288 Wiwa 0, , , , , , , , , , , ,4837 0, , ,46121 Wounan 0,2003 0, ,1292 0, , , , , , , , ,293 0, , ,22299 Xavante 0, , , , , , , ,6282 0, , , , , , ,1172 Yamamadi 0, , , ,6815 0, , , , ,25 0, , , , ,6803 Yaminawa 0, , , , , , , , , , , , , , ,28123 Yanomama 0, , , , , , , ,019 0, ,0509 0, , , , ,25178 Yanomama Brasil 0, , , , , , , , , , , , , , ,36932 Yanomami 0, , ,5202 0, , ,6599 0, , , , , , , , ,60983 Yanomamö 0, ,3269 0, , , , , , , , , , , , ,18581 Yuko-Yukpa 0, , , , ,7791 0, , , , , , , , ,66416 Yuracaré 0, , , ,3463 0,1081 0, , , , , ,2093 0, , , ,04257 Zenu 0, , ,0051 0, , , , , , , , , , , ,08816 Zoro 0, , , , , , , , , ,1685 0,0582 0, , , ,36138 Apêndice XXVII D: Matriz de Fst entre populações da região centro norte da América do Sul (biom42) com quatro haplogrupos de DNA mitocondrial
231 232 Movima Bolívia Murui- Muinane Ngöbé Nukak Paez Parakanã Pasto Piaroa Movima 0 Bolívia Murui- 0, Muinane Ngobe 0, , Nukak 0, ,1599 0, Paez 0, , , , Parakana -0,0028-0, , , , Pasto 0, , , , ,1061 0, Piaroa 0, ,0282 0, , , , , Piaroa 0, , , , , , , , Colombia Pilaga 0, , , , , ,0276 0, , , Formosa Poturujara 0,1622 0, , , ,0572 0,0858 0, , ,0513 0, (Zoe) 0, , , , , , , , , , , (Arequipa) 0, ,1649 0,3279 0, , , , , , , , , (Tayacaja) 0, , , , , , , ,0002 0,0756 0, , ,0224 0, , , , , , ,2644 0, , , , , , , , Bolivia / Nmatsiqueng a (San Martin) 0, , , , , , , , , ,0312 0, , , ,0224 0,07553 Siona 0, , ,01 0, , , , , , , , , , , ,43232 Tayacaja Peru 0, , , , , , , ,0002 0,0756 0, , ,0224 0, , ,15955 Teribe 0, , , , ,1413 0, , , , , , , , , ,47301 Teribe2 0, ,5921 0,2312 0, , ,6648 0,401 0, , , , , ,7014 0, ,70922 Tikuna 0, , , , , , , , , , , , , , ,32996 Tikuna Brasil 0, ,0324 0, , , , , , , , , , , , ,44392 Tikuna 0, , , , , , , , , ,028 0, , , , ,30564 Colombia Toba 0,2485 0, , , , , , , , ,0011 0, , , , ,32275 Toba (chaco) 0, , , , , , , , , , , , , , ,09277 Toba 0, , , , ,2198 0, , , , ,0417 0, , , , ,19151 (Formosa) Tucano 0, , , , , , , ,025 0,1945 0, , , , , ,36476 Tule-Cuna 0,2404 0, , , , , , , , , , , , , ,24261 Waiãpi 0, , , , , , , , , , , , , ,2639 0,56881 Wapishana Brasil 0, , , , , , ,4366 0,1151 0, , , , , , ,42761 Waunana 0, ,1291 0, , , , , , , , , , , , ,05532 Wayuu 0, , ,3537 0, , , , , , , , , , ,1758 0,37868 Wayuu Colombia 0,1054 0, , , , , , , , , , , , , ,16172 Wiwa -0, ,107 0,4672 0, , , , , , , , ,3279 0, , ,52749 Wounan 0, , , , , , , , , , , , ,2265 0, ,29298 Xavante 0, , , , , , , , , ,2473 0, , , , ,01212 Yamamadi 0, , , , , , ,7619 0, , , , , , , ,67859 Yaminawa 0, , , , , , , , , , , , ,3947 0, ,4563 Yanomama 0, , , , ,1482-0, , , , , , , , , ,27359 Yanomama Brasil -0,0144-0, , , , , , , , ,0506 0, , , , ,37867 Yanomami 0, , , , , , , , , , , , , , ,59636 Yanomamö 0, , , , , , , , ,3519 0, , , , , ,08664 Yuko-Yukpa 0, , , , , , ,913 0, , , , ,3503 0, , ,29335 Yuracaré 0, , , , , , , , ,112 0, , , , ,0493 0,16615 Zenu 0, , , , , ,0096 0, , , , , , , , ,09898 Zoro 0, ,0985 0, , , , , , , , , , , , ,44156 Piaroa Colombia Pilagá Formosa Poturujara (Zoé) (Arequipa) (Tayacaja) Bolivia Apêndice XXVII E: Matriz de Fst entre populações da região centro norte da América do Sul (biom42) com quatro haplogrupos de DNA mitocondrial / Nmatsiqueng a (San Martin) Siona Tayacaja Peru Teribe Teribe2 Tikúna Tikúna Brasil Tikúna Colombia Toba Toba (chaco) Toba (Formosa) Tucano Tule-Cuna Waiãpi / Nmatsiqueng a (San Martin) 0 Siona 0, Tayacaja Peru 0,0224 0, Teribe 0, , , Teribe2 0,6572 0, , , Tikuna 0, , , , , Tikuna Brasil 0, , , , , , Tikuna 0, , , , , , , Colombia Toba 0, , , , , , , , Toba (chaco) -0, , , , , ,155 0,1936 0, , Toba 0, , , ,2542 0, , , , , , (Formosa) Tucano 0, , , , , , , , , , , Tule-Cuna 0,2045 0, , , , , , , , , , , Waiãpi 0, , ,2639 0, , , , , , , , , , Wapishana Brasil 0, , , , ,7928 0, , ,1065-0, , ,0748 0, , , Waunana 0, , , , , , , , , , , ,1894 0, , ,31974 Wayuu 0, ,2959 0,1758 0, , , , , , , , , , , ,38564 Wayuu Colombia 0, ,1962 0, , , ,1232 0, , ,2114 0, , , , ,3431 0,29704 Wiwa 0, ,4205 0, , , , , , , , , , ,2264 0, ,48156 Wounan 0, , , , , , , , , , ,1653 0, , , ,31488 Xavante 0,1248 0, , , , , , , , , , , , , ,5212 Yamamadi 0, , , , , , , , , ,5258 0, , , ,68923 Yaminawa 0, , , , , , , , , , , , , , ,41037 Yanomama 0, , , , , , , , , , , , , , ,3158 Yanomama Brasil 0, , , , , , , , , , , , ,2544 0, ,1937 Yanomami 0, ,6253 0, , , , , ,2029 0, , , , , , ,55652 Yanomamö 0,0622 0, , , , , , , , , , , , , ,25748 Yuko-Yukpa 0, , , , , , , , , , , , , ,8944 Yuracaré 0, , ,0493 0, , , , , , , , , , , ,2826 Zenu 0, , , , , , , , , , , , , ,361 0,24459 Zoro 0, , , , , , , , , ,1654 0, , , , ,01109 Wapishana Brasil Apêndice XXVII F: Matriz de Fst entre populações da região centro norte da América do Sul (biom42) com quatro haplogrupos de DNA mitocondrial
232 233 Wayuu Yanomama Waunana Wayuu Colombia Wiwa Wounan Xavante Yamamadi Yaminawa Yanomama Brasil Yanomami Yanomamö Yuko-Yukpa Yuracaré Zenu Zoro Waunana 0 Wayuu 0, Wayuu Colombia 0, , Wiwa 0, , , Wounan 0, , , , Xavante 0, ,4933 0, , , Yamamadi 0, , , , , , Yaminawa 0,3985 0, , , , , , Yanomama 0, ,0209 0, , , , , , Yanomama Brasil 0, , ,1141 0, , , , , , Yanomami 0, , , , , ,68 0, , , , Yanomamö -0, ,2041 0, , , , , , ,1115 0, , Yuko-Yukpa 0, , , , ,7385 0, , , , , , Yuracaré 0, , , , ,0386 0, , , , , , , , Zenu 0, ,0798-0, , ,0364 0, , , , , , , , , Zoro 0, , , , , , , , , , , , , , , Apêndice XXVII G: Matriz de Fst entre populações da região centro norte da América do Sul (biom42) com quatro haplogrupos de DNA mitocondrial
233 Ache Ancash Arara Arequipa Peru Assurini Assurini2 Atacameno Atacameno2 Atacamenos Awá-Guajá Aymara Aymara2 Baníwa Boruca Bribi-Cabecar Cayapa Cayapa Ecuador Chimane Chimila Choroti Choroti2 Coreguaje Curripaco Embera Embera2 Embera3 Gaviao Guahibo Guahibo-Sikuani Guambiano Guane-Butaregua Guatuso Guayabero Guaymi Huetar Huetar2 Huitoto Ignaciano Ijka-Arhuaco Ingano Colombia Kanamarí Katuena Katukina Katukina2 Kaxináwa Kayapó Kogui Kraho Kuna Kuna2 Kuna3 Macushi Makiritare Marúbo Marúbo Brasil Mataco (Chaco) Mataco (Salta) Mataco (Wichí) Argentina Mataco (Wichí) Formosa Mosetén Bolivia Movima Bolívia Murui-Muinane Ngöbé Nukak Paez Parakanã Pasto Piaroa Piaroa Colombia Pilagá Formosa Poturujara (Zoé) (Arequipa) (Tayacaja) Bolivia / Nmatsiquenga (San Martin) Siona Tayacaja Peru Teribe Teribe2 Tikúna Tikúna Brasil Tikúna Colombia Toba Toba (chaco) Toba (Formosa) Tucano Tule-Cuna Waiãpi Wapishana Brasil Waunana Wayuu Wayuu Colombia Wiwa Wounan Xavante Yamamadi Yaminawa Yanomama Yanomama Brasil Yanomami Yanomamö Yuko-Yukpa Yuracaré Zenu Zoro Ache Ancash Arara Arequipa Peru Assurini Assurini2 Atacameno Atacameno2 Atacamenos Awá- Guajá Aymara Aymara2 Baníwa Boruca Bribi- Cabecar 0 0, , , , , , , , , , , , ,7066 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,1981 0, , ,2315 0, , , , , , , , , , , , , , , , , , , , , , , ,4176 0, , , , , , , , , , , , , , , ,0536 0, , , , , , , , ,0784 0,261 0, , , , , , , , , ,0554 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0104 0, , , ,0301 0, , , ,1032 0, , , , , , , , , , , , , , , , , , ,207-0, , , ,5234 0, , , , , , , , , , , , , , , , ,0669 0, , , ,2024-0, , , , , , ,3831 0, , , , , ,7067 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,5305 0,3912 0, , , , , , , , , , , , ,2955 0, , , , , , , ,6274 0, , , , , , , , , , , , , , , , , ,0847 0, , , , ,0162 0, , , , , , , , , , , , , , , , , , , , , , , , , , , ,7187 0,4179 0, , , , , , , , , , , , , , , , ,2589 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,2388 0, , , , , ,7107 0, , , , , ,3444 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,1746 0, , , , , , ,5642 0,5397 0, , , , , , , , , , , , , , , , , , , ,1013 0, ,1712 0, , , , , , , , , , , , , , , , , , , , , , , , ,6205 0, , , , , ,6721 0, , , , , , , , , , , , , , , , , , , , , ,3584 0, , , , , , , , , , , , , , , , , , , , , ,2379 0, , , , , ,3765 0, , , , , , ,0136 0, , , , , , , , ,4729 0, ,4685 0, , , , ,482 0, ,1998 0, , , , , , ,0577 0, , , ,037 0, , , , ,5533 0, , , , , , , , , , , , , , ,3813 0, , , ,2988 0, , , , , , , , , , , , , , , , , , , , , , , , ,1899 0, , , , , , , , , , , , , , , , , , , , , , , , , ,4844 0,422 0,5962 0, , , , , , , , ,6261 0, , , , , , , , , ,0199 0,7 0, , , , , , , , , , , , , , , , , , , ,4644 0, , , , , , , , , , , , , ,1689 0,1332 0, , , , , , , , , , , ,0524 0, , , , , ,0242 0, , , , , , , ,0256 0, , , , , , , , , , ,4245 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,5251 0,3279 0, , , , , , , , , , , ,705 0, , , , , , ,4897 0, , , , , , ,524 0, ,2817 0, , , ,6022 0,4082 0, ,4008 0, , , ,0405 0, , , , , , , , , , , , , , , , , , , , , , , , ,1875 0, , , , , , , , , , , ,0486 0, , ,1875 0, , , , , , , , , , , , , , , , , , , , , , , , , ,0805-0, , , , , , , , , , , , , , , ,0383 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,1981-0, , , ,0181 0, , , , , , ,1403 0, , , , , ,1214 0, , , , , , , , ,0198 0, , , , , , , , , , , , , , , , , , , , , , , , , ,2619 0, , , , , , , , , , , ,0181 0, , , , , , ,1403 0, , , , , ,1214 0, , , , , , , , , , , ,44 0, , , , , , ,7014 0, , , , , , , , , , , , , ,0115 0, , , ,3122 0, , , ,156 0, , , , ,287 0, , , , , , , , , , , , , , , , , , , ,2508 0, ,4803 0, , , , , , , , , , , , , , ,66 0, , ,2777 0,2408 0, , , , , , , , , , ,2883 0, , , , , , , , , ,0092 0, , , , , , ,141 0, , , , , , , , , ,3249 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,4632 0, , , , , , , , , , , , , , , , , ,1808 0,0359 0, , , , , , , , , , , , , , , , , , ,628 0, , , , , , , , , , , , , ,3889 0, , ,0504 0, , , , , , , , , , , , , , ,2554 0,5197 0, , , , ,2265 0, , , , ,283 0, , , , , , , , , , , ,0067-0, ,0052 0, , ,0484 0, , , , , , , , , , , , , , , , , , , ,3947 0, , , , , , , , , , , , ,1219 0, , , , , , , , , , , , , , , , , , , ,3439 0, , , , , , , , , ,3328 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,4661 0, , , , , , , ,1703 0, ,5094 0,7284 0, , , , , , , , , , ,409 0, ,0247 0, , , , , , , , , , , , , ,0921 0, , , , , , , , , , , , , , , , , ,34998 Apêndice XXVIII A: Matriz de Fst entre populações da região centro norte da América do Sul (biom42) com cinco haplogrupos de DNA mitocondrial 234
234 Cayapa Cayapa Chimane Chimila Choroti Choroti2 Coreguaj Curripaco Embera Embera2 Embera3 Gaviao Guahibo Guahibo- Guambiano Ecuador e Sikuani Cayapa 0 Cayapa Ecuador 0, Chimane 0, , Chimila 0, , , Choroti 0, ,0365 0, , Choroti2 0, , , , Coreguaje 0, , ,3301 0, , , Curripaco 0, ,1101-0, , , , , Embera 0, , , , , ,2661 0, , Embera2 0, , , , ,0267 0, ,0109 0, Embera3 0, , , , , , , , , , Gaviao 0, , , , , , , , , , , Guahibo 0, ,1476 0, , , ,1798 0, , , , , , Guahibo-Sikuani 0,1357 0,1455 0, , , , , , , ,1874 0, , , Guambiano 0, , , , , , , ,5298 0, , , , , , Guane-Butaregua 0, , , , , , ,1609 0, , , , , , ,49938 Guatuso 0, , , , , , , ,174-0, ,2616 0, , , , ,65625 Guayabero 0, , , , , , , , , , , , , , ,37216 Guaymi 0, , , , , , , , , , , , , , ,56452 Huetar 0,2262 0, ,2647 0, , , , ,1955 0, , , , , , ,54399 Huetar2 0, , , , , , , , , , , , , , ,55042 Huitoto 0, , , , , , , ,2604 0, , , , , , ,28047 Ignaciano 0, , ,1157 0, , , , , , , , , , , ,17809 Ijka-Arhuaco 0, , ,3516 0, , , , , , ,3528 0, , , , ,59842 Ingano Colombia 0, , , , , , , , ,2955 0, , , ,183 0, ,22785 Kanamarí 0, , , , , , , , , , , , , , ,21901 Katuena 0, , , , , , , , , ,1708 0, , , , ,13421 Katukina 0, , , , , , , , ,4088 0, , , , , ,10116 Katukina2 0, , , , , , , , , , , , , , ,60821 Kaxináwa 0, , , , , , ,1207 0,2311 0, , , , , , ,16654 Kayapó 0, ,1107-0, , , , , , , , , , ,2918 0, ,53211 Kogui 0, , , , , , , , , , , , ,0064 0, ,11318 Kraho 0, , , , ,008 0, , , , , , , , ,43044 Kuna 0, , , , ,6028 0, , ,4285 0, , , , , , ,76951 Kuna2 0, , , , , , , , , , , , ,2671 0, ,57808 Kuna3 0,2113 0, , , , ,3676 0, , , , , , , , ,61836 Macushi 0, , , , , , , , , , , , , , ,2082 Makiritare 0, , , , , , , , , ,3157 0, , , ,3276-0,03214 Marúbo 0, , , , , ,0523 0, , , , , , , , ,08177 Marúbo Brasil 0, , , ,6191 0, , , , , , , , , , ,00352 Mataco (Chaco) 0, , , , ,0576 0, , , , , , , , , ,45633 Mataco (Salta) 0, , , , , , , ,3718 0, , , , , ,46762 Mataco (Wichí) Argentina 0, , , , , , , , , , , , , ,47058 Mataco (Wichí) Formosa 0, , , , , , , , , , , , , ,33264 Mosetén Bolivia 0, , ,0355 0, , , , , , , , , , , ,51014 Movima Bolívia 0, , , , , , , , , , , , , , ,01466 Murui-Muinane 0,1226 0, , , ,031-0,0132 0, , , , , , , , ,13144 Ngöbé 0, , , ,1655 0,2456 0, , ,0041-0, , , , , , ,55781 Nukak 0, , , , , , , , , , , , , , ,01347 Paez 0, , , , , , ,2496 0, , , , ,4297 0, , ,32051 Parakanã 0,132 0, , , , , , , , , , ,2252 0, , ,11187 Pasto 0, , , , , , , , , ,0347 0,1476 0, ,2183 0, ,55593 Piaroa 0, , , ,369 0, , , , , , ,1033 0, , , ,20698 Piaroa Colombia 0, , , , , , , , , ,1592 0,2644 0, , , ,42652 Pilagá Formosa 0, , , ,4843-0,0139-0, , , , , , , , , ,22728 Poturujara (Zoé) 0, , , , , , , , , , , , , , , ,073 0, , , , , , , ,0258 0, , ,2913 0, ,32784 (Arequipa) 0, , , , , , , ,3831 0, , ,3912 0, , ,46597 (Tayacaja) 0, ,0029 0,0939 0, , , , , , ,0746 0, , , ,28545 Bolivia 0, , , , , , , , , , , , , , ,54686 / Nmatsiquenga (San Martin) 0, , ,0934 0, , , , , ,0246 0, , ,3632 0, ,43103 Siona 0, , , , , , ,4201 0, , , , , , , ,54658 Tayacaja Peru 0, ,0029 0,0939 0, , , , , , ,0746 0, , , ,28545 Teribe 0, , , , , , ,4975 0, , , , , , , ,62234 Teribe2 0, , , , ,6364 0, , , , , , , , , ,78775 Tikúna 0, , , , , , , , , , , , , , ,15945 Tikúna Brasil 0, , , , ,1815 0, , , , ,2499 0, , , , ,23854 Tikúna Colombia 0, , , , ,0863 0, ,1046 0, , , , , , , ,1253 Toba 0, , , , ,0287 0, , , , ,154 0, , ,3867 0, ,41768 Toba (chaco) 0, , , , , ,2583 0, , , , , ,3074 0, ,37414 Toba (Formosa) 0, , , , , , , , , , , , , ,38397 Tucano 0, , , , , , , , , , , , , , ,10321 Tule-Cuna 0, , , , , , , , , ,0361 0, , , ,0676 0,34691 Waiãpi 0, , , , , , , , ,0742 0, , , , , ,62282 Wapishana Brasil 0, , , , ,1332 0, , , , , , , , ,4389 0,46981 Waunana 0, , , , , , , ,2517 0, , , ,4172 0, , ,37363 Wayuu 0, , , ,5115 0, , , , , , ,1504 0, , , ,05907 Wayuu Colombia 0, , , , , , ,0776 0, , , , , ,1038 0, ,1948 Wiwa 0, , , , , , ,0234 0, , , , , ,0393 0, ,00486 Wounan 0,1342 0, , , , , , , , ,1434 0, , , , ,11444 Xavante 0,1567 0, , , , , , ,2366 0, , ,1193 0, , , ,65483 Yamamadi 0, , , ,833 0,5369 0, , , , , , , , , ,05062 Yaminawa 0, , , , , , , ,2008 0, , , ,4047 0,0007 0, ,34053 Yanomama 0,1518 0, , , , , , ,2521 0,3361 0, , , , , ,08344 Yanomama Brasil 0, , , , , ,0648 0, , , , , , , , ,05457 Yanomami 0, , , , , , ,0228 0, , , , , , ,4442-0,00346 Yanomamö 0, , , , , , , , , , , , , , ,29501 Yuko-Yukpa 0, , , , , , , , , , , ,8489 0, , ,90879 Yuracaré 0, ,0268 0, , , , , , , , , , , , ,30005 Zenu 0, , , , , , , ,229 0, , , , , ,22936 Zoro 0, ,0568 0, ,4911 0, , , ,3422 0, , , , , , ,38338 Apêndice XXVIII B: Matriz de Fst entre populações da região centro norte da América do Sul (biom42) com cinco haplogrupos de DNA mitocondrial 235
235 236 Guane- Guatuso Guayabero Guaymi Huetar Huetar2 Huitoto Ignaciano Ijka- Ingano Kanamarí Katuena Katukina Katukina2 Kaxináwa Butaregua Arhuaco Colombia Guane-Butaregua 0 Guatuso 0, Guayabero 0, , Guaymi 0,2988 0, , Huetar 0, , , , Huetar2 0, , , ,0973-0, Huitoto 0, , ,162 0, , , Ignaciano 0, , , ,2331 0, , , Ijka-Arhuaco 0, , , , , , , , Ingano Colombia 0, ,4035 0, , , , , , , Kanamarí 0, , , , , , , ,0057 0, , Katuena 0, , , , , , , , , , , Katukina 0, , , , , , , , , , , , Katukina2 0, , , , , , , , , , , , , Kaxináwa 0, , ,0834 0, , , , ,0686 0, , , , , , Kayapó 0, , , , , , , , , , , , ,3526 0,1386 0,24732 Kogui 0, , , , , , , , ,347 0, , , ,1441 0,3935-0,01141 Kraho 0, , , , , , , , , , , ,2037 0, ,2512 0,20608 Kuna 0, , , , , , , , , , , , , , ,43512 Kuna2 0, , , , , , , , , , , , , , ,26646 Kuna3 0, , , ,0187 0,1106 0, , , , , , , , , ,30347 Macushi 0, , , , , , , , , , , , , , ,11302 Makiritare 0, , , , , , , , , , , ,0275 0, , ,01295 Marúbo 0, , , , , ,4041 0, , , ,1127 0, , , ,466 0,13642 Marúbo Brasil 0, , , , ,3915 0, , , , , , , , ,4911 0,06711 Mataco (Chaco) 0, , , , , , ,0928 0, , , , , , , ,32888 Mataco (Salta) -0, ,4733 0, , , ,4123 0,2698 0, , , , , , , ,36074 Mataco (Wichí) Argentina -0, , ,2955 0,3333 0, , , ,1867 0, , , ,2891 0, , ,37391 Mataco (Wichí) Formosa 0, , , , , , , , ,468 0,0216 0, , , , ,2333 Mosetén Bolivia 0, , , , , ,2764 0, , , , , , , ,1758 0,25973 Movima Bolívia 0, , , , , , , , , , , , , , ,07384 Murui-Muinane 0, , , , ,2779 0, , , , , , , , , ,07204 Ngöbé 0, , ,0715-0, , , , , , , , , , , ,2475 Nukak 0, , , , , ,5964 0, , ,6374 0, , , , , ,21355 Paez 0, , , , , , , , , , , , ,2142 0, ,00576 Parakanã 0, , , , , , , , , , , , , , ,07103 Pasto 0, , , , , , , , , , ,0754 0, , , ,18705 Piaroa 0, , , , ,15 0,1595-0, , , , , , ,0008 0, ,03207 Piaroa Colombia 0, , , , , , ,0196 0, , , , , , , ,1203 Pilagá Formosa 0, ,4107 0, , ,308 0, , ,0491 0, , , , , , ,16994 Poturujara (Zoé) 0, ,2329 0, ,1717 0, , , , , , , , , ,1816 0, , , , , , , , , ,4254 0, , , , , ,22801 (Arequipa) 0, , ,2589 0, , , , , , , , , , , ,33071 (Tayacaja) 0, , , , , , , , , , , , , , ,14523 Bolivia 0, , , , , , , , , , , ,375 0, , ,40131 / Nmatsiquenga (San Martin) -0, , , , ,3453 0,3553 0, , , , , , , , ,29147 Siona 0, , , ,0387 0, ,0417 0, , , , , , , ,0686 0,15323 Tayacaja Peru 0, , , , , , , , , , , , , , ,14523 Teribe 0, , , , , , , , , , , , , , ,26457 Teribe2 0,6417 0, , , ,2158 0, , , , , , ,5193 0, , ,4672 Tikúna 0, , , , , , , , ,2731 0, , , ,0136 0,272 0,06618 Tikúna Brasil 0,3161 0, , , , , , , , , , , ,0241 0, ,1532 Tikúna Colombia 0, , , , , , , , , , , , , , ,08663 Toba 0, , , , , , , , , ,192 0, ,114 0,0969 0, ,29904 Toba (chaco) -0, , , , , , , , , ,078 0, , , , ,23341 Toba (Formosa) 0, , , , , , ,0527 0, , ,1286 0, , , , ,19267 Tucano 0, , , , , , , , , ,111 0, , , , ,14111 Tule-Cuna 0, , , , , , , , , , , ,1183 0, , ,05497 Waiãpi 0, ,0503 0, , , , , ,3991 0, , , , ,4358 0, ,27281 Wapishana Brasil 0, ,609 0,3417 0, , , ,0722 0, , , , , , ,5327 0,36079 Waunana 0, , , , , , , , ,5789 0, , , , , ,30656 Wayuu 0, , , , , , , , , , , , , , ,00379 Wayuu Colombia 0, , ,0951 0, , , , , , , ,0025 0, ,1314 0, ,05549 Wiwa 0, , , , , , , , , , ,1184 0, , , ,00852 Wounan 0, , , , , , , , , , , , , , ,01381 Xavante 0, , , , , , , , , , , , , , ,50555 Yamamadi 0, , , , ,6879 0, , , , , , , , , ,32196 Yaminawa 0, , , , , , , , , , , , , , ,01411 Yanomama 0, ,4248 0, , , , , , , , , , , , ,04533 Yanomama Brasil 0, , , , ,4092 0, , , ,5196 0, , ,0258-0, , ,13448 Yanomami 0, , , ,5349 0, , , , , , , , , , ,18755 Yanomamö 0,0911 0, , , , , , ,0541 0, , , , ,2323 0, ,27143 Yuko-Yukpa 0, , , , , , , , , ,6008 0,7326 0, , , ,85789 Yuracaré 0, , , , , , ,1288 0, , , , , ,1595 0, ,05647 Zenu 0, , , , , , , , , , , , , , ,10194 Zoro 0, , ,2624 0, , , , , , , ,1446 0, , , ,25303 Apêndice XXVIII C: Matriz de Fst entre populações da região centro norte da América do Sul (biom42) com cinco haplogrupos de DNA mitocondrial
236 237 Kayapó Kogui Kraho Kuna Kuna2 Kuna3 Macushi Makiritare Marúbo Marúbo Brasil Mataco (Chaco) Mataco (Salta) Mataco (Wichí) Argentina Mataco Mosetén (Wichí) Bolivia Formosa Kayapó 0 Kogui 0, Kraho -0, , Kuna 0, ,5493 0, Kuna2 0, , , , Kuna3 0, , , ,1198-0, Macushi 0, , ,1498 0, , , Makiritare 0, , , , , , , Marúbo 0, , , ,6623 0, , , , Marúbo Brasil 0, , , , , , , , , Mataco (Chaco) 0, , , ,6054 0,404 0, , , , , Mataco (Salta) 0, , , ,596 0, , , , , , , Mataco (Wichí) Argentina 0, , , , , , , , , , , , Mataco (Wichí) Formosa 0, , , , , , ,0731 0, , , ,1279 0, , Mosetén Bolivia -0, , , , , , , , , , , ,106 0, , Movima Bolívia 0, , , , , , , ,0469-0, , , , , , ,37898 Murui-Muinane 0, , , , , ,4176-0, , , , , ,1701 0, , ,2023 Ngöbé 0, , , , , , , , , ,466 0, , , , ,08781 Nukak 0,5244 0, , , , , , , , ,1217 0, , , , ,49438 Paez 0, , , , , , , , , , , , , , ,24013 Parakanã 0,2183 0, , , , , , , , , , , , , ,21343 Pasto -0, , , ,3557-0, , , , , , , ,2825 0, , ,02928 Piaroa 0, , , , , , , , , , , , , , ,15677 Piaroa Colombia 0,2347 0, , , , , , , , , , ,3286 0, , ,25179 Pilagá Formosa 0, , , , , , , , ,0291 0,1025 0, , , , ,16508 Poturujara (Zoé) 0, ,15 0, , , , , , , , , , , , , , , , , , , , , , , , , , , ,08169 (Arequipa) 0, , , , ,3813 0, , , , , , , , , ,07698 (Tayacaja) 0, ,2479 0, , , ,2804-0, , , , , ,0735 0, , ,09102 Bolivia 0, , , , , , , , , ,4816 0, , , , ,07103 / Nmatsiquenga (San Martin) 0, , , , , , , , , , , , , , ,08607 Siona 0, , , , , , , , , , , ,3895 0, , ,1685 Tayacaja Peru 0, ,2479 0, , , ,2804-0, , , , , ,0735 0, , ,09102 Teribe 0, , , , , ,03 0, , , , , , , , ,21053 Teribe2 0, , , , , ,68 0, , , , , , , ,5307 Tikúna 0, , , , , , , , , , , , , , ,25578 Tikúna Brasil 0, , , , , ,4622-0, , , , , , , , ,35063 Tikúna Colombia 0, , , , , ,3996-0, , , , , ,2185 0, , ,252 Toba 0, , , , , , , , , , , , , , ,27132 Toba (chaco) 0,0837 0, , , , , , , , ,2344 0,036 0, , , ,07114 Toba (Formosa) 0, , , , , , , , ,1557 0, , , ,0787 0,0652 0,08791 Tucano 0, , , , , , , , ,0595-0, , , , , ,32355 Tule-Cuna 0, , ,0552 0, ,0602 0, ,1506 0, , , , , , , ,05959 Waiãpi 0, , , , , , , , , , , , ,4861 0, ,346 Wapishana Brasil 0, , ,3205 0, ,5156 0, , , , , , , , , ,36384 Waunana 0, ,3813 0, , , , , , , , , , , , ,16909 Wayuu 0, , , , , , , , , , , , , , ,29825 Wayuu Colombia 0, , ,0289 0, , , ,0799 0, ,1108 0, , , , , ,1067 Wiwa 0, , , , , , , , , , , , , , ,43086 Wounan 0,2003 0, ,1292 0, , , , , , , , , ,2918 0, ,20646 Xavante 0, , , , , , , ,6282 0, , , , , , ,11737 Yamamadi 0, , , ,6815 0, , , , ,25 0, , , , ,64811 Yaminawa 0, , , , , , , , , , , , , , ,25978 Yanomama 0, , , , , , , ,019 0, ,0509 0, ,2564 0, , ,23443 Yanomama Brasil 0, , , , , , , , , , , ,2822 0, , ,34776 Yanomami 0, , , , , , , , , , , , , , ,48808 Yanomamö 0, ,3269 0, , , , , , , , , , , , ,17641 Yuko-Yukpa 0, , , , ,7791 0, , , , , ,3043 0, , ,63993 Yuracaré 0, , , , , , ,0883 0,1791 0, , , , , , ,03218 Zenu 0, , , , , , , , ,1008 0, ,1782 0, , , ,07282 Zoro 0, , , , , , , , , ,1685 0,0582 0, , , ,3413 Apêndice XXVIII D: Matriz de Fst entre populações da região centro norte da América do Sul (biom42) com cinco haplogrupos de DNA mitocondrial
237 238 Movima Bolívia Murui- Muinane Ngöbé Nukak Paez Parakanã Pasto Piaroa Piaroa Colombia Pilagá Poturujara Formosa (Zoé) (Arequipa) (Tayacaja) Bolivia Movima Bolívia 0 Murui-Muinane 0, Ngöbé 0, , Nukak 0, , , Paez 0, , , , Parakanã -0,0028-0, , , , Pasto 0, , , , ,1061 0, Piaroa 0, , , , , , , Piaroa Colombia 0, , , , , , , , Pilagá Formosa 0, , , , , ,0279 0, , , Poturujara (Zoé) 0,1622 0, , , ,0572 0,0858 0, , ,0513 0, , , , , , ,0503 0, ,0774 0, , , (Arequipa) 0, , ,3279 0, , , , , , ,0805 0, , (Tayacaja) 0,1734 0,0198 0, , , , , , , , , , , Bolivia 0, ,2643 0, , , ,2644 0, , , , , , , , / Nmatsiquenga (San Martin) 0, , , , , , , , , , ,2358-0, , , ,07771 Siona 0, , ,01 0, , , , , , , , ,2869 0, , ,43232 Tayacaja Peru 0,1734 0,0198 0, , , , , , , , , , , , ,15831 Teribe 0, , , , ,1413 0, , , , , , , , , ,47301 Teribe2 0, , ,2312 0, , ,6648 0,401 0, , ,542 0, , ,7014 0, ,70922 Tikúna 0, , , , , , , , , , , , , ,0699 0,32996 Tikúna Brasil 0, , , , , , , , , , , , , ,0949 0,44392 Tikúna Colombia 0, , , , , , , , , , , , , , ,30564 Toba 0,2485 0, , , , , , , , , , ,0506 0, , ,32275 Toba (chaco) 0, , ,2472 0, , , , , , , , , , ,0034 0,09789 Toba (Formosa) 0, , , , ,2198 0, , , , , , , , ,0171 0,19151 Tucano 0, , , , , , , ,025 0,1945 0, , , , , ,36476 Tule-Cuna 0, , , , , , , , , , , ,1464 0, , ,23569 Waiãpi 0, , , , , , , , , , , , , , ,56881 Wapishana Brasil 0, , , , , , ,4366 0,1151 0, , , , , , ,42761 Waunana 0, , , , , , , , , , , , , , ,05532 Wayuu 0, ,0561 0,3537 0, , , , , , ,1465 0, ,2138 0, ,1707 0,37868 Wayuu Colombia 0, , , , , , , , , , ,1365 0, , ,075 0,15962 Wiwa -0, ,1065 0,4672 0, , , , , , , , , , , ,52749 Wounan 0, , , , , , , , , , , , ,2265 0, ,29298 Xavante 0, ,3626 0, , , , , , , , , , , , ,01212 Yamamadi 0, , , , , , ,7619 0, , , , ,4467 0, , ,67859 Yaminawa 0, , , , , , , , , , , , ,3947 0, ,4563 Yanomama 0, , , , ,1482-0, , , , , , , , , ,27359 Yanomama Brasil -0,0144-0, , , , , , , , , , , , ,1221 0,37867 Yanomami 0,0113 0, , , , , , , , , , , , , ,50293 Yanomamö 0, , , , , , , , ,3519 0, , ,033 0, ,1077 0,08664 Yuko-Yukpa 0, , , , , , ,913 0, , , , , , , ,29335 Yuracaré 0, ,0661 0, , , ,0653 0, , , , ,0779 0, , , ,16427 Zenu 0, , , , , , , , , , , ,0264 0, , ,10252 Zoro 0, , , , , , , , , , , , , , ,44156 Apêndice XXVIII E: Matriz de Fst entre populações da região centro norte da América do Sul (biom42) com cinco haplogrupos de DNA mitocondrial
238 239 / Nmatsiquenga (San Martin) Siona Tayacaja Peru Teribe Teribe2 Tikúna Tikúna Brasil Tikúna Colombia Toba Toba (chaco) Toba (Formosa) Tucano Tule- Cuna Waiãpi Wapishana Brasil / Nmatsiquenga (San Martin) 0 Siona 0, Tayacaja Peru 0, , Teribe 0, , , Teribe2 0,6334 0, , , Tikúna 0, , ,0699 0, , Tikúna Brasil 0, , ,0949 0, , , Tikúna Colombia 0, , , , , , , Toba 0, , , , , , , , Toba (chaco) -0, , ,0034 0, , , , , , Toba (Formosa) 0, , ,0171 0,2542 0, , , , , , Tucano 0, , , , , , , , , ,1336 0, Tule-Cuna 0, , , , , , , , , , ,1031 0, Waiãpi 0, , , , , , , , , , , , , Wapishana Brasil 0, , , , ,7928 0, , ,1065-0, , ,0748 0, , , Waunana 0, , , , , , , , , , , ,1894 0, , ,31974 Wayuu 0, ,2959 0,1707 0, , , , , , ,2412 0, , , , ,38564 Wayuu Colombia 0, , ,075 0, , , , , , , , , ,0428 0, ,28846 Wiwa 0, ,4205 0, , , , , , , , , , , , ,48156 Wounan 0, , , , , , , , , , ,1653 0, , , ,31488 Xavante 0, , , , , , , , , , , , , , ,5212 Yamamadi 0, , , , , , , , , ,5258 0, , , ,68923 Yaminawa 0, , , , , , , , , , , , ,0321 0, ,41037 Yanomama 0, , , , , , , , , , , , , , ,3158 Yanomama Brasil 0, , ,1221 0, , , , , , ,1773 0, , , , ,1937 Yanomami 0,4184 0, , , , , , , , , , , , , ,44363 Yanomamö 0, , ,1077 0, , , , , , , , , , , ,25748 Yuko-Yukpa 0, , , , , , , , , , , , , ,8944 Yuracaré 0, , , , , , , , , , , , , , ,27202 Zenu 0, , , , , , , , , , , , ,0586 0, ,23128 Zoro 0, , , , , , , , , , , , , , ,01109 Apêndice XXVIII F: Matriz de Fst entre populações da região centro norte da América do Sul (biom42) com cinco haplogrupos de DNA mitocondrial Waunana Wayuu Wayuu Wiwa Wounan Xavante Yamamadi Yaminawa Yanomama Yanomama Yanomami Yanomamö Yuko- Yuracaré Zenu Zoro Colombia Brasil Yukpa Waunana 0 Wayuu 0, Wayuu Colombia 0, , Wiwa 0, , , Wounan 0, , , , Xavante 0, ,4933 0, , , Yamamadi 0, , , , , , Yaminawa 0,3985 0, , , , , , Yanomama 0, ,0209 0,0013 0, , , , , Yanomama Brasil 0, , , , , , , , , Yanomami 0, ,0767 0, , ,1317 0, , , , , Yanomamö -0, ,2041 0, , , , , , ,1115 0, , Yuko-Yukpa 0, , , , ,7385 0, , , , , , Yuracaré 0, , ,0041 0, , , , , , ,1822 0, , , Zenu 0, , , ,1693 0,0359 0, , , , , , , , , Zoro 0, , ,2626 0, , , , , , , , , , , , Apêndice XXVIII G: Matriz de Fst entre populações da região centro norte da América do Sul (biom42) com cinco haplogrupos de DNA mitocondrial
239 Figura esquemática da localização original dos apêndices XXIX e XXX A-L da matriz de DNA mitocondrial de das populações da América do Sul (biom74).
240 Ache Ancash Aonikenk Arara Arequipa Peru Assurini Assurini2 Atacameno Atacameno2 Atacamenos Awa-Guaja Aymara Aymara2 Baniwa Boruca Ache Ancash Aonikenk Arara Arequipa Peru Assurini Assurini2 Atacameno Atacameno2 Atacamenos Awa-Guaja Aymara Aymara2 Baniwa Boruca Bribi-Cabecar Cayapa Cayapa Ecuador Chimane Chimila Choroti Choroti2 Coreguaje Curripaco Embera Embera2 Embera3 Fueguian Argentina Gaviao Guahibo Guahibo-Sikuani Guambiano Guane- Butaregua Guatuso Guayabero Guaymi Huetar Huetar2 Huilliche Huilliche2 Huilliche Chile Huitoto Ignaciano Ijka-Arhuaco Ingano Colombia Kanamari Katuena Katukina Katukina2 Kaweskar Kaxinawa Kayapo Kogui Kraho Kuna Kuna2 Kuna3 Macushi Makiritare Mapuche2 Mapuche (Chile) 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,25476 Apêndice XXIX A: Matriz de Fst entre populações da América do Sul (biom74) com quatro haplogrupos de DNA mitocondrial
241 Mapuche Anecon Grande Ache Ancash Aonikenk Arara Arequipa Peru Assurini Assurini2 Atacameno Atacameno2 Atacamenos Awa-Guaja Aymara Aymara2 Baniwa 0, , , , , , , , , , , , , , ,08419 Boruca 242 Mapuche Argentina Marubo Marubo Brasil Mataco (Chaco) Mataco (Salta) Mataco (Wichi) Argentina Mataco (Wichi) Formosa Moseten Bolivia Movima Bolívia Murui-Muinane Ngobe Nukak Paez Parakana Pasto Pehuenche Butalelbum Pehuenche Trapa Trapa Peneunche Chile 2 Piaroa Piaroa Colombia Pilaga Formosa Poturujara (Zoe) (Arequipa) (Tayacaja) Bolivia / Nmatsiquenga (San Martin) Selknam Siona Tayacaja Peru Tehuelche Argentina Teribe Teribe2 Tikuna Tikuna Brasil Tikuna Colombia Toba Toba (chaco) Toba (Formosa) Tucano Tule-Cuna Waiapi Wapishana Brasil Waunana Wayuu Wayuu Colombia Wiwa Wounan Xavante Yaghan Chile Yamamadi Yamana (Yaghan) Yaminawa Yanomama Yanomama Brasil Yanomami Yanomamo Yuko-Yukpa Yuracare Zenu Zoro 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,37059 Apêndice XXIX B: Matriz de Fst entre populações da América do Sul (biom74) com quatro haplogrupos de DNA mitocondrial
242 243 Bribi-Cabecar Cayapa Cayapa Chimane Chimila Choroti Choroti2 Coreguaje Curripaco Embera Embera2 Embera3 Fueguian Gaviao Ecuador Argentina Guahibo Bribi-Cabecar 0,00000 Cayapa 0, ,00000 Cayapa Ecuador 0, , ,00000 Chimane -0, , , ,00000 Chimila 0, , , , ,00000 Choroti 0, , , , , ,00000 Choroti2 0, , , , , , ,00000 Coreguaje 0, , , , , , , ,00000 Curripaco -0, , , , , , , , ,00000 Embera 0, , , , , , , , , ,00000 Embera2-0, , , , , , , , , , ,00000 Embera3 0, , , , , , , , , , , ,00000 Fueguian Argentina 0, , , , , , , , , , , , ,00000 Gaviao 0, , , , , , , , , , , , , ,00000 Guahibo 0, , , , , , , , , , , , , , ,00000 Guahibo- Sikuani 0, , , , , , , , , , , , , , ,19053 Guambiano 0, , , , , , , , , , , , , , ,20555 Guane- Butaregua 0, , , , , , , , , , , , , , ,41389 Guatuso 0, , , , , , , , , , , , , , ,27765 Guayabero 0, , , , , , , , , , , , , , ,10367 Guaymi -0, , , , , , , , , , , , , , ,23548 Huetar 0, , , , , , , , , , , , , , ,24308 Huetar2 0, , , , , , , , , , , , , , ,24503 Huilliche 0, , , , , , , , , , , , , , ,31345 Huilliche2 0, , , , , , , , , , , , , , ,30136 Huilliche Chile 0, , , , , , , , , , , , , , ,31944 Huitoto 0, , , , , , , , , , , , , , ,19428 Ignaciano 0, , , , , , , , , , , , , , ,12785 Ijka-Arhuaco 0, , , , , , , , , , , , , , ,18843 Ingano Colombia 0, , , , , , , , , , , , , , ,18866 Kanamari 0, , , , , , , , , , , , , , ,06678 Katuena 0, , , , , , , , , , , , , , ,08576 Katukina 0, , , , , , , , , , , , , , ,17569 Katukina2 0, , , , , , , , , , , , , , ,24274 Kaweskar 0, , , , , , , , , , , , , , ,51380 Kaxinawa 0, , , , , , , , , , , , , , ,03005 Kayapo -0, , , , , , , , , , , , , , ,29180 Kogui 0, , , , , , , , , , , , , , ,00640 Kraho 0, , , , , , , , , , , , , , ,26358 Kuna 0, , , , , , , , , , , , , , ,36043 Kuna2 0, , , , , , , , , , , , , , ,26710 Kuna3 0, , , , , , , , , , , , , , ,28963 Macushi 0, , , , , , , , , , , , , , ,20614 Makiritare 0, , , , , , , , , , , , , , ,05885 Mapuche2 0, , , , , , , , , , , , , , ,21416 Mapuche (Chile) 0, , , , , , , , , , , , , , ,20674 Apêndice XXIX C: Matriz de Fst entre populações da América do Sul (biom74) com quatro haplogrupos de DNA mitocondrial
243 Mapuche Anecon Grande Mapuche Argentina Bribi-Cabecar Cayapa Cayapa Chimane Chimila Choroti Choroti2 Coreguaje Curripaco Embera Embera2 Embera3 Fueguian Gaviao Ecuador Argentina Guahibo 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,23856 Marubo 0, , , , , , , , , , , , , , ,21688 Marubo Brasil 0, , , , , , , , , , , , , , ,14015 Mataco (Chaco) Mataco (Salta) Mataco (Wichi) Argentina Mataco (Wichi) Formosa Moseten Bolivia Movima Bolívia Murui- Muinane 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,15289 Ngobe 0, , , , , , , , , , , , , , ,25739 Nukak 0, , , , , , , , , , , , , , ,24302 Paez 0, , , , , , , , , , , , , , ,00374 Parakana 0, , , , , , , , , , , , , , ,15434 Pasto -0, , , , , , , , , , , , , , ,21830 Pehuenche Butalelbum Pehuenche Trapa Trapa Peneunche Chile 2 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,27288 Piaroa 0, , , , , , , , , , , , , , ,10234 Piaroa Colombia Pilaga Formosa Poturujara (Zoe) 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,30348 (Arequipa) (Tayacaja) Bolivia / Nmatsiqueng a (San Martin) 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,37674 Selknam 0, , , , , , , , , , , , , , ,28031 Siona 0, , , , , , , , , , , , , , ,16499 Tayacaja Peru 0, , , , , , , , , , , , , , ,21767 Tehuelche Argentina 0, , , , , , , , , , , , , , ,34399 Teribe 0, , , , , , , , , , , , , , ,25852 Teribe2 0, , , , , , , , , , , , , , ,37612 Tikuna 0, , , , , , , , , , , , , , ,12279 Tikuna Brasil 0, , , , , , , , , , , , , , ,22791 Tikuna Colombia 0, , , , , , , , , , , , , , ,15678 Toba 0, , , , , , , , , , , , , , ,38670 Toba (chaco) 0, , , , , , , , , , , , , , ,32314 Toba (Formosa) 0, , , , , , , , , , , , , , ,26688 Tucano 0, , , , , , , , , , , , , , ,22404 Tule-Cuna 0, , , , , , , , , , , , , , ,08916 Waiapi 0, , , , , , , , , , , , , , ,27042 Wapishana Brasil 0, , , , , , , , , , , , , , ,43447 Waunana 0, , , , , , , , , , , , , , ,35892 Wayuu 0, , , , , , , , , , , , , , ,03391 Wayuu Colombia 0, , , , , , , , , , , , , , ,10501 Wiwa 0, , , , , , , , , , , , , , ,03930 Wounan 0, , , , , , , , , , , , , , ,02657 Xavante 0, , , , , , , , , , , , , , ,50908 Yaghan Chile 0, , , , , , , , , , , , , , ,30246 Yamamadi 0, , , , , , , , , , , , , , ,31462 Yamana (Yaghan) 0, , , , , , , , , , , , , , ,30285 Yaminawa 0, , , , , , , , , , , , , , ,00070 Yanomama 0, , , , , , , , , , , , , , ,09694 Yanomama Brasil 0, , , , , , , , , , , , , , ,20881 Yanomami 0, , , , , , , , , , , , , , ,28785 Yanomamo 0, , , , , , , , , , , , , , ,32178 Yuko-Yukpa 0, , , , , , , , , , , , , , ,76302 Yuracare 0, , , , , , , , , , , , , , ,10354 Zenu 0, , , , , , , , , , , , , , ,16640 Zoro 0, , , , , , , , , , , , , , ,32391 Apêndice XXIX D: Matriz de Fst entre populações da América do Sul (biom74) com quatro haplogrupos de DNA mitocondrial 244
244 245 Guahibo- Sikuani Guambiano Guane- Guatuso Guayabero Guaymi Huetar Huetar2 Huilliche Huilliche2 Huilliche Butaregua Chile Huitoto Ignaciano Ijka-Arhuaco Ingano Colombia Guahibo- Sikuani 0,00000 Guambiano 0, ,00000 Guane- Butaregua 0, , ,00000 Guatuso 0, , , ,00000 Guayabero 0, , , , ,00000 Guaymi 0, , , , , ,00000 Huetar 0, , , , , , ,00000 Huetar2 0, , , , , , , ,00000 Huilliche 0, , , , , , , , ,00000 Huilliche2 0, , , , , , , , , ,00000 Huilliche Chile 0, , , , , , , , , , ,00000 Huitoto 0, , , , , , , , , , , ,00000 Ignaciano 0, , , , , , , , , , , , ,00000 Ijka-Arhuaco -0, , , , , , , , , , , , , ,00000 Ingano Colombia 0, , , , , , , , , , , , , , ,00000 Kanamari 0, , , , , , , , , , , , , , ,03626 Katuena 0, , , , , , , , , , , , , , ,13414 Katukina 0, , , , , , , , , , , , , , ,18982 Katukina2 0, , , , , , , , , , , , , , ,33468 Kaweskar 0, , , , , , , , , , , , , , ,49313 Kaxinawa 0, , , , , , , , , , , , , , ,13124 Kayapo 0, , , , , , , , , , , , , , ,12816 Kogui 0, , , , , , , , , , , , , , ,20185 Kraho 0, , , , , , , , , , , , , , ,01444 Kuna 0, , , , , , , , , , , , , , ,56440 Kuna2 0, , , , , , , , , , , , , , ,32745 Kuna3 0, , , , , , , , , , , , , , ,39836 Macushi 0, , , , , , , , , , , , , , ,09668 Makiritare 0, , , , , , , , , , , , , , ,16573 Mapuche2 0, , , , , , , , , , , , , , ,06687 Mapuche (Chile) 0, , , , , , , , , , , , , , ,09394 Apêndice XXIX E: Matriz de Fst entre populações da América do Sul (biom74) com quatro haplogrupos de DNA mitocondrial
245 Mapuche Anecon Grande Mapuche Argentina Guahibo- Sikuani Guambiano Guane- Guatuso Guayabero Guaymi Huetar Huetar2 Huilliche Huilliche2 Huilliche Huitoto Butaregua Chile Ignaciano Ijka-Arhuaco Ingano Colombia 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,06577 Marubo 0, , , , , , , , , , , , , , ,11957 Marubo Brasil 0, , , , , , , , , , , , , , ,17399 Mataco (Chaco) Mataco (Salta) Mataco (Wichi) Argentina Mataco (Wichi) Formosa Moseten Bolivia Movima Bolívia Murui- Muinane 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,04982 Ngobe 0, , , , , , , , , , , , , , ,28159 Nukak 0, , , , , , , , , , , , , , ,17740 Paez 0, , , , , , , , , , , , , , ,20176 Parakana 0, , , , , , , , , , , , , , ,02236 Pasto 0, , , , , , , , , , , , , , ,21739 Pehuenche Butalelbum Pehuenche Trapa Trapa Peneunche Chile 2 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,22927 Piaroa 0, , , , , , , , , , , , , , ,08227 Piaroa Colombia Pilaga Formosa Poturujara (Zoe) 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,05591 (Arequipa) (Tayacaja) Bolivia / Nmatsiqueng a (San Martin) 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,11122 Selknam 0, , , , , , , , , , , , , , ,25827 Siona -0, , , , , , , , , , , , , , ,28557 Tayacaja Peru 0, , , , , , , , , , , , , , ,08719 Tehuelche Argentina 0, , , , , , , , , , , , , , ,22667 Teribe 0, , , , , , , , , , , , , , ,36641 Teribe2 0, , , , , , , , , , , , , , ,58958 Tikuna 0, , , , , , , , , , , , , , ,16175 Tikuna Brasil 0, , , , , , , , , , , , , , ,24753 Tikuna Colombia 0, , , , , , , , , , , , , , ,11021 Toba 0, , , , , , , , , , , , , , ,20398 Toba (chaco) 0, , , , , , , , , , , , , , ,09220 Toba (Formosa) 0, , , , , , , , , , , , , , ,13660 Tucano 0, , , , , , , , , , , , , , ,11817 Tule-Cuna 0, , , , , , , , , , , , , , ,10625 Waiapi 0, , , , , , , , , , , , , , ,43977 Wapishana Brasil 0, , , , , , , , , , , , , , ,30399 Waunana 0, , , , , , , , , , , , , , ,01988 Wayuu 0, , , , , , , , , , , , , , ,07384 Wayuu Colombia 0, , , , , , , , , , , , , , ,01640 Wiwa 0, , , , , , , , , , , , , , ,17889 Wounan 0, , , , , , , , , , , , , , ,04048 Xavante 0, , , , , , , , , , , , , , ,21555 Yaghan Chile 0, , , , , , , , , , , , , , ,28301 Yamamadi 0, , , , , , , , , , , , , , ,36893 Yamana (Yaghan) 0, , , , , , , , , , , , , , ,28490 Yaminawa 0, , , , , , , , , , , , , , ,22474 Yanomama 0, , , , , , , , , , , , , , ,01203 Yanomama Brasil 0, , , , , , , , , , , , , , ,11876 Yanomami 0, , , , , , , , , , , , , , ,31262 Yanomamo 0, , , , , , , , , , , , , , ,02276 Yuko-Yukpa 0, , , , , , , , , , , , , , ,61397 Yuracare 0, , , , , , , , , , , , , , ,04146 Zenu 0, , , , , , , , , , , , , , ,02564 Zoro 0, , , , , , , , , , , , , , ,30307 Apêndice XXIX F: Matriz de Fst entre populações da América do Sul (biom74) com quatro haplogrupos de DNA mitocondrial 246
246 247 Kanamari Katuena Katukina Katukina2 Kaweskar Kaxinawa Kayapo Kogui Kraho Kuna Kuna2 Kuna3 Macushi Makiritare Mapuche2 Mapuche (Chile) Kanamari 0,00000 Katuena 0, ,00000 Katukina 0, , ,00000 Katukina2 0, , , ,00000 Kaweskar 0, , , , ,00000 Kaxinawa 0, , , , , ,00000 Kayapo 0, , , , , , ,00000 Kogui 0, , , , , , , ,00000 Kraho 0, , , , , , , , ,00000 Kuna 0, , , , , , , , , ,00000 Kuna2 0, , , , , , , , , , ,00000 Kuna3 0, , , , , , , , , , , ,00000 Macushi 0, , , , , , , , , , , , ,00000 Makiritare 0, , , , , , , , , , , , , ,00000 Mapuche2 0, , , , , , , , , , , , , , ,00000 Mapuche (Chile) 0, , , , , , , , , , , , , , , ,00000 Apêndice XXIX G: Matriz de Fst entre populações da América do Sul (biom74) com quatro haplogrupos de DNA mitocondrial
247 Mapuche Anecon Grande Mapuche Argentina Kanamari Katuena Katukina Katukina2 Kaweskar Kaxinawa Kayapo Kogui Kraho Kuna Kuna2 Kuna3 Macushi Makiritare Mapuche2 Mapuche (Chile) 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,01909 Marubo 0, , , , , , , , , , , , , , , ,02861 Marubo Brasil 0, , , , , , , , , , , , , , , ,00792 Mataco (Chaco) Mataco (Salta) Mataco (Wichi) Argentina Mataco (Wichi) Formosa Moseten Bolivia Movima Bolívia Murui- Muinane 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,03465 Ngobe 0, , , , , , , , , , , , , , , ,36433 Nukak 0, , , , , , , , , , , , , , , ,16493 Paez 0, , , , , , , , , , , , , , , ,23531 Parakana -0, , , , , , , , , , , , , , , ,04827 Pasto 0, , , , , , , , , , , , , , , ,30502 Pehuenche Butalelbum Pehuenche Trapa Trapa Peneunche Chile 2 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,01836 Piaroa -0, , , , , , , , , , , , , , , ,01351 Piaroa Colombia Pilaga Formosa Poturujara (Zoe) 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,09694 (Arequipa) (Tayacaja) Bolivia / Nmatsiqueng a (San Martin) 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,13288 Selknam 0, , , , , , , , , , , , , , , ,01420 Siona 0, , , , , , , , , , , , , , , ,33412 Tayacaja Peru 0, , , , , , , , , , , , , , , ,05719 Tehuelche Argentina 0, , , , , , , , , , , , , , , ,02749 Teribe 0, , , , , , , , , , , , , , , ,40592 Teribe2 0, , , , , , , , , , , , , , , ,55325 Tikuna 0, , , , , , , , , , , , , , , ,03584 Tikuna Brasil 0, , , , , , , , , , , , , , , ,04438 Tikuna Colombia 0, , , , , , , , , , , , , , , ,01135 Toba 0, , , , , , , , , , , , , , , ,03921 Toba (chaco) 0, , , , , , , , , , , , , , , ,10339 Toba (Formosa) 0, , , , , , , , , , , , , , , ,09918 Tucano 0, , , , , , , , , , , , , , , ,03480 Tule-Cuna 0, , , , , , , , , , , , , , , ,20071 Waiapi 0, , , , , , , , , , , , , , , ,40146 Wapishana Brasil 0, , , , , , , , , , , , , , , ,09857 Waunana 0, , , , , , , , , , , , , , , ,14815 Wayuu 0, , , , , , , , , , , , , , , ,10257 Wayuu Colombia -0, , , , , , , , , , , , , , , ,09411 Wiwa 0, , , , , , , , , , , , , , , ,14825 Wounan -0, , , , , , , , , , , , , , , ,08206 Xavante 0, , , , , , , , , , , , , , , ,36611 Yaghan Chile 0, , , , , , , , , , , , , , , ,03430 Yamamadi 0, , , , , , , , , , , , , , , ,28213 Yamana (Yaghan) 0, , , , , , , , , , , , , , , ,03668 Yaminawa 0, , , , , , , , , , , , , , , ,22081 Yanomama 0, , , , , , , , , , , , , , , ,06120 Yanomama Brasil 0, , , , , , , , , , , , , , , ,01197 Yanomami 0, , , , , , , , , , , , , , , ,20628 Yanomamo 0, , , , , , , , , , , , , , , ,11186 Yuko-Yukpa 0, , , , , , , , , , , , , , , ,65151 Yuracare -0, , , , , , , , , , , , , , , ,13791 Zenu 0, , , , , , , , , , , , , , , ,07668 Zoro 0, , , , , , , , , , , , , , , ,10531 Apêndice XXIX H: Matriz de Fst entre populações da América do Sul (biom74) com quatro haplogrupos de DNA mitocondrial 248
248 Mapuche Anecon Grande Mapuche Argentina Mapuche Anecon Grande 0,00000 Mapuche Marubo Argentina -0, ,00000 Marubo 0, , ,00000 Marubo Brasil 0, , , ,00000 Mataco (Chaco) Mataco (Salta) Mataco (Wichi) Argentina Mataco (Wichi) Formosa Moseten Bolivia Movima Bolívia Murui- Muinane Marubo Brasil Mataco Mataco (Chaco) (Salta) 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,00000 Mataco Mataco Moseten Movima Murui- Ngobe Nukak Paez Parakana Pasto (Wichi) (Wichi) Bolivia Bolivia Muinane Argentina Formosa 0, , , , , , , , , , ,00000 Ngobe 0, , , , , , , , , , , ,00000 Nukak 0, , , , , , , , , , , , ,00000 Paez 0, , , , , , , , , , , , , ,00000 Parakana -0, , , , , , , , , , , , , , ,00000 Pasto 0, , , , , , , , , , , , , , , ,00000 Pehuenche Butalelbum Pehuenche Trapa Trapa Peneunche Chile 2 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,39717 Piaroa 0, , , , , , , , , , , , , , , ,13573 Piaroa Colombia Pilaga Formosa Poturujara (Zoe) 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,21258 (Arequipa) (Tayacaja) Bolivia / Nmatsiqueng a (San Martin) 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,24503 Selknam 0, , , , , , , , , , , , , , , ,47522 Siona 0, , , , , , , , , , , , , , , ,05531 Tayacaja Peru -0, , , , , , , , , , , , , , , ,13431 Tehuelche Argentina 0, , , , , , , , , , , , , , , ,38450 Teribe 0, , , , , , , , , , , , , , , ,03563 Teribe2 0, , , , , , , , , , , , , , , ,40100 Tikuna 0, , , , , , , , , , , , , , , ,23066 Tikuna Brasil 0, , , , , , , , , , , , , , , ,33609 Tikuna Colombia 0, , , , , , , , , , , , , , , ,26615 Toba 0, , , , , , , , , , , , , , , ,35938 Toba (chaco) -0, , , , , , , , , , , , , , , ,19089 Toba (Formosa) 0, , , , , , , , , , , , , , , ,11992 Tucano 0, , , , , , , , , , , , , , , ,36928 Tule-Cuna 0, , , , , , , , , , , , , , , ,01654 Waiapi 0, , , , , , , , , , , , , , , ,12654 Wapishana Brasil 0, , , , , , , , , , , , , , , ,43660 Waunana 0, , , , , , , , , , , , , , , ,34889 Wayuu 0, , , , , , , , , , , , , , , ,29901 Wayuu Colombia 0, , , , , , , , , , , , , , , ,15076 Wiwa 0, , , , , , , , , , , , , , , ,44124 Wounan 0, , , , , , , , , , , , , , , ,19545 Xavante 0, , , , , , , , , , , , , , , ,40855 Yaghan Chile 0, , , , , , , , , , , , , , , ,48398 Yamamadi 0, , , , , , , , , , , , , , , ,76190 Yamana (Yaghan) 0, , , , , , , , , , , , , , , ,48522 Yaminawa 0, , , , , , , , , , , , , , , ,11138 Yanomama 0, , , , , , , , , , , , , , , ,27137 Yanomama Brasil 0, , , , , , , , , , , , , , , ,39251 Yanomami 0, , , , , , , , , , , , , , , ,63696 Yanomamo 0, , , , , , , , , , , , , , , ,32316 Yuko-Yukpa 0, , , , , , , , , , , , , , , ,91300 Yuracare 0, , , , , , , , , , , , , , , ,01621 Zenu 0, , , , , , , , , , , , , , , ,15283 Zoro 0, , , , , , , , , , , , , , , ,33595 Apêndice XXIX I: Matriz de Fst entre populações da América do Sul (biom74) com quatro haplogrupos de DNA mitocondrial 249
249 250 Pehuenche Butalelbum Pehuenche Trapa Trapa Peneunche Chile 2 Pehuenche Butalelbum 0,00000 Pehuenche Peneunche Trapa Trapa Chile 2 0, , , , ,00000 Piaroa Piaroa 0, , , ,00000 Piaroa Colombia Pilaga Formosa Poturujara (Zoe) 0, , , , ,00000 Piaroa Pilaga Poturujara Colombia Formosa (Zoe) 0, , , , , , , , , , , , , , , , , , , , ,00000 (Arequipa) (Tayacaja) Bolivia / Nmatsiqueng a (San Martin) 0, , , , , , , , ,00000 (Arequipa) (Tayacaja) Bolivia 0, , , , , , , , , , , , , , , , , , , , ,00000 / Nmatsiquenga (San Martin) 0, , , , , , , , , , , ,00000 Selknam Siona Selknam 0, , , , , , , , , , , , ,00000 Siona 0, , , , , , , , , , , , , ,00000 Tayacaja Peru Tayacaja Peru 0, , , , , , , , , , , , , , ,00000 Tehuelche Argentina 0, , , , , , , , , , , , , , ,06817 Teribe 0, , , , , , , , , , , , , , ,24922 Teribe2 0, , , , , , , , , , , , , , ,41769 Tikuna 0, , , , , , , , , , , , , , ,07114 Tikuna Brasil 0, , , , , , , , , , , , , , ,09722 Tikuna Colombia 0, , , , , , , , , , , , , , ,05465 Toba 0, , , , , , , , , , , , , , ,01292 Toba (chaco) 0, , , , , , , , , , , , , , ,00086 Toba (Formosa) 0, , , , , , , , , , , , , , ,01877 Tucano 0, , , , , , , , , , , , , , ,08548 Tule-Cuna 0, , , , , , , , , , , , , , ,09859 Waiapi 0, , , , , , , , , , , , , , ,26390 Wapishana Brasil 0, , , , , , , , , , , , , , ,07805 Waunana 0, , , , , , , , , , , , , , ,12235 Wayuu 0, , , , , , , , , , , , , , ,17580 Wayuu Colombia 0, , , , , , , , , , , , , , ,08147 Wiwa 0, , , , , , , , , , , , , , ,25667 Wounan 0, , , , , , , , , , , , , , ,11582 Xavante 0, , , , , , , , , , , , , , ,21555 Yaghan Chile 0, , , , , , , , , , , , , , ,16861 Yamamadi 0, , , , , , , , , , , , , , ,42114 Yamana (Yaghan) 0, , , , , , , , , , , , , , ,17178 Yaminawa 0, , , , , , , , , , , , , , ,17391 Yanomama 0, , , , , , , , , , , , , , ,12696 Yanomama Brasil 0, , , , , , , , , , , , , , ,12548 Yanomami 0, , , , , , , , , , , , , , ,38014 Yanomamo 0, , , , , , , , , , , , , , ,10775 Yuko-Yukpa 0, , , , , , , , , , , , , , ,49938 Yuracare 0, , , , , , , , , , , , , , ,04930 Zenu 0, , , , , , , , , , , , , , ,04564 Zoro 0, , , , , , , , , , , , , , ,08405 Apêndice XXIX J: Matriz de Fst entre populações da América do Sul (biom74) com quatro haplogrupos de DNA mitocondrial
250 251 Tehuelche Argentina Tehuelche Argentina 0,00000 Teribe 0, ,00000 Teribe2 0, , ,00000 Tikuna 0, , , ,00000 Teribe Teribe2 Tikuna Tikuna Brasil Tikuna Toba Colombia Tikuna Brasil 0, , , , ,00000 Tikuna Colombia 0, , , , , ,00000 Toba -0, , , , , , ,00000 Toba (chaco) 0, , , , , , , ,00000 Toba (Formosa) 0, , , , , , , , ,00000 Toba (chaco) Toba Tucano Tule-Cuna Waiapi Wapishana (Formosa) Brasil Tucano 0, , , , , , , , , ,00000 Tule-Cuna 0, , , , , , , , , , ,00000 Waiapi 0, , , , , , , , , , , ,00000 Wapishana Brasil -0, , , , , , , , , , , , ,00000 Waunana Wayuu Waunana 0, , , , , , , , , , , , , ,00000 Wayuu 0, , , , , , , , , , , , , , ,00000 Wayuu Colombia 0, , , , , , , , , , , , , , ,03107 Wiwa 0, , , , , , , , , , , , , , ,04823 Wounan 0, , , , , , , , , , , , , , ,02762 Xavante 0, , , , , , , , , , , , , , ,49330 Yaghan Chile 0, , , , , , , , , , , , , , ,22394 Yamamadi 0, , , , , , , , , , , , , , ,18963 Yamana (Yaghan) 0, , , , , , , , , , , , , , ,22471 Yaminawa 0, , , , , , , , , , , , , , ,11781 Yanomama 0, , , , , , , , , , , , , , ,02090 Yanomama Brasil 0, , , , , , , , , , , , , , ,07361 Yanomami 0, , , , , , , , , , , , , , ,12657 Yanomamo 0, , , , , , , , , , , , , , ,20410 Yuko-Yukpa 0, , , , , , , , , , , , , , ,84463 Yuracare 0, , , , , , , , , , , , , , ,10092 Zenu 0, , , , , , , , , , , , , , ,07980 Zoro 0, , , , , , , , , , , , , , ,31224 Apêndice XXIX K: Matriz de Fst entre populações da América do Sul (biom74) com quatro haplogrupos de DNA mitocondrial Wayuu Colombia Wayuu Wiwa Colombia 0,00000 Wiwa 0, ,00000 Wounan 0, , ,00000 Xavante 0, , , ,00000 Yaghan Chile 0, , , , ,00000 Wounan Xavante Yaghan Chile Yamamadi Yamana Yaminawa Yanomama Yanomama (Yaghan) Brasil Yamamadi 0, , , , , ,00000 Yamana (Yaghan) 0, , , , , , ,00000 Yaminawa 0, , , , , , , ,00000 Yanomama 0, , , , , , , , ,00000 Yanomama Brasil 0, , , , , , , , , ,00000 Yanomami 0, , , , , , , , , , ,00000 Yanomamo 0, , , , , , , , , , , ,00000 Yanomami Yanomamo Yuko-Yukpa Yuracare Zenu Yuko-Yukpa 0, , , , , , , , , , , , ,00000 Yuracare 0, , , , , , , , , , , , , ,00000 Zenu -0, , , , , , , , , , , , , , ,00000 Zoro 0, , , , , , , , , , , , , , , ,00000 Apêndice XXIX L: Matriz de Fst entre populações da América do Sul (biom74) com quatro haplogrupos de DNA mitocondrial Zoro
251 252 Ache Ancash Aonikenk Arara Arequipa Peru Assurini Assurini2 Atacameno Atacameno2 Atacamenos Awa-Guaja Aymara Aymara2 Baniwa Boruca Ache Ancash Aonikenk Arara Arequipa Peru Assurini Assurini2 Atacameno Atacameno2 Atacamenos Awa-Guaja Aymara Aymara2 Baniwa Boruca Bribi-Cabecar Cayapa Cayapa Ecuador Chimane Chimila Choroti Choroti2 Coreguaje Curripaco Embera Embera2 Embera3 Fueguian Argentina Gaviao Guahibo Guahibo-Sikuani Guambiano Guane-Butaregua Guatuso Guayabero Guaymi Huetar Huetar2 Huilliche Huilliche2 Huilliche Chile Huitoto Ignaciano Ijka-Arhuaco Ingano Colombia 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,11442 Kanamari Katuena Katukina Katukina2 Kaweskar Kaxinawa Kayapo Kogui Kraho Kuna Kuna2 Kuna3 Macushi Makiritare Mapuche2 Mapuche (Chile) 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,25476 Apêndice XXX A: Matriz de Fst entre populações da América do Sul (biom74) com cinco haplogrupos de DNA mitocondrial
252 Ache Ancash Aonikenk Arara Arequipa Peru Assurini Assurini2 Atacameno Atacameno2 Atacamenos Awa-Guaja Aymara Aymara2 Baniwa Boruca Mapuche Anecon Grande Mapuche Argentina Marubo Marubo Brasil Mataco (Chaco) Mataco (Salta) Mataco (Wichi) Argentina Mataco (Wichi) Formosa Moseten Bolivia Movima Bolívia Murui-Muinane Ngobe Nukak Paez Parakana Pasto Pehuenche Butalelbum Pehuenche Trapa Trapa Peneunche Chile 2 Piaroa Piaroa Colombia Pilaga Formosa Poturujara (Zoe) (Arequipa) (Tayacaja) Bolivia / Nmatsiquenga (San Martin) Selknam Siona Tayacaja Peru Tehuelche Argentina Teribe Teribe2 Tikuna Tikuna Brasil Tikuna Colombia Toba Toba (chaco) Toba (Formosa) Tucano Tule-Cuna Waiapi Wapishana Brasil Waunana Wayuu Wayuu Colombia Wiwa Wounan Xavante Yaghan Chile Yamamadi Yamana (Yaghan) Yaminawa Yanomama Yanomama Brasil 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , Yanomami Yanomamo Yuko-Yukpa Yuracare Zenu Zoro 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,37059 Apêndice XXX B: Matriz de Fst entre populações da América do Sul (biom74) com cinco haplogrupos de DNA mitocondrial
253 254 Bribi-Cabecar Cayapa Cayapa Chimane Chimila Choroti Choroti2 Coreguaje Curripaco Embera Embera2 Embera3 Fueguian Gaviao Ecuador Argentina Guahibo Bribi-Cabecar 0,00000 Cayapa 0, ,00000 Cayapa Ecuador 0, , ,00000 Chimane -0, , , ,00000 Chimila 0, , , , ,00000 Choroti 0, , , , , ,00000 Choroti2 0, , , , , , ,00000 Coreguaje 0, , , , , , , ,00000 Curripaco -0, , , , , , , , ,00000 Embera 0, , , , , , , , , ,00000 Embera2 0, , , , , , , , , , ,00000 Embera3 0, , , , , , , , , , , ,00000 Fueguian Argentina 0, , , , , , , , , , , , ,00000 Gaviao 0, , , , , , , , , , , , , ,00000 Guahibo 0, , , , , , , , , , , , , , ,00000 Guahibo-Sikuani 0, , , , , , , , , , , , , , ,16331 Guambiano 0, , , , , , , , , , , , , , ,20555 Guane-Butaregua 0, , , , , , , , , , , , , , ,41389 Guatuso 0, , , , , , , , , , , , , , ,27765 Guayabero 0, , , , , , , , , , , , , , ,11738 Guaymi -0, , , , , , , , , , , , , , ,23548 Huetar 0, , , , , , , , , , , , , , ,24308 Huetar2 0, , , , , , , , , , , , , , ,24503 Huilliche 0, , , , , , , , , , , , , , ,31345 Huilliche2 0, , , , , , , , , , , , , , ,30136 Huilliche Chile 0, , , , , , , , , , , , , , ,31944 Huitoto 0, , , , , , , , , , , , , , ,19428 Ignaciano 0, , , , , , , , , , , , , , ,12531 Ijka-Arhuaco 0, , , , , , , , , , , , , , ,18843 Ingano Colombia 0, , , , , , , , , , , , , , ,18300 Kanamari 0, , , , , , , , , , , , , , ,06678 Katuena 0, , , , , , , , , , , , , , ,08576 Katukina 0, , , , , , , , , , , , , , ,16839 Katukina2 0, , , , , , , , , , , , , , ,24274 Kaweskar 0, , , , , , , , , , , , , , ,51380 Kaxinawa 0, , , , , , , , , , , , , , ,03005 Kayapo -0, , , , , , , , , , , , , , ,29180 Kogui 0, , , , , , , , , , , , , , ,00640 Kraho 0, , , , , , , , , , , , , , ,26358 Kuna 0, , , , , , , , , , , , , , ,36043 Kuna2 0, , , , , , , , , , , , , , ,26710 Kuna3 0, , , , , , , , , , , , , , ,28963 Macushi 0, , , , , , , , , , , , , , ,20614 Makiritare 0, , , , , , , , , , , , , , ,05885 Mapuche2 0, , , , , , , , , , , , , , ,20693 Mapuche (Chile) 0, , , , , , , , , , , , , , ,20674 Apêndice XXX C: Matriz de Fst entre populações da América do Sul (biom74) com cinco haplogrupos de DNA mitocondrial
254 Mapuche Anecon Grande Mapuche Argentina Bribi-Cabecar Cayapa Cayapa Chimane Chimila Choroti Choroti2 Coreguaje Curripaco Embera Embera2 Embera3 Fueguian Gaviao Ecuador Argentina Guahibo 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,22665 Marubo 0, , , , , , , , , , , , , , ,21688 Marubo Brasil 0, , , , , , , , , , , , , , ,14015 Mataco (Chaco) 0, , , , , , , , , , , , , , ,39812 Mataco (Salta) 0, , , , , , , , , , , , , , ,41139 Mataco (Wichi) Argentina Mataco (Wichi) Formosa 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,29698 Moseten Bolivia -0, , , , , , , , , , , , , , ,30485 Movima Bolívia 0, , , , , , , , , , , , , , ,13416 Murui-Muinane 0, , , , , , , , , , , , , , ,15146 Ngobe 0, , , , , , , , , , , , , , ,25739 Nukak 0, , , , , , , , , , , , , , ,24302 Paez 0, , , , , , , , , , , , , , ,00374 Parakana 0, , , , , , , , , , , , , , ,15434 Pasto -0, , , , , , , , , , , , , , ,21830 Pehuenche Butalelbum Pehuenche Trapa Trapa 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,25502 Peneunche Chile 2 0, , , , , , , , , , , , , , ,27288 Piaroa 0, , , , , , , , , , , , , , ,10234 Piaroa Colombia 0, , , , , , , , , , , , , , ,18391 Pilaga Formosa 0, , , , , , , , , , , , , , ,24625 Poturujara (Zoe) 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,29130 (Arequipa) (Tayacaja) 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,21281 Bolivia 0, , , , , , , , , , , , , , ,42899 / Nmatsiquenga (San Martin) 0, , , , , , , , , , , , , , ,36320 Selknam 0, , , , , , , , , , , , , , ,26245 Siona 0, , , , , , , , , , , , , , ,16499 Tayacaja Peru 0, , , , , , , , , , , , , , ,21281 Tehuelche Argentina 0, , , , , , , , , , , , , , ,34399 Teribe 0, , , , , , , , , , , , , , ,25852 Teribe2 0, , , , , , , , , , , , , , ,37612 Tikuna 0, , , , , , , , , , , , , , ,12279 Tikuna Brasil 0, , , , , , , , , , , , , , ,22791 Tikuna Colombia 0, , , , , , , , , , , , , , ,15678 Toba 0, , , , , , , , , , , , , , ,38670 Toba (chaco) 0, , , , , , , , , , , , , , ,30740 Toba (Formosa) 0, , , , , , , , , , , , , , ,26688 Tucano 0, , , , , , , , , , , , , , ,22404 Tule-Cuna 0, , , , , , , , , , , , , , ,08775 Waiapi 0, , , , , , , , , , , , , , ,27042 Wapishana Brasil 0, , , , , , , , , , , , , , ,43447 Waunana 0, , , , , , , , , , , , , , ,35892 Wayuu 0, , , , , , , , , , , , , , ,03391 Wayuu Colombia 0, , , , , , , , , , , , , , ,10380 Wiwa 0, , , , , , , , , , , , , , ,03930 Wounan 0, , , , , , , , , , , , , , ,02657 Xavante 0, , , , , , , , , , , , , , ,50908 Yaghan Chile 0, , , , , , , , , , , , , , ,27792 Yamamadi 0, , , , , , , , , , , , , , ,31462 Yamana (Yaghan) 0, , , , , , , , , , , , , , ,30285 Yaminawa 0, , , , , , , , , , , , , , ,00070 Yanomama 0, , , , , , , , , , , , , , ,09694 Yanomama Brasil 0, , , , , , , , , , , , , , ,20881 Yanomami 0, , , , , , , , , , , , , , ,22739 Yanomamo 0, , , , , , , , , , , , , , ,32178 Yuko-Yukpa 0, , , , , , , , , , , , , , ,76302 Yuracare 0, , , , , , , , , , , , , , ,10298 Zenu 0, , , , , , , , , , , , , , ,16179 Zoro 0, , , , , , , , , , , , , , ,32391 Apêndice XXX D: Matriz de Fst entre populações da América do Sul (biom74) com cinco haplogrupos de DNA mitocondrial 255
255 256 Guahibo- Sikuani Guambiano Guane- Guatuso Guayabero Guaymi Huetar Huetar2 Huilliche Huilliche2 Huilliche Butaregua Chile Huitoto Ignaciano Ijka-Arhuaco Ingano Colombia Guahibo-Sikuani 0,00000 Guambiano 0, ,00000 Guane- Butaregua 0, , ,00000 Guatuso 0, , , ,00000 Guayabero -0, , , , ,00000 Guaymi 0, , , , , ,00000 Huetar 0, , , , , , ,00000 Huetar2 0, , , , , , , ,00000 Huilliche 0, , , , , , , , ,00000 Huilliche2 0, , , , , , , , , ,00000 Huilliche Chile 0, , , , , , , , , , ,00000 Huitoto 0, , , , , , , , , , , ,00000 Ignaciano 0, , , , , , , , , , , , ,00000 Ijka-Arhuaco 0, , , , , , , , , , , , , ,00000 Ingano Colombia 0, , , , , , , , , , , , , , ,00000 Kanamari 0, , , , , , , , , , , , , , ,03117 Katuena 0, , , , , , , , , , , , , , ,12564 Katukina 0, , , , , , , , , , , , , , ,15884 Katukina2 0, , , , , , , , , , , , , , ,32189 Kaweskar 0, , , , , , , , , , , , , , ,47739 Kaxinawa 0, , , , , , , , , , , , , , ,12432 Kayapo 0, , , , , , , , , , , , , , ,12214 Kogui 0, , , , , , , , , , , , , , ,19522 Kraho 0, , , , , , , , , , , , , , ,01346 Kuna 0, , , , , , , , , , , , , , ,54668 Kuna2 0, , , , , , , , , , , , , , ,31904 Kuna3 0, , , , , , , , , , , , , , ,38931 Macushi 0, , , , , , , , , , , , , , ,08737 Makiritare 0, , , , , , , , , , , , , , ,15754 Mapuche2 0, , , , , , , , , , , , , , ,05906 Mapuche (Chile) 0, , , , , , , , , , , , , , ,08871 Apêndice XXX E: Matriz de Fst entre populações da América do Sul (biom74) com cinco haplogrupos de DNA mitocondrial
256 Guahibo- Sikuani Guambiano Guane- Guatuso Guayabero Guaymi Huetar Huetar2 Huilliche Huilliche2 Huilliche Butaregua Chile Huitoto Ignaciano Ijka-Arhuaco Ingano Colombia Mapuche Anecon Grande Mapuche Argentina 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,05715 Marubo 0, , , , , , , , , , , , , , ,11270 Marubo Brasil 0, , , , , , , , , , , , , , ,16406 Mataco (Chaco) 0, , , , , , , , , , , , , , ,23581 Mataco (Salta) 0, , , , , , , , , , , , , , ,12854 Mataco (Wichi) Argentina Mataco (Wichi) Formosa 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,02160 Moseten Bolivia 0, , , , , , , , , , , , , , ,11235 Movima Bolívia 0, , , , , , , , , , , , , , ,12723 Murui-Muinane 0, , , , , , , , , , , , , , ,04185 Ngobe 0, , , , , , , , , , , , , , ,27334 Nukak 0, , , , , , , , , , , , , , ,17371 Paez 0, , , , , , , , , , , , , , ,19408 Parakana 0, , , , , , , , , , , , , , ,01737 Pasto 0, , , , , , , , , , , , , , ,20661 Pehuenche Butalelbum Pehuenche Trapa Trapa Peneunche Chile 2 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,22288 Piaroa 0, , , , , , , , , , , , , , ,07463 Piaroa Colombia 0, , , , , , , , , , , , , , ,26032 Pilaga Formosa 0, , , , , , , , , , , , , , ,04707 Poturujara (Zoe) 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,04802 (Arequipa) (Tayacaja) 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,07808 Bolivia 0, , , , , , , , , , , , , , ,11281 / Nmatsiquenga (San Martin) 0, , , , , , , , , , , , , , ,09585 Selknam 0, , , , , , , , , , , , , , ,22063 Siona 0, , , , , , , , , , , , , , ,27348 Tayacaja Peru 0, , , , , , , , , , , , , , ,07808 Tehuelche Argentina 0, , , , , , , , , , , , , , ,21705 Teribe 0, , , , , , , , , , , , , , ,35372 Teribe2 0, , , , , , , , , , , , , , ,57168 Tikuna 0, , , , , , , , , , , , , , ,15546 Tikuna Brasil 0, , , , , , , , , , , , , , ,23694 Tikuna Colombia 0, , , , , , , , , , , , , , ,10423 Toba 0, , , , , , , , , , , , , , ,19200 Toba (chaco) 0, , , , , , , , , , , , , , ,07800 Toba (Formosa) 0, , , , , , , , , , , , , , ,12860 Tucano 0, , , , , , , , , , , , , , ,11100 Tule-Cuna 0, , , , , , , , , , , , , , ,09413 Waiapi 0, , , , , , , , , , , , , , ,42526 Wapishana Brasil 0, , , , , , , , , , , , , , ,29045 Waunana 0, , , , , , , , , , , , , , ,02224 Wayuu 0, , , , , , , , , , , , , , ,07073 Wayuu Colombia 0, , , , , , , , , , , , , , ,01702 Wiwa 0, , , , , , , , , , , , , , ,17064 Wounan 0, , , , , , , , , , , , , , ,03784 Xavante 0, , , , , , , , , , , , , , ,21151 Yaghan Chile 0, , , , , , , , , , , , , , ,23698 Yamamadi 0, , , , , , , , , , , , , , ,35679 Yamana (Yaghan) 0, , , , , , , , , , , , , , ,27353 Yaminawa 0, , , , , , , , , , , , , , ,21425 Yanomama 0, , , , , , , , , , , , , , ,01153 Yanomama Brasil 0, , , , , , , , , , , , , , ,11296 Yanomami 0, , , , , , , , , , , , , , ,21977 Yanomamo 0, , , , , , , , , , , , , , ,02443 Yuko-Yukpa 0, , , , , , , , , , , , , , ,60080 Yuracare 0, , , , , , , , , , , , , , ,03492 Zenu 0, , , , , , , , , , , , , , ,02421 Zoro 0, , , , , , , , , , , , , , ,29148 Apêndice XXX F: Matriz de Fst entre populações da América do Sul (biom74) com cinco haplogrupos de DNA mitocondrial 257
257 258 Kanamari Katuena Katukina Katukina2 Kaweskar Kaxinawa Kayapo Kogui Kraho Kuna Kuna2 Kuna3 Macushi Makiritare Mapuche2 Mapuche (Chile) Kanamari 0,00000 Katuena 0, ,00000 Katukina 0, , ,00000 Katukina2 0, , , ,00000 Kaweskar 0, , , , ,00000 Kaxinawa 0, , , , , ,00000 Kayapo 0, , , , , , ,00000 Kogui 0, , , , , , , ,00000 Kraho 0, , , , , , , , ,00000 Kuna 0, , , , , , , , , ,00000 Kuna2 0, , , , , , , , , , ,00000 Kuna3 0, , , , , , , , , , , ,00000 Macushi 0, , , , , , , , , , , , ,00000 Makiritare 0, , , , , , , , , , , , , ,00000 Mapuche2 0, , , , , , , , , , , , , , ,00000 Mapuche (Chile) 0, , , , , , , , , , , , , , , ,00000 Apêndice XXX G: Matriz de Fst entre populações da América do Sul (biom74) com cinco haplogrupos de DNA mitocondrial
258 Mapuche Anecon Grande Mapuche Argentina Kanamari Katuena Katukina Katukina2 Kaweskar Kaxinawa Kayapo Kogui Kraho Kuna Kuna2 Kuna3 Macushi Makiritare Mapuche2 Mapuche (Chile) 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,02168 Marubo 0, , , , , , , , , , , , , , , ,02861 Marubo Brasil 0, , , , , , , , , , , , , , , ,00792 Mataco (Chaco) 0, , , , , , , , , , , , , , , ,12368 Mataco (Salta) 0, , , , , , , , , , , , , , , ,19076 Mataco (Wichi) Argentina Mataco (Wichi) Formosa 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,09808 Moseten Bolivia 0, , , , , , , , , , , , , , , ,26035 Movima Bolívia 0, , , , , , , , , , , , , , , ,03966 Murui-Muinane -0, , , , , , , , , , , , , , , ,02955 Ngobe 0, , , , , , , , , , , , , , , ,36433 Nukak 0, , , , , , , , , , , , , , , ,16493 Paez 0, , , , , , , , , , , , , , , ,23531 Parakana -0, , , , , , , , , , , , , , , ,04827 Pasto 0, , , , , , , , , , , , , , , ,30502 Pehuenche Butalelbum Pehuenche Trapa Trapa Peneunche Chile 2 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,01836 Piaroa -0, , , , , , , , , , , , , , , ,01351 Piaroa Colombia 0, , , , , , , , , , , , , , , ,16351 Pilaga Formosa 0, , , , , , , , , , , , , , , ,00511 Poturujara (Zoe) 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,09099 (Arequipa) (Tayacaja) 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,05595 Bolivia 0, , , , , , , , , , , , , , , ,28649 / Nmatsiquenga (San Martin) 0, , , , , , , , , , , , , , , ,12425 Selknam 0, , , , , , , , , , , , , , , ,00766 Siona 0, , , , , , , , , , , , , , , ,33412 Tayacaja Peru 0, , , , , , , , , , , , , , , ,05595 Tehuelche Argentina 0, , , , , , , , , , , , , , , ,02749 Teribe 0, , , , , , , , , , , , , , , ,40592 Teribe2 0, , , , , , , , , , , , , , , ,55325 Tikuna 0, , , , , , , , , , , , , , , ,03584 Tikuna Brasil 0, , , , , , , , , , , , , , , ,04438 Tikuna Colombia 0, , , , , , , , , , , , , , , ,01135 Toba 0, , , , , , , , , , , , , , , ,03921 Toba (chaco) 0, , , , , , , , , , , , , , , ,09622 Toba (Formosa) 0, , , , , , , , , , , , , , , ,09918 Tucano 0, , , , , , , , , , , , , , , ,03480 Tule-Cuna 0, , , , , , , , , , , , , , , ,19244 Waiapi 0, , , , , , , , , , , , , , , ,40146 Wapishana Brasil 0, , , , , , , , , , , , , , , ,09857 Waunana 0, , , , , , , , , , , , , , , ,14815 Wayuu 0, , , , , , , , , , , , , , , ,10257 Wayuu Colombia -0, , , , , , , , , , , , , , , ,09082 Wiwa 0, , , , , , , , , , , , , , , ,14825 Wounan -0, , , , , , , , , , , , , , , ,08206 Xavante 0, , , , , , , , , , , , , , , ,36611 Yaghan Chile 0, , , , , , , , , , , , , , , ,02563 Yamamadi 0, , , , , , , , , , , , , , , ,28213 Yamana (Yaghan) 0, , , , , , , , , , , , , , , ,03668 Yaminawa 0, , , , , , , , , , , , , , , ,22081 Yanomama 0, , , , , , , , , , , , , , , ,06120 Yanomama Brasil 0, , , , , , , , , , , , , , , ,01197 Yanomami 0, , , , , , , , , , , , , , , ,14640 Yanomamo 0, , , , , , , , , , , , , , , ,11186 Yuko-Yukpa 0, , , , , , , , , , , , , , , ,65151 Yuracare -0, , , , , , , , , , , , , , , ,13254 Zenu 0, , , , , , , , , , , , , , , ,07282 Zoro 0, , , , , , , , , , , , , , , ,10531 Apêndice XXX H: Matriz de Fst entre populações da América do Sul (biom74) com cinco haplogrupos de DNA mitocondrial 259
259 Mapuche Anecon Grande Mapuche Argentina Mapuche Anecon Grande 0,00000 Mapuche Marubo Argentina -0, ,00000 Marubo 0, , ,00000 Marubo Brasil 0, , , ,00000 Marubo Brasil Mataco Mataco (Chaco) (Salta) Mataco (Chaco) 0, , , , ,00000 Mataco (Salta) 0, , , , , ,00000 Mataco (Wichi) Argentina Mataco (Wichi) Formosa 0, , , , , , ,00000 Mataco (Wichi) Mataco (Wichi) Moseten Bolivia Argentina Formosa 0, , , , , , , ,00000 Moseten Bolivia 0, , , , , , , , ,00000 Movima Bolivia Movima Bolívia 0, , , , , , , , , ,00000 Murui-Muinane 0, , , , , , , , , , ,00000 Ngobe 0, , , , , , , , , , , ,00000 Murui- Ngobe Nukak Paez Parakana Pasto Muinane Nukak 0, , , , , , , , , , , , ,00000 Paez 0, , , , , , , , , , , , , ,00000 Parakana -0, , , , , , , , , , , , , , ,00000 Pasto 0, , , , , , , , , , , , , , , ,00000 Pehuenche Butalelbum Pehuenche Trapa Trapa Peneunche Chile 2 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,39717 Piaroa 0, , , , , , , , , , , , , , , ,13573 Piaroa Colombia 0, , , , , , , , , , , , , , , ,13968 Pilaga Formosa -0, , , , , , , , , , , , , , , ,24621 Poturujara (Zoe) 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,20125 (Arequipa) (Tayacaja) 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,13121 Bolivia 0, , , , , , , , , , , , , , , ,30495 / Nmatsiquenga (San Martin) 0, , , , , , , , , , , , , , , ,22943 Selknam 0, , , , , , , , , , , , , , , ,43611 Siona 0, , , , , , , , , , , , , , , ,05531 Tayacaja Peru -0, , , , , , , , , , , , , , , ,13121 Tehuelche Argentina 0, , , , , , , , , , , , , , , ,38450 Teribe 0, , , , , , , , , , , , , , , ,03563 Teribe2 0, , , , , , , , , , , , , , , ,40100 Tikuna 0, , , , , , , , , , , , , , , ,23066 Tikuna Brasil 0, , , , , , , , , , , , , , , ,33609 Tikuna Colombia 0, , , , , , , , , , , , , , , ,26615 Toba 0, , , , , , , , , , , , , , , ,35938 Toba (chaco) -0, , , , , , , , , , , , , , , ,17763 Toba (Formosa) 0, , , , , , , , , , , , , , , ,11992 Tucano 0, , , , , , , , , , , , , , , ,36928 Tule-Cuna 0, , , , , , , , , , , , , , , ,01376 Waiapi 0, , , , , , , , , , , , , , , ,12654 Wapishana Brasil 0, , , , , , , , , , , , , , , ,43660 Waunana 0, , , , , , , , , , , , , , , ,34889 Wayuu 0, , , , , , , , , , , , , , , ,29901 Wayuu Colombia 0, , , , , , , , , , , , , , , ,14619 Wiwa 0, , , , , , , , , , , , , , , ,44124 Wounan 0, , , , , , , , , , , , , , , ,19545 Xavante 0, , , , , , , , , , , , , , , ,40855 Yaghan Chile 0, , , , , , , , , , , , , , , ,43303 Yamamadi 0, , , , , , , , , , , , , , , ,76190 Yamana (Yaghan) 0, , , , , , , , , , , , , , , ,48522 Yaminawa 0, , , , , , , , , , , , , , , ,11138 Yanomama 0, , , , , , , , , , , , , , , ,27137 Yanomama Brasil 0, , , , , , , , , , , , , , , ,39251 Yanomami 0, , , , , , , , , , , , , , , ,52333 Yanomamo 0, , , , , , , , , , , , , , , ,32316 Yuko-Yukpa 0, , , , , , , , , , , , , , , ,91300 Yuracare 0, , , , , , , , , , , , , , , ,01855 Zenu 0, , , , , , , , , , , , , , , ,14542 Zoro 0, , , , , , , , , , , , , , , ,33595 Apêndice XXX I: Matriz de Fst entre populações da América do Sul (biom74) com cinco haplogrupos de DNA mitocondrial 260
260 261 Pehuenche Butalelbum Pehuenche Trapa Trapa Pehuenche Butalelbum 0,00000 Pehuenche Peneunche Trapa Trapa Chile 2 0, ,00000 Peneunche Chile 2 0, , ,00000 Piaroa Piaroa Pilaga Poturujara Colombia Formosa (Zoe) (Arequipa) (Tayacaja) Bolivia / Selknam Siona Nmatsiqueng a (San Martin) Tayacaja Peru Piaroa 0, , , ,00000 Piaroa Colombia 0, , , , ,00000 Pilaga Formosa 0, , , , , ,00000 Poturujara (Zoe) 0, , , , , , , , , , , , , , ,00000 (Arequipa) (Tayacaja) 0, , , , , , , , , , , , , , , , , , ,00000 Bolivia 0, , , , , , , , , , ,00000 / Nmatsiquenga (San Martin) 0, , , , , , , , , , , ,00000 Selknam 0, , , , , , , , , , , , ,00000 Siona 0, , , , , , , , , , , , , ,00000 Tayacaja Peru 0, , , , , , , , , , , , , , ,00000 Tehuelche Argentina 0, , , , , , , , , , , , , , ,06763 Teribe 0, , , , , , , , , , , , , , ,24432 Teribe2 0, , , , , , , , , , , , , , ,40939 Tikuna 0, , , , , , , , , , , , , , ,06990 Tikuna Brasil 0, , , , , , , , , , , , , , ,09490 Tikuna Colombia 0, , , , , , , , , , , , , , ,05324 Toba 0, , , , , , , , , , , , , , ,01355 Toba (chaco) 0, , , , , , , , , , , , , , ,00340 Toba (Formosa) 0, , , , , , , , , , , , , , ,01710 Tucano 0, , , , , , , , , , , , , , ,08285 Tule-Cuna 0, , , , , , , , , , , , , , ,08932 Waiapi 0, , , , , , , , , , , , , , ,25861 Wapishana Brasil 0, , , , , , , , , , , , , , ,07767 Waunana 0, , , , , , , , , , , , , , ,12098 Wayuu 0, , , , , , , , , , , , , , ,17070 Wayuu Colombia 0, , , , , , , , , , , , , , ,07500 Wiwa 0, , , , , , , , , , , , , , ,24926 Wounan 0, , , , , , , , , , , , , , ,11235 Xavante 0, , , , , , , , , , , , , , ,21335 Yaghan Chile 0, , , , , , , , , , , , , , ,13916 Yamamadi 0, , , , , , , , , , , , , , ,41219 Yamana (Yaghan) 0, , , , , , , , , , , , , , ,16774 Yaminawa 0, , , , , , , , , , , , , , ,16888 Yanomama 0, , , , , , , , , , , , , , ,12345 Yanomama Brasil 0, , , , , , , , , , , , , , ,12210 Yanomami 0, , , , , , , , , , , , , , ,29969 Yanomamo 0, , , , , , , , , , , , , , ,10770 Yuko-Yukpa 0, , , , , , , , , , , , , , ,49107 Yuracare 0, , , , , , , , , , , , , , ,04362 Zenu 0, , , , , , , , , , , , , , ,03908 Zoro 0, , , , , , , , , , , , , , ,08312 Apêndice XXX J: Matriz de Fst entre populações da América do Sul (biom74) com cinco haplogrupos de DNA mitocondrial
261 262 Tehuelche Argentina Tehuelche Argentina 0,00000 Teribe 0, ,00000 Teribe2 0, , ,00000 Tikuna 0, , , ,00000 Teribe Teribe2 Tikuna Tikuna Brasil Tikuna Toba Colombia Tikuna Brasil 0, , , , ,00000 Tikuna Colombia 0, , , , , ,00000 Toba -0, , , , , , ,00000 Toba (chaco) 0, , , , , , , ,00000 Toba (Formosa) 0, , , , , , , , ,00000 Toba (chaco) Toba Tucano Tule-Cuna Waiapi Wapishana (Formosa) Brasil Tucano 0, , , , , , , , , ,00000 Tule-Cuna 0, , , , , , , , , , ,00000 Waiapi 0, , , , , , , , , , , ,00000 Wapishana Brasil -0, , , , , , , , , , , , ,00000 Waunana Wayuu Waunana 0, , , , , , , , , , , , , ,00000 Wayuu 0, , , , , , , , , , , , , , ,00000 Wayuu Colombia 0, , , , , , , , , , , , , , ,03105 Wiwa 0, , , , , , , , , , , , , , ,04823 Wounan 0, , , , , , , , , , , , , , ,02762 Xavante 0, , , , , , , , , , , , , , ,49330 Yaghan Chile 0, , , , , , , , , , , , , , ,19641 Yamamadi 0, , , , , , , , , , , , , , ,18963 Yamana (Yaghan) 0, , , , , , , , , , , , , , ,22471 Yaminawa 0, , , , , , , , , , , , , , ,11781 Yanomama 0, , , , , , , , , , , , , , ,02090 Yanomama Brasil 0, , , , , , , , , , , , , , ,07361 Yanomami 0, , , , , , , , , , , , , , ,07670 Yanomamo 0, , , , , , , , , , , , , , ,20410 Yuko-Yukpa 0, , , , , , , , , , , , , , ,84463 Yuracare 0, , , , , , , , , , , , , , ,09802 Zenu 0, , , , , , , , , , , , , , ,07789 Zoro 0, , , , , , , , , , , , , , ,31224 Apêndice XXX K: Matriz de Fst entre populações da América do Sul (biom74) com cinco haplogrupos de DNA mitocondrial Wayuu Wiwa Colombia Wayuu Colombia 0,00000 Wiwa 0, ,00000 Wounan 0, , ,00000 Xavante 0, , , ,00000 Yaghan Chile 0, , , , ,00000 Wounan Xavante Yaghan Chile Yamamadi Yamana Yaminawa Yanomama Yanomama (Yaghan) Brasil Yamamadi 0, , , , , ,00000 Yamana (Yaghan) 0, , , , , , ,00000 Yaminawa 0, , , , , , , ,00000 Yanomama 0, , , , , , , , ,00000 Yanomama Brasil 0, , , , , , , , , ,00000 Yanomami 0, , , , , , , , , , ,00000 Yanomamo 0, , , , , , , , , , , ,00000 Yanomami Yanomamo Yuko-Yukpa Yuracare Zenu Yuko-Yukpa 0, , , , , , , , , , , , ,00000 Yuracare 0, , , , , , , , , , , , , ,00000 Zenu -0, , , , , , , , , , , , , , ,00000 Zoro 0, , , , , , , , , , , , , , , ,00000 Apêndice XXX L: Matriz de Fst entre populações da América do Sul (biom74) com cinco haplogrupos de DNA mitocondrial Zoro
262 Figura esquemática da localização original dos apêndices XXIII e XXIV A-E da matriz de DNA de populações da América do Norte (biom63)
263 Akimal Oodham Anasazi ancient Apache Bella Coola Bella Coola2 Bella Coola3 Bella Coola4 Cherokee Cherokee Oklahoma Red Cross Cherokee Stillwell Cheyenne/Araphoe Cheyenne/Araphoe2 Chickasaw Chickasaw/Choctaw Chippewa Chippewa Turtle Muntain Chippewa Wisconsin Chippewa/Kickapoo Choctaw Chumash Cochimi Creek (Muskoke) Creek/Seminole Havasupai/Hualapai/Yavapai/ Mojave hokan (central coast) Hokan Northern Hopi Kiliwa Kiliwa/Paipai Kiowa Kumiai Mohawk Mohawk2 Muskoke Oklahoma Navajo Nuu-Chah-Nulth Ojibwa Manitoulin Island Ojibwa North Ontario Ojibwa/Chippewa Paiute N./Shoshoni Paiute/Shoshone Pawnee Penutian California Penutian California2 Pima Pima2 Pima3 Pyramid Lake ancient Quechan/Cocopa Sahaptian Plateau Salinan/Chumash Salish Plateau Seminole Seri Siouan Sioux Sisseton/Wapheton Sioux Stillwater Marsh ancient Takik Taono Oodham (Papago) Uto-Aztecan California Uto-Aztecan Central Uto-Aztecan Northern Washo Yakama Yakima Yok-Utian Yuman Yuman (Hokan) Yuman Delta Yuman Pai Yuman River Zuni Zuni (penutian) Akimal Anasazi Apache Bella Coola Bella Coola2 Bella Coola3 Bella Coola4 Cherokee Cherokee Cherokee Cheyenne/ Cheyenne/ Chickasaw Chickasaw/ Chippewa Oodham ancient Oklahoma Stillwell Red Cross Arapahoe Araphoe2 Choctaw 0 0, , , , , , ,4704 0, , , , , ,0376-0, , ,3791 0, , ,0106 0, , , , , ,3203 0, , , , , , , , , , , , , , , , , , , , , , ,0578 0,0357 0, , , , , , , , , , , , , , , , , , , , , , , , ,0906 0, , , , , , , , , , , , , , , , , , , , , , ,1366 0, , , , , , , , , , ,0271 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,4211-0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0919 0, , , , , , , , , , , , , , , , , , ,0052 0,2187 0,0774 0, , , , , , , , , , , , , , , , , , , , , , , , , , ,0411 0,0045 0, , , ,0089 0, , ,0525 0, , , , , , , , , , , , ,058 0, , , , , ,5016 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0388 0, ,45 0, , , , , , , , , , , , , , , , ,042 0, , , , , , , , , , , , ,1762 0,325 0,2084-0, , , , ,0209 0, , , , , , , , , , , , , , , ,2773 0, , , , , , , , , , , , ,0182 0, ,261 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0211 0, , , , , ,0216 0, , , , , ,0539 0, , , , , , , , , , ,2067 0, , , , , , , , , , , , , , , ,1026 0, , ,0875 0, , , , , , , ,2221 0, , ,2335 0, ,1907 0, , , ,2166 0, , , , , , , , , , , , ,1653 0, , , , , , ,1712 0,1796 0, ,113 0, , ,0546 0, , , , , , , , , , , , , ,1681 0, , , , , , , , , , , , , , , , , , , , ,3253 0, , ,3614 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,3603-0, ,0571-0, , , ,0805 0, , , , , , , , ,3087 0, , , , , ,2611 0, ,2863-0, , , , , , , , ,1895 0, ,2569 0, , , , , ,0298 0, , , , , , , , , , , , , , , , , , ,0411 0,0045 0, , , ,0089 0, , ,0525 0, , , , , , , , , , , , , , , , , , , , , , , ,3389 0, , , , ,2823-0, , , , ,4389 0, , , , , , , , , , ,5516 0, , ,168-0, , , ,0309-0, , , , , , , , , , , , , , ,0468 0, , ,1366 0, , , , ,0048 0,0363 0,3038 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0731-0, ,0602 0, , , , , , , , , , , , , , , ,2854 0, , , , , , , ,0707 0, , , ,0743-0, , ,3253 0, , ,3614 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,1652 0, , , , , , , ,3591 0, , , , , , , , , , , , , , ,3445 0, , , , , , , , , , , , , ,2567 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,4081-0,0135 0, , , , , , , , ,0747 0, ,3533 0,5201 0,3915 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,2294 0, , , , ,1232 0, ,3844 0, , , , , , , , , , , , , , , , , , , , , , , , , , ,23576 Apêndice XXXI A: Matriz de Fst entre populações da América do Norte (biom63) com quatro haplogrupos de DNA mitocondrial
264 265 Chippewa Turtle Muntain Chippewa Chippewa/ Choctaw Chumash Cochimi Creek Creek/ Havasupai/ Wisconsin Kickapoo (Muskoke) Seminole Hualapai/ Yavapai/ Mojave hokan (central coast) Hokan Hopi Kiliwa Kiliwa/Paipai Kiowa Northern Chippewa Turtle Muntain 0 Chippewa Wisconsin 0, Chippewa/Kickapoo -0, , Choctaw 0, , , Chumash 0, , , , Cochimi 0, , , , , Creek (Muskoke) 0, , , , , , Creek/Seminole -0,0003 0, , ,0656-0, , , Havasupai/Hualapai/ Yavapai/Mojave 0,2196 0, , , ,2977-0, , , hokan (central coast) 0, ,1149 0, ,0668-0, , , , , Hokan Northern 0, ,2121 0,2929 0, , , , , , , Hopi 0, , , , , ,2611 0, , , , , Kiliwa 0, , ,5241 0, ,578 0, , , , , , Kiliwa/Paipai 0, , , , , , , , , ,439 0, , , Kiowa -0, ,0934-0, , , , , , , , , , , , Kumiai 0, , , , , , , , , , , , , , ,37784 Mohawk -0, , , , , , , , , , , ,5443 0, , ,18919 Mohawk2-0, ,0774-0, , , , ,1059 0, , ,0344 0, , , ,4317-0,1505 Muskoke Oklahoma 0, , , , ,008 0, , , , , , , , , ,04915 Navajo 0, , , , , , , , , , , , , , ,06965 Nuu-Chah-Nulth 0, , ,0617 0,0907-0, , , , , ,0383 0, , ,4685 0, ,07052 Ojibwa Manitoulin Island 0, , , , , , , , , , , , , , ,163 Ojibwa North Ontario 0, , , , , , , , , , , , ,7899 0, ,05078 Ojibwa/Chippewa -0, , , , , , , , , , , , , , ,19019 Paiute N./Shoshoni 0, , , , , , , , , , , , , , ,37583 Paiute/Shoshone 0, , , , ,1696 0, , , , , , , , ,4372 0,23873 Pawnee 0, , ,1456 0, , , ,118 0, , , , , , ,2262 0,1446 Penutian California 0, , , , ,1982 0, , , , ,0711-0, , , , ,29218 Penutian California2 0, , , , , , , , , , , , , , ,24745 Pima 0, , ,3046 0, , , , , , , , , , , ,31148 Pima2 0, , , , , , , , , , , , ,2724 0, ,25848 Pima3 0,2727 0, , , , , , , , , ,0877 0, , , ,26815 Pyramid Lake ancient 0, , , , , , , , , , , , , , ,33143 Quechan/Cocopa 0, , , , ,4362 0, , ,3102-0, ,3165 0, , , ,0678 0,4357 Sahaptian Plateau 0, , , , , , , , , , , , , , ,23873 Salinan/Chumash 0, ,1149 0, ,0668-0, , , , , ,1 0, , , ,439-0,07287 Salish Plateau 0, ,215 0, , , , , , , , , , , , ,14876 Seminole -0, , , , , , , , , , , ,4442 0, , ,06337 Seri 0, , , , , ,1672 0, , ,2244 0, , , , ,7383 0,46538 Siouan -0, , ,0138 0, ,0491 0, , ,022 0,2039-0, , , , , ,13535 Sioux Sisseton/ Wapheton Sioux -0, , ,0116 0,0168 0, ,2099 0, , , , , , , , ,13114 Stillwater Marsh ancient 0, , , ,4417 0, , , , , , , ,372 0, , ,4004 Takik 0, , , , , , , , , , , , , , ,20516 Taono Oodham (Papago) 0, , , , , , , , , , , , , , ,34105 Uto-Aztecan California 0, , , , , , , , , , , , ,4113 0, ,19301 Uto-Aztecan Central 0, , ,3046 0, , , , , , , , , , , ,31148 Uto-Aztecan Northern 0, , , , , , , , , , , , , , ,33723 Washo 0, , ,3137 0, , , , , , , , , , , ,30307 Yakama 0, , , , , , , , , , , ,0059 0,0652 0, ,44557 Yakima 0, , , ,4311 0, , , , , , , , ,0745 0,0404 0,42676 Yok-Utian 0, , , , , , , , , , , ,3217 0, , ,21347 Yuman 0, , ,3396 0, , ,0196 0, , , , , , , , ,3599 Yuman (Hokan) 0, , , , , , ,2755 0, , ,2993 0,1056 0, , , ,3776 Yuman Delta 0, , , , , , , , , , , , , , ,35348 Yuman Pai 0, , , , , , , , , , , , , , ,37315 Yuman River 0, , , , , , , , , ,2986 0, , , , ,39752 Zuni 0, , , , , , , , , , , , , , ,48068 Zuni (penutian) 0, , , , , ,1119 0, , , , , , , ,0383 0,3623 Apêndice XXXI B: Matriz de Fst entre populações da América do Norte (biom63) com quatro haplogrupos de DNA mitocondrial
265 266 Kumiai Mohawk Mohawk2 Muskoke Navajo Nuu-Chah- Ojibwa Ojibwa North Ojibwa/ Paiute N./ Paiute/ Pawnee Penutian Penutian Pima Oklahoma Nulth Manitoulin Ontario Island Chippewa Shoshoni Shoshone California California2 Kumiai 0 Mohawk 0, Mohawk2 0, , Muskoke Oklahoma 0,2839 0, , Navajo 0, , , , Nuu-Chah-Nulth 0, ,041 0, , , Ojibwa Manitoulin Island 0, , , , ,1263 0, Ojibwa North Ontario 0, , , , , , , Ojibwa/Chippewa 0, , , ,158 0, , , , Paiute N./Shoshoni 0, ,3723 0, , , , ,3091 0, , Paiute/Shoshone 0,2087 0, , , , , , , , , Pawnee 0, , , , ,0643 0,158 0, , , , , Penutian California 0, , , , , , , ,4853 0, , , , Penutian California2 0, , , , , , , , , , , , , Pima -0, , ,272 0,2642 0, , , , , , , , , , Pima2-0, , , , , , , , , , , , , , ,02382 Pima3-0, , , , , , , , , , , , , , ,00959 Pyramid Lake ancient 0, , , , , , , , , , , ,2048-0, , ,29113 Quechan/Cocopa -0, , , ,3063 0, , ,2977 0, ,4556 0, , , , , ,01742 Sahaptian Plateau -0, , , ,1229 0, , , , , , , , , , ,00067 Salinan/Chumash 0, , ,0344-0, , ,0383 0, , , , , , ,0711 0, ,25165 Salish Plateau 0, , , ,1103 0, , , , , , , , , , ,05865 Seminole 0, , , , , , , , , , , , , , ,3095 Seri 0, , , ,3759 0, , , , , , , , , , ,27058 Siouan 0, , , , ,0361 0, , , , , , , , , ,27317 Sioux Sisseton/Wapheton Sioux 0, , , , , , , ,0682-0, , , ,0636 0, , ,259 Stillwater Marsh ancient 0,3075 0, , , , , , ,5558 0, , , , , , ,29702 Takik 0, ,3175 0, , , , , , , , , , , , ,13491 Taono Oodham (Papago) -0, , , , , , , , ,4014 0,1835 0, , , , ,02216 Uto-Aztecan California 0,0917 0, , , , , , ,5028 0, , , , ,104 0, ,08453 Uto-Aztecan Central -0, , ,272 0,2642 0, , , , , , , , , ,1748-0,02778 Uto-Aztecan Northern 0, ,3428 0, , , , ,2732 0, ,3875-0, , , , , ,16734 Washo -0, , , , , , , ,5174 0, , , , , , ,0177 Yakama 0, , , , , , ,3413 0, , , , , , ,0834 0,09948 Yakima 0, , , , , , ,3272 0, , , , , , , ,0944 Yok-Utian 0, , , , , , , , , , , , , ,0459 0,21026 Yuman -0, ,361 0, , ,2644 0, , , , , , , , , ,01778 Yuman (Hokan) -0, , , , , , , , , , , , , , ,01489 Yuman Delta -0, , , , , , , , , ,225 0, ,1739 0, , ,03043 Yuman Pai -0, , , , , ,3131 0, , ,4055 0, , , , ,1627-0,00898 Yuman River -0, , , , , , , ,5867 0, , , , , , ,02958 Zuni 0, , , ,3023 0, , , , , , , , , , ,10189 Zuni (penutian) 0, , , , , , , , , , , , , , ,06741 Apêndice XXXI C: Matriz de Fst entre populações da América do Norte (biom63) com quatro haplogrupos de DNA mitocondrial
266 267 Pima2 Pima3 Pyramid Lake ancient Quechan/ Cocopa Sahaptian Plateau Salinan/ Chumash Salish Plateau Seminole Seri Siouan Sioux Sisseton/ Wapheton Sioux Stillwater Marsh ancient Takik Taono Uto-Aztecan Oodham California (Papago) Pima2 0 Pima3-0, Pyramid Lake ancient 0, , Quechan/Cocopa 0, , , Sahaptian Plateau 0,0041 0, , , Salinan/Chumash 0, , , ,3165 0, Salish Plateau 0,0734 0, , , , , Seminole 0,2955 0, ,3001 0, , , , Seri 0, , , , , , , , Siouan 0,2498 0, , , , , , , , Sioux Sisseton/Wapheton Sioux 0, , , , , , , , , , Stillwater Marsh ancient 0, , ,0481 0,3205 0,0628 0, , , , , , Takik 0, , , , , , , , , , , , Taono Oodham (Papago) -0, , , , , , , , , , , , , Uto-Aztecan California 0, , , , , , ,1164 0, , , , , , , Uto-Aztecan Central -0, , , , , , , ,3095 0, , ,259 0, , , ,08453 Uto-Aztecan Northern 0, , , , , , , , , , , , ,1258 0, ,05211 Washo -0, , , , , , ,052 0, , , , , , , ,02472 Yakama 0, , , , , , , , , ,3276 0, , , , ,19421 Yakima 0,1313 0, , , , , , ,3244 0, , , , , , ,17788 Yok-Utian 0,1986 0, , , , , , , , , , , , , ,03622 Yuman -0,0101 0, , , ,0164 0, , ,3432 0, , , , , , ,11149 Yuman (Hokan) 0, , , , , ,2993 0, , , , ,3011 0,3148 0, , ,13282 Yuman Delta -0, , , ,0167 0, , , , ,2572 0, , , , , ,07348 Yuman Pai 0, , , , , , , , , , , , , , ,14905 Yuman River -0, , , , , ,2986 0, , , , , , , , ,1113 Zuni 0, , , , , , ,0119 0, , , , , , , ,28981 Zuni (penutian) 0, , , , , , , , , ,2433 0, , , , ,22436 Apêndice XXXI D: Matriz de Fst entre populações da América do Norte (biom63) com quatro haplogrupos de DNA mitocondrial Uto-Aztecan Central Uto-Aztecan Northern Washo Yakama Yakima Yok-Utian Yuman Yuman (Hokan) Yuman Delta Yuman Pai Yuman River Zuni Zuni (penutian) Uto-Aztecan Central 0 Uto-Aztecan Northern 0, Washo -0,0177 0, Yakama 0, , , Yakima 0,0944 0, , , Yok-Utian 0, , , , , Yuman -0, , , , , , Yuman (Hokan) -0, , , , , , , Yuman Delta -0, , ,0237 0, , , , , Yuman Pai -0, , , , , , , , , Yuman River -0, , , , , , , , , , Zuni 0, , , , , , , , , , , Zuni (penutian) 0, , , , , , , , ,1099-0, , , Apêndice XXXI E: Matriz de Fst entre populações da América do Norte (biom63) com quatro haplogrupos de DNA mitocondrial
267 268 Akimal O odham Anasazi ancient Apache Bella Coola Bella Coola2 Bella Coola3 Bella Coola4 Cherokee Cherokee Oklahoma Red Cross Cherokee Stillwell Cheyenne/Arapahoe Cheyenne/Araphoe2 Chickasaw Chickasaw/Choctaw Chippewa Chippewa Turtle Muntain Chippewa Wisconsin Chippewa/Kickapoo Choctaw Chumash Cochimi Creek (Muskoke) Creek/Seminole Havasupai/Hualapai/ Yavapai/Mojave hokan (central coast) Hokan Northern Hopi Kiliwa Kiliwa/Paipai Kiowa Kumiai Mohawk Mohawk2 Muskoke Oklahoma Navajo Nuu-Chah-Nulth Ojibwa Manitoulin Island Ojibwa North Ontario Ojibwa/Chippewa Paiute N./Shoshoni Paiute/Shoshone Pawnee Penutian California Penutian California2 Pima Pima2 Pima3 Pyramid Lake Quechan/Cocopa Sahaptian Plateau Salinan/Chumash Salish Plateau Seminole Seri Siouan Sioux Sisseton/ Wapheton Sioux Stillwater Marsh ancient Takik Taono O odham (Papago) Uto-Aztecan California Uto-Aztecan Central Uto-Aztecan Northern Washo Yakama Yakima Yok-Utian Yuman Yuman (Hokan) Yuman Delta Yuman Pai Yuman River Zuni Zuni (penutian) Akimal Anasazi Apache O odham ancient Bella Coola Bella Coola2 Bella Coola3 0 0, , , , , , , , , , , , ,0376-0, , , , , , , , , , , ,2455 0, ,3177 0, Bella Cherokee Cherokee Cherokee Cheyenne/ Cheyenne/ Chickasaw Chickasaw/ Chippewa Coola4 Oklahoma Red Cross Stillwell Arapahoe Araphoe2 Choctaw 0, , , ,1537 0, , , , , , , , , , , ,0935 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,0081 0,0635 0, , ,0089 0, , , , , , , , , , , ,1568 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,4211-0, , , ,268 0, , , , , ,3052 0, , , , , , , , , , , , , , , , , , , , , , , , ,0919 0, , , , , , , , , , , , , , , , , , , ,1143 0, , , , ,2259 0, , ,0355 0, , , , , , , , , ,1067 0, , , , ,2446 0, , ,044 0, ,0411 0,0045 0,2067 0, , , , , , , , , , , , , , , , , , , , , , , , ,5016 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , ,1032 0, , , , , , , , ,3805 0, ,0388 0, , ,281 0, , ,0281 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,2851 0, , , , , , , , , , , , , , , , , , , , ,0146 0, ,0182 0, ,2378 0, , , , , , , ,2269 0, , , , , , , , , , ,1421 0, ,0245 0, , , , , , , , , ,1341 0, , , , , , , , , , , , , , , , , , , , , , , ,036 0, , , , , , , ,1202 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,1907 0, , , , , , , , , , , , , , , , , , , , , , , ,1712 0, , ,113 0, , ,0546 0,0825 0, , , , , , , , ,2181 0, , , , , ,2995 0, , , , , , , , , , , , , , , ,1819-0, , ,3253 0,3099 0, ,3614 0, , , , , , , , , , , , , , , , , , , , , , , , , , , ,2938 0, , , , ,0547-0, , , , , ,1379 0, , , , , , ,3087 0, , , , , ,2611 0, , , ,0317 0, , , , , , , , , , , , , , ,0186 0, , , , , , , , , , , , ,1168 0,2446 0, , ,044 0, ,0411 0,0045 0,2067 0, , , , , , , , , , , , , , , , , , , , , , , , , ,0527 0, , , ,3126 0, , , ,1166 0,2823-0, , , , ,4389 0, , , , , , , , , , , ,2094 0, , , , , , , , , , , , , ,0066 0, ,2467 0,1395-0, , , , , , , ,197 0, , , , ,079 0, , , , , , , , , , , , , , ,2962 0, , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , , ,2854 0, , , , , , , , , , ,3387 0, , , ,3253 0,3099 0, ,3614 0, , , , , , , , , , , , , , , , , , , , , , ,3508 0, , , , , , , , , , , , , , , , , , , , , , , , ,2652 0, , , , , , , , , , , , , , , , ,2659 0, , , , ,2049 0, ,2567 0, , , , , , , ,1091 0, , , , , , , , , , , ,1462 0, , , , , , , , , , , , , ,4081 0, , , , , , , , ,0295 0, , , ,5201 0,3915 0, , , , , , , ,387 0,1799 0,0041 0, , , , , , , , , , , , , , , , , , , , , , , , , , , ,3958 0, , , ,3844 0, , , , , , , ,3023 0,3036-0, , , ,0836-0, , ,3002 0, , , , , , , , , , ,23744 Apêndice XXXII A: Matriz de Fst entre populações da América do Norte (biom63) com cinco haplogrupos de DNA mitocondrial
268 269 Chippewa Turtle Muntain Chippewa Wisconsin Chippewa Choctaw Chumash Cochimi /Kickapoo Creek (Muskoke) Creek/ Seminole Havasupai/ Hualapai/ Yavapai/ Mojave hokan Hokan (central Northern coast) Hopi Kiliwa Kiliwa/ Paipai Chippewa Turtle Muntain 0 Chippewa Wisconsin 0, Chippewa/Kickapoo -0,0024 0, Choctaw 0, , , Chumash 0, , , , Cochimi 0, , , , , Creek (Muskoke) 0, , , , , , Creek/Seminole 0, , , , , , , Havasupai/Hualapai/ Yavapai/Mojave 0, , , , ,2977-0, , , hokan (central coast) 0, , , ,0668-0, , , , , Hokan Northern 0, , , , , , , , , , Hopi 0, , ,4166 0, , ,2611 0, , , , , Kiliwa 0,4799 0, , , ,578 0, , , , , , Kiliwa/Paipai 0, ,4108 0, , , , , , , ,439 0, , , Kiowa 0, , , ,1898 0, , , , , , ,0725 0, , , Kumiai 0, , , , , , , , , , ,1002 0, , , ,34507 Mohawk 0, , , , , , ,1197-0, , , , , , ,4597-0,12278 Mohawk2-0, , , , , , ,1059 0, , ,0344 0, , , ,4317 0,07672 Muskoke Oklahoma 0, , , , ,008 0, , , , , , , , , ,11881 Navajo 0,0356 0, , , , , , , , , , , , , ,15203 Nuu-Chah-Nulth 0, , , , , , , , , , , , , , ,02437 Ojibwa Manitoulin Island 0, , , , , , ,1047-0, , , , , , , ,10134 Ojibwa North Ontario 0, , , , , ,367 0, , , , , , , , ,0234 Ojibwa/Chippewa 0, , , , , , , , , ,0586 0, , , , ,08946 Paiute N./Shoshoni 0, ,3007 0, , , , , , , , , , , , ,34073 Paiute/Shoshone 0, , , , ,157 0, , , , , , , , , ,13571 Pawnee 0, , , , , , ,118 0, , , , , , ,2262 0,16667 Penutian California 0, , , , ,1982 0, , ,1316 0, ,0711-0, , , , ,2628 Penutian California2 0, , , , , , , , , , ,0726 0, , , ,2272 Pima 0, , , , , , , , , , , , , , ,30578 Pima2 0, , , , , , , , , , , , ,2724 0, ,26457 Pima3 0, , , , , , , , , , , , , , ,26931 Pyramid Lake 0, ,2801 0, , , , , , , , , , , , ,29693 Quechan/Cocopa 0, ,2591 0, , , , , , , ,2899 0, , , , ,34999 Sahaptian Plateau 0,2362 0, , , , , , , , , , , , , ,20849 Salinan/Chumash 0, , , ,0668-0, , , , , ,1 0, , , ,439 0,03848 Salish Plateau 0, , , , , , , , , , , , , , ,1686 Seminole -0, , , , , , , , , , , ,4442 0, , ,1232 Seri 0, , , , , ,1672 0, , ,2244 0, ,3459 0, , ,7383 0,41748 Siouan -0, ,0867-0, , , , , , , , , , , , ,0049 Sioux Sisseton/Wapheton Sioux -0, , , , , , ,074 0, , ,0024 0, , , , ,04862 Stillwater Marsh ancient 0, , , ,4417 0, , , , , , , ,372 0, , ,35337 Takik 0, , , , , , , , , , ,0139 0, , , ,20917 Taono O odham (Papago) 0, ,234 0,2812 0, , , , , , , , , , , ,32353 Uto-Aztecan California 0, , , , , , , , , , , , ,4113 0, ,18902 Uto-Aztecan Central 0, , , , , , , , , , , , , , ,30578 Uto-Aztecan Northern 0, , , , , , , , , , , , , , ,30783 Washo 0, , , , , , , , , , , , , , ,28543 Yakama 0, ,3044 0, , , , , , , ,2334 0, , , , ,35588 Yakima 0, ,3228 0,3196 0,4311 0, , , , , , , , ,0745 0,0404 0,38928 Yok-Utian 0, , , , , , , , , , , ,3217 0, , ,202 Yuman 0,3107 0, , , , ,0196 0, ,2764-0, , , , , , ,35179 Yuman (Hokan) 0, , , , , , ,2755 0, , ,2993 0, , , , ,36491 Yuman Delta 0, , ,2827 0, , , , , , , , , , , ,33369 Yuman Pai 0, , , , , , , , , , , , , , ,35118 Yuman River 0, , , , , , , , , ,2986 0, , , , ,36688 Zuni 0, , ,3204 0, , , , , , , , , , , ,44458 Zuni (penutian) 0,2469 0,287 0, , , ,1119 0, , , , ,1478 0, , ,0383 0,34392 Apêndice XXXII B: Matriz de Fst entre populações da América do Norte (biom63) com cinco haplogrupos de DNA mitocondrial Kiowa
269 270 Kumiai Mohawk Mohawk2 Muskoke Oklahoma Navajo Nuu- Chah- Nulth Ojibwa Manitoulin Island Ojibwa North Ontario Ojibwa/ Paiute N./ Chippewa Shoshoni Paiute/ Pawnee Shoshone Penutian Penutian California California 2 Pima Kumiai 0 Mohawk 0, Mohawk2 0, , Muskoke Oklahoma 0,2839 0, , Navajo 0, , , , Nuu-Chah-Nulth 0, , , , , Ojibwa Manitoulin Island 0, , , , , , Ojibwa North Ontario 0, , , , , , , Ojibwa/Chippewa 0, , , , , , , , Paiute N./Shoshoni 0, , , , , , , , , Paiute/Shoshone 0, , , , , , , , , , Pawnee 0, , , , , ,1422 0, , , , , Penutian California 0, , , , , , , , , , , , Penutian California2 0, , , , , , , , , , , , , Pima -0, ,2924 0,272 0,2642 0, , , , , , , , , , Pima2-0, , , , , , , , ,2804 0, , , , , ,02382 Pima3-0, , , , ,2321 0, , , , , , , , , ,00933 Pyramid Lake 0, , , , , , , , , , , ,2048-0, , ,29113 Quechan/Cocopa -0, , , , , , , ,4502 0, , , ,1127 0,2052 0, ,01914 Sahaptian Plateau -0, , , ,1229 0, , , ,3771 0, , , , , , ,00067 Salinan/Chumash 0, , ,0344-0, , , , , ,0586 0, , , ,0711 0, ,25165 Salish Plateau 0, , , ,1103 0, , , , , , , , , , ,05865 Seminole 0, , , , , , ,1279 0, , , , , , , ,3095 Seri 0, , , ,3759 0,4683 0, , , , , , , , , ,27058 Siouan 0, , , , , , , , , , , , ,2052 0, ,25177 Sioux Sisseton/Wapheton Sioux 0, , , , ,0329 0, , , , , , ,0588 0,2281 0, ,2522 Stillwater Marsh ancient 0,3075 0, , , , , ,2908 0, , , , , , , ,29702 Takik 0, , , , , , , , ,2739 0, , , , , ,13491 Taono O odham (Papago) -0, , , , , , , , , ,1835 0, , , , ,02216 Uto-Aztecan California 0,0917 0, , , , , , , ,2574 0, , , ,104 0, ,08453 Uto-Aztecan Central -0, ,2924 0,272 0,2642 0, , , , , , , , , ,1748-0,02778 Uto-Aztecan Northern 0, , , , , , , , , , , , , , ,16734 Washo -0, , , , , , , , , , , , , , ,0177 Yakama 0, ,3132 0, , , , , , , , , , , , ,09025 Yakima 0, , , , , ,2895 0, , ,3629 0, , , , , ,0944 Yok-Utian 0, , , , , , , , , , , , , ,0459 0,21026 Yuman -0, , , , , , ,2618 0, , , ,2238 0, , , ,01778 Yuman (Hokan) -0, , , , , , , , , , ,2343 0, , , ,01489 Yuman Delta -0, , , , , , , , , ,225 0, ,1739 0, , ,03043 Yuman Pai -0, , , , , , , , , , , , , ,1627-0,00898 Yuman River -0, , , , , ,3108 0, , , , , , , , ,02958 Zuni 0, , , ,3023 0, , ,3216 0, ,3698 0, , , , , ,10189 Zuni (penutian) 0, , , , ,1216 0, , , , , , , , , ,06741 Apêndice XXXII C: Matriz de Fst entre populações da América do Norte (biom63) com cinco haplogrupos de DNA mitocondrial
270 271 Pima2 Pima3 Pyramid Lake Quechan/ Cocopa Sahaptian Plateau Salinan/ Chumash Salish Plateau Seminole Seri Siouan Sioux Sisseton/ Wapheton Sioux Stillwater Marsh ancient Takik Taono Uto- O odham Aztecan (Papago) California Pima2 0 Pima3-0, Pyramid Lake 0, , Quechan/Cocopa 0, ,0189 0, Sahaptian Plateau 0,0041 0,0198 0, , Salinan/Chumash 0, , , ,2899 0, Salish Plateau 0,0734 0, , , , , Seminole 0,2955 0, ,3001 0, , , , Seri 0, , , , , , , , Siouan 0, , ,2443 0, , , , , , Sioux Sisseton/Wapheton Sioux 0, , , , , ,0024 0, , , , Stillwater Marsh ancient 0, , ,0481 0, ,0628 0, , , , ,2915 0, Takik 0, , , , , , , , , , ,2522 0, Taono O odham (Papago) -0, , , , , , , , , , , , , Uto-Aztecan California 0, , , , , , ,1164 0, , ,2093 0, , , , Uto-Aztecan Central -0, , , , , , , ,3095 0, , ,2522 0, , , ,08453 Uto-Aztecan Northern 0, , , , , , , , , , , , ,1258 0, ,05211 Washo -0, , , , , , ,052 0, , , , , , , ,02472 Yakama 0, , , , , ,2334 0, , , , , ,1662 0, , ,17146 Yakima 0,1313 0, , , , , , ,3244 0, , , , , , ,17788 Yok-Utian 0,1986 0, , , , , , , , , , , , , ,03622 Yuman -0,0101 0, , , ,0164 0, , ,3432 0, , , , , , ,11149 Yuman (Hokan) 0, ,017 0, ,0234 0, ,2993 0, , , , , ,3148 0, , ,13282 Yuman Delta -0, , , , , , , , ,2572 0, , , , , ,07348 Yuman Pai 0, ,0341 0, , , , , , , , , , , , ,14905 Yuman River -0, , , ,0403 0, ,2986 0, , , , , , , , ,1113 Zuni 0, ,1537 0, , , , ,0119 0, , , , , , , ,28981 Zuni (penutian) 0, , , , , , , , , , , , , , ,22436 Apêndice XXXII D: Matriz de Fst entre populações da América do Norte (biom63) com cinco haplogrupos de DNA mitocondrial Uto- Uto- Aztecan Aztecan Central Northern Washo Yakama Yakima Yok-Utian Yuman Yuman (Hokan) Yuman Delta Yuman Pai Yuman River Zuni Zuni (penutian) Uto-Aztecan Central 0 Uto-Aztecan Northern 0, Washo -0,0177 0, Yakama 0, , , Yakima 0,0944 0, , , Yok-Utian 0, , , , , Yuman -0, , , , , , Yuman (Hokan) -0, , , ,0761 0, , , Yuman Delta -0, , ,0237 0, , , , , Yuman Pai -0, , , , , , , , , Yuman River -0, , , , , , , , , , Zuni 0, , , , , , , , , , , Zuni (penutian) 0, , , ,0163 0, , , , ,1099-0, , , Apêndice XXXII E: Matriz de Fst entre populações da América do Norte (biom63) com cinco haplogrupos de DNA mitocondrial
271 Apêndice XXXIII a XXXV: Figura esquemática da localização original dos apêndices XXXIII a XXXV A-AC das matrizes de distância geográfica e distância fisiográfica.
Filogeografia e Demografia
 Aula 10 Filogeografia e Demografia (princípios e ferramentas estatísticas) Filogeografia O que é? Ferramentas Aplicações Filogeografia: O que é? O campo da ciência que estuda os princípios e processos
Aula 10 Filogeografia e Demografia (princípios e ferramentas estatísticas) Filogeografia O que é? Ferramentas Aplicações Filogeografia: O que é? O campo da ciência que estuda os princípios e processos
estuda a distribuição dos organismos sobre a superfície da Terra.
 BIOGEOGRAFIA BIOGEOGRAFIA estuda a distribuição dos organismos sobre a superfície da Terra. tenta explicar os padrões e a variação entre os indivíduos, as populações, as espécies e as comunidades por toda
BIOGEOGRAFIA BIOGEOGRAFIA estuda a distribuição dos organismos sobre a superfície da Terra. tenta explicar os padrões e a variação entre os indivíduos, as populações, as espécies e as comunidades por toda
Descreve a história da vida na Terra Investiga os processos responsáveis por essa história
 Aula 1 Evolução Biologia Evolutiva x Evolução Biológica O termo Evolução biológica refere-se ao processo de modificação e surgimento das espécies na Terra Biologia Evolutiva refere-se à disciplina que
Aula 1 Evolução Biologia Evolutiva x Evolução Biológica O termo Evolução biológica refere-se ao processo de modificação e surgimento das espécies na Terra Biologia Evolutiva refere-se à disciplina que
Universidade Estadual do Rio Grande do Sul Curso Superior de Tecnologia em Gestão Ambiental Biologia Aplicada Aula 7
 Universidade Estadual do Rio Grande do Sul Curso Superior de Tecnologia em Gestão Ambiental Biologia Aplicada Aula 7 Professor Antônio Ruas 1. Créditos: 60 2. Carga horária semanal: 4 3. Semestre: 1 4.
Universidade Estadual do Rio Grande do Sul Curso Superior de Tecnologia em Gestão Ambiental Biologia Aplicada Aula 7 Professor Antônio Ruas 1. Créditos: 60 2. Carga horária semanal: 4 3. Semestre: 1 4.
Origem da variação. Conceitos importantes. Variação Genética e Evolução. Deriva. Seleção. Mutação. Migração
 Origem da variação Conceitos importantes Variação genética -> variantes alélicos São originados por mutação e intensificados por mecanismos recombinatórios e sexuais Diversidade ou variabilidade genética:
Origem da variação Conceitos importantes Variação genética -> variantes alélicos São originados por mutação e intensificados por mecanismos recombinatórios e sexuais Diversidade ou variabilidade genética:
Recursos Genéticos Vegetais
 Recursos Genéticos Vegetais Caracterização x Avaliação Caracterização Alta herdabilidade Pouca influência ambiental Avaliação Baixa herdabilidade Muita influência ambiental Caracterização: Caracterização
Recursos Genéticos Vegetais Caracterização x Avaliação Caracterização Alta herdabilidade Pouca influência ambiental Avaliação Baixa herdabilidade Muita influência ambiental Caracterização: Caracterização
Microssatélites. Marcadores Genômicos. Metodologia Molecular. Evolução Humana - Módulo 5. O passado humano 07/02/2015. DNA Mitocondrial ~17 Kb
 Evolução Humana - Módulo 5 O passado humano Metodologia Molecular Extração de ADN, armazenamento, catalogação Genética Coleção de tecido/espécime PCR Análise dos dados Bioinformática Dados de sequenciamento/genotipagem
Evolução Humana - Módulo 5 O passado humano Metodologia Molecular Extração de ADN, armazenamento, catalogação Genética Coleção de tecido/espécime PCR Análise dos dados Bioinformática Dados de sequenciamento/genotipagem
a) do DNAmt, transmitido ao longo da linhagem materna, pois, em cada célula humana, há várias cópias dessa molécula.
 01 - (ENEM) Uma vítima de acidente de carro foi encontrada carbonizada devido a uma explosão. Indícios, como certos adereços de metal usados pela vítima, sugerem que a mesma seja filha de um determinado
01 - (ENEM) Uma vítima de acidente de carro foi encontrada carbonizada devido a uma explosão. Indícios, como certos adereços de metal usados pela vítima, sugerem que a mesma seja filha de um determinado
O MODELO DE HARDY-WEINBERG
 Modelo simples de genética de populações: modelo de Hardy-Weinberg (Hardy 1908; Weinberg 1908). Embora faça vários pressupostos simplificadores que não são realistas, ele se mostra bastante útil para descrever
Modelo simples de genética de populações: modelo de Hardy-Weinberg (Hardy 1908; Weinberg 1908). Embora faça vários pressupostos simplificadores que não são realistas, ele se mostra bastante útil para descrever
Universidade Federal de Santa Maria PET - Biologia. Marcela Dambrowski dos Santos
 Universidade Federal de Santa Maria PET - Biologia Marcela Dambrowski dos Santos Introdução Hemoglobina A Hemoglobina S Anemia falciforme Traço falciforme Malária Hipótese da malária Origem e dispersão
Universidade Federal de Santa Maria PET - Biologia Marcela Dambrowski dos Santos Introdução Hemoglobina A Hemoglobina S Anemia falciforme Traço falciforme Malária Hipótese da malária Origem e dispersão
GENÉTICA DE POPULAÇÕES
 GENÉTICA DE POPULAÇÕES FATORES MICROEVOLUTIVOS: ENDOGAMIA PROF. MACKS WENDHELL GONÇALVES ENDOGAMIA DEME Deme é uma coleção de indivíduos intercruzantes da mesma espécie que vive em proximidade suficiente
GENÉTICA DE POPULAÇÕES FATORES MICROEVOLUTIVOS: ENDOGAMIA PROF. MACKS WENDHELL GONÇALVES ENDOGAMIA DEME Deme é uma coleção de indivíduos intercruzantes da mesma espécie que vive em proximidade suficiente
Marcadores Moleculares
 Marcadores Moleculares Marcadores Moleculares Marcadores Genéticos Características polimórficas: marcadores de variabilidade genética entre indivíduos, populações, espécies e taxons superiores. Marcadores
Marcadores Moleculares Marcadores Moleculares Marcadores Genéticos Características polimórficas: marcadores de variabilidade genética entre indivíduos, populações, espécies e taxons superiores. Marcadores
Origem da variação. Conceitos importantes. Variabilidade genética. Variabilidade Genética. Variação genética e Evolução
 Variabilidade genética Origem da variação Professor Fabrício R Santos fsantos@icb.ufmg.br Departamento de Biologia Geral, UFMG 2011 Conceitos importantes Variação genética: variantes alélicos originados
Variabilidade genética Origem da variação Professor Fabrício R Santos fsantos@icb.ufmg.br Departamento de Biologia Geral, UFMG 2011 Conceitos importantes Variação genética: variantes alélicos originados
CST 304-3: Fundamentos de Ecologia e de Modelagem Ambiental Aplicados à conservação da Biodiversidade
 CST 304-3: Fundamentos de Ecologia e de Modelagem Ambiental Aplicados à conservação da Biodiversidade O REINO NEOTROPICAL AL U N A : Y H AS M I N M E N D E S D E M O U R A REINO NEOTROPICAL AMÉRICA LATINA
CST 304-3: Fundamentos de Ecologia e de Modelagem Ambiental Aplicados à conservação da Biodiversidade O REINO NEOTROPICAL AL U N A : Y H AS M I N M E N D E S D E M O U R A REINO NEOTROPICAL AMÉRICA LATINA
ECOLOGIA II RELATÓRIO 1-2ªPARTE. Trabalho realizado por:
 ECOLOGIA II RELATÓRIO 1-2ªPARTE Genética de populações Trabalho realizado por: Eva Firme, nº15471 Licª. Ensino de Ciências da Natureza Maio de 2006 INTRODUÇÃO Uma população é a reunião de famílias com
ECOLOGIA II RELATÓRIO 1-2ªPARTE Genética de populações Trabalho realizado por: Eva Firme, nº15471 Licª. Ensino de Ciências da Natureza Maio de 2006 INTRODUÇÃO Uma população é a reunião de famílias com
I. ANTES DE ASSISTIR AO VÍDEO, COMENTAR OS SEGUINTES PONTOS EM CLASSE:
 TÍTULO: ÁFRICA: A ORIGEM DO HOMEM DURAÇÃO: 50 GRAUS: 6-8 EXTENSÃO CURRICULAR: CIÊNCIAS NATURAIS, GEOGRAFIA, HISTÓRIA, MATEMÁTICA, NUTRIÇÃO, LÍNGUA PORTUGUESA, EXPRESSÃO ORAL E ESCRITA. I. ANTES DE ASSISTIR
TÍTULO: ÁFRICA: A ORIGEM DO HOMEM DURAÇÃO: 50 GRAUS: 6-8 EXTENSÃO CURRICULAR: CIÊNCIAS NATURAIS, GEOGRAFIA, HISTÓRIA, MATEMÁTICA, NUTRIÇÃO, LÍNGUA PORTUGUESA, EXPRESSÃO ORAL E ESCRITA. I. ANTES DE ASSISTIR
Evolução Molecular. "Nothing in Biology Makes Sense Except in the Light of Evolution. Theodosius Dobzhansky
 "Nothing in Biology Makes Sense Except in the Light of Evolution Theodosius Dobzhansky Evolução Evolução Evolução Genótipo + Ambiente = Fenótipo Parental F1 F2 Evolução Evolução = mudança (características
"Nothing in Biology Makes Sense Except in the Light of Evolution Theodosius Dobzhansky Evolução Evolução Evolução Genótipo + Ambiente = Fenótipo Parental F1 F2 Evolução Evolução = mudança (características
Evolução Def. usual Biologicamente frequências gênicas populações
 Evolução A palavra evolução vem do Latim evolvere que significa Desenvolver ou Estender. A Def. usual: progresso, desenvolvimento, melhora. Biologicamente: evolução é a mudança nas propriedades (frequências
Evolução A palavra evolução vem do Latim evolvere que significa Desenvolver ou Estender. A Def. usual: progresso, desenvolvimento, melhora. Biologicamente: evolução é a mudança nas propriedades (frequências
A evolução é o processo de transformações hereditárias e adaptações que vem ocorrendo nos seres vivos desde que surgiram no planeta Terra.
 Fixismo (Criacionismo) - Até o século XVIII - Todos os seres vivos foram criados por um ser supremo Deus. - Uma vez criadas as espécies eram imutáveis (fixas). ADAPTAÇÃO O termo adaptação refere-se ao
Fixismo (Criacionismo) - Até o século XVIII - Todos os seres vivos foram criados por um ser supremo Deus. - Uma vez criadas as espécies eram imutáveis (fixas). ADAPTAÇÃO O termo adaptação refere-se ao
MARCADORES MOLECULARES
 MARCADORES MOLECULARES Genética Melhoramento na agricultura até séc. XIX arte e seleção inconsciente 1900s - Descoberta dos princípios genéticos 1920-50 - Melhoramento genético científico 1970-80 - Utilização
MARCADORES MOLECULARES Genética Melhoramento na agricultura até séc. XIX arte e seleção inconsciente 1900s - Descoberta dos princípios genéticos 1920-50 - Melhoramento genético científico 1970-80 - Utilização
Diversidade genética de populações (princípios e
 Aula 8 Diversidade genética de populações (princípios e ferramentas estatísticas) População Um grupo de indivíduos da mesma espécie que potencialmente podem se acasalar, produzindo descendência, e vivem
Aula 8 Diversidade genética de populações (princípios e ferramentas estatísticas) População Um grupo de indivíduos da mesma espécie que potencialmente podem se acasalar, produzindo descendência, e vivem
Genética de Populações. Ricardo L. R. de Souza Depto de Genética - UFPR
 Genética de Populações Ricardo L. R. de Souza Depto de Genética - UFPR Populações Genética de populações a palavra população geralmente não se refere a todos os indivíduos de uma espécie, mas sim a um
Genética de Populações Ricardo L. R. de Souza Depto de Genética - UFPR Populações Genética de populações a palavra população geralmente não se refere a todos os indivíduos de uma espécie, mas sim a um
A PERIODIZAÇÃO HISTÓRICA. Paleolítico: Pedra Lascada Mesolítico: Pedra Polida Neolítico: Idade dos Metais
 A PRÉ-HISTÓRIA A PERIODIZAÇÃO HISTÓRICA Paleolítico: Pedra Lascada Mesolítico: Pedra Polida Neolítico: Idade dos Metais UTENSÍLIOS DE PEDRA LASCADA Lascas cortantes UTENSÍLIOS DE PEDRA POLIDA Anzóis de
A PRÉ-HISTÓRIA A PERIODIZAÇÃO HISTÓRICA Paleolítico: Pedra Lascada Mesolítico: Pedra Polida Neolítico: Idade dos Metais UTENSÍLIOS DE PEDRA LASCADA Lascas cortantes UTENSÍLIOS DE PEDRA POLIDA Anzóis de
SUBESTRUTURA POPULACIONAL E FLUXO GÊNICO
 SUBESTRUTURA POPULACIONAL E FLUXO GÊNICO AULA 5 Mariana Fonseca Rossi mfonsecarossi@gmail.com RELEMBRANDO... Equilíbrio de Hardy-Weiberng: RELEMBRANDO... Equilíbrio de Hardy-Weiberng: Frequência dos genótipos
SUBESTRUTURA POPULACIONAL E FLUXO GÊNICO AULA 5 Mariana Fonseca Rossi mfonsecarossi@gmail.com RELEMBRANDO... Equilíbrio de Hardy-Weiberng: RELEMBRANDO... Equilíbrio de Hardy-Weiberng: Frequência dos genótipos
O CONTINENTE AMERICANO
 O CONTINENTE AMERICANO Possuindo uma área de 42.560.270 Km2 o continente americano é o segundo maior continente da Terra ocupando 28 % das terras emersas, ficando atrás apenas do continente asiático. Mapa-múndi
O CONTINENTE AMERICANO Possuindo uma área de 42.560.270 Km2 o continente americano é o segundo maior continente da Terra ocupando 28 % das terras emersas, ficando atrás apenas do continente asiático. Mapa-múndi
GOIÂNIA, / / PROFESSOR: FreD. DISCIPLINA: Biologia TOOOP SÉRIE: 9º. ALUNO(a):
 GOIÂNIA, / / 2016 PROFESSOR: FreD DISCIPLINA: Biologia TOOOP SÉRIE: 9º ALUNO(a): Lista de Exercícios P1 4º Bimestre Prova dia 21/10 No Anhanguera você é + Enem Antes de iniciar a lista de exercícios leia
GOIÂNIA, / / 2016 PROFESSOR: FreD DISCIPLINA: Biologia TOOOP SÉRIE: 9º ALUNO(a): Lista de Exercícios P1 4º Bimestre Prova dia 21/10 No Anhanguera você é + Enem Antes de iniciar a lista de exercícios leia
Ecologia de Populações e Comunidades
 Ecologia de Populações e Comunidades Profa. Isabel Belloni Schmidt Dept. Ecologia UnB isabels@unb.br Biogeografia O que é Ecologia? O que é Biogeografia Estudo de padrões de distribuição e abundância de
Ecologia de Populações e Comunidades Profa. Isabel Belloni Schmidt Dept. Ecologia UnB isabels@unb.br Biogeografia O que é Ecologia? O que é Biogeografia Estudo de padrões de distribuição e abundância de
Sistemas de Acasalamento. Acasalamento ao acaso. Acasalamento ao acaso. O ciclo de vida de uma população. Pressupostos de Hardy Weinberg.
 Pressupostos de Hardy Weinberg Produção de alelos: 1 locus autossômico 2 alelos sem mutação 1ª Lei de Mendel União de alelos: Sistema de acasalamento aleatório Tamanho populacional infinito Troca genética
Pressupostos de Hardy Weinberg Produção de alelos: 1 locus autossômico 2 alelos sem mutação 1ª Lei de Mendel União de alelos: Sistema de acasalamento aleatório Tamanho populacional infinito Troca genética
Sibele Borsuk
 Genética de populações Sibele Borsuk sibele@ufpel.tche.br A Genética de Populações trata das frequências alélicas e genotípicas nas populações e as forças capazes de alterar essas frequências ao longo
Genética de populações Sibele Borsuk sibele@ufpel.tche.br A Genética de Populações trata das frequências alélicas e genotípicas nas populações e as forças capazes de alterar essas frequências ao longo
Biogeografia Histórica. Cenozóico
 Biogeografia Histórica Cenozóico Ao final do Mesozóico: Todos os continentes afastados Oceanos em expansão Continentes em deriva latitudinal e/ou longitudinal Revolução climática nos continentes Choque
Biogeografia Histórica Cenozóico Ao final do Mesozóico: Todos os continentes afastados Oceanos em expansão Continentes em deriva latitudinal e/ou longitudinal Revolução climática nos continentes Choque
Genética de Populações. Ricardo L. R. de Souza Depto de Genética - UFPR
 Genética de Populações Ricardo L. R. de Souza Depto de Genética - UFPR Populações Genética de populações a palavra população geralmente não se refere a todos os indivíduos de uma espécie, mas sim a um
Genética de Populações Ricardo L. R. de Souza Depto de Genética - UFPR Populações Genética de populações a palavra população geralmente não se refere a todos os indivíduos de uma espécie, mas sim a um
AS PAISAGENS NATURAIS E A FORMAÇÃO DO TERRITÓRIO DA AMÉRICA ANGLO SAXÔNICA
 AS PAISAGENS NATURAIS E A FORMAÇÃO DO TERRITÓRIO DA AMÉRICA ANGLO SAXÔNICA FORMAÇÃO DO TERRITÓRIO A colonização na América do Norte iniciou-se depois quando comparada ao processo de colonização da América
AS PAISAGENS NATURAIS E A FORMAÇÃO DO TERRITÓRIO DA AMÉRICA ANGLO SAXÔNICA FORMAÇÃO DO TERRITÓRIO A colonização na América do Norte iniciou-se depois quando comparada ao processo de colonização da América
Genética de Populações e Evolução
 Genética de Populações e Evolução Populações Genética de populações a palavra população geralmente não se refere a todos os indivíduos de uma espécie, mas sim a um grupo de indivíduos da mesma espécie
Genética de Populações e Evolução Populações Genética de populações a palavra população geralmente não se refere a todos os indivíduos de uma espécie, mas sim a um grupo de indivíduos da mesma espécie
POR QUE A EVOLUÇÃO BIOLÓGICA É UMA TEORIA CIENTÍFICA E NÃO UMA LEI CIENTÍFICA?
 POR QUE A EVOLUÇÃO BIOLÓGICA É UMA TEORIA CIENTÍFICA E NÃO UMA LEI CIENTÍFICA? O que seria uma lei científica? Lei científica é uma explicação para um fenômeno natural que apresenta regularidade, de tal
POR QUE A EVOLUÇÃO BIOLÓGICA É UMA TEORIA CIENTÍFICA E NÃO UMA LEI CIENTÍFICA? O que seria uma lei científica? Lei científica é uma explicação para um fenômeno natural que apresenta regularidade, de tal
HERANÇA MONOGÊNICA OU MENDELIANA
 HERANÇA MONOGÊNICA OU MENDELIANA Herança monogênica Determinada por um gene apenas; Segregação Familiar; Genótipo: constituição individual para um determinado locus. Fenótipo: é o conjunto das características
HERANÇA MONOGÊNICA OU MENDELIANA Herança monogênica Determinada por um gene apenas; Segregação Familiar; Genótipo: constituição individual para um determinado locus. Fenótipo: é o conjunto das características
ACASALAMENTO PREFERENCIAL
 ACASALAMENTO PREFERENCIAL A endogamia representa um desvio do acasalamento ao acaso no qual o parentesco biológico entre indivíduos afeta a probabilidade de se acasalar. Indivíduos também podem ter sua
ACASALAMENTO PREFERENCIAL A endogamia representa um desvio do acasalamento ao acaso no qual o parentesco biológico entre indivíduos afeta a probabilidade de se acasalar. Indivíduos também podem ter sua
GENÉTICA DE POPULAÇÃO
 GENÉTICA DE POPULAÇÃO Eng. Agr. Msc. Franco Romero Silva Muniz Doutorando em Genética e Melhoramento de Soja Departamento de Produção Vegetal UNESP Jaboticabal/SP Molecular e Biotecnologia Quantitativa
GENÉTICA DE POPULAÇÃO Eng. Agr. Msc. Franco Romero Silva Muniz Doutorando em Genética e Melhoramento de Soja Departamento de Produção Vegetal UNESP Jaboticabal/SP Molecular e Biotecnologia Quantitativa
Curso de Informática Biomédica BG-054 Genética. Prof. Ricardo Lehtonen R. de Souza
 Curso de Informática Biomédica BG-054 Genética Prof. Ricardo Lehtonen R. de Souza Bibliografia Fundamentos de Genética / D. Peter Snustad, Michael J. Simmons Introdução a genética/ Anthony J.F. Griffiths...
Curso de Informática Biomédica BG-054 Genética Prof. Ricardo Lehtonen R. de Souza Bibliografia Fundamentos de Genética / D. Peter Snustad, Michael J. Simmons Introdução a genética/ Anthony J.F. Griffiths...
Ligação, permuta e mapas genéticos: ligação e permuta genética, estimativa da freqüência de permuta
 Universidade Federal de Pelotas FAEM - DZ Curso de Zootecnia Genética Aplicada à Produção Animal Ligação, permuta e mapas genéticos: ligação e permuta genética, estimativa da freqüência de permuta Após
Universidade Federal de Pelotas FAEM - DZ Curso de Zootecnia Genética Aplicada à Produção Animal Ligação, permuta e mapas genéticos: ligação e permuta genética, estimativa da freqüência de permuta Após
A JORNADA DA HUMANIDADE O Povoamento da Terra
 A JORNADA DA HUMANIDADE O Povoamento da Terra A Fundação Bradshaw, em associação com Stephen Oppenheimer, apresenta a jornada global virtual do homem moderno nos últimos 160.000 anos. O mapa mostra pela
A JORNADA DA HUMANIDADE O Povoamento da Terra A Fundação Bradshaw, em associação com Stephen Oppenheimer, apresenta a jornada global virtual do homem moderno nos últimos 160.000 anos. O mapa mostra pela
GENÉTICA DE POPULAÇÕES. Prof. Piassa
 GENÉTICA DE POPULAÇÕES Prof. Piassa DEFINIÇÃO Estudo da distribuição e frequência dos alelos determinantes de características de uma população. EQUILÍBRIO DE HARDY-WEINBERG 1908 Definição: Na ausência
GENÉTICA DE POPULAÇÕES Prof. Piassa DEFINIÇÃO Estudo da distribuição e frequência dos alelos determinantes de características de uma população. EQUILÍBRIO DE HARDY-WEINBERG 1908 Definição: Na ausência
Evolução Estocasticidade (Acaso) e Determinismo (Seleção natural)
 Evolução Estocasticidade (Acaso) e Determinismo (Seleção natural) Os fatores evolutivos e a dinâmica populacional Equilíbrio de Hardy-Weinberg (EHW) (p + q) 2 = p 2 + 2pq + q 2 Professor Fabrício R Santos
Evolução Estocasticidade (Acaso) e Determinismo (Seleção natural) Os fatores evolutivos e a dinâmica populacional Equilíbrio de Hardy-Weinberg (EHW) (p + q) 2 = p 2 + 2pq + q 2 Professor Fabrício R Santos
Deriva Genética. (Genetic drift)
 Deriva Genética (Genetic drift) => Primeiros trabalhos demonstrando a existência de deriva genética, em 1954, pelos Profs. Warwick Kerr e S. Wright: Um dos fundadores da Teoria Sintética da Evolução i)
Deriva Genética (Genetic drift) => Primeiros trabalhos demonstrando a existência de deriva genética, em 1954, pelos Profs. Warwick Kerr e S. Wright: Um dos fundadores da Teoria Sintética da Evolução i)
Tamanho populacional 31/08/2010. Evolução Estocasticidade (Acaso) e Determinismo (Seleção natural) Relação entre o Censo (N) e tamanho efetivo (Ne)
 Evolução Estocasticidade (Acaso) e Determinismo (Seleção natural) Equilíbrio de Hardy-Weinberg (EHW) Os fatores evolutivos e a dinâmica populacional (p + q) 2 = p 2 + 2pq + q 2 Professor Fabrício R. Santos
Evolução Estocasticidade (Acaso) e Determinismo (Seleção natural) Equilíbrio de Hardy-Weinberg (EHW) Os fatores evolutivos e a dinâmica populacional (p + q) 2 = p 2 + 2pq + q 2 Professor Fabrício R. Santos
Genética de Populações Equilíbrio de Hardy-Weinberg
 Genética de Populações Equilíbrio de Hardy-Weinberg Clique para assistir 1. (PUC RS) Para responder à questão, analise o gráfico, que representa diferentes classes fenotípicas de uma característica genética
Genética de Populações Equilíbrio de Hardy-Weinberg Clique para assistir 1. (PUC RS) Para responder à questão, analise o gráfico, que representa diferentes classes fenotípicas de uma característica genética
MARCADORES MOLECULARES
 ESALQ/USP MARCADORES MOLECULARES Base genética dos marcadores e usos no melhoramento de plantas e em estudos de diversidade genética e conservação Departamento de Genética ESTUDO DIRIGIDO 1. O que são
ESALQ/USP MARCADORES MOLECULARES Base genética dos marcadores e usos no melhoramento de plantas e em estudos de diversidade genética e conservação Departamento de Genética ESTUDO DIRIGIDO 1. O que são
Estudo da diversidade com sequências de DNA
 Estudo da diversidade com sequências de DNA estudo do DNA por sequenciação extracção de DNA PCR do fragmento desejado sequenciação estudo do DNA por sequenciação sequenciação 1 2 3 indivíduo A indivíduo
Estudo da diversidade com sequências de DNA estudo do DNA por sequenciação extracção de DNA PCR do fragmento desejado sequenciação estudo do DNA por sequenciação sequenciação 1 2 3 indivíduo A indivíduo
MARCADORES MOLECULARES: DO MELHORAMENTO A CONSERVAÇÃO. Aula 10. Maria Carolina Quecine Departamento de Genética
 MARCADORES MOLECULARES: DO MELHORAMENTO A CONSERVAÇÃO Aula 10 LGN232 Genética Molecular Maria Carolina Quecine Departamento de Genética mquecine@usp.br RELEMBRANDO. kit de genética molecular ENZIMAS DE
MARCADORES MOLECULARES: DO MELHORAMENTO A CONSERVAÇÃO Aula 10 LGN232 Genética Molecular Maria Carolina Quecine Departamento de Genética mquecine@usp.br RELEMBRANDO. kit de genética molecular ENZIMAS DE
Universidade Estadual de Maringá - UEM
 Universidade Estadual de Maringá - UEM Disciplina: Biologia Molecular 6855 T1 e T2 Ciências Biológicas DNA forense Profa. Dra. Maria Aparecida Fernandez TIPAGEM MOLECULAR Somos parecidos no entanto com
Universidade Estadual de Maringá - UEM Disciplina: Biologia Molecular 6855 T1 e T2 Ciências Biológicas DNA forense Profa. Dra. Maria Aparecida Fernandez TIPAGEM MOLECULAR Somos parecidos no entanto com
O que precisamos saber sobre ESPECIAÇÃO?
 Universidade Federal do Paraná Departamento de Genética Setor Ciências Biológicas O que precisamos saber sobre ESPECIAÇÃO? danielbruschi@ufpr.br Prof. Dr. Daniel Pacheco Bruschi Modificado de Moritz, 2002.
Universidade Federal do Paraná Departamento de Genética Setor Ciências Biológicas O que precisamos saber sobre ESPECIAÇÃO? danielbruschi@ufpr.br Prof. Dr. Daniel Pacheco Bruschi Modificado de Moritz, 2002.
Dispersão. Biogeografia. Maratona de Biogeografia Fundamentos de Ecologia e Modelagem Ambiental Aplicados à Conservação da Biodiversidade
 Biogeografia Dispersão Maratona de Biogeografia Fundamentos de Ecologia e Modelagem Ambiental Aplicados à Conservação da Biodiversidade Vivian F. Renó 29/03/2012 O que é dispersão? Movimento de um organismo
Biogeografia Dispersão Maratona de Biogeografia Fundamentos de Ecologia e Modelagem Ambiental Aplicados à Conservação da Biodiversidade Vivian F. Renó 29/03/2012 O que é dispersão? Movimento de um organismo
17/04/2017. Prof. Leonardo F. Stahnke
 Prof. Leonardo F. Stahnke Algumas características são determinadas por genes localizados em cromossomos diferentes. Nesse caso, a segregação dos alelos ocorre de forma independente durante a meiose. Há,
Prof. Leonardo F. Stahnke Algumas características são determinadas por genes localizados em cromossomos diferentes. Nesse caso, a segregação dos alelos ocorre de forma independente durante a meiose. Há,
Biologia e Ciências Naturais. Prof. Paulo Henrique Mueller.
 Prof. Paulo Henrique Mueller GENÉTICA É a ciência que estuda o material hereditário e os mecanismos de transmissão ao longo das gerações. É o processo pelo qual as características são passadas dos genitores
Prof. Paulo Henrique Mueller GENÉTICA É a ciência que estuda o material hereditário e os mecanismos de transmissão ao longo das gerações. É o processo pelo qual as características são passadas dos genitores
GENÉTICA. Profª Fernanda Toledo
 GENÉTICA Profª Fernanda Toledo O que é Genética? É a ciência que estuda os genes e sua transmissão para as gerações futuras. As características são transmitidas de pais para filhos. Essa atividade é coordenada
GENÉTICA Profª Fernanda Toledo O que é Genética? É a ciência que estuda os genes e sua transmissão para as gerações futuras. As características são transmitidas de pais para filhos. Essa atividade é coordenada
LGN GENÉTICA. Aula 2 - Genética da Transmissão I. Antonio Augusto Franco Garcia Filipe Inácio Matias Marianella F. Quezada Macchiavello
 LGN 215 - GENÉTICA Aula 2 - Genética da Transmissão I Antonio Augusto Franco Garcia Filipe Inácio Matias Marianella F. Quezada Macchiavello Departamento de Genética Escola Superior de Agricultura Luiz
LGN 215 - GENÉTICA Aula 2 - Genética da Transmissão I Antonio Augusto Franco Garcia Filipe Inácio Matias Marianella F. Quezada Macchiavello Departamento de Genética Escola Superior de Agricultura Luiz
Ligação e Recombinação Gênica Elaboração de Mapas Cromossômicos QTLs e sua detecção
 Ligação e Recombinação Gênica Miguel H.A. Santana mhasantana@usp.br Genética Básica e Evolução (ZVM 0215) Quarta, 21 de Setembro 2016 Visão geral Meta Importância dos princípios que regem a diversidade
Ligação e Recombinação Gênica Miguel H.A. Santana mhasantana@usp.br Genética Básica e Evolução (ZVM 0215) Quarta, 21 de Setembro 2016 Visão geral Meta Importância dos princípios que regem a diversidade
Herança das características complexas
 Herança das características complexas Genética Mendeliana Os sete traços que Mendel observava em suas ervilhas eram os seguintes: 1. forma ou aspecto da semente: lisa ou rugosa 2. cor da semente: verde
Herança das características complexas Genética Mendeliana Os sete traços que Mendel observava em suas ervilhas eram os seguintes: 1. forma ou aspecto da semente: lisa ou rugosa 2. cor da semente: verde
1) ESTREITO DE BERING: A América começou a ser povoada quando os primeiros grupos humanos vindos da Ásia, atravessaram o Estreito de Bering.
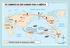 1) ESTREITO DE BERING: A América começou a ser povoada quando os primeiros grupos humanos vindos da Ásia, atravessaram o Estreito de Bering. Essa travessia ocorreu na ultima glaciação, período em que o
1) ESTREITO DE BERING: A América começou a ser povoada quando os primeiros grupos humanos vindos da Ásia, atravessaram o Estreito de Bering. Essa travessia ocorreu na ultima glaciação, período em que o
Seleção Natural. A grande originalidade de Darwin foi a proposta de seleção natural -> essencialmente ligada à demografia de populações.
 Seleção Natural Não é um processo aleatório. A grande originalidade de Darwin foi a proposta de seleção natural -> essencialmente ligada à demografia de populações. Seleção natural => mecanismo chave no
Seleção Natural Não é um processo aleatório. A grande originalidade de Darwin foi a proposta de seleção natural -> essencialmente ligada à demografia de populações. Seleção natural => mecanismo chave no
Lista de Recuperação Não rasure os testes, não use branquinho à tinta.
 Data: /10/14 Bim.: 3º Nome: 9 ANO Nº Disciplina: Biologia Professora: Ângela Valor da Prova / Atividade: 2,0 Nota: Objetivo / Instruções: Lista de Recuperação Não rasure os testes, não use branquinho à
Data: /10/14 Bim.: 3º Nome: 9 ANO Nº Disciplina: Biologia Professora: Ângela Valor da Prova / Atividade: 2,0 Nota: Objetivo / Instruções: Lista de Recuperação Não rasure os testes, não use branquinho à
As Teorias Evolutivas. Princípios da Teoria de Lamarck. Fundamentos da Evolução Biológica. Ideias Evolucionistas - Lamarckismo
 Fundamentos da Evolução Biológica As Teorias Evolutivas Várias teorias evolutivas surgiram, mas destacam-se se as teorias de Lamarck e de Darwin. O EVOLUCIONISMO, OU TEORIA DA EVOLUÇÃO, É A EXPLICAÇÃO
Fundamentos da Evolução Biológica As Teorias Evolutivas Várias teorias evolutivas surgiram, mas destacam-se se as teorias de Lamarck e de Darwin. O EVOLUCIONISMO, OU TEORIA DA EVOLUÇÃO, É A EXPLICAÇÃO
Aula 2: Genética da Transmissão I
 LGN215 - Genética Geral Aula 2: Genética da Transmissão I Antonio Augusto Franco Garcia Maria Marta Pastina Primeiro semestre de 2011 Piracicaba SP Conceitos Essenciais A existência de genes pode ser deduzida
LGN215 - Genética Geral Aula 2: Genética da Transmissão I Antonio Augusto Franco Garcia Maria Marta Pastina Primeiro semestre de 2011 Piracicaba SP Conceitos Essenciais A existência de genes pode ser deduzida
Forças evolutivas. Definição de Evolução. Deriva Genética. Desvios de Hardy-Weinberg
 Definição de Evolução A definição operacional de evolução em nível de deme é mudanças na freqüência alélica ou genotípica. Forças evolutivas Fatores ou processos que podem alterar a freqüência alélica
Definição de Evolução A definição operacional de evolução em nível de deme é mudanças na freqüência alélica ou genotípica. Forças evolutivas Fatores ou processos que podem alterar a freqüência alélica
BG-053 Genética Curso de Nutrição
 BG-053 Genética Curso de Nutrição Prof. Ricardo Lehtonen R. de Souza http://www.ufpr.br/~lehtonen polimorfismos.ufpr@gmail.com Bibliografia Fundamentos de Genética / D. Peter Snustad, Michael J. Simmons
BG-053 Genética Curso de Nutrição Prof. Ricardo Lehtonen R. de Souza http://www.ufpr.br/~lehtonen polimorfismos.ufpr@gmail.com Bibliografia Fundamentos de Genética / D. Peter Snustad, Michael J. Simmons
Colégio Santa Dorotéia
 Colégio Santa Dorotéia Disciplinas: Geografia e História ORIENTAÇÃO DE ESTUDOS Ano: 5º - Ensino Fundamental - Data: 4 / 4 / 2019 CONTEÚDO DA AVALIAÇÃO DE GEOGRAFIA: O planeta Terra e sua superfície. Os
Colégio Santa Dorotéia Disciplinas: Geografia e História ORIENTAÇÃO DE ESTUDOS Ano: 5º - Ensino Fundamental - Data: 4 / 4 / 2019 CONTEÚDO DA AVALIAÇÃO DE GEOGRAFIA: O planeta Terra e sua superfície. Os
A teoria sintética da evolução
 A teoria sintética da evolução De 1900 até cerca de 1920, os adeptos da genética mendeliana acreditavam que apenas as mutações eram responsáveis pela evolução e que a seleção natural não tinha importância
A teoria sintética da evolução De 1900 até cerca de 1920, os adeptos da genética mendeliana acreditavam que apenas as mutações eram responsáveis pela evolução e que a seleção natural não tinha importância
PROCESSO SELETIVO Edital 02/2017
 MINISTÉRIO DA EDUCAÇÃO UNIVERSIDADE FEDERAL DE GOIÁS INSTITUTO DE CIÊNCIAS BIOLÓGICAS PROGRAMA DE PÓS-GRADUAÇÃO EM GENÉTICA E BIOLOGIA MOLECULAR - PGBM INSTRUÇÕES (LEIA ATENTAMENTE) PROCESSO SELETIVO Edital
MINISTÉRIO DA EDUCAÇÃO UNIVERSIDADE FEDERAL DE GOIÁS INSTITUTO DE CIÊNCIAS BIOLÓGICAS PROGRAMA DE PÓS-GRADUAÇÃO EM GENÉTICA E BIOLOGIA MOLECULAR - PGBM INSTRUÇÕES (LEIA ATENTAMENTE) PROCESSO SELETIVO Edital
Evolução Estocasticidade (Acaso) e Determinismo (Seleção natural)
 Evolução Estocasticidade (Acaso) e Determinismo (Seleção natural) Os fatores evolutivos e a dinâmica populacional Equilíbrio de Hardy-Weinberg (EHW) (p + q) 2 = p 2 + 2pq + q 2 Professor Fabrício R Santos
Evolução Estocasticidade (Acaso) e Determinismo (Seleção natural) Os fatores evolutivos e a dinâmica populacional Equilíbrio de Hardy-Weinberg (EHW) (p + q) 2 = p 2 + 2pq + q 2 Professor Fabrício R Santos
Origens da genética e trabalho de Mendel
 Origens da genética e trabalho de Mendel Hereditariedade Gregor Mendel (1865) - Leis da hereditariedade A Pureza dos Gametas A Segregação Independente Associou conhecimentos práticos sobre cultivo de plantas
Origens da genética e trabalho de Mendel Hereditariedade Gregor Mendel (1865) - Leis da hereditariedade A Pureza dos Gametas A Segregação Independente Associou conhecimentos práticos sobre cultivo de plantas
Marco G E O G R A F I A ESPAÇO NATURAL EVOLUÇÃO GEOLÓGICA DA TERRA
 VEST Marco @vestmapamental G E O G R A F I A ESPAÇO NATURAL EVOLUÇÃO GEOLÓGICA DA TERRA A Terra se originou há cerca de 4,6 bilhões de anos, muito depois da formação do Universo, que se supõe tenha surgido
VEST Marco @vestmapamental G E O G R A F I A ESPAÇO NATURAL EVOLUÇÃO GEOLÓGICA DA TERRA A Terra se originou há cerca de 4,6 bilhões de anos, muito depois da formação do Universo, que se supõe tenha surgido
2. O ângulo de incidência é i = 45 0 e o índice de refração do ar é 1. Determine:
 Aluno(a):_ Código: Série: 1ª Turma: Data: / / 03. Sob um ângulo de incidência de 60 0, faz-se incidir sobre a superfície de um material transparente um raio de luz monocromática. Observa-se que o raio
Aluno(a):_ Código: Série: 1ª Turma: Data: / / 03. Sob um ângulo de incidência de 60 0, faz-se incidir sobre a superfície de um material transparente um raio de luz monocromática. Observa-se que o raio
Genética de Populações. Prof. Anderson Moreira
 Genética de Populações Prof. Anderson Moreira Genética de Populações É o estudo do conjunto de genes de uma população em dado momento (pool gênico), detectando suas variações ou sua estabilidade (equilíbrio
Genética de Populações Prof. Anderson Moreira Genética de Populações É o estudo do conjunto de genes de uma população em dado momento (pool gênico), detectando suas variações ou sua estabilidade (equilíbrio
BIO-103 Biologia evolutiva
 BIO-103 Biologia evolutiva Excerto da apostila de Genética de populações para as disciplinas BIO-305 e BIO-212 (1986-2006), vários autores. EQUILÍBRIO DE HARDY-WEINBERG Um dos aspectos importantes do estudo
BIO-103 Biologia evolutiva Excerto da apostila de Genética de populações para as disciplinas BIO-305 e BIO-212 (1986-2006), vários autores. EQUILÍBRIO DE HARDY-WEINBERG Um dos aspectos importantes do estudo
GENÉTICA DE POPULAÇÕES
 GENÉTICA DE POPULAÇÕES Conceito geral de populações Freqüências alélicas e genotípicas Equilíbrio de Hardy-Weinberg Estrutura genética de populações Fatores que afetam o equilíbrio de H-W: mutação, seleção,
GENÉTICA DE POPULAÇÕES Conceito geral de populações Freqüências alélicas e genotípicas Equilíbrio de Hardy-Weinberg Estrutura genética de populações Fatores que afetam o equilíbrio de H-W: mutação, seleção,
Biologia Professor Leandro Gurgel de Medeiros
 Biologia Professor Leandro Gurgel de Medeiros Genética Clássica 1. Conceito: É a ciência voltada para o estudo da hereditariedade, bem como da estrutura e função dos genes. Características Fundamentais
Biologia Professor Leandro Gurgel de Medeiros Genética Clássica 1. Conceito: É a ciência voltada para o estudo da hereditariedade, bem como da estrutura e função dos genes. Características Fundamentais
Genética de Populações. Prof. Ricardo Lehtonen R. de Souza
 Genética de Populações Prof. Ricardo Lehtonen R. de Souza E-mail: ricardo.lehtonen@gmail.com http://www.ufpr.br/~lehtonen VARIAÇÃO EM POPULAÇÕES NATURAIS A maioria das características varia pelo menos
Genética de Populações Prof. Ricardo Lehtonen R. de Souza E-mail: ricardo.lehtonen@gmail.com http://www.ufpr.br/~lehtonen VARIAÇÃO EM POPULAÇÕES NATURAIS A maioria das características varia pelo menos
SISTEMA ANGLO DE ENSINO Prova Geral P-04 G A B A R I T O
 SISTEMA ANGLO DE ENSINO Prova Geral P-0 Tipo M1-06/2009 G A B A R I T O 01. D 10. E 19. D 28. C 37. A 02. A 11. A 20. C 29. A 38. A 03. C 12. B 21. B 30. C 39. A 0. B 13. B 22. C 31. D 0. C 0. C 1. B 23.
SISTEMA ANGLO DE ENSINO Prova Geral P-0 Tipo M1-06/2009 G A B A R I T O 01. D 10. E 19. D 28. C 37. A 02. A 11. A 20. C 29. A 38. A 03. C 12. B 21. B 30. C 39. A 0. B 13. B 22. C 31. D 0. C 0. C 1. B 23.
Teste de Hipóteses em Genética. Professora Lupe Furtado Alle
 Teste de Hipóteses em Genética Professora Lupe Furtado Alle Email: lupealle@gmail.com AS HIPÓTESES GENÉTICAS Após a redescoberta do trabalho de Mendel: Muitos estudos foram realizados para descobrir o
Teste de Hipóteses em Genética Professora Lupe Furtado Alle Email: lupealle@gmail.com AS HIPÓTESES GENÉTICAS Após a redescoberta do trabalho de Mendel: Muitos estudos foram realizados para descobrir o
31/10/2011. Sibele Borsuk. Supondo que o número de indivíduos em uma
 3/0/0 Genética de populações Sibele Borsuk sibele@ufpel.tche.br A Genética de Populações trata das frequências alélicas e genotípicas nas populações e as forças capazes de alterar essas frequências ao
3/0/0 Genética de populações Sibele Borsuk sibele@ufpel.tche.br A Genética de Populações trata das frequências alélicas e genotípicas nas populações e as forças capazes de alterar essas frequências ao
Origem da variação. Conceitos importantes. Diversidade Genética. Variação genética
 Variação genética Origem da variação Professor Fabrício R Santos fsantos@icb.ufmg.br Departamento de Biologia Geral, UFMG Variação fenotípica hereditária Plasticidade fenotípica Densidade populacional
Variação genética Origem da variação Professor Fabrício R Santos fsantos@icb.ufmg.br Departamento de Biologia Geral, UFMG Variação fenotípica hereditária Plasticidade fenotípica Densidade populacional
GEOLOGIA GERAL E TIPOS DE VEGETAÇÕES MUNDIAIS. Prof ª Gustavo Silva de Souza
 GEOLOGIA GERAL E TIPOS DE VEGETAÇÕES MUNDIAIS Prof ª Gustavo Silva de Souza O que é a Geologia? A palavra geologia vem do grego e significa: ESTUDO DA TERRA. Geólogo é como chamamos quem estuda a geologia.
GEOLOGIA GERAL E TIPOS DE VEGETAÇÕES MUNDIAIS Prof ª Gustavo Silva de Souza O que é a Geologia? A palavra geologia vem do grego e significa: ESTUDO DA TERRA. Geólogo é como chamamos quem estuda a geologia.
SELEÇÃO DE MESTRADO 2018 PROGRAMA DE PÓS-GRADUAÇÃO EM ECOLOGIA E EVOLUÇÃO INSTITUTO DE BIOLOGIA ROBERTO ALCANTARA GOMES/UERJ
 PROGRAMA DE PÓS-GRADUAÇÃO EM ECOLOGIA E EVOLUÇÃO INSTITUTO DE BIOLOGIA ROBERTO ALCANTARA GOMES/UERJ PROVA ESPECÍFICA ECOLOGIA E EVOLUÇÃO SELEÇÃO DE MESTRADO 2018 NOME: NÚMERO DE INSCRIÇÃO ASSINATURA: Questão
PROGRAMA DE PÓS-GRADUAÇÃO EM ECOLOGIA E EVOLUÇÃO INSTITUTO DE BIOLOGIA ROBERTO ALCANTARA GOMES/UERJ PROVA ESPECÍFICA ECOLOGIA E EVOLUÇÃO SELEÇÃO DE MESTRADO 2018 NOME: NÚMERO DE INSCRIÇÃO ASSINATURA: Questão
AULA Nº 4. Neste tópico começamos a falar dos aspectos quantitativos da coleta, uma vez
 AULA Nº 4 Neste tópico começamos a falar dos aspectos quantitativos da coleta, uma vez que até aqui tratamos dos aspectos qualitativos. Para tanto teremos que apreender alguns conceitos de genética de
AULA Nº 4 Neste tópico começamos a falar dos aspectos quantitativos da coleta, uma vez que até aqui tratamos dos aspectos qualitativos. Para tanto teremos que apreender alguns conceitos de genética de
Aula 9: Genética de Populações
 LGN215 - Genética Geral Aula 9: Genética de Populações Prof. Dr. Antonio Augusto Franco Garcia Monitora: Maria Marta Pastina Piracicaba - SP Genética de populações Genética de populações: fornece informações
LGN215 - Genética Geral Aula 9: Genética de Populações Prof. Dr. Antonio Augusto Franco Garcia Monitora: Maria Marta Pastina Piracicaba - SP Genética de populações Genética de populações: fornece informações
Teste de Hipóteses em Genética. Professora Lupe Furtado Alle
 Teste de Hipóteses em Genética Professora Lupe Furtado Alle Email: lupealle@gmail.com AS HIPÓTESES GENÉTICAS Após a redescoberta do trabalho de Mendel: Muitos estudos para descobrir o mecanismo de herança
Teste de Hipóteses em Genética Professora Lupe Furtado Alle Email: lupealle@gmail.com AS HIPÓTESES GENÉTICAS Após a redescoberta do trabalho de Mendel: Muitos estudos para descobrir o mecanismo de herança
HISTÓRIA INDÍGENA AULA 2 ANTES DO BRASIL...
 HISTÓRIA INDÍGENA AULA 2 ANTES DO BRASIL... Evolução de hominídios I Pesquisas arqueológicas indicam que os seres humanos descendem de uma linha de primatas que viviam na região central da África. Existem
HISTÓRIA INDÍGENA AULA 2 ANTES DO BRASIL... Evolução de hominídios I Pesquisas arqueológicas indicam que os seres humanos descendem de uma linha de primatas que viviam na região central da África. Existem
ACASALAMENTO PREFERENCIAL
 ACASALAMENTO PREFERENCIAL A endogamia representa um desvio do acasalamento ao acaso no qual o parentesco biológico entre indivíduos afeta a probabilidade de se acasalar. Indivíduos também podem ter sua
ACASALAMENTO PREFERENCIAL A endogamia representa um desvio do acasalamento ao acaso no qual o parentesco biológico entre indivíduos afeta a probabilidade de se acasalar. Indivíduos também podem ter sua
RESUMOS ELABORADOS PELOS ALUNOS DO. Tema Paleontologia
 RESUMOS ELABORADOS PELOS ALUNOS DO Conceitos mais importantes: Tema Paleontologia Paleontologia: é uma das ciências que estuda a evolução da vida na Terra ao longo do tempo geológico; Fóssil: são restos
RESUMOS ELABORADOS PELOS ALUNOS DO Conceitos mais importantes: Tema Paleontologia Paleontologia: é uma das ciências que estuda a evolução da vida na Terra ao longo do tempo geológico; Fóssil: são restos
PRÉ-HISTÓRIA. PERÍODO QUE VAI DO SURGIMENTO DO HOMEM (± 4 MILHÕES DE ANOS ATRÁS) À INVENÇÃO DA ESCRITA (±4.000 a.c.)
 PERÍODO QUE VAI DO SURGIMENTO DO HOMEM (± 4 MILHÕES DE ANOS ATRÁS) À INVENÇÃO DA ESCRITA (±4.000 a.c.) A EVOLUÇÃO DA ESPÉCIE HUMANA: NOME PERÍODO CRÂNIO LOCAL CARACTERÍSTICA Australopithecus 4,2 1,4 milhões
PERÍODO QUE VAI DO SURGIMENTO DO HOMEM (± 4 MILHÕES DE ANOS ATRÁS) À INVENÇÃO DA ESCRITA (±4.000 a.c.) A EVOLUÇÃO DA ESPÉCIE HUMANA: NOME PERÍODO CRÂNIO LOCAL CARACTERÍSTICA Australopithecus 4,2 1,4 milhões
KIT DE RECUPERAÇÃO 1º SEMESTRE/2017 8º ANO A/ B GEOGRAFIA. Profºs. Marcelo e Fernando
 KIT DE RECUPERAÇÃO 1º SEMESTRE/2017 8º ANO A/ B GEOGRAFIA Profºs. Marcelo e Fernando Aluno (a):... Rua São Leopoldo 326-356 - Belenzinho - São Paulo-SP - CEP 03055-000 Telefones: (11) 2692 1196 - (11)
KIT DE RECUPERAÇÃO 1º SEMESTRE/2017 8º ANO A/ B GEOGRAFIA Profºs. Marcelo e Fernando Aluno (a):... Rua São Leopoldo 326-356 - Belenzinho - São Paulo-SP - CEP 03055-000 Telefones: (11) 2692 1196 - (11)
Figura 1 Figura 2 ALOTRÓPICA Figura 3 SIMPÁTRICA
 ANEXO AULA 07 Figura 1 TIPOS DE ESPECIAÇÃO Figura 2 ALOTRÓPICA Figura 3 SIMPÁTRICA Figura 4 PARAPÁTRICA Figura 5 HIBRIDAÇÃO EXERCÍCIOS 1-(FUVEST - 2005) Devido ao aparecimento de uma barreira geográfica,
ANEXO AULA 07 Figura 1 TIPOS DE ESPECIAÇÃO Figura 2 ALOTRÓPICA Figura 3 SIMPÁTRICA Figura 4 PARAPÁTRICA Figura 5 HIBRIDAÇÃO EXERCÍCIOS 1-(FUVEST - 2005) Devido ao aparecimento de uma barreira geográfica,
BioApoio FREQUÊNCIA ALÉLICA ANOS
 1. (Ufpe 2012) O princípio de Hardy- Weinberg tem sido utilizado pelos evolucionistas como uma importante ferramenta para compreender as frequências gênicas nas populações dos seres vivos. Sobre esse assunto,
1. (Ufpe 2012) O princípio de Hardy- Weinberg tem sido utilizado pelos evolucionistas como uma importante ferramenta para compreender as frequências gênicas nas populações dos seres vivos. Sobre esse assunto,
Conceitos Iniciais Pré-História. Prof.ª Maria Auxiliadora
 Conceitos Iniciais Pré-História Prof.ª Maria Auxiliadora Conceitos Iniciais Histor (grego) = Investigação = História História = Fatos ==//== Historiografia = Interpretação da história Cronologia Biênio
Conceitos Iniciais Pré-História Prof.ª Maria Auxiliadora Conceitos Iniciais Histor (grego) = Investigação = História História = Fatos ==//== Historiografia = Interpretação da história Cronologia Biênio
Marcadores Moleculares
 Marcadores Moleculares Marcadores Genéticos Características polimórficas: marcadores de variabilidade genética entre indivíduos, populações, espécies e taxons superiores. Marcadores moleculares: características
Marcadores Moleculares Marcadores Genéticos Características polimórficas: marcadores de variabilidade genética entre indivíduos, populações, espécies e taxons superiores. Marcadores moleculares: características
As terras submersas e a água no globo terrestre
 As terras submersas e a água no globo terrestre CONTINENTES Blocos continentais que resultaram da divisão do continente único: Pangeia. Quatro grandes massas continentais: Continente Tríplipe (África,
As terras submersas e a água no globo terrestre CONTINENTES Blocos continentais que resultaram da divisão do continente único: Pangeia. Quatro grandes massas continentais: Continente Tríplipe (África,
2 vertical: 5 letras, plural. 1 vertical: 11 letras
 1 vertical: 11 letras São organismos originados da alteração molecular do DNA. 2 vertical: 5 letras, plural Fatores que condicionam as características genéticas de um organismo, sendo um proveniente do
1 vertical: 11 letras São organismos originados da alteração molecular do DNA. 2 vertical: 5 letras, plural Fatores que condicionam as características genéticas de um organismo, sendo um proveniente do
Lista de Exercícios - Monitorias
 Monitoria (Biologia - Biologia Molecular) - data (27/06) 01 (UNEAL 2013) Muito tem sido aprendido sobre como as instruções genéticas escritas em um alfabeto de apenas 4 letras os quatros diferentes nucleotídeos
Monitoria (Biologia - Biologia Molecular) - data (27/06) 01 (UNEAL 2013) Muito tem sido aprendido sobre como as instruções genéticas escritas em um alfabeto de apenas 4 letras os quatros diferentes nucleotídeos
Variação ambiental Poligenes
 Fenótipo é qualquer característica mensurável. Genótipos Mendelianos são sempre discretos, mas fenótipos podem ser discretos ou contínuos. Ronald A. Fisher Encerrou toda oposição séria ao Mendelismo, e
Fenótipo é qualquer característica mensurável. Genótipos Mendelianos são sempre discretos, mas fenótipos podem ser discretos ou contínuos. Ronald A. Fisher Encerrou toda oposição séria ao Mendelismo, e
Pré-História. Inicia-se com o aparecimento do gênero Homo e vai até cerca de a.c., período em que a escrita surgiu.
 Pré-História Inicia-se com o aparecimento do gênero Homo e vai até cerca de 3.100 a.c., período em que a escrita surgiu. Os conhecimentos que temos da Pré- História são derivados de fragmentos obtidos
Pré-História Inicia-se com o aparecimento do gênero Homo e vai até cerca de 3.100 a.c., período em que a escrita surgiu. Os conhecimentos que temos da Pré- História são derivados de fragmentos obtidos
