Biologia Molecular 2011/2012
|
|
|
- Leandro Padilha Van Der Vinne
- 7 Há anos
- Visualizações:
Transcrição
1 Componentes Químicos da Célula As células são constituídas por elementos químicos tais como: oxigénio, carbono, hidrogénio, azoto, fósforo e enxofre e representam aproximadamente 99% do seu peso: Água compõe 70% do volume celular. Dissolve e transporta materiais na célula e participa em inúmeras reacções bioquímicas; Sais minerais reguladores químicos; Hidratos de Carbono têm a função de fornecer energia através de oxidações e participar em estruturas celulares; Lípidos compostos insolúveis em água e solúveis em éter, como a acetona e o clorofórmio. Participam no fornecimento de energia através da oxidação; Proteínas constituem os polipeptídios (cadeias de aminoácidos), cuja função é na participar na estrutura celular, na defesa (anticorpos), no transporte de iões e moléculas e na catalisação de reacções químicas; Ácidos Nucleicos: o Ácido Desoxirribonucleico (DNA) responsável pela transmissão hereditária das características; o Ácido Ribonucleico (RNA) controla a síntese de proteínas. Trifosfato de Adenosina (ATP) armazena energia nas ligações fosfato; Elementos químicos: como o sódio, o magnésio, o cálcio, o potássio, o ferro, o cloro, o iodo, o bromo e o zinco, estão presentes em pequenas quantidades nas células (4% do total), e desempenham funções igualmente importantes. Estes componentes são classificados em inorgânicos (água e minerais) e orgânicos (ácidos nucleicos, hidratos de carbono, lípidos e proteínas). Molécula da Água Os seres vivos são constituídos por H 2O, iões inorgânicos, pequenas moléculas e macromoléculas. A molécula da água é uma molécula polar possui um dipolo eléctrico (sistema de cargas de igual intensidade e de sinais opostos, separadas entre si por uma distância muito curta) que resulta da geometria da molécula e da diferença de electronegatividade entre o oxigénio e o hidrogénioesta molécula é constituída por 2 átomos de hidrogénio e 1 de oxigénio. Os átomos de hidrogénio estão ligados ao de oxigénio através de ligações covalentes simples (O-H), e é devido às suas cargas, que a molécula H2O tem a sua conformação própria, ou seja, os seus constituintes têm um arranjo específico. Assim, os átomos de hidrogénio afastam-se um do outro (pois têm ambos carga positiva) e aproximam-se do átomo de oxigénio (pois tem carga negativa). Os electrões da ligação química estão dispostos assimetricamente pois são mais atraídos pelo núcleo do oxigénio do que pelo núcleo do hidrogénio. No dipólo natural da molécula de água a carga é apenas parcial pois é mais fraca que a carga de um electrão ou protão, tendo, cada zona polar da ligação O-H, um momento dipolar. É, desta forma, que a molécula de água tem uma região electropositiva e outra electronegativa. 1
2 A polaridade da água faz com que ela seja capaz de estabilizar cargas eléctricas nos solutos (constante dieléctrica relativamente alta) fazendo diminuir a sua atracção ou repulsão, possibilitando assim a ligação com outras moléculas. Pontes de Hidrogénio e as Ligações da Molécula de Água Ligações ou Pontes de Hidrogénio é um tipo de ligação não covalente entre o átomo electropositivo de hidrogénio com átomos electronegativos (O ou N), ou seja, são um tipo de interacção electrostática entre moléculas que ocorre entre as moléculas que têm o átomo de hidrogénio ligado a átomos muito electronegativos, como é o oxigénio. Estas ligações estabelecem-se devido ao facto de as moléculas serem fortemente polares. A ligação é tanto mais forte quanto maior for a polaridade da ligação covalente. É através deste tipo de ligações de hidrogénio que as moléculas de H2O estabelecem ligações fortes entre elas (formando água) ou então ligações com outras moléculas. Na ligação entre as 2 moléculas de H2O, a ligação de hidrogénio é feita entre um átomo de hidrogénio de uma molécula (átomo dador) e o átomo de oxigénio de outra molécula (átomo receptor). Nas várias ligações de hidrogénio que se podem realizar entre as diversas moléculas, este átomo comportase sempre como átomo dador, enquanto o átomo de oxigénio ou azoto, ao qual o hidrogénio se liga, é sempre o átomo receptor. As ligações / pontes de hidrogénio mais importantes a nível biológico são entre: o grupo hidróxido de um álcool e água; o grupo carboxilo de uma acetona e água; grupos peptídicos nos polipeptídios; as bases complementares de DNA. Água como Solvente A água é a substância em maior concentração nas células (70%), bastante usada como solvente. A água é assim, o solvente para muitas substâncias, pois como essas substâncias apresentam grupos polares estabelecem ligações com as moléculas de hidrogénio e destroem a sua estrutura. Assim a solubilidade das substâncias hidrofílicas (as que são atraídas pela água) é explicada pois a água hidrata os seus componentes e destrói a sua estrutura. Os iões positivos são hidratados pelos átomos de oxigénio, e os iões negativos são hidratados pelos átomos de hidrogénio. Existem ainda substâncias hidrofóbicas (sem afinidade com a água, logo não solúveis como é o caso dos grupos apolares e lípidos) e anfipáticas (com características hidrofílicas e hidrofóbicas, caso dos fosfolípidos) Biomoléculas polares, apolares e anfipáticas Moléculas polares moléculas que estabelecem ligações com outras que têm uma carga diferente à que essa molécula possui, ou seja, são moléculas com grupos de átomos capazes de estabelecer ligações com a água. 2
3 Os grupos polares mais importantes são a glicose, glicina, aspártato, lactato e glicerina. Moléculas apolares moléculas que estabelecem ligações entre dois átomos iguais, com igual carga negativa, ou seja, são moléculas com grupos de átomos incapazes de estabelecer ligações com a água. Moléculas anfipáticas moléculas que têm na sua estrutura grupos com características polares, que têm afinidade com a água, mas também apresentam grupos com características não polares. São o caso das moléculas de fenilalanina e o de fosfatodicolina. Comportamento da água A água intervém nas ligações dos complexos enzima-substrato. A molécula de H 2O forma uma cadeia ordenada de água à volta do substrato e da enzima, quando ainda estão separados, formando interacções com esses componentes. Quando o substrato tiver para se ligar à enzima formando o complexo enzimasubstrato, a água necessita de sair rapidamente do meio destes dois componentes para permitir a sua ligação, assim há uma dispersão de água, causada pela interacção da enzima e do substrato no momento anterior à sua ligação. Esta interacção é depois estabilizada pelas pontes de hidrogénio, os iões, e interacções hidrofóbicas. Movimentos da água nas células Célula num meio isotónico a célula está num meio onde a concentração do meio extracelular é igual à concentração do meio intracelular, pelo que não há movimentação de água. As dimensões da célula mantêm-se iguais. Célula num meio hipertónico a célula está num meio onde o meio extracelular tem maior concentração que o meio intracelular, pelo que a célula vai permitir a saída de água. A célula diminui o seu tamanho, ficando plasmolisada. Célula num meio hipotónico - a célula está num meio onde o meio extracelular tem menor concentração que o meio intracelular, pelo que a célula vai permitir a entrada de água criando pressão na sua membrana citoplasmática. A célula aumenta o seu tamanho com a entrada sucessiva de água. A célula animal pode rebentar pois não tem parede celular sofrendo lise; a célula vegetal não rebenta pois tem parede esquelética. Aquoporinas proteínas inseridas na membrana citoplasmática e através das quais se dá rapidamente a passagem de água. Aparecem em grandes quantidades em células que necessitam de fazer sair água rapidamente das células, como é o caso das células dos rins. 3
4 Ácido Nucleicos Os ácidos nucleicos são polímeros de nucleotídios, ou seja, constituem cadeias de moléculas (nucleotídios) unidas quimicamente, que controlam a actividade celular. Nucleotídio são as subunidades dos ácidos nucleicos, constituídos por uma base azotada ligada a um açúcar e a um (ou mais) grupo(s) fosfato(s). Se provocarmos a hidrólise de um nucletídio haverá libertação do fosfato e assim forma-se um nucleosídio. Os ácidos nucleicos são importantes porque suportam a informação genética. São estas macromoléculas que determinam a formação e o desenvolvimento de todas as formas de vida: a sua estrutura primária contém (sob a forma de sequencia de nucleótidos) a informação necessária para a síntese dos milhares de proteínas que regulam todas as funções vitais. Há dois tipos de ácidos nucleicos: DNA (ácido desoxirribonucleico) e RNA (ácido ribonucleico). As estruturas dos ácidos nucleicos Os ácidos nucleicos são formados por sequencias de nucleótidos. Cada nucleótido tem a seguinte estrutura: Base Azotada: as bases azotadas que entram na constituição dos ácidos nucleicos são derivadas das estruturas purina ou pirimidina, São portanto, bases púricas ou pirimídicas, consoante derivem de uma outra estrutura. É a partir dos aminoácidos que se sintetizam as bases purinas e pirimidinas. Bases azotadas do DNA: Adenina Citosina Guanina Guanina Timina Bases azotadas do RNA: Adenina Citosina Guanina Uracilo Bases Púricas (com dois aneis heterocíclico): Adenina e Bases Pirimídicas (com um anel heterocíclico): Citosina Timina (DNA), Uracilo (RNA) Açucar ou Pentose: O açúcar, dos nucleotídios dos ácidos nucleicos, é uma pentose, ou seja, um açúcar de 5 carbonos, que pode aparecer em dois tipos: β-d-ribose presente no ácido ribonucleico, tem um átomo de oxigénio no carbono-2; β-d-2-desoxirribose presente no ácido desoxirribonucleico, não tem o átomo de oxigénio no carbono-2. Assim, as pentoses dos ácidos nucleicos podem ser de dois tipos: Ribose (no RNA) e desoxirribose (no DNA) 4
5 Grupo Fosfato: Os grupos fosfato juntam-se, normalmente, ao carbono-5 da ribose ou da desoxirribose. Os fosfatos tornam a carga, do nucleotídio, negativa. Nas células existem nucleotídios com 1,2 e 3 fosfatos, sendo mono-, di- e trifosfatos. Estes grupos de fosfatos são comuns nas células e cada uma das moléculas apresenta funções bem definidas. O DNA e o RNA só têm 1 fosfato na sua molécula. Nucleotídios e suas Funções Os vários nucleotídios que se podem formar são capazes de ter diversas funções, mas todas elas se encontram bem definidas e especificadas, podem: Transportar energia química através do grupo fosfato. É o caso do nucleosídio Adenina com 3 fosfatos, ou seja, a adenosina trifosfato (ATP); Formar combinações com outros grupos, originando coenzimas. É o caso da coenzima A (CoA) e dos transportadores de monossacarídios (UDPG); Ser usadas como moléculas sinalizadoras específicas da célula. É o caso da cyclic AMP (camp). Ligações fosfodiester Fosfodiester são ligações características dos ácidos nucleicos, ou seja, são ligações que acontecem entre as moléculas de nucleotídios. Estas ligações são formadas em 3 5. Os nucleotídios conseguem ligar-se uns aos outros através de ligações fosfodiester entre o carbono-5 e o carbono-3, para formar os ácidos nucleicos. A sequência linear de nucleotídios num ácido nucleico é, normalmente, representada pela abreviatura de 1 letra, com o fim em 5. 5
6 Composição e Estrutura do DNA O DNA é consituido por um fosfato, base azotada e uma pentose Desoxirribose. É uma molécula longa e helicoidal. Trata-se de um polímero e os seus monómeros são os nucleotidos. O DNA apresenta uma estrutura secundária sob a forma de dupla hélice (Watson e Crick, 1953), formado por duas cadeias complementares antiparalelas. As bandas laterais da hélice são formadas por moléculas de fosfato, que alternam com moléculas de desoxirribose, e os centrais são pares de bases ligados entre si por pontes de hidrogénio. A especificidade de ligações entre as bases é chamada de complementaridade de bases: Adenina liga-se à timina por 2 ligações de hidrogénio (A = T) Guanina liga-se à citosina por 3 ligações de hidrogénio (G C) As cadeias de DNA são cadeias anti-paralelas pois são iniciadas por uma extremidade 5 e terminam em 3. Assim, à extremidade 3 de uma cadeia corresponde a extremidade 5 da outra. Assim, por exemplo, a sequência de bases na cadeia da esquerda é AGTC e a sequência da cadeia complementar (à direita) é GACT. Numa molécula de DNA duas cadeias anti-paralelas que são complementares na sua sequência de nucleotídios estão emparelhadas numa dupla hélice, com cerca de 10 pares de nucleotídios, por volta. A dupla hélice deixa expostas superfícies que podem ser reconhecidas por proteínas específicas, duma forma dependente da sequência, mas sem necessidade de abrir a dupla cadeia O interior hidrofóbico da molécula de DNA é essencial porque estabiliza e protege as ligações de hidrogénio das bases complementares; estas ligações não se formariam se as bases estivessem rodeadas de moléculas de água, pois cada uma dessas ligações seria facilmente substituída por pontes de H com a água. O DNA possui timina em vez de uracilo por uma questão de segurança, ou seja, é uma forma de prevenir possíveis mutações. A citosina (que se liga à guanina) é susceptível de sofrer uma alteração espontânea e originar uracilo (que se liga à adenina). Assim, se o uracilo fosse constituinte do DNA, quando a citosina se convertesse em DNA faria com que os sistemas de reparação do DNA dificilmente reconhecessem essa alteração, por isso, tendo o DNA a timina, caso haja a transformação da citocina para uracilo, este nucleotídio é facilmente reconhecido pelas enzimas, sendo possível a excisão da mutação na sequência. As extremidades da cadeia de DNA não são iguais: uma extremidade tem um grupo 5 -PO4 livre, a outra um grupo 3 -OH livre Compreensão da Molécula de DNA 1940 Erwin Chargoff Adenina = Timina Guanina = Citosina Purinas Pirimidinas (A+G) (T+C) 6
7 1950 Rosalind Franklin e Maurice Wilkins Fizeram estudos relativos à interpretação de radiogramas de difracção dos raios-x através de DNA cristalizado, tendo concluído que a molécula tem uma estrutura helicoidal 1953 James Watson e Francis Crick Apresentam uma proposta de um modelo de dupla hélice para a estrutura do DNA. Segundo Watson e Crick, o DNA é uma longa molécula em forma de dupla hélice. As bandas laterais da hélice são formadas por moléculas de fosfato, que alternam com moléculas de desoxirribose, e os degraus centrais são pares de bases ligados entre si por pontes de hidrogénio. A especificidade de ligações hidrogénio entre as bases é chamada de complementaridade de bases. As duas cadeias de dupla hélice desenvolvem-se em sentidos opostos. À extremidade 3 de uma cadeia corresponde a extremidade 5 da outra, são cadeias antiparalelas. A estrutura do DNA é a mesma em todas as espécies e é universal no mundo vivo. Composição e Estrutura do RNA O ácido ribonucleico, RNA, é um polímero de nucleotídios, geralmente em cadeias simples, formado por moléculas de dimensões muito inferiores à dimensões das moléculas de DNA. O RNA pode dobrar e formar ligações entre os seus nucleotídios, mas continua a ser uma cadeia simples. Estas interacções não-convencionais permitem que a molécula de RNA se converta numa estrutura tridimensional que é determinada pela sequência de nucleotídios. O RNA tem na sua constituição: um grupo fosfato, uma ribose em vez da desoxirribose presente no DNA, o que lhe confere uma desvantagem estrutural pois torna-se menos resistente à hidrólise) e uma base azotada que pode ser adenina, guanina, citosina ou uracilo. As ligações feitas são: A = C e G U. Como se observa, o RNA contém a base uracilo que difere da timina, a base equivalente no DNA. As pontes de hidrogénio que ligam os nucleotídios do RNA são iguais às do DNA. O RNA pode ter várias funções, de entre as quais a função enzimática (riboenzimas). Duplicação do DNA: Mecanismos de Duplicação Num processo de replicação da molécula de DNA é necessário que as duas cadeias de DNA se separem, temporariamente. Isto permite que cada cadeia sirva de base complementar. Assim, forma-se uma cadeia nova com molde na cadeia antiga e é feita sempre por complementaridade, originando 2 moléculas iguais.para a separação das pontes de hidrogénio, tem de existir uma hidrolização das mesmas. Existem 2 mecanismos diferentes para a replicação do DNA: Mecanismo Conservativo as 2 cadeias separam-se e sintetizam-se novas cadeias. Após a duplicação, as 2 cadeias antigas voltam-se a unir e ficam na célula-mãe, enquanto as 2 cadeias recém-formadas associam-se e vão para a nova célula formada; 7
8 Mecanismo Semi-conservativo após a duplicação, cada uma das moléculas aparece com 1 cadeia nova e 1 cadeia antiga. Ou seja, é uma hipótese de auto duplicação do DNA, na qual, cada uma das 2 cadeias da molécula serve de molde à síntese de uma nova cadeia complementar, constituindo-se duas moléculas idênticas. Cada uma destas moléculas conserva assim uma cadeia da molécula original, ou seja, há uma réplica integral de cada uma das cadeias constituintes da molécula original através da polimerização ordenada de nucleotídios, segundo a regra de complementaridade de bases. Para comprovar qual o tipo de mecanismo que o DNA usa para se replicar usou-se se a experiencia de Meleson-Stahl Interpretação da experiência: 1) As bactérias de E.coli foram cultivadas num meio de cultura contendo um isótopo leve de azoto 14 N. As bactérias produziram bases azotadas contendo 14 N que ficaram integradas no seu DNA. O DNA, depois de extraído, foi centrifugado, verificando-se que ficava na parte superior do tubo DNA leve 2) As bactérias E. coli foram cultivadas num meio de cultura contendo um isótopo pesado de 15 N. As bactérias produziram bases azotadas contendo 15 N que ficaram integradas no seu DNA. O DNA depois de extraído, foi centrifugado, verificando-se que ficava no fundo do tubo DNA pesado 3) As bactérias que foram cultivadas em 15 N são agora transferidas para um meio com 14 N. O azoto 14 N é incorporado no seu DNA e o que se obtem é uma molécula de DNA com uma cadeia com 15 N e outra com 14 N, isto é, apresenta uma densidade intermédia entre o DNA só com 15 N e o DNA só com 14 N DNA híbrido Em suma, os resultados desta experiencia apoiam o modelo de replicação semi-conservativa que se processa segundo a regra da complementaridade de bases. A replicação semi-conservativa ocorre da seguinte forma: 1. As duas cadeias da dupla hélice, na presença de enzimas específicas (DNA-polimerases) separam-se por ruptura das ligações hidrogénio; 8
9 2. Cada uma das cadeias serve de molde à formação de uma cadeia complementar, sendo utilizados nucleotídios, que existem livres na célula 3. Formam-se, simultaneamente, duas cadeias novas de desoxirribonucleótidos de acordo com a regra de complementaridade de bases. Estas novas cadeias são complementares das duas cadeias originais, sendo cada uma anti-paralela em relação à que lhe serviu de molde. Neste processo de replicação semiconservativa as novas moléculas de DNA formadas são idênticas à molécula original, sendo portadoras de uma cadeia antiga e de uma cadeia recém-formada. Síntese Química de DNA A adição de um desoxirribonucleótido ao fim 3 da cadeia de polinucleótidos é a reacção fundamental para a síntese de DNA. O emparelhamento entre um desoxirribonucleosídeo de trifosfato livre e uma cadeia de DNA existente leva à formação de uma nova cadeia de DNA, o que implica que haja uma sequência de nucleotídios complementares. Os nucleotídios livres na célula para a duplicação do DNA são o trifosfato, no entanto, só entram na síntese monofosfatos por complementaridade, logo, os outros 2 fosfatos são libertados sob a forma de pirofosfato. DNA Polimerase Kornberg liderou a equipa que demonstrou que era a DNA polimerase a responsável pelo estabelecimento da ligação entre a cadeia antiga (molde) e a nova, na duplicação de DNA. DNA polimerase catalisa uma nova cadeia de DNA durante a replicação do DNA, usando uma cadeia já existente como molde. A DNA polimerase apresenta locais de ligação e locais de entrada dos novos nucleotídios. Esta é única, só com uma extremidade 3 livre, pelo que a síntese só se realiza de 5 para 3, originando problemas no modelo. Durante a duplicação, as cadeias vão-se separando, formando uma forquilha de duplicação onde são adicionados nucleotídios livres, formando, por complementaridade, a nova cadeia. A DNA polimerase fixa-se a uma das cadeias de DNA e vai incorporando desoxinucleosídios trifosfatos para formar a nova cadeia, que após estar na posição devida, liberta os pirofosfatos, permanecendo na cadeia nova apenas o nucleotídio correcto. Propriedades gerais das DNA polimerases: Necessidade absoluta de cadeia molde Necessidade absoluta de um polinucleótidopré-existente (normalmente um iniciador ou primer) Síntese na direcção 5 -> 3 9
10 Síntese Contínua e Síntese Descontínua (Síntese semidescontinua do DNA) A síntese de cada uma cada uma das novas cadeias de DNA através das cadeias molde faz-se de diferente forma pela DNA polimerase, devido ao facto de esta só sintetizar de 5 para 3. Assim, o processo de DNA não é idêntico para as 2 cadeias antigas: Processo de síntese contínua (cadeia leading) realiza-se na cadeia 3 -> 5 Processo de síntese descontínua (cadeia lagging) realiza-se na cadeia de 5 -> 3. Os fragmentos de Okasaki são a explicação para a duplicação da cadeia 5 -> 3 da molécula molde. Na síntese contínua, o processo ocorre com a adição contínua de nucleotídios seguidos, até ao fim da sequência. Na síntese descontínua, a formação da nova cadeia tem uma série de pequenos fragmentos que são subsequentemente unidos. Esta cadeia, orientada de 5 -> 3 não pode ser sintetizada nessa direcção, logo a síntese procede-se de uma forma não contínua, numa série de passos repetidos. A descontinuidade produz uma série de pequenos fragmentos de DNA (fragmentos de Okasaki) complementares à cadeia original. Posteriormente, estes fragmentos são unidos através de uma enzima, a DNA ligase (enzima capaz de unir 2 porções de DNA), formando uma cadeia contínua e completando a replicação. Assim, e como a DNA polimerase não consegue iniciar o processo de síntese pois não é capaz de sintetizar os 2 primeiros nucleotídios da sequência existe a DNA primase que é uma RNA polimerase. A DNA primase é então uma enzima que cria uma pequena sequência iniciadora necessária à replicação do DNA. É porntato, uma RNA polimerase que sintetiza este primer, formado por RNA. Assim, as primases vão iniciar o processo de replicação, levando à síntese de um pequeno fragmento de RNA, o iniciador (primer). O primer é uma sequência de RNA, que não apresenta mais de 10 nucleotídios, e que é sintetizada para a DNA polimerase ser capaz de se fixar à cadeia de DNA e iniciar a sua actividade. O facto de o primer ser RNA não tem qualquer interesse pois a DNA polimerase só necessita de uma extremidade 3 para se fixar e iniciar o processo. No processo descontínuo existem vários iniciadores por parte da DNA primase, de modo a que a DNA polimerase se possa fixar em 3, em todos os fragmentos de Okasaki. Assim, quando um primer é sintetizado pela DNA primase, a DNA polimerase fixa-se nesse primer para iniciar um novo fragmento de Okasaki. Quando a sua acção termina e acaba a síntese do fragmento de DNA, outra DNA polimerase retira os fragmentos de RNA iniciadores. e depois a DNA ligase intervém com a capacidade de ligar os fragmentos e formar a cadeia de DNA completa. É necessário a ligação de proteínas à cadeia nuclear para que se fixe e mantenha a DNA polimerase na cadeia até todos os fragmentos de Okasaki estarem sintetizados. 10
11 DNA Ligase A DNA Ligase cataliza a formação de ligações fosfodiester entre grupos terminais 5 -fosfato e 3 hidroxilo justapostos, em moléculas de DNA em cadeia dupla. DNA Helicase A DNA helicase (uma enzima) é um complexo proteico com moléculas de ATP associadas, que quebra as ligações de hidrogénio entre as bases azotadas (purinas ou pirimidinas) de ambas as cadeias de DNA, fazendo com que estas se separem antes da acção da DNA polimerase. Esta enzima move-se ao longo da cadeia dupla de DNA utilizando energia da hidrólise do grupo fosfato do ATP para separar as duas cadeias da molécula. A separação das cadeias de DNA é importante para a sua replicação. Ao funcionarem durante o processo de replicação, as helicases recebem ajuda de umas proteínas chamadas topoisomerases, que desenrolam a cadeia, diminuindo a tensão à medida que as helicases avançam, facilitando assim o seu trabalho. Depois de aberta, a cadeia dupla de DNA não se volta a ligar devido à acção de enzimas SSB, que mantêm a cadeia aberta, para poder ser replicada. Proteínas Destabilizadoras da Hélice e DNA Topoisomerase No processo de duplicação, após a actuação da helicase, a molécula de DNA tem a tendência de voltar a estabelecer a ligação de dupla hélice, para isso as proteínas destabilizadoras da hélice ligam-se à cadeia impedindo que estas voltem a formar as pontes de hidrogénio, e permitem a duplicação da molécula, pois mantêm as cadeias separadas de modo a que a DNA polimerase possa actuar. As DNA topoisomerases são enzimas que cortam temporariamente ligações fosfodiester fazendo com que não haja movimento de rotação. Têm então como função impedir a rotação da molécula de DNA quando esta está a ser separada, impedindo grandes gastos de energia. As proteínas vão quebrar a molécula de DNA permitindo que a parte cortada pare a rotação e a molécula de DNA permaneça estática. Após a síntese da molécula, as proteínas degradam-se e a molécula de DNA restitui-se. A DNA polimerase e as primases formam um complexo enzimático. A DNA polimerase α inicia o processo de síntese mas é rapidamente substituída, por uma DNA polimerase δ, após ter sintetizado um pequeno fragmento iniciador, continuando a síntese com este ultimo tipo de DNA polimerase. Telomerase Há medida que as células se dividem e os organismos envelhecem, as extremidades do DNA não vão ser sintetizadas, fazendo com que as células entrem em recessão, acabando por morrer. A telomerase é a enzima que acrescenta, em cada uma das extremidades do DNA um bocado de RNA, aumentando a molécula em comprimento. Desta forma, o primer fixa-se nesse segmento, garantindo que toda a molécula de DNA se sintetize. 11
12 Apesar de a telomerase existir, esta deixa de actuar ao longo do tempo com o envelhecimento das células até ficar inactiva, e assim a molécula de DNA não é completamente sintetizada, entrando em cinestesia. Desta forma, a enzima telomerase garante que a replicação do DNA seja completa e da molécula toda, enquanto é capaz de desempenhar a sua função em pleno. Duplicação do DNA Geral Duplicação do DNA em Procariontes e em Eucariontes : Origens da Duplicação Procariontes Existe uma só origem de duplicação (ou replicação) que origina duas forquilhas de replicação que progridem em direcções opostas. Esta zona de duplicaçã é reconhecida por uma combinação de proteínas, numa zona essencialmente de adenina e timina, pois como é uma ligação dupla a sua separação é mais simples. São duplicações mais rápidas que nos eucariontes, pois a molécula de DNA nestes seres é mais pequena. Assim, a duplicação apenas se inicia num único local, em toda a molécula e as forquilhas de replicação ocorrem apenas em duas direcções a partir de um mesmo local. Os cromossomas são circulares e mais complexos, uma vez que têm um papel importante na duplicação e no crescimento celular. A replicação é bastante rápida. Eucariontes A duplicação nos seres eucariontes tem que ser regulada de forma muito precisa para assegurar que o genoma inteiro é duplicado uma só vez em cada ciclo celular. Há várias origens da duplicação, visto que a molécula de DNA, nestes seres, é significativamente maior.. Existem vários pontos onde a duplicação se pode iniciar, de modo que a duplicação seja rápida o suficiente para a sobrevivência da célula. A quantidade de DNA duplicado por cada origem de replicação é relativamente pequena A forquilha de replicação movimenta-se em ambas direcções a partir de múltiplas origens de duplicação, num cromossoma eucariótico. Foram estas múltiplas origens de replicação que permitiram a expansão do tamanho do genoma durante a evolução. 12
13 As etapas de replicação do DNA são em traços gerais comuns aos três domínios dos seres vivos (bactéria, archea, eucariotas), variando contudo em complexidade. Alteração do DNA e Principais Mecanismos de Reparação Diariamente, ocorrem várias alterações no DNA, cuja maioria é reversível pois a célula possui mecanismos de reparação da molécula, no entanto algumas persistem, ou não são detectadas, e vão originar verdadeiras mutações. O DNA é a única macromolécula na célula que é reparada. Os principais mecanismos de reparação do DNA assentam no facto de a informação genética existir em duas cópias: uma modificação numa cadeia pode ser reparada através da informação contida na outra. Alterações espontâneas alterações ao nível do genoma humano, que ocorrem espontaneamente, devido a um qualquer erro na replicação do DNA aquando a mitose; Despurinação perda de bases púricas, obtendo-se um nucleotídio sem a base azotada. Leva à perda de guanina ou de adenina; Desaminação perda do grupo amina (NH3), pelo que afecta as bases que apresentam um grupo amina, levando a que a citosina se converta em uracilo,, ou a erros entre outras bases; Alteração das bases alteração de uma base por outra por acção de radicais livres de O 2; Dimerização de bases pirimidicas (como as timinas) estabelecendo-se ligações entre duas bases adjacentes da cadeia, formando um dímero. São induzidas por radiação UV Quebras nas ligações fosfodiester ou nos anéis de desoxirribose Ligaçoes covalentes entre as cadeias complementares Formação de aductos As alterações mais frequentes que causam sérios danos ao DNA são a desaminação e a despurinação, sendo estes erros espontâneos. A desaminação da citosina em uracilo é particularmente frequente. As células são capazes de reparar estes erros beneficiando da presença da timina no DNA. Os mecanismos de alterações são regulados por enzimas. Assim, quando existe um erro, este é reconhecido pelas respectivas enzimas que tentam ultrapassar esse mesmo erro. Excisão de uma base há a remoção da base errada por enzimas específicas (glicosilase remove o uracilo, mas existem enzimas para remover todos os grupos), de seguida, as DNA polimerases de reparação introduzem o nucleotídio correcto e as ligases ligam-no ao resto da cadeia; Excisão de um nucleotídio removem um segmento de Nucleotídios que contém as bases incorrectas e volta a sintetizar esse mesmo segmento correctamente. De seguida, as ligases ligam o segmento de novo à cadeia. Transcrição e Tradução Transcrição e Tradução são os processos através dos quais as células lêm (ou expressam) as instruções genéticas contidas nos genes. 13
14 A informação contida nos genes é expressa através da transcrição e tradução de modo diferencial. Transcrição Transcrição é o processo de formação do RNA a partir do DNA. A síntese de RNA inicia-se em sequências especiais do DNA chamadas promotores. Uma sequencia promotora é uma sequencia assimétrica que se encontra numa das cadeias do DNA, e que marca onde as RNApolimerases devem iniciar a síntese e em que direcção a devem continuar. O RNA formado é o mrna que tem como função informar ao trna a ordem correcta dos aminoácidos a serem sintetizados em proteínas. A transcrição realiza-se no núcleo, ocorrendo a polimerização de ribonucleotidios segundo a regra da complementaridade de bases. Ao dar-se a transcrição, só uma das cadeias de DNA é utilizada como molde (não são necessárias DNA helicases). O complexo enzimático RNA-polimerase fixa-se sobre uma certa sequencia de DNA, desliza ao longo dela, provocando a sua abertura e iniciando-se a transcrição da informação. Depois da enzima actuar, as cadeias voltam a enrolar-se. Ao encaminha-se para o DNA, a RNA polimerase, vai ligar-se a uma região especifica, designada por promotor, através de um factor sigma (proteína) que auxilia a enzima a encontrar o promotor. Depois de transcrever essa zona chega ao local de terminação (local em que acaba a transcrição) onde se dá a conformação do RNA deixando por isso a RNA polimerase de ter afinidade e acabando por se libertar. O processo de transcrição apresenta pontos de semelhança com a replicação do DNA. Intervém a enzima RNA polimerase que é muito menos exigente que a DNA polimerase, pois é capaz de se ligar directamente a qualquer parte de DNA, e também, de separar a dupla hélice para ter uma única cadeia molde, assim sendo, é capaz de iniciar o processo e de formar ligações fosfodiester, no entanto, à semelhança da DNA polimerase, só sintetiza de 5 para 3. A molécula de RNA transcrita é menor que a cadeia de DNA que é transcrita, pois é só um gene funcional dessa cadeia que é transcrito e não toda a sua sequência. Da transcrição originam-se vários tipos de RNA, ou seja, as sequências de DNA não codificam o mesmo tipo de RNA. mrna RNA mensageiro, codifica as proteínas; rrna RNA ribossómico, forma as estruturas básicas dos ribossomas e catalisa a síntese de proteínas; trna RNA de transferência, intervém na síntese de proteínas como adaptador entre o mrna e os aminoácidos; snrna small nuclear RNA, tem variadas funções nos processos nucleares e na transcrição; 14
15 snorna small nucleolar RNA, usado no processamento e modificação química do rrna, sendo importante na transcrição; scarna small cajal RNA, usada para modificar snorna e snrna; mirna microrna, regulador da expressão genética, que bloquea a translação de mrna selectivos, tendo um papel importante na regulação da expressão genética; sirna small interfering RNA, desliga a expressão genética, induzindo a degradação de mrna selectivos e na estabilização de estruturas cromossómicas compactas, tendo um papel importante na regulação genética; Outros RNA intervêm em vários processos celulares. Podem ligar-se vários RNA polimerase à mesma sequência de DNA, transcrevendo-se sequencialmente a molécula, para originar várias cadeias de RNA do mesmo gene. Diferentes tipos de RNApolimerase: Procariontes: um único tipo de RNApolimerase Eucariontes: três tipos de RNA polimerase, 1) RNApolimerase I: sintetiza o RNA 2) RNApolimerase II: sintetiza o RNA mensageiro 3) RNApolimerase III: sintetiza o trna e o RNA nuclear e citoplasmático. Transcrição em procariontes Todos os genes dos procariontes são transcritos pela acção de uma única RNA polimerase. Existe um factor σ (proteico) que se liga à cadeia de DNA para depois ser possível o reconhecimento do promotor, ou seja, de uma sequência específica, inicia-se a transcrição, de modo a que seja detectado pela RNA polimerase e através da qual se inicia a actividade de transcrição. Este RNA liberta-se para o núcleo da célula, não se associando à sequência de DNA, por isso é uma molécula livre para desempenhar a sua função. A escolha da cadeia para ser transcrita é determinada pela ligação do promotor e pelo facto de a transcrição ser só de 5 -> 3. Policistrónico característica do mrna, na qual a mesma sequência de mrna tem a capacidade de codificar mais que uma proteína. Inicio da síntese de RNA: Ligação da holoenzima de RNA polimerase à região promotora Desenrolamento dos dois filamentos de DNA filamento molde Formação de ligações fosfodiester entre os primeiros ribonucleotidios da cadeia. Promotor: pequena sequencia de nucleotidios reconhecida pela RNApolimerase como o ponto onde se deve ligar ao DNA para iniciar a transcrição. Ocorre numa posição especifica antes do gene ser transcrito. 15
16 Promotores apresentam sequencias consenso reconhecidas pela enzima. Essas sequencias ocorrem a cerca de 10 a 35 pares de nucleotidios antes do sitio da transcrição. Sequencias de consenso: -35:5 -TTGACA-3-10:5 -TATAAT-3 Elongação do RNA: Ocorre dentro da bolha de transcrição A polimerase desenrola continuamente o DNA que está à sua frente e enrola o DNA que está atrás, ou seja, o local que já foi transcrito A cadeia de RNA nascente vai-se desligando do DNA à medida que a RNApolimerase se move pela dupla hélice. A região de pareamento DNA/RNA é muito curta (cerca de 3 pares de bases) Terminação do RNA: Ocorre quando a RNA polimerase encontra um sinal de término o complexo de transcrição dissocia-se e há libertaão da molécula de RNA Existem duas maneiras de terminar a transcrição em procariontes. Uma é denominada terminação intrínseca e a outra depende do terminador Rho, uma helicase de DNA/RNA hexomérica. Terminação intrínseca: A RNA polimerase reconhece uma sequencia especifica de DNA denominada por terminador. Estas sequencias normalmente contêm sequencias autocomplementares que formam uma estrutura em forma de ansa no RNA, causando assim a paragem da enzima e consequentemente terminando a transcrição. Terminador Rho: é necessária uma proteína adicional (Rho) para permitir que a RNA polimerase reconheça os sinais de terminação. O terminador Rho dissocia o RNA do complexo RNApolimerase-DNAmolde. Todo o processo ocorre no citoplasma Depois de terminada a transcrição o mrna é logo sujeito a um processo de tradução, até podem ocorrer os dois processos em simultâneo porque a síntese de proteínas ocorre no mesmo compartimento celular. O mrna pode conter informação para diferentes genes policistrónico. Transcrição nos eucariontes Os genes são transcritos pela acção de 3 RNA polimerase diferentes, dependendo do tipo de RNA que é codificado: RNA I, II e III. TFIIH funciona como helicase. É o factor proteico que divide as cadeias para se iniciar a transcrição. Cinases adicionam grupos fosfatos ao substrato. Fosforila a RNA polimerase. Complexo TATA sequência de DNA contida no promotor. 16
17 Sem que o complexo iniciador da transcrição, que é formado pelas enzimas TFIIE e TFIIH, e as cinases estejam ligados à polimerase, esta não é activada e não avança, assim a transcrição não e iniciada. A transcrição termina quando a RNA polimerase encontra uma sequência específica, abandonando o DNA e libertando o RNA. Há outras proteínas que necessitam de se ligar à RNA polimerase para regularem a transcrição da molécula. Os antibióticos interferem na transcrição: Rifampicina inibe as ligações fosfodiester; Actinomicina D inibe ligações ao DNA; Amanitina inibe a acção da RNA polimerase II. A transcrição acontece no núcleo da célula. O DNA primário proveniente da transcrição não é funcional, pelo que tem de sofrer alterações, ou seja, tem de ser processado. Este processamento consiste na remoção dos intrões (sequências não codificantes de aminoácidos), tornando-se funcional. Assim, migra para o citoplasma, nomeadamente, para os ribossomas, onde será traduzido. Monocistrónico característica do mrna, pelo que cada mrna só traduz uma única proteína. Inicio da Síntese do RNA: A polimerase tem de reconhecer um promotor de entre várias sequências que não são promotores; tem de separar a dupla hélice do DNA (feito pela TFIIH) para expor a cadeia molde e iniciar a síntese de RNA. RNA polimerase precisa de factores de transcrição para iniciar a síntese de RNA. Estes factores de transcrição ligam-se à região promotora e formam um complexo de iniciação para a ligação da RNApolimerase e inicio da transcrição Os promotores reconhecidos pela RNApolimerase II são elementos curtos conservados antes do local de transcrição TATA box: TATAAAA na posição 30 Predominam nucleotidios de timina e adenina. O facto de ser constituída por estes nucleotidios confere-lhe uma relativa fragilidade, permitindo a ruptura de pontes de hidrogénio entre os pares A=T das cadeias complementares de DNA e a interacção com factores de transcrição 17
18 específicos dando passagem à RNApolimerase, que assim se posiciona sobre o molde de DNA apto a ser transcrito. CAAT box: GGCCAATCT na posição 80 Os factores de transcrição também são necessários para o inicio da transcrição. O grupo de factores de transcrição que ajuda a iniciar a transcrição de genes chama-se TFII. Cada factor contem uma outra letra na sua designação, TFFIA, TFFIB, TFFID, TFIIE, TFIIF, TFIIH, TFIIJ, formam um complexo no DNA que recruta a RNA polimerase e promove a iniciação da trasncrição. Elongação do RNA: Dá-se a limpeza do promotor, que consiste na remoção de alguns factores da região do promotor. A RNApolimerase vai avançando ao longo do DNA, sintetizando a cadeia de RNA A TIIF permanece associada à RNA polimerase A actividade da RNApolimerase II é muito aumentada por factores de elongação (EF)- Estes factores suprimem a pausa ou paragem ocasional do complexo RNApolimerase-TFIIF Terminação do RNA: A terminação da transcrição é determinada pelas características das regiões especificas do DNA Para terminar a transcrição, a formação de ligações fosfodiester deve ser interrompida Quando o nucleotidio é adicionado à cadeia de RNA, a bolha de transcrição colapsa. O DNA retoma a dupla hélice e a cadeia de RNA assim como a RNApolimerase são libertadas. A terminação da transcrição pela RNApolimerase II não está ainda totalmente percebida. A transcrição ocorre ao nível nuclear. O produto da transcrição é o mrna, e este atravessa o invólucro nuclear e é no citoplasma que é traduzido. Processamento do RNA nos Eucariontes Nos eucariontes, copiar a informação genética contida numa molécula de DNA molde numa RNA transcrito não resulta na síntese de uma molécula de mrna. A molécula de RNA originada pela transcrição denomina-se 45s pré-mrna que tem de ser convertida num RNA maduro através de um mecanismo denominado processamento do RNA (capping, splicing e formação terminal 3 de mrna). O processamento do pre-rrna necessita da acção de proteínas e de RNAs que estão localizadas no nucléolo. O nucléolo contém mais de 300 proteínas e de cerca de 200 pequenos RNAs nucleolares (snornas) que actuam no processamento do pre-rrna. Os snornas encontram-se habitualmente em complexos com proteínas denominando-se snornps. Estes ligam-se ao pre-rrna para desempenharam diversas funções Só os mrna correctamente processados são exportados para o citosol através dos poros nucleares. Alguns snornas são responsáveis pelas clivagens no pré-rrna, mas a maioria actua na síntese de rrna estando envolvido nas modificações das bases do pré-rrna. Capping 18
19 Este processo consiste na adição de uma guanosina metilada na posição 7 à extremidade 5 da molécula de RNA. Imediatamente após formando, o cap liga-se a duas proteínas, CBP20 e CBP80, formando um complexo de ligação ao cap (CBC) este ajuda o RNA a ser apropriadamente processado e exportado para o citoplasma. Após o transporte para o citoplasma, as proteínas nucleares ligadas ao cap são substituídas pelo factor de iniciação da tradução citoplasmatico, elf-4e Desempenha um papel importante no encaixe da subunidade 40s dos ribossomas no mrna A função do capping é proteger o RNA da degradação. Só ocorre no mrna ajudando a célula a distinguilo dos outros tipos de RNA Poliadenilação A função da poliadenilação é facilitar o transporte de moléculas de mrna para o citoplasma, estabilizar as moléculas de mrna no citoplasma (encurtamento da cauda poli(a) está associado com a degradação do mrna) e facilitar a tradução ao permitir uma intensificação do reconhecimento do mrna pelos ribossomas. A sequencia AAUUAAA é o elemento principal para assinalar a clivagem 3 A clivagem ocorre num sitio especifico localizado 15 a 30 nucelotidios a jusante do elemento AAUAAA São adicionados cerca de 200 nucleotidios de adenina pela enzima polia polimerase Spliccing A maioria dos genes de mamíferos são interrompidos por sequências não codificadas (intrões). De modo a gerar uma mensagem funcional a partir do DNA molde tem que ocorrer spliccing, que consiste na excisão de intrões durante o processo de transcrição. As restantes sequencias que permanecem no mrna constituem os exões (normalmente mais curtos que os intrões). Essa excisão de intrões do mrna é um evento muito importante e requer uma extrema precisão das moléculas envolvidas no processo. A exclusão ou o acréscimo de um único nucleotido num exão pode levar a uma alteração da fase de leitura e à produção de uma proteína completamente diferente da original ou mesmo defeituosa. Os intrões são removidos do RNA percursor por enzimas constituídas por um complexo de proteínas e RNA e designado por pequenas partículas nucleares ribonucleoproteicas (snrnp s). Um grupo de snrnp s liga-se ao RNA em cada intrao, corta-o e liga-o de novo à cadeia de RNA libertanto o intrão. As snrnp s são um grande complexo dinâmico denominado spliceossoma, que se forma para remover cada intrão do pré-mrna, pois identifica as terminações 5 e 3 do intrão. Exões e Intrões Os genes têm mais intrões que exões. Os intrões são removidos por recurso a snrna (a maior parte). Os processamentos mais complexos são em mrna de eucariontes e em trna de bactérias e eucariontes. 19
20 Após ser transcrita uma certa sequência de DNA, é produzido um pré-mrna que necessita de ser processado. Este processamento consiste na excisão de intrões pelas snrnp (small nuclear ribonucleoproteins), que faz parte de um mecanismo do RNA splicing. Mecanismos de excisão de intrões Auto-excisão de intrões há activação de 1 nucleotídio extra de guanina que quebra uma ligação fosfodiester, a outra é quebrada pela própria molécula de RNA. o Grupo I - Actua em alguns rrna, mrna e trna do núcleo mitocôndrial e de cloroplastos. Ribozimas ácidos nucleicos que têm a capacidade de remover os próprios intrões; o Grupo II actua em alguns mrna de mitocôndrias e cloroplastos de fungos, algas e plantas; Excisão defeituosa a alteração de nucleotídios faz com que um gene seja reconhecido como um intrão, sendo removido, provocando uma alteração na proteína Excisão alternativa nem todos os intrões são removidos e originam diferentes mrna pelo que surgem diferentes proteínas; o Tropomiosina proteína relacionada com a contracção dos músculos. De acordo com o tipo de músculo em que se encontra, a tropomiosina tem constituições ligeiramente diferentes, que resultam desta excisão alternativa. Processamento de rrna O primeiro RNA a ser transcrito é um precursor do RNA ribossómico (rrna) que não é funcional, sendo quimicamente modificado de modo a dar origem a 3 tipos de RNA. Este processamento do precursor, que ocorre no nucléolo, consiste: Adição de um grupo CH3 (nucleotídios metilados); Aparecimento de uracilos anormais. O snorna estabelece ligações com o precursor, quebrando-o e dando origem aos 3 tipos de rrna, que se associam a proteínas e formam as subunidades dos ribossomas. As proteínas são sintetizadas no citoplasma e entram no núcleo pelos poros, enquanto os ribossomas são sintetizados ao nível do nucléolo. Passam pelos poros, novamente, para o citoplasma e realizam a síntese proteica Processamento de trna Retira-se o intrão e a extremidade 5. Este trna inicialmente não é uma molécula funcional, pelo que a extremidade 3 é alterado pela adição de uma sequência de 1 nucleotídio de adenina e 2 de citosina. É à extremidade 3 que se liga o aminoácido que o TRNA transporta. Nucleotídios anómalos no trna: 20
21 2 grupos metil adicionado à guanina, 2 hidrogénios adicionados ao uracilo; Sulfato substitui o oxigénio num uracilo; Deaminação de uma adenina. Tradução da informação genética (Síntese Proteica) As células de um organismo têm todas a mesma informação genética mas têm diferentes proteínas, ou seja, certos genes são expressos numa célula e noutras não. Pode também acontecer a síntese de uma proteína e ela ser degradada, tornando-se, assim, inactiva. Neste processo há a transformação da mensagem contida no mrna numa sequência de aminoácidos que constituem a cadeia polipetidica. Existem vários intrevenientes. De entre os intervenientes, fazemos especial referência aos ribossomas e ao trna Do DNA para o RNA a informação é transportada pelo mensageiro. Do RNA para as proteínas são necessários adaptadores que adaptem a linguagem do RNA para a proteína, que só é possível através de um processo de tradução proteica. Os codões não reconhecem directamente o aminoácido que especificam, pelo que, para a tradução ocorrer, depende de uma molécula adaptadora que reconhece e se liga a um codão e a um aminoácido (noutro local da sua molécula). Esta molécula adaptadora é o trna. Código Genético A transferência da linguagem do DNA contida na sequência de nucleotídios para a linguagem das proteínas, expressa na sequência de aminoácidos, envolve a codificação dos 22 aminoácidos a partir de quatro nucleotídios diferentes. 3 Nucleotídios consecutivos de DNA constituem o codogene, tripleto que representa a mais pequena unidade de mensagem genética necessária à codificação de um aminoácido. São apenas sequências de 3 nucleotídios, mas a partir destes existem possibilidades suficientes para codificar os aminoácidos conhecidos. Essas sequências vão permitir codificar a ordenação de séries de aminoácidos que caracterizam diversas proteínas. Assim, um código de tripletos forma 1 codão. O código genético é: Código degenerado: contém redundâncias; não existe uma relação unívoca entre o codão e o aa. Existem diferentes codões para o mesmo aa, mas não há nenhuma aa que codifique mais do que um codão. É universal: há uma linguagem que é comum a quase todas as células desde os organismos mais simples aos mais complexos. Não é ambíguo: a um tripleto de nucleótidos corresponde um aminoácido e só um, sempre o mesmo É redundante: significa que vários codões podem codificar o mesmo aa 21
22 Tem codões de finalização (UAA, UAG, UGA): estes codões aparentemente sem sentido, representam sinais de fim de síntese e não codificam aa. Há tripletos com dupla função; O terceiro nucleótido de cada codão é o menos específico; Tem codões sinónimos codões que codificam proteínas iguais. Metionina e triptofano codificados por um só codão Tem um codão de iniciação: AUG (metionina Há 3 grelhas de leitura possíveis para uma determinada sequência de DNA, no entanto, a única possível na realidade é determinada pelo codão de iniciação (AUG) e a sua localização. trna Tem uma conformação tridimensional característica devido ao emparelhamento de bases entre diferentes regiões da molécula. Uma das extremidades forma o anti-codão (sequência de 3 nucleotídios que emparelham com o codão complementar do mrna). Na outra extremidade existe o local de ligação do aminoácido. Os trna são sintetizados como um precursor que é processado, sofrendo remoção de sequências, substituição dos 3 nucleotídios 3 e modificações de bases. Modificações dos nucleotídios do trna: 2 grupos metil adicionados à guanina; 2 grupos hidrogénios adicionados ao uracilo; Sulfato substituiu o oxigénio no uracilo; Desaminação da adenina. A associação do trna ao aminoácido correcto depende das aminoacil-trna sintetases, onde 20 enzimas reconhecem o trna e estabelecem ligações com gastos de energia, ou seja, estabelecem uma ligação covalente entre cada aminoácido e as moléculas de trna apropriadas. Esta reconhece o trna adequado pela sequência do anti-codão e sequências específicas do braço aceitador do trna. Esta actividade depende da hidrólise de ATP pois há a formação de uma ligação energética cuja energia é utilizada posteriormente para a formação da ligação peptídica. Muitos antibióticos utilizados têm a função de intervir com a síntese proteica das bactérias, sem interferir na síntese das proteínas do organismo onde estão aplicados. Estes antibióticos matam as bactérias por inibição da sua síntese proteica devido à ligação no local de ligação dos ribossomas ao trna. Tradução Aminoacil-tRNA Sintetase e Activação dos Aminoácidos Este processo implica o funcionamento sequencial por parte de 2 adaptadores: o aminoacil-trna sintetases e o trna. Aminoacil trna sintetases catalizam a reacção de ligação dos aminoácidos ao trna. O funcionamento destes adaptadores resulta numa selecção de cada aminoácido tendo em conta o seu codão. 22
23 Desta forma os aminoácidos são activados para constituir a cadeia polipeptídica e, consequentemente, a proteína. O reconhecimento do trna pela aminoacil-trna sintetases ocorre ao nível do anti-codão e do braço onde se liga o aminoácido. A sintetase é capaz de reconhecer codões mal sintetizados, rejeitando essas proteínas, ou seja, as aminoaciltrna sintetases são capazes de corrigir a adição de um aminoácido errado, sendo este rejeitado, numa zona de edição da molécula. O anti-codão do trna emparelha com o codão do mrna. Tradução do mrna Esta ocorre de 5 -> 3. Inicia-se num codão AUG (codão de iniciação) ao qual se liga um trna especial (trna iniciador) fixo no aminoácido metionina (posteriormente removida por uma protease). O fim da tradução é indicado pela presença dos codões stop (UAA, UAG, UGA). A cadeia polipeptídica tem início na extremidade amina e cresce pela adição de aminoácidos à extremidade C-terminal utilizando a energia da ligação covalente ao trna. Início da tradução Há ligação do trna iniciador à extremidade 5 do mrna com a ajuda de proteínas (factores de iniciação). Este inicio é determinado por uma série de proteínas, e é a subunidade pequena dos ribossomas a responsável por este processo até à fixação do codão de iniciação. Assim, é a ligação do mrna e de um trna iniciador que transporta a metionina (nos eucariontes) e a formilmetionina (nos procariontes) até à subunidade pequena de um ribossoma. A subunidade grande de um ribossoma liga-se ao conjunto. O ribossoma está assim funcional. Alongamento (continuação) Fase de tradução dos codões sucessivos e da ligação dos aminoácidos. Um novo trna, que transporta um segundo aminoácido, liga-se ao segundo codão. Há a formação de uma 1ª ligação peptídica entre o aminoácido que ele transporta e a metionina. O ribossoma avança 3 bases (um codão). O processo repete-se ao longo do mrna. 23
24 A tradução é sempre feita com gastos de energia e a associação de várias proteínas Finalização Consiste na ligação de um factor de libertação a um codão stop. Os codões de finalização (UAA, UAG, UGA) não têm nenhum anticodão complementar. Quando o ribossoma chega a um destes codões a síntese acaba. A cadeia polipeptídica destaca-se passando por várias transformações. As subunidades dos ribossomas separam-se e ficam livres para iniciar outro processo de tradução de uma nova proteína. Esta síntese é um processo simples e rápido para garantir a sobrevivência da célula. Ribossomas Para uma tradução rápida do mrna para proteína é requerida uma molécula capaz de migrar ao longo da cadeia de mrna, capturar o trna complementar a cada codão e ligar os aminoácidos para formar uma cadeia, este processo é realizado pelo ribossomas. Os ribossomas são organelos que se podem encontrar livres no citoplasma ou associados a membranas do reticulo A grande quantidade de ribossomas no citoplasma é devido à grande capacidade que a célula tem de ter para sintetizar a grande quantidade de proteínas que necessita para as suas funções, garantindo que estas sejam contínuas. Estes ribossomas são constituídos por 2 subunidades, uma grande e outra pequena, que são construídas no núcleo e exportadas para o citoplasma, mais especificamente, estas subunidades são montadas no nucléolo e exportadas para o citoplasma onde se unem apenas quando estão a sintetizar uma proteína a partir da informação de um mrna. Subunidade pequena emparelha o trna com o codão do mrna; Subunidade grande catalisa a formação das ligações peptídicas. As 2 subunidades associam-se uma à outra sobre uma molécula de mrna próxima da sua extremidade 5. A capacidade que o ribossoma tem, de se mover ao longo do mrna e traduzir a sequência de nucleotídios numa sequência de aminoácidos, deve-se à utilização de trna adaptadores. Os ribossomas possuem 4 locais de ligação para moléculas de RNA, que ligam as 2 subunidades: 1 local para o mrna; 3 locais para o trna: o Local A: ligação do aminoacil trna; o Local P: ligação do peptidil-trna: o Local E local de saída do trna 24
25 O ribossoma procariótico está bem estudado pelo que o número de proteínas que possui é bem conhecido, por outro lado, o ribossoma eucariótico não está bem estudado, pelo que não se sabe exactamente o número de proteínas de cada subunidade que possui. Muitos antibióticos matam as bactérias por inibição da sua síntese proteica p devido à ligação ao local de ligação dos ribossomas ao trna. Polirribossomas Normalmente, ocorre uma iniciação múltipla de síntese proteica em cada molécula de mrna que sofre tradução, por isso mal um ribossoma avança na molécula, liga-se outro à extremidade 5 da mesma cadeia. Assim, cada molécula de mrna a sofrer tradução aparece ligada a uma série de ribossomas permitindo a síntese simultânea de várias moléculas de proteínas a partir de uma única molécula de mrna, facto pelo qual a capacidade se designa de polirribossomas. Os ribossomas ligam-se em polirribossomas, de modo a que a mesma cadeia seja sintetizada várias vezes ao mesmo tempo, sendo o processo de síntese, rápido e eficaz. Regulação da Expressão de Genes As células são todas diferentes apesar de possuírem o mesmo DNA, pois só um conjunto de genes são expressos numa determinada célula, num determinado momento. Esta diferenciação celular é muitas vezes um processo irreversível. Os mecanismos que controlam a expressão dos genes operam a vários níveis. Todos os seres vivos possuem proteínas comuns a todas as células, que são necessárias às vias metabólicas normais: house keeping genes, como por exemplo as proteínas estruturais dos cromossomas, RNA polimerases, proteínas ribossomais, proteínas do citoesqueleto, proteínas envolvidas em processos metabólicos básicos, etc Há no entanto, também, proteínas específicas, como por exemplo a hemoglubina, e que variam consoante o tipo de células. Para a expressão de um conjunto específico de genes, por determinados tipos de células, originam-se proteínas específicas. Assim, a concentração de uma proteína pode ser regulada a vários níveis. Alguns house keeping genes expressam-se regularmente, no entanto a expressão, de outros genes, varia em resposta a moléculas sinal que modificam a proteína tornando-a activa, sendo alvo de uma expressão regulada. Controlo positivo quando a transcrição do operão é permitida. Controlo negativo quando a transcrição do operão é inibida. Expressão de Genes A expressão de genes pode ser regulada a vários níveis nos organismos eucariontes: 1) Ao nível do gene -> controlo da transcrição 25
26 2) Ao nível do processamento do RNA 3) Selecção de mrnas: transporte para o citosol e localização 4) Controlo da tradução 5) Degradação de alguns RNAs no citosol 6) Processamento (activação e desactivação) e degradação de proteínas Sequências e Proteínas Reguladoras da Transcrição A transcrição dos genes é regulada por proteínas reguladoras que se ligam a sequências de DNA específicas: interruptores genéticos. Determinadas sequências de nucleótidos, geralmente menos de 20 nucleótidos, funcionam como locais de reconhecimento aos quais se ligam proteínas reguladoras que vão ligar ou desligar genes. Operões proteínas que regulam a expressão dos genes. Ligando-se a um gene, conseguem inibir ou produzir a sua transcrição, ou seja, tornam inactivo ou funcional a expressão de um determinado gene. São unidades genéticas de transcrição que incluem genes estruturais e pelo menos 2 genes reguladores, o promotor e o operador. Gene regulador informação genética para a síntese de uma macromolécula reguladora do funcionamento do DNA, é o gene responsável pela produção de uma proteína que funciona como repressor impedindo a transcrição dos genes estruturais. Este gene é constantemente transcrito e traduzido. Operador porção de DNA, entre o promotor e os genes estruturais, onde se pode ligar uma molécula que regula a transcrição, ou seja, onde se liga o repressor, impedindo a transcrição dos genes estruturais. Promotor sequência de DNA à qual se liga a RNA polimerase para iniciar a transcrição dos genes estruturais, desde que o operador esteja livre, sem o repressor. Genes estruturais genes que codificam a estrutura primária de proteínas, ou seja, codificam a produção das proteínas necessárias ao metabolismo do organismo. Repressor proteína halostérica. Impede que a RNA polimerase se ligue ao promotor. Desta forma, um gene possui: Local de iniciação da transcrição; Local de ligação da RNA polimerase; 26
27 Sequências reguladoras muito curtas em procariontes e muito longas e complexas em eucariontes. Regulação da Expressão de Genes Procarióticos Operão da Lactose (lac) A molécula indutora promove o funcionamento dos genes estruturais, ou seja, só quando a molécula indutora (lactose) está presente é que a transcrição ocorre. Assim, se há presença de lactose, esta liga-se ao repressor, alterando a sua forma e inactiva-o, dando-se a transcrição da sequência de DNA. Este operão é constituído por 3 genes estruturais: lactose permease; β- galactosidase; tiogalactosídae e transacetilase. Controlo Positivo do Operão lac pelo camp-cap CAP proteína activadora do catabolismo. camp (adenosina-3-5 monofosfato) funciona como molécula sinal, produzida em maior quantidade quando não há glucose no meio, ou seja, aumenta quando a glucose diminui. Complexo camp-cap activador da transcrição aumenta a afinidade da RNA polimerase. Quando a glucose está presente, a molécula camp tem uma concentração baixa na célula e: Não existe lactose, a transcrição do operão é inibida; Há lactose presente na célula, existe a transcrição do operão mas em pequena quantidade. Quando a glucose não está presente, a molécula camp tem uma concentração elevada na célula e há lactose presente na mesma, pelo que há a transcrição do operão em grandes quantidades. Regulação da Expressão de Genes Procarióticos Operão do Triptofano O triptofano é um aminoácido que pode ser produzido através de uma cadeia de síntese que mobiliza várias enzimas e surge como resultado da actividade dos genes estruturais. No entanto, o aminoácido também pode surgir do meio externo, não necessitando de ser sintetizado e por isso não é vantajoso produzir as enzimas necessárias à sua síntese. A molécula, quando presente, liga-se ao repressor, activando-o, ou seja, a molécula (como o triptofano) promove o bloqueio da transcrição. 27
28 A proteína repressora do triptofano tem uma determinada configuração pelo que, na ausência do triptofano, a molécula é inactivada. Com o triptofano ligado, a configuração do repressor muda, e pode ligar-se ao promotor. Regulação da Expressão de Genes Procarióticos Operão da Arabinose (ara) A arabinose é um açúcar que para ser utilizado pelas bactérias necessita de 3 proteínas, codificadas por 3 genes do operão da arabinose. A proteína C é a proteína que regula a expressão do operão. Mecanismos de Regulação da Transcrição de Genes em Procariontes Regulação negativa existe uma proteína repressora que bloqueia a transcrição da proteína impedindo a sua síntese. Regulação positiva só na presença de uma proteína é que é activado o complexo que permite a fixação da RNA polimerase e, logo, a síntese da proteína. Regulação da Expressão de Genes em Eucariontes A regulação da expressão de genes é mais complexa nos eucariontes devido à organização do DNA nos cromossomas. É a associação dos histonas ao DNA e o maior, ou menor, grau de compactação da cromatina, que faz com que os promotores estejam encobertos no interior destas estruturas condensadas e seja dificultada a ligação da RNA polimerase ao promotor, assim, existe DNA em regiões de cromatina condensada que se encontra inacessível às proteínas necessárias para se iniciar a transcrição. Para além dos factores inibidores e promotores, necessitam de existir factores iniciadores da transcrição e a presença de proteínas reguladoras (que se podem ligar, ou não, à sequência reguladora). Para a transcrição, 28
29 a molécula de DNA necessita de sofrer modificações para que todas as proteínas entrem em contacto umas com as outras e, também, com o mediador. Para iniciar a transcrição, os factores iniciadores da transcrição associam-se num complexo que separa as cadeias para servirem de molde e fosforilam a RNA polimerase para ficar activa. Quando o promotor está condensado na cromatina e nos histonas, são necessários complexos, de modo a tornar acessível o gene e a transcrevê-lo. Este complexos podem ser complexos remodeláveis da cromatina (complexo TATA), proteínas que eliminam ou substituem os histonas ou enzimas. Assim, para iniciar estes processos tem de existir uma alteração química dos histonas. Mecanismos de Regulação da Expressão por Proteínas Activadoras Mecanismos de Regulação da Expressão por Proteínas Repressoras As proteínas repressoras inibem a transcrição de vários modos: Competição pelo promotor quando o repressor se liga ao promotor, não se liga a RNA polimerase; As 2 proteínas interagem e bloqueiam a acção da RNA polimerase quando o repressor e o activador têm locais de ligação próximos e a superfície de activação interage com o repressor, impedindo a transcrição por parte da RNA polimerase; Interacção directa com o factor de transcrição D a interacção com o primeiro factor de transcrição impede a ligação de outras proteínas; Complexos remodeladores da cromatina associam-se induzem a condensação da cromatina e impedem o acesso ao promotor; Activação de desacetulases pelo repressor; Activação de metilases proteínas que adicionam grupos metil aos histonas provocando a adição de proteínas que impedem a ligação da RNA polimerase; 29
30 Metilação das citocinas do DNA adicionam um grupo metil que leva à associação de proteínas que bloqueiam o acesso da RNA polimerase. Regulação Pós-Transcrição Riboswitch RNA que pode regular a transcrição e impedir a produção de mrna. Da excisão alternativa de intrões originam-se diferentes proteínas. As alterações, que existem na estrutura do RNA são pontuais e devidas a erros na sequência. Regulação da Tradução / Pós-Tradução Inibição da tradução do mrna; Degradação das proteínas (proteossomas) quando uma proteína é sintetizada com algum defeito a célula marca-a com unidades de ubiquitina, sendo enviada para o proteossoma (complexo de proteases especializadas na degradação de proteínas) de modo a ser destruída Regulação por Interferência Mediada por RNA (RNAi) RNAi processo de silenciamento dos genes provocadas por pequenas moléculas de RNA. Intervêm fragmentos de RNA muito pequenos (sirna e mirna) que se encontram na célula. mirna são codificados por genes nas células, nomeadamente por moléculas codificadas no DNA nuclear e transcritas pela RNA polimerase II. Associam-se a proteínas e migram para o citoplasma onde são cortados em pequenos fragmentos que se associam, parcialmente, ao DNA, impedindo que o mrna seja transcrito. Interferem em processos de diferenciação, proliferação celular e de apoptose. Estão codificadas no genoma da célula. sirna moléculas estranhas às células. Levam à associação do RNA a mrna provocando a degradação destes últimos. São moléculas introduzidas nas células por um factor externo. Actualmente, o modo mais utilizado para impedir a transcrição de proteínas é fabricar fragmentos de sirna, para se introduzir na célula e inibir a sua transcrição. DNA Recombinante DNA recombinante é o DNA que resulta da junção não natural de DNA, proveniente de 2 fontes diferentes. Na tecnologia do DNA recombinante utilizam-se técnicas específicas, nas quais se recorre a enzimas de restrição (endonucleases) para cortar o DNA em pontos específicos e a ligases do DNA para reconstruir a molécula, de modo a inserir uma porção de DNA, natural ou sintético, noutro DNA estranho. Técnicas utilizadas na tecnologia do DNA recombinante: 30
31 Corte do DNA em locais específicos utilizando enzimas de restrição o que facilita o isolamento e manipulação dos genes. Electroforese em gel de agarose a separação electroforética é o método mais utilizado para estimar o tamanho do fragmento de DNA ou RNA. Clonagem utilizando vectores / PCR uma molécula é copiada de maneira a gerar muitas moléculas iguais. Hibridação capacidade que uma sequência de ácido nucleico tem para se ligar a uma sequência complementar e serve para identificar uma determinada sequência. Sequenciação determinação da sequência de bases que permite identificar genes e deduzir a sequência das moléculas. Estas técnicas, que resultam na produção de DNA recombinante, foram possíveis devido à descoberta de 2 tipos de enzimas: Enzimas de restrição cortam o DNA em locais específicos gerando um conjunto reprodutível de fragmentos de DNA; DNA ligases restabelecem as ligações fosfodiester entre os grupos fosfato 5 e o hidroxilo 3, numa reacção dependente de energia biológica (ATP). Enzimas de Restrição Verificou-se que algumas bactérias resistiam aos fagos (bacteriófagos vírus que destroem bactérias) porque apresentavam enzimas de restrição que eliminavam o DNA estranho que os fagos inseriam dentro do seu organismo. Enzimas de restrição / Endonucleases enzimas que fragmentam o DNA por hidrólise em locais específicos. Reconhecem uma dada sequência de nucleotídios, fragmentando o DNA sempre que essa mesma sequência é encontrada. Estas são isoladas de bactérias, por isso, o seu nome tem origem no nome do organismo do qual foi isolada, ou seja, apresenta 3 letras em itálico que correspondem ao nome do organismo seguido da sua estirpe e da ordem de identificação (EcoRI Escherichia coli, RY13). Locais de restrição sequencia de nucleotídios (4 a 8) que as enzimas de restrição reconhecem, cortando o DNA nessa mesma sequência. A mesma enzima de restrição corta sempre na mesma sequência de nucleotídios em qualquer DNA, mas depende do organismo onde actua, ou seja, no mesmo organismo, as enzimas de restrição cortam no mesmo local, originando sempre o mesmo conjunto de fragmentos, mas noutro indivíduo origina fragmentos diferentes. As bactérias possuem um sistema que impede que as enzimas de restrição do organismo cortem o seu próprio DNA, ou seja, são protectores de CH3 que impedem a destruição do DNA próprio da bactéria, mas induzem a destruição do DNA desconhecido. 31
32 Este sistema é constituído por enzimas de restrição e metilases (grupos de nucleotídios onde a enzima de restrição actua). Sequência palindrómicas locais de restrição onde a sequência das duas cadeias é igual quando lida de 5 para 3. As enzima de restrição podem cortar o DNA: A direito originam extremidades em cadeia dupla que são mais difíceis de ligar; Em ziguezague cortam a partir de 3 ou de 5, originando extremidades coesivas de cadeia única. Permite o emparelhamento de caudas de diferentes fragmentos de DNA cortados com a mesma enzima, estes fragmentos podem depois ser unidos pela DNA ligase com o consumo de ATP (se as ligações não emparelharem podem-se modificar as extremidades de modo a torná-las compatíveis). Isosesquizómeros e Isocaudâmeros Isosesquizómeros endonucleases de restrição provenientes de organismos diferentes que reconhecem e cortam de igual forma a mesma sequência de bases, ou seja, têm nomes diferentes por virem de organismos diferentes, mas reconhecem a mesma sequência de DNA. São enzimas diferentes que podem ter configurações diferentes, mas cuja função é semelhante por terem a configuração do centro activo semelhante. Isocaudâmeros endonucleases que originam extremidades coesivas compatíveis, ou seja, são endonucleases distintas, cujas extremidades coesivas são compatíveis apesar de serem originadas por organismos diferentes. Factores que Influenciam a Actividade das Enzimas Temperatura Tampão (ph) Electroforese em Gel de Agarose A electroforese em gel de agarose é utilizada na análise de fragmentos de restrição (ou outros fragmentos de DNA), nos quais há separação das moléculas de DNA, de acordo com o seu tamanho, por migração, num campo eléctrico, através de um gel poroso (a velocidade de migração é inversamente proporcional ao logaritmo do tamanho dos fragmentos). O DNA (básico) adquire carga negativa, devido ao grupo fosfato, sendo atraído para o pólo positivo, ou seja, migra, ao longo do gel, no sentido do pólo positivo. 32
33 A agarose é um polissacarídeo extraído de algas, formando um gel com poros maiores do que a policrilamida, dependendo da sua concentração. Quanto menor a concentração de agarose, maior são os poros do gel, e vice-versa. A policrilamida é utilizada normalmente para separar moléculas que diferem pouco em tamanho (ex: 1 nucleótido, géis de sequenciação) O tamanho dos fragmentos de DNA é possível estimar em função da concentração. Na electroforese em gel de agorose usamos Tampao da Amostra (contém glicerol para tornar a amostra mais densa e azul de bromofenol que é um corante que permite visualizar a corrida electroforética) e Tampão TAE. A visualização de bandas é feita normalmente por adição de brometo de etídio que fluoresce através de luz UV. Uma alternativa ao uso de brometo de etidio (que é mutagénico e carcinogénico) é o GelRed(Biotium) ou o SYBR Safe DNA Gel Stain que têm uma reduzida mutagenecidade e a sensibilidade de detecção é semelhante à do brometo de etídio. Clonagem Para estudar genes ou fragmentos de DNA é necessário obter muitas cópias desse gene ou fragmento. Este fragmento de DNA é ligado a um vector (molécula de DNA capaz de se replicar quando introduzida numa células hospedeira). Clone origina-se quando, da multiplicação da célula hospedeira, resultam muitas cópias do fragmento de DNA que foi inserido no vector. Sendo este o processo básico de clonagem. Vectores de clonagem: Bacteriófagos lambda vírus com DNA pequeno que realizam uma multiplicação lítica do vector, juntamente, com o fragmento inserido; Plasmídios ocorrem naturalmente nas células de algumas bactérias, sendo vectores de DNA circular que não codificam informação importante para a célula. Realizam uma multiplicação, juntamente, com a célula hospedeira; Cosmídios são vectores híbridos, artificiais, derivados de fagos e plasmídios, que permitem clonar fragmentos de DNA maiores. Características comuns aos vectores: Origem de replicação permite a sua multiplicação no hospedeiro; 33
34 Local de clonagem múltipla locais onde as enzimas de restrição cortam o DNA e onde é possível introduzir o fragmento de DNA a clonar; Marcador de selecção do gene que codifica. Plasmídio Plasmídios são pequenas moléculas de DNA circular, de cadeia dupla, com a capacidade de replicação independente do genoma da bactéria e que são distribuídos pelas células-filhas. Ocorrem naturalmente nas bactérias, leveduras e alguns eucariontes, podendo, cada célula, apresentar mais que um plasmídio. A molécula possui uma origem de replicação, um gene resistente ao antibiótico, e uma região em que o DNA pode ser inserido, esta região apresenta várias zonas de restrição de modo a facilitar o corte no DNA. A Escherichia coli é um dos organismos, preferencialmente, utilizados em clonagem. Os plasmideos introduzidos em E.coli sofrem transformação. A transformação é um processo que pode ocorrer naturalmente mas em laboratório faz-se um tratamento prévio de maneira a tornar as células competentes, isto é, capazes de incorporarem o DNA. Se a introdução for feita em células eucarióticas animais o processo chama-se transfecção e não transformação. A E.coli é muito usada em clonagem pois, após a inserção do fragmento de DNA a clonar, no plasmídeo, este organismo recolhe-os, naturalmente, e estes entram na célula, replicando-se facilmente, no entanto para este processo ser mais eficiente são utilizadas técnicas para tornar a sua entrada mais rápida. Corta-se o plasmídio e o DNA a clonar, com a mesma enzima de restrição; Juntam-se as extremidades coesivas e o DNA insere-se no plasmídio; O plasmídio é misturado com as células das bactérias e um choque térmico aumenta a permeabilidade da membrana da bactéria, facilitando a entrada do plasmídio; A bactéria entra em contacto com o antibiótico; Sobrevivem as que têm o plasmídio e as outras morrem; Induz-se o crescimento e replicação, obtendo-se muitas células bacterianas resistentes e com vários clones de DNA; Destrói-se as bactérias e extrai-se o plasmídio, cortando-se com a enzima de restrição o DNA clonado, e separa-se esse DNA do plasmídio, por electroforese. A partir de 5 kb, a clonagem torna-se mais complicada, apesar da capacidade máxima de clonagem ser 10 kb. pgem vector comercial, apesar de ocorrerem naturalmente na célula. 34
35 Fagos como Vectores de Clonagem Os fagos derivados do fago lambda podem ser: Fagos de inserção o DNA é metido dentro do genoma do fago, têm uma capacidade de clonagem de 15 kb; Fagos de substituição é removida parte do genoma do fago para inserir o DNA, apresentam maior capacidade de clonagem, de kb. A técnica consiste em: Inserir-se o DNA no vírus e restitui-se a sua estrutura; Libertar-se o vírus na placa, que vai infectar as bactérias, reproduzindo-se; Provocar-se a dissolução da membrana celular dos vírus e restituir-se o DNA clonado; Nesta técnica utilizam-se bactérias de estirpes não resistente ao vírus, de modo a garantir que a técnica é sucedida e as bactérias não resistem ao vírus. Cosmídios São vectores híbridos, derivados de plasmídios e fagos, pelo que têm um processo idêntico ao destes, pelo que é, também, necessário um vírus para infectar as bactérias. Têm uma capacidade até 45 kb. Corta-se, o DNA a clonar, com enzimas de restrição; Liga-se esse DNA ao cosmídio; Empacota-se em partículas fágicas; Os vírus infectam as bactérias; Os cosmídios replicam-se dentro de células bacterianas; Estes cosmídios são isolados e manuseados como plasmídios. Vectores de Expressão Os vectores de expressão são desenvolvidos para a produção em grande quantidade de proteínas celulares raras. São semelhantes aos vectores de clonagem mas possuem promotores fortes que fazem com que um gene introduzido ao seu lado seja activamente transcrito. Os promotores são pequenas sequências que antecedem o local de clonagem, não codificante, que controlam a ligação de proteínas ou RNA polimerase, ou seja, controlam a transcrição do gene. 35
36 A sobreexpressão é produzida pela grande quantidade de mrna do gene clonado, existindo grande quantidade de proteína. É permitida através do promotor que activa a transcrição contínua. Forma agregados insolúveis (corpos de inclusão) relativamente fáceis de purificar. É utilizada a Escherichia coli (sistema in vivo para produzir hormona de crescimento) e a Saccharomyces cerevisiae (para produzir insulina). Sistema de expressão: Introduz-se a sequência de DNA pretendida, após o promotor forte do vector; É promovida a transcrição de RNA em grandes quantidades; Forma-se a proteína necessária em grandes quantidades, sendo obtida através da sua extracção. Biblioteca Genómica Biblioteca genómica colecção de clones dos fragmentos de DNA que representam todo o genoma de um determinado organismo. Ou seja, em vez de e clonar uma porção de DNA que permite a clonagem de todo o genoma do organismo, clonam-se vários fragmentos de DNA cortados, que juntos formam todo o genoma. A biblioteca genómica surge do conjunto desses fragmentos todos clonados. Extrai-se o DNA dos tecidos; Parte-se o DNA genómico total em pedaços que possam ser manipulados, através da digestão com uma enzima de restrição; Clonam-se todos os fragmentos, através das várias técnicas já referidas. Com o genoma completo pode-se saber o funcionamento total do organismo. Biblioteca de cdna Biblioteca de cdna colecção de clones das moléculas de DNA complementares de todos os mrna presentes num determinado tecido. Representa apenas o conjunto de genes que está a ser expresso, num determinado momento, num determinado tecido. É importante, pois em vez de ser clonado todo o DNA é apenas clonado o DNA produzido pelo mrna (cdna), obtendo-se o código já desprovido de intrões, ou seja, cada clone representará um gene sem intrões. Extrai-se o RNA total do tecido; Purifica-se o mrna; Sintetiza-se o cdna através da transcriptase reversa, que faz o processo contrário do mrna, ou seja, sintetiza o cdna a partir do mrna. É convertido cdna a partir do mrna funcional que não apresenta intrões, só as partes funcionais do gene que se pretende clonar; 36
37 Liga-se o cdna a uma sequência de restrição, em cada extremidade; Clonam-se os fragmentos através da ligação do cdna a um vector e com transformação da célula hospedeira. Enquanto, na biblioteca genómica, todo o DNA é fragmentado para ser clonado, na biblioteca de cdna, só o gene que é transcrito, num determinado conjunto de células, é que é clonado. Reacção em Cadeia da Polimerase (PCR) O PCR é um método de amplificação (de criação de múltiplas cópias) que realiza a replicação selectiva e rápida de uma sequência específica de nucleotídios a partir de uma mistura complexa de DNA (amplificação específica), ou seja, faz a multiplicação de uma sequência de DNA pretendida. A amplificação de genes in vitro, ao contrário da clonagem in vitro, tem bastantes aplicações (detecção de mutações, infecções ou vírus, medicina forense). Componentes necessários para a reacção em cadeia da polimerase: DNA que serve de molde e se vai multiplicar. Pode ser extraída de qualquer parte do organismo, sem ser necessária a extracção de células vivas, mas quanto mais puro for, mais fácil é a sua multiplicação; Oligonucleótidos de iniciação (primers F e R) permitem a ligação da DNA polimerase para efectuar a replicação; dntp s Tampão (Mg2+) DNA polimerase (Taq polimerase) enzima que replica o DNA, e que é bastante resistente às variações de temperatura. Uma reacção de PCR corresponde a um conjunto de ciclos, envolvendo cada um desses ciclos: 1) Desnaturação da dupla hélice é conseguida com a incubação do DNA a uma temperatura de 95ºC, formando 2 cadeias simples a partir de uma dupla hélice. Com o aumento da temperatura, a dupla hélice desnatura e forma 2 cadeias simples; 2) Emparelhamento dos iniciadores é necessário diminuir a temperatura (55ºC) para ocorrer a ligação dos primers. Ligação com os primers; 3) Polimerização (síntese) de DNA / Extensão ocorre na temperatura óptima de actividade da Taq polimerase (70ºC). Esta liga-se à região dos iniciadores e promove a polimerização da cadeia de DNA, servindo a outra como molde. A polimerase inicia a cópia de DNA a partir de um primer. 37
38 O processo realiza-se em vários ciclos (20 a 40x), ou seja, repete-se o processo até se obter muitas cópias do DNA. Estes são feitos num termociclador. Podem ser originadas moléculas de DNA de tamanhos ligeiramente diferentes, devido à posição dos primers. Pode-se utilizar um fragmento particular de DNA de uma célula. O molde pode ser: DNA obtendo-se uma cópia de DNA; RNA obtendo-se uma cópia de cdna, sem intrões, sendo necessária a adição da transcriptase reversa para sintetizar cdna. Factores que Afectam o PCR Quantidade e qualidade do DNA molde; Oligonucleótidos de iniciação (primers F e R) Mg2+ - os iões de magnésio formam complexos solúveis com os dntp s e com o DNA, sendo um factor importante para a acção da polimerase; DNA polimerase Taq polimerase, específica para PCR s longos; dntp s; Desenho dos oligonucleótidos de iniciação (primes Forwrd e Reverse) existem programas bioinformáticos para desenhar primers, no entanto deve-se verificar sempre se são a melhor opção segundo: o tamanho; a especificidade; a sequência (evitando sequências repetidas); 50% de citosina e guanina; evitar complementaridade entre os 2 primers; extremidade 5 deve ter mais guanina / citosina; evitar guaninas nas extremidades 3 ; Temperaturas e tempo de emparelhamento; Tempo de extensão; Número de ciclos. Aplicações do PCR Detecção da infecção pelo vírus HIV se o indivíduo estiver infectado aparece, na electroforese, uma banda que num indivíduo, de controlo, saudável não apresenta. Este processo é repetido várias vezes e completada com outros testes antes de ser totalmente confirmada. É utilizada a transcriptase reversa no processo pois necessita-se de sintetizar cdna a partir de mrna. Medicina forense na análise do DNA de indivíduos diferentes, em regiões altamente variáveis de DNA, de modo a identificar-se um determinado indivíduo através de uma amostra. 38
39 Genome Walking Genome walking faz-se uma combinação das técnicas utilizadas e permite obter regiões adjacentes e regiões já conhecidas. DNA genómico é cortado com enzimas de restrição, que cortam a direito; A todos os fragmentos obtidos ligam-se adaptadores (molécula de DNA em cadeia dupla); Amplificação por PCR utilizando AP1 (oligo dentro do adaptador) e GSP1 (oligo dentro da região já conhecida); Faz-se a clonagem e sequenciação dos produtos. Técnicas de Hibridação do DNA Para sabermos a localização de um gene num cromossoma, em que células ocorre transcrição de um gene ou semelhanças entre genomas de diferentes organismos usa-se a hibridação. A hibridição consiste no emparelhamento entre cadeias simples de ácidos nucleicos com sequências complementares (DNA/DNA, DNA/RNA ou RNA/RNA) As técnicas que assentam na hibridação de sondas de DNA implicam a utilização de sequências complementares / alvo, motivo pela qual a complementaridade entre bases se encontra subjacente à técnica. Técnicas que implicam hibridação: Rastreio de bibliotecas; Hibridação in situ; Southern / Northern blotting; PCR; DNA microarrays. Sonda pequena sequência de DNA usada na detecção de uma sequência de nucleotídios complementares (alvo). Esta vai emparelhar e descobrir uma zona de DNA, total ou parcialmente, complementar. A sonda é obtida por síntese química, PCR, etc e pode ser marcada com nucleótidos com fósforo radioactivo (32P) e detectada por autorradiografia ou outro tipo de marcação que pode ser detectada quimicamente ou através de fluorescência. Podem ser sondas homólogas (do mesmo organismo) ou heterólogas (de organismos diferentes, ou seja, é retirada de um organismo diferente, do organismo do qual se aplica). Quanto mais restringentes forem as condições, maior será a especificidade da hibridação. Os factores que afectam a hibridação são: 39
40 Temperatura (ex. 65ºC para 100% de homologia) ph (ph neutro baixa restrigência) Concentração de sais (concentração elevada favorece hibridação) Presença de desnaturantes (> conc. > rest.) Tamanho da sonda Hibridação de Ácidos Nucleicos No rastreio a bibliotecas de ácidos nucleicos é utilizado um ácido nucleico. A chave para detectar ácidos nucleicos específicos é a capacidade de emparelhamento entre cadeias complementares de RNA ou de DNA. Quando submetidas a elevadas temperatura ou a um ambiente alcalino, as cadeias complementares de DNA desnaturam e originam 2 cadeias simples. Ao ser removido o agente desnaturante, estas cadeias renaturam, de acordo com a complementaridade das suas bases azotadas (hibridação). Esta hibridação permite detectar sequências de DNA / RNA que são complementares a uma determinada sequência conhecida, que pode ser um gene ou uma molécula clonada, e tem de ser marcada radioactivamente ou com um composto que facilite a sua detecção visual e, assim, funcionar como sonda. A sonda desnaturada vai hibridar com as sequências de DNA / RNA que lhe são complementares, para tal a sequência deve ser bem conhecida. Estas sondas serão detectadas em virtude da marcação dos híbridos sonda. A sonda procura as sequências de DNA para emparelhamento, de modo a realizar-se a hibridação. Técnica de Southern-Blotting Técnica desenvolvida por Edward Southern que permite identificar um fragmento de DNA específico numa mistura complexa. O southern-blotting pode ser utilizado para saber quantos clones do gene existe no DNA, também pode ser utilizado para o conhecimento sobre a organização dos genes. Para além disso, utiliza-se em testes de paternidade e investigação forense 1) digestão do DNA com uma enzima de restrição 2) separação dos fragmentos por electroforese 3) desnaturação a ph básico e transferência para uma membrana de nitrocelulose/nilon 40
41 4) incubação da membrana com uma sonda com marcação (radioactiva, fluorescente ou outra) 5) Detecção ex. autorradiografia (marcação radiactividade) a membrana é justaposta a 1a película fotográfica e deixada a expor Northern blotting Detecta um RNA específico numa mistura de RNAs Permite analisar se determinado gene está activo, através da detecção, ou não, da sequência pela sonda, e a intensidade da banda dá informação sobre a concentração de RNA. Assim, permite analisar se ocorre transcrição de genes e o padrão de acumulação do seu mrna.a desnaturação do RNA faz-se com formaldeído, segue-se electroforese, transferência e autorradiografia ou outra forma de detecção. Técnica de Western-Blotting Permite detectar uma proteína específica numa cultura / extracto de proteínas, hibridadas por anticorpos (sonda), no qual o primário detecta a proteína e o secundário detecta o anticorpo primário, servindo, desta forma, para tornar o sinal de detecção mais forte. As proteínas podem ser separadas por tamanho ou por tamanho e quantidade. A técnica permite comparar os níveis de acumulação de uma dada proteína em diferentes situações. Hibridação in situ Hibridação in situ envolve hibridação de um fragmento de ácido nucleico a uma preparação cromossómica de DNA desnaturado ou a uma secção de tecido de RNA. 41
42 FISH (fluorescence in situ hybridization) técnica citogenética utilizada para detectar e localizar a presença ou ausência de uma sequência de DNA específica ou um cromossoma. DNA Microarrays São utilizados para estudar a transcrição de muitos genes simultaneamente. A principal aplicação desta tecnologia é a possibilidade de se identificar uma sequência (genética ou mutação genética) e a possibilidade de se determinar níveis de expressão genética e a sua abundância. Esta tecnologia promete monitorizar todo o genoma num único chip para que os investigadores possam obter uma melhor ideia das interacções simultâneas que existem entre milhares de genes. Pode apresentar vários formatos: em cdna ou em oligonucleótidos. Os DNA microarrays permitem a análise da expressão, ou seja, permite a observação dos genes a funcionarem juntos, bem como os genes que a célula activa ou inactiva. Assim: 1) Fragmentos de genes são fixados a uma lâmina 2) mrnas são recolhidos transformados em cdnas e marcados com fluorocromos diferentes 3) Segue-se hibridação com os fragmentos da lâmina 4) Lavagem 5) Observação Sequenciação Genómica Qualquer método de sequenciação de DNA inicia-se com uma população de um fragmento de DNA laboratorial bem definido e com uma base conhecida na extremidade. Princípio de sequenciação obter uma população de moléculas, com uma extremidade inicial comum conhecida e com outra extremidade final diferente, como o último nucleotídio é diferente, originam-se fragmentos de diferentes tamanhos. 42
43 Através de uma fraccionação por electroforese, capaz de separar moléculas que diferem num único nucleotídio, consegue-se estabelecer a sequência de nucleotídios, através da análise desses resultados. Método Didesóxi de Sanger Este método utiliza DNA polimerase para fazer cópias parciais do fragmento de DNA a sequenciar Produzir uma população de moléculas, com uma extremidade inicial igual e com uma extremidade final de diferentes tamanhos, que acabam sempre na mesma base. A reacção tem o DNA a sequenciar; desoxirribonucleótidos trifosfato (dntps); DNA polimerase e um primer para a polimerase poder começar. São feitas 4 reacções, cada uma com uma pequena % de um didesoxirribonucleótido (ddntp) de cada base que fazem terminar a polimerização. Por ex: na reacção com ddatp são produzidos fragmentos parciais que terminam sempre em A. Cada mistura é então separada num gel de sequenciamento por eletroforese para se detectar cada um dos nucleotídeos presentes na sequência de DNA lida. Gel de poliacrilamida separa fragmentos que diferem apenas um nucleótido. A sequenciação pode ser feita com marcadores fluorescentes. Biologia Sintética Biologia sintética construção de vida sintética que aparece na sequência de grandes avanços tecnológicos, ou seja, consiste na concepção e construção de novas partes, módulos e circuitos que não existam no mundo natural e, também, re-desenhar sistemas biológicos já existentes, de modo a que desempenhem novas funções. Os avanços neste campo, levaram a um desenvolvimento de nanotecnologia e de novas técnicas de manipulação de átomos e moléculas, bem como o estabelecimento de modelos computacionais e síntese em larga escala de DNA. Esta biologia reúne uma variedade de campos científicos. É possível a construção de genes, mas a construção de genomas inteiros permanece só na teoria. Não é um processo de clonagem pois não se copia um determinado organismo, pelo contrário, induz-se a sua síntese do nada. Esta síntese de DNA reduz significativamente o tempo necessário para produzir os organismos geneticamente modificados. Últimos anos verificou-se a síntese e utilização rotineira de oligonucleótideos para PCR (primers) ou como sondas (Southern blot) -> fragmentos < 200 bp e de cadeia simples Actualmente já se produzem fragmentos 3-4kb e cadeia dupla; algumas empresas prometem até 40 kb (pelo menos 1 fragmento de 35 kb já foi produzido) Craig Venter foi capaz de produzir sinteticamente um vírus em 14 dias, apesar desta produção apresentar alguns erros. Utilizando um computador, as sequencias nucleotídicas disponíveis e encomendando fragmentos de DNA é teoricamente possível construir genes e até genomas inteiros, assim no futuro genomas simples poderão 43
44 ser rotineiramente sintetizados ou reconfigurados de modo desempenharem funções específicas. As principais potenciais aplicações da biologia sintética são a produção de energia, bio-remediação, indústria farmacêutica, agricultura etc A biologia sintética dá-nos a possibilidade de ler o código genética bem com a possibilidade de escrever (uma letra de cada vez) o código genético e assim criar vida artificial. Como seria de esperar isto levanta enormes problemas éticos. A biologia sintética trabalha ao nível do codão (abaixo do nível de gene), identificando codões para construir novos conjuntos de informação. Devido à quantidade de codões existentes, é possível escolher codões quando se quer exprimir um determinado aminoácido, consoante as características necessárias, tendo-se em conta padrões de optimização do codão. Existem 64 codões (43) mas só 20 aminoácidos. Deste modo é possível escolher vários codões quando se quer exprimir um determinado aa (optimização do codão). Ex. um det. codão pode funcionar melhor em bactérias e outro em plantas, embora ambas produzam o mesmo aa. O código genético não é tão linear como o código de um computador, os genes actuam em rede produzindo proteínas que promovem ou suprimem a expressão de outros produtos génicos (sist. de regulação que controla a quantidade e/ou altura em que determinada substância é produzida) No código genético os genes actuam em rede produzindo proteínas que promovem, ou suprimem, a expressão de outros produtos génicos, podendo, então, falar-se de uma via genética (em vez de metabólica). Esta via para ser alterada, necessita que várias áreas diferentes sejam alteradas e colocadas num cromossoma sintético. Princípios básicos da Biologia Sintética I) Utilização de partes biológicas padronizadas e bem caracterizadas II) Hierarquização III) Síntese química de DNA -> sequências que não existem na natureza Partes Padronizadas Bio-legos / Partes padronizadas sequências de nucleotídios com características específicas, que podem funcionar como: Promotor - que liga ou desliga as várias reacções; Sensor de várias partículas e condições; Gene modificado. 44
45 Estas sequências são porções de DNA desenhadas para desempenhar uma função específica e que são compatível com outros bio-legos de modo a poderem construir-se módulos e circuitos que depois serão integrados num chassis. MIT base de dados e repositório de partes biológicas padronizadas, de modo a que possam ser utilizadas por outros e montadas para gerar sistemas genéticos mais complexos (módulos e circuitos). Chassis Chassis têm de ser organismos bem estudados com o genoma totalmente conhecido (Escherichia coli, Bacillus subtilis, levedura). São organismos que servem de suporte para a síntese dos bio-legos. Principais Áreas de Investigação em Biologia Sintética Genomas mínimos conjunto necessário de genes que um organismo necessita para viver, em determinadas condições. Inactivando os genes 1 a 1, consegue-se concluir que determinado organismo, em determinadas condições, pode sobreviver com cerca de x genes. Estes estudos são utilizados para se desenvolver organismos semelhantes aos originais mas com menos genes de modo a serem mais simples e capazes de serem utilizados como chassis, na biologia sintética. Bio-legos utilizados com o objectivo de serem construídos sistemas biológicos mais simples a partir de peças (bio-legos) que estão bem descritas e caracterizadas (cujo comportamento é previsível). Células artificiais objectivo de criar uma protocell que necessita de ter um metabolismo que capte e gere energia, na qual a molécula armazene informação, e possuam uma membrana que delimite a célula. A membrana artificial utilizada é uma monocamada de lípidos (mais simples e de fácil manuseamento). Desenvolveram-se PNA (em vez de DNA) que possuem péptidos (em vez de monossacarídios), têm as mesmas bases, mas são mais seguros em termos de risco biológico. Modificação das vias genéticas / metabólicas de modo a produzir compostos de interesse comercial. Expansão do código genético criam moléculas diferentes, mas que como o DNA, reproduzem e transmitem a informação genética (moléculas com mais bases). Este DNA expandido produziria menos risco que o DNA normal. Química da vida A composição dos seres vivos difere da composição do ambiente inorgânico circundante. Apesar disso, os principais constituintes são a água, o carbono e as macromoléculas (polissacarídios, lípídos, proteínas, ácidos nucleicos), de entre as quais, as principais em termos de estrutura e função são as proteínas. 45
46 Proteínas As moléculas que põe a célula a funcionar e assumem a maioria das suas capacidades são as proteínas As proteínas são polímeros de aminoácidos. São constituídas por combinações de 20 aminoácidos diferentes. Assumem funções de suporte estrutural, protecção, catálise, transporte, defesa, regulação e movimento. Têm uma forma tridimensional específica e necessária para as suas funções específicas. Funções das proteínas: Enzimáticas as células possuem milhares de eximas diferentes. Ex: pepsina, DNA polimerase; Estruturais colagénio e elastina, queratina; Transportadoras hemoglobina, bacterirodopsina; Motoras miosina, cinesina; Armazenamento ferritina (fígado), albumina do ovo; Sinalização insulina; Receptoras rodopsina; Regulação repressor da lactose; Outras (protecção, defesa, ect ) Aminoácidos São constituídos por um grupo carboxílico e um grupo amina ligados ao mesmo carbono. Possuem ainda ligado a esse carbono uma cadeia lateral R e um hidrogénio. A variedade de aminoácidos resulta da cadeia lateral (que difere de aa para aa) ligada ao mesmo carbono, que lhes conferem propriedades particulares sendo responsáveis pela sua especificidade. Os aminoácidos são agrupados com o tipo de grupo R que possuem. O H 2N constitui o grupo amina e o COOH constitui o grupo carboxilo. Esta molécula é neutra mas em solução aquosa torna-se polar, com H 2N a passar para H 3N com carga positiva e o COOH a passar para 46
47 COO com carga negativa. No geral, de acordo com as características das suas cadeias laterais R, os aminoácidos podem ter carga positiva (básicos), carga negativa (ácidos), polares ou apolares. Os aminoácidos possuem um carbono assimétrico que origina dois enantiómeros D e L. Só os L- aminoácidos fazem parte das proteínas. Aminoácidos hidrofílicos aminoácidos atraídos pelas moléculas de água: aminoácidos básicos, aminoácidos polares sem carga no grupo R, aminoácidos ácidos. Aminoácidos hidrofóbicos aminoácidos repelidos pelas moléculas de água. No ponto isoelétrico (onde o ph da solução para o qual a carga absoluta do aa é nula) não há migração. Ligação Peptídica Uma cadeia polipeptídica tem, no início, uma extremidade amina e, no fim, uma extremidade ácido carboxilo: À extremidade inicial de H2N designa-se por N-terminal, enquanto a extremidade final de COOH designase por C-terminal. A ligação peptídica é uma ligação carbono-azoto, apolar, planar e que não permite rotações. A ligação deve-se ao facto do carbono e do azoto serem mais fortes que o hidrogénio a que estão ligados. Apenas as ligações Cα N e Cα C permitem rotação, influenciando os ângulos da estrutura e determinando a sua configuração. Assim, a conformação da cadeia é determinada pelos ângulos de rotação destas ligações para cada aminoácido, que por sua vez são determinados pelas interacções que se estabelecem entre átomos próximos e distantes na cadeia. Em solução aquosa, a proteína sofre alterações fazendo com que os grupos apolares fiquem para dentro, interagindo entre si, e os grupos polares para fora, ou seja, as proteínas podem dobrarse numa conformação compacta em que as cadeias laterais polares orientam-se para o exterior interagindo com a água, e em que as cadeias laterais apolares ficam enterradas no interior interagindo entre elas. Esta característica é devida aos 3 tipos de ligações não-covalentes que ajudam as proteínas a adoptar uma conformação tridimensional correspondente ao estado mínimo de energia, onde cada ligação destas é fraca mas a formação conjunta das ligações é forte. As pontes de hidrogénio estabelecem-se entre: átomos de 2 ligações peptídicas; átomos de 1 ligação peptídica com uma cadeia lateral; 2 cadeias laterais. Estrutura das Proteínas A estrutura das proteínas e a grande variedade de formas é o factor que influencia as suas múltiplas funções. 47
48 O estabelecimento de uma estrutura tridimensional das proteínas resulta das ligações que permitem a rotação da cadeia polipeptídica, da distribuição das cadeias laterais polares e apolares, das pontes de hidrogénio e de disulfito e das interacções hidrofóbicas e forças. Os tipos de ligações que ajudam a proteína a adoptar conformação tridimensional são : Pontes de Hidrogénio Forças de Van der Walls: ocorre quando dois átomos estão muito próximos Interacções hidrofóbicas: tendem a minimizar o contacto com moléculas de água que vêm para o exterior. Pontes dissulfito/ditiol ou dissulfureto: ligação entre as mesmas ou diferentes cadeias; ligação covalente forte. As proteínas podem perder a sua conformação, entrando em desnaturação. No entanto, se forem removidas as condições desnaturantes (como por exemplo: calor, ph extremo, solventes orgânicos, ureia e detergentes), ocorre a renaturação da força nativa. Assim, a informação para o arranjo tridimensional das proteínas reside na sua sequência de aminoácidos. Possuem 4 níveis de organização da estrutura das proteínas: Estrutura primária A estrutura primária das proteínas consiste na sequencia específica de aminoácidos da cadeia polipeptídica, a qual é ditada pela informação contida nos genes. Cada tipo de proteína tem uma sequencia única de aa, a qual vai induzir os níveis subsequentes de organização deste tipo de macromoléculas. Estrutura secundária As cadeias da estrutura primária podem dispor-se paralelamente e ligar-se entre si por pontes de hidrogénio formando uma estrutura em Folha β pregueada, que é um tipo de estrutura secundári. Por outro lado, as cadeias peptídicas podem enrolar-se em Hélice α, devido ao estabelecimento de pontes de hidrogénio entre gupos amina e carboxilico de aminoácidos diferentes. Hélice α a cadeia polipeptídica enrola-se numa espiral em que o grupo carbonilo de cada ligação estabelece uma ponte de hidrogénio com o azoto de outra, 4 resíduos à frentes. Assim, a cadeia forma um cilindro do qual se salientam as cadeias laterais; Folha β estrutura planar composta pelo alinhamento de 2 ou mais cadeias β, que são segmentos curtos quase totalmente estendidos da cadeia polipeptídica. Possui 2 cadeias antiparalelas e a folha β fica pregueada. As cadeias laterais projectam-se para um lado e outro das 2 faces da folha. A folha β pode apresentar-se como anti-paralela ou paralela; Curvas segmentos de 4 resíduos em forma de U estabilizados por uma ponte de hidrogénio, que redireccionam a cadeia. Estrutura terciária Quando a estrutura se dobra sobre si própria, fica com uma forma globular. É assim a conformação global da cadeia polipeptídica (arranjo tridimensional), estabilizada por pontes de hidrogénio, ligações iónicas, interacções hidrofóbicas e pontes de dissulfito; 48
49 Domínios regiões de um polipeptídeo com uma forma compacta que podem estar associados a uma função específica. Podem ser constituídos por diferentes exões (Bacillus thurigiensis Toxina BT); CAP proteína que regula a expressão de genes em bactérias. É responsável pela activação do operão lac na ausência de glucose, pois detecta a diminuição dos valores de ATP. Estrutura quaternária Várias cadeias globulares podem estabelecer ligações entre si, constituindo uma estrutura quartenária. Esta estrutura é estabilizada por pontes de hidrogénio, ligações iónicas, interacções hidrofóbicas e pontes dissulfito. Hemoglobina tetrâmero com 2 cópias de α-globina e 2 cópias de β-globina, e em que cada subunidade possui um grupo heme. Rubisco proteína mais abundante à face da terra, que faz a fixação do CO2 na fotossíntese. Função das Proteínas A função das proteínas depende, quase sempre, da sua capacidade para ligar outras moléculas (ligandos) com elevada especificidade. Estas ligações são não-covalentes. Devido à formação de múltiplas interacções fracas, possuem uma grande capacidade de ligação que exige a coincidência dos contornos do ligando com a proteína e complementaridade química (especificidade). Assim, o ligando e o local de ligação complementam-se (ao nível da forma e de grupos químicos) com grande especificidade e estabelecem, entre si, múltiplas interacções. O local de ligação consiste numa cavidade, na superfície da proteína, formada por um arranjo particular de aminoácidos. Este surge devido à conformação tridimensional que forma concavidades na superfície da proteína e contém um arranjo característico de cadeias laterais de aminoácidos que pertencem a regiões separadas da cadeia tornando-se mais próximos quando estão na sua conformação tridimensional. A proteína depois de produzida fica inactiva, e só é activada para realizar a sua função, quando necessária. Anticorpos - são exemplos característicos de interacção proteína / ligando. Estas pertencem à família das imunoglobulinas. Estas interacções possuem 2 características: Afinidade força da ligação proteína / ligando; Especificidade capacidade de ligar uma molécula em preferência de outras. Modificação Química e Processamento Após a síntese, as proteínas podem sofrer modificações químicas e processamento, onde recusam as sequências que não interessam. 49
50 Modificação química processo pelo qual passam quase todas as proteínas onde há ligação de um grupo químico à terminação N ou C ou a uma cadeia lateral, de modo a darem a especificidade à proteína. Esta pode consistir numa alteração da actividade, tempo de vida ou localização celular das proteínas. Acetilação adição de grupo acetil (CH3CO) ao grupo amino do aa. da extremidade amino Fosforilação de resíduos de serina, treonina e tirosina (regulação da actividade por fosforilação/desfosforilação). Consite na adição de grupos fosfato à cadeia polipéptidica Glicosilação ligação de cadeias polissacarídicas Adição de grupos prostéricos compostos químicos que podem estar ligados à proteína; Processamento processo que consiste na remoção de segmentos da cadeia por acção de proteases, são mecanismos de activação ou inactivação irreversíveis. Ciclo de Vida das Proteínas Enzimas Quase todas as reacções numa célula são catalisadas por uma classe de proteínas que promove alterações nos seus ligandos (enzimas). Catalisadores aumentam a velocidade de reacção e são energeticamente favoráveis; Diminuem a energia de activação e não alteram o equilíbrio final da reacção; Catalisam a formação ou ruptura de ligações covalentes; Ligam-se a um ou mais ligandos (substratos) que os convertem em produto; Aumentam a concentração local dos substratos, colocando-os na posição correcta para que a reacção ocorra e a energia de ligação contribua para a catálise. Classes funcionais das enzimas: Hidrolases enzimas que catalisam reacções de clivagem hidrolítica; 50
51 Nucleases degradam ácidos nucleicos por hidrólise das ligações entre nucleotídios; Proteases degradam proteínas por hidrólise das ligações peptídicas; Sintases catalisam a síntese de moléculas, por condensação de 2 mais pequenas; Isomerases catalisam o rearranjo de ligações dentro de uma molécula; Polimerases catalisam reacções de polimerisação, Cínases catalisam a adição de grupos fosfato a moléculas ou a proteínas; Fosfatases catalisam a remoção hidrolítica de um grupo fosfato; Oxido-redutases catalisam reacções em que uma molécula é reduzida e outra é oxidada; ATPases hidrolisam ATP. A velocidade da reacção, com a presença de enzimas, será maior à medida que aumenta a concentração de substrato. O ph e a temperatura influenciam a actividade das enzimas e a função das proteínas pois alteram o seu arranjo tridimensional, excepto as que apresentam pontes de sulfito pois a sua estrutura é mais rígida. Cofactores enzimáticos moléculas não proteicas que as enzimas requerem para a sua actividade. Grupos prostéricos grupos químicos permanentemente ligados à proteína que fazem parte do local activo (retinal, grupo heme); Coenzimas ligam-se às enzimas, temporariamente, e sofrem alteração durante o processo de catálise, são necessárias à actividade das enzimas; Inibição da actividade enzimática 1) Reversivel: rápida dissociação do complexo enzima-inibidor a) Competitiva: inibidor compete com o substrato para se ligar ao mesmo local activo da enzima. Se substrato aumentar em relação a inibidor, há mais probabilidades do substrato se ligar ao local activo. A velocidade de reacção não é muito alterada b) Não-competitiva: local de ligação do substrato diferente do local de ligação do inibidor. Não interfere o aumento da concentração do substrato nem do inibidor. c) Mista: certos inibidores tanto se ligam à enzima como ao complexo enzima-substrato. 2) Irreversivel: inibidores combinam-se ou destroem o local activo da enzima 51
52 Chaperones / Estabelecimento da Conformação Tridimensional das Proteínas Este processo começa durante a síntese da proteína, após o surgimento da cadeia polipeptídica dos ribossomas. Chaperones moleculares (hsp70) proteínas encarregadas de gerir a conformação das proteínas recém formadas. É um processo eficaz pois estes vão-se ligando e desligando, com gastos de energia, a todas as proteínas em nascimento, impedindo a ligação de outras moléculas, que mudem a sua conformação. Ligam-se às proteínas e estabilizam formas distendidas ou parcialmente dobradas evitando a degradação e facilitando o estabelecimento da conformação estável. Quando as proteínas não adquirem a sua conformação correcta, ou a perdem, são marcadas e enviadas para as chaperoninas. Chaperoninas (hso60) actuam directamente no estabelecimento da conformação tridimensional. Actuam após a síntese completa da cadeia polipeptídica e formam uma câmara de isolamento (GroEL bactérias / TCiP vertebrados). Os chaperones moleculares e as chaperoninas podem funcionar sequencialmente. As proteínas com conformação incorrecta são marcadas para a degradação, ou seja, se mesmo depois de passarem pelas chaperoninas as proteínas não tiverem a sua conformação específica, os mecanismos de vigilância encarregam-se da sua degradação, tanto no meio intracelular (lisossomas ou via da ubiquitina), como no meio extracelular (proteases digestivas). Priões São proteínas com carácter infeccioso. Desencadeiam doenças a nível neurodegenerativo. A forma citosólica dita normal é PrP C mas que pode adquirir conformação alternativa - PrP SC que tende a formar agregados que se depositam nas membranas citoplasmáticas. Esta alteração pode acontecer por exemplo devido a uma substituição de um aa. Estas proteínas alteradas acumulam-se nas células do sistema nervoso e não são alteradas ou degeneradas pelas proteases. Via da Ubiquitina Quando uma proteína é reconhecida por proteínas e deve ser degradada, a ubiquitina (proteína) vai marcala e leva-la para uma câmara onde se degradará a proteína em questão, pois tem uma conformação incorrecta. Adição de uma cadeia de ubiquitina a uma cadeia lateral de uma lisina interna da proteína a degradar; Degradação por proteossomas complexo multimérico cilíndrico constituído por proteases e que é limitado por cap (tampas). 52
53 Tecnicas para Separação, Purificação e Caracterização de Proteínas Purificação e Caracterização de Proteínas A purificação é necessária para a determinação da estrutura e do mecanismo de funcionamento de uma proteína. Para a separação de proteínas utiliza-se a cromatografia ou a electroforese. Para a caracterização das proteínas conta-se com a sequenciação de aminoácidos, espectrometria de massa, cristalografia de raios X ou ressonância magnética. Existe um fraccionamento celular, para este processo, que consiste na homogeneização e centrifugação. Depois efectua-se uma separação. Fraccionamento Celular Permite estudar e caracterizar diferentes fracções subcelulares. Homogeneização rebentamento das células. Centrifugação separação das diferentes fracções subcelulares. Diferencial ou Fraccionada através de várias centrifugações a diferentes velocidades; Assim: baseia-se nas diferenças de velocidade com que as partículas sedimentam no fundo de um tubo de centrifugação. Sujeitas a uma determinada força de centrifugação, as estruturas relativamente grandes, densas e pesadas, sedimentam mais rapidamente. Deste modo, usando-se forças e durações de centrifugação crescentes, e retirando de cada vez, o sedimento que se concentra no fundo do tubo será possível obter suspensões de núcleos, depois de mitocôndrias, cloroplastos, lisossomas e peroxissomas, depois separam-se os pequenos microssomas, depois ainda, os ribossomas livres. A fracção sobrante, que não possui quase totalmente estruturas, é o citossol. 53
54 Centrifugação em gradiente de densidade: a centrifugação diferencial não permite separar por exemplo as mitocôndrias, dos cloroplastos dos lisossomas e dos peroxissomas. Estes organelos sedimentam conjuntamente, se bem que tenham densidades diferentes. Nestes casos, emprega-se a técnica de fraccionamento em gradiente de densidades. Consiste em sujeitar uma suspensão de partículas com diferentes densidades, a uma força centrífuga constante, mas num meio de densidade gradualmente variável, de uma extremidade à outra do tubo. Empregam-se diversas substâncias para criar, num tubo de centrifugação, um gradiente de densidades; a mais comum é a sacarose. Nestas condições, partículas com densidades diferentes, deslocar-se-ão no campo de forças e situarse-ão em níveis de densidade da solução que igualem a sua própria densidade. Deste modo, é possível separar organelos como as mitocôndrias, cloroplastos, os lisossomas e os peroxissomas. Electroforese A electroforese permite a separação das proteínas ao mover-se através de uma matriz porosa, sob a acção de um campo eléctrico, onde as proteínas vão mover-se a velocidades diferentes consoante a sua carga e tamanho. SDS PAGE (poli-acrilamida gel electroforese) As proteínas são separadas em função da sua massa. A separação das proteínas é feita de acordo com a massa por limitação do tamanho dos poros do gel. A SDS confere às proteínas cargas negativas de modo a serem atraídas para o pólo negativo. Não separa moléculas com a mesma massa. Faz-se assim, primeiro um tratamento prévio das proteínas com SDS e aquecimento. O SDS é um detergente usado para desnaturar proteínas com várias subunidades e para cobrir as moléculas da proteína com cargas negativas. Deste modo, a carga intrínseca à proteína é mascarada,e a razao carga/massa torna-se constante. Depois, adiciona-se mercaptoetanol que vai hidrolisar as ligações dissulfureto que possam existir nas proteínas. Faz-se depois a electroforese. É usado azul de coomassie para corar o gel quando a quantidade de proteínas é elevada e o nitrato de prato quando a quantidade de proteínas é baixa. A velocidade de separação é inversamente proporcional à massa das proteínas. Como este processo não separa moléculas com a mesma massa, para separar essas moléculas é necessário explorar a carga usando para isso a electroforese bidimensional. Electroforese bidimensional há separação das proteínas pela carga (focagem isoeléctrica) seguida de separação pela massa (SDS PAGE). Focagem isoeléctrica as proteínas migram de acordo com a sua carga sob o efeito de um campo eléctrico ao longo de um gradiente de ph, ou seja, as proteínas são separadas de acordo com o seu ponto isoeléctrico (ph para a carga da proteína é 0). 54
55 Detecção de Proteínas Específicas Western Blotting ou Imunotransferência Espectrometria de massa A Espectrometria de massa é um método de determinação precisa de massas moleculares. Permite determinar a massa de proteínas ou de polipeptídios resultantes da sua clivagem enzimática ou química; permite também determinar a sequência de aa. do polipeptídio Esta informação é utilizada para identificar a proteína, pesquisando bases de dados de sequências de nucleótidos e de proteínas. Também é utilizada para determinar o tipo e localização das modificações pós-traducionais das proteínas. A maioria dos espectrómetros de massa possuem: Uma fonte de ionização (produz iões moléculas carregadas) Um ou mais analisadores de massa (separa os iões) Um detector (conta iões) Cromatografia Líquida em Coluna Separação das proteínas, ao passar através de uma matriz sólida permeável (esta matriz está contida numa coluna e é constituída por esferas de gel - fase estacionária) e empurrada por um fluxo de solvente (com o tampão adequado em fase móvel). Assim, as proteínas vão mover-se a velocidades diferentes consoante as suas interacções com a fase estacionária e a fase móvel. Baseia-se em: Cromatografia de troca iónica princípio de separação por carga; A matriz tem de ser tratada com solventes especiais que vão conferir carga positiva ou negativa às esferas (recobertas com radicais NH 3 + ou COO - ). Depois as amostras de proteínas passam pela coluna e ficam agarradas às esferas com carga oposta e as que não conseguem ligar-se saem e são recolhidas. Depois, para recolher as proteínas que ficaram agarradas às esferas passa-se na coluna uma solução de NaCl que se vai ligar ao sitio onde estava a proteína eluição da coluna Cromatografia de filtração em gel princípio de separação pelo tamanho (acordo com a massa); 55
COMPOSIÇÃO QUÍMICA DOS ÁCIDOS NUCLEICOS
 COMPOSIÇÃO QUÍMICA DOS ÁCIDOS NUCLEICOS Unidade básica dos Ácidos Nucleicos Existem apenas 4 bases em cada um dos ácidos nucleicos DNA DNA e RNA RNA Ácido fosfórico Ácido fosfórico Pentose Desoxirribose
COMPOSIÇÃO QUÍMICA DOS ÁCIDOS NUCLEICOS Unidade básica dos Ácidos Nucleicos Existem apenas 4 bases em cada um dos ácidos nucleicos DNA DNA e RNA RNA Ácido fosfórico Ácido fosfórico Pentose Desoxirribose
A síntese de DNA tem como objetivo replicar, de modo exato, o genoma. Já a síntese de RNA está relacionada com a própria expressão gênica.
 A síntese de DNA tem como objetivo replicar, de modo exato, o genoma. Já a síntese de RNA está relacionada com a própria expressão gênica. O processo de síntese de RNA, a partir de um molde de DNA, é denominado
A síntese de DNA tem como objetivo replicar, de modo exato, o genoma. Já a síntese de RNA está relacionada com a própria expressão gênica. O processo de síntese de RNA, a partir de um molde de DNA, é denominado
Biologia Molecular (BM) 2009/2010. Ana Cristina Ribeiro Gomes
 Biologia Molecular (BM) 2009/2010 Principais Constituintes Químicos da Célula Componentes Químicos da Célula As células são constituídas por elementos químicos tais como: oxigénio, carbono, hidrogénio,
Biologia Molecular (BM) 2009/2010 Principais Constituintes Químicos da Célula Componentes Químicos da Célula As células são constituídas por elementos químicos tais como: oxigénio, carbono, hidrogénio,
Genes. Informam para proteínas. Formam o GENOMA. Constituídas por monómeros: a.a. Codificados pelo código genético
 SÍNTESE PROTEICA São fragmentos de DNA (bases azotadas) que informam para uma característica Formam o GENOMA Genes Constituem os cromossomas Informam para proteínas Constituídas por monómeros: a.a. Codificados
SÍNTESE PROTEICA São fragmentos de DNA (bases azotadas) que informam para uma característica Formam o GENOMA Genes Constituem os cromossomas Informam para proteínas Constituídas por monómeros: a.a. Codificados
Do D.N.A. à síntese proteica. Descoberta do D.N.A. e da sua estrutura
 Do D.N.A. à síntese proteica Descoberta do D.N.A. e da sua estrutura Do DNA ao organismo! Para percorrer este caminho temos que responder à questão que se segue: Onde se localiza a informação genética?
Do D.N.A. à síntese proteica Descoberta do D.N.A. e da sua estrutura Do DNA ao organismo! Para percorrer este caminho temos que responder à questão que se segue: Onde se localiza a informação genética?
Geralmente é arredondado e único por célula, mas existem núcleos com outras formas e células com mais de um núcleo
 Núcleo Celular Geralmente é arredondado e único por célula, mas existem núcleos com outras formas e células com mais de um núcleo Núcleo Celular Algumas células não têm núcleo (são anucleadas), como as
Núcleo Celular Geralmente é arredondado e único por célula, mas existem núcleos com outras formas e células com mais de um núcleo Núcleo Celular Algumas células não têm núcleo (são anucleadas), como as
Ácidos Nucléicos Duplicação do DNA e Síntese de PROTEÍNAS. Paulo Dutra
 Ácidos Nucléicos Duplicação do DNA e Síntese de PROTEÍNAS Paulo Dutra ÁCIDOS NUCLEICOS Nucleotídeos É a unidade formadora dos ácidos nucléicos: DNA e RNA. É composto por um radical fosfato, uma pentose
Ácidos Nucléicos Duplicação do DNA e Síntese de PROTEÍNAS Paulo Dutra ÁCIDOS NUCLEICOS Nucleotídeos É a unidade formadora dos ácidos nucléicos: DNA e RNA. É composto por um radical fosfato, uma pentose
Replicação do DNA e Cromossomos
 Replicação do DNA e Cromossomos Características básicas da replicação do DNA In Vivo É semiconservativa, Inicia-se em origens únicas Geralmente é bidirecional a partir de cada origem de replicação. A replicação
Replicação do DNA e Cromossomos Características básicas da replicação do DNA In Vivo É semiconservativa, Inicia-se em origens únicas Geralmente é bidirecional a partir de cada origem de replicação. A replicação
Ficha Teórica de Apoio
 ESCOLA SECUNDÁRIA/3 JOAQUIM DE ARAÚJO Disciplina de Biologia/Geologia Ano Lectivo 2009-2010 Ficha Teórica de Apoio Assunto: Síntese Proteica EXPRESSÃO DA INFORMAÇÃO GENÉTICA BIOSSÍNTESE DE PROTEÍNAS O
ESCOLA SECUNDÁRIA/3 JOAQUIM DE ARAÚJO Disciplina de Biologia/Geologia Ano Lectivo 2009-2010 Ficha Teórica de Apoio Assunto: Síntese Proteica EXPRESSÃO DA INFORMAÇÃO GENÉTICA BIOSSÍNTESE DE PROTEÍNAS O
Cat Ca ab a olismo Anab a ol o ismo
 A acção das enzimas Metabolismo celular É o conjunto de reacções químicas que ocorrem numa célula. Catabolismo moléculas complexas são convertidas em moléculas mais simples, com libertação de energia.
A acção das enzimas Metabolismo celular É o conjunto de reacções químicas que ocorrem numa célula. Catabolismo moléculas complexas são convertidas em moléculas mais simples, com libertação de energia.
constituídos por moléculas orgânicas de Estas são formadas por um número
 Constituintes básicos de uma célula Moléculas da vida Todos os seres vivos, logo, as suas células, são constituídos por moléculas orgânicas de grandes dimensões macromoléculas. Estas são formadas por
Constituintes básicos de uma célula Moléculas da vida Todos os seres vivos, logo, as suas células, são constituídos por moléculas orgânicas de grandes dimensões macromoléculas. Estas são formadas por
BIOQUÍMICA E BIOLOGIA MOLECULAR 1º S_2013_2014_2º Teste
 BIOQUÍMICA E BIOLOGIA MOLECULAR 1º S_2013_2014_2º Teste 18/01/2014 (Duração: 1h,30 m) Nome do Aluno: Nº: Curso: Cada uma das questões de escolha múltipla (1 à 40) tem a cotação de 0,5 valores. Será descontado
BIOQUÍMICA E BIOLOGIA MOLECULAR 1º S_2013_2014_2º Teste 18/01/2014 (Duração: 1h,30 m) Nome do Aluno: Nº: Curso: Cada uma das questões de escolha múltipla (1 à 40) tem a cotação de 0,5 valores. Será descontado
A matéria constituinte dos seres vivos. Biologia Humana
 A matéria constituinte dos seres vivos Biologia Humana Quais os compostos químicos que constituem os seres vivos? Compostos inorgânicos água e sais minerais Compostos orgânicos glícidos, lípidos, prótidos,
A matéria constituinte dos seres vivos Biologia Humana Quais os compostos químicos que constituem os seres vivos? Compostos inorgânicos água e sais minerais Compostos orgânicos glícidos, lípidos, prótidos,
Como se transmite a informação contida no DNA? Como se traduz a informação que se encontra num gene? Como se passa de um gene para uma característica?
 Como se transmite a informação contida no DNA? Como se traduz a informação que se encontra num gene? Como se passa de um gene para uma característica? A síntese de proteínas resume-sena transformação da
Como se transmite a informação contida no DNA? Como se traduz a informação que se encontra num gene? Como se passa de um gene para uma característica? A síntese de proteínas resume-sena transformação da
AMINOÁCIDOS E PROTEÍNAS
 AMINOÁCIDOS E PROTEÍNAS Origem grego (protos) primeira, mais importante A palavra proteína que eu proponho vem derivada de proteos, porque ela parece ser a substância primitiva ou principal da nutrição
AMINOÁCIDOS E PROTEÍNAS Origem grego (protos) primeira, mais importante A palavra proteína que eu proponho vem derivada de proteos, porque ela parece ser a substância primitiva ou principal da nutrição
Sociedade, Tecnologia e Ciência. DR1 - O Elemento, Graça Mendonça. Núcleo Gerador 7 DNA
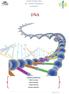 DNA Trabalho realizado por: Rafael Lourenço Fábio Rodrigues Miguel Almeida Rodrigo Sambento 1 á g i n a Índice INTRODUÇÃO... 3 DNA... 4 Organização do DNA na célula... 4 Elementos básicos dos ácidos nucleicos...
DNA Trabalho realizado por: Rafael Lourenço Fábio Rodrigues Miguel Almeida Rodrigo Sambento 1 á g i n a Índice INTRODUÇÃO... 3 DNA... 4 Organização do DNA na célula... 4 Elementos básicos dos ácidos nucleicos...
Membranas Biológicas. Profª Eleonora Slide de aula
 Membranas Biológicas Profª Eleonora Slide de aula Membranas Biológicas Todas as células possuem na sua periferia uma membrana, a qual fornece a barreira necessária que separa o lado interno do lado externo.
Membranas Biológicas Profª Eleonora Slide de aula Membranas Biológicas Todas as células possuem na sua periferia uma membrana, a qual fornece a barreira necessária que separa o lado interno do lado externo.
LIPÍDIOS. 1º ano Pró Madá
 LIPÍDIOS 1º ano Pró Madá O QUE SÃO? Também chamados de gorduras Compostos com estrutura molecular variada principalmente, por moléculas de hidrogênio, oxigênio, carbono Característica principal insolubilidade
LIPÍDIOS 1º ano Pró Madá O QUE SÃO? Também chamados de gorduras Compostos com estrutura molecular variada principalmente, por moléculas de hidrogênio, oxigênio, carbono Característica principal insolubilidade
Biologia Celular e Molecular:
 Disciplina: Biologia Celular e Molecular: Estrutura e Fisiologia da Célula Os Ácidos Nucleicos Os ácidos nucleicos são as maiores moléculas encontradas no mundo vivo e responsáveis pelo controle dos processos
Disciplina: Biologia Celular e Molecular: Estrutura e Fisiologia da Célula Os Ácidos Nucleicos Os ácidos nucleicos são as maiores moléculas encontradas no mundo vivo e responsáveis pelo controle dos processos
A CÉLULA. Natércia Charruadas 2011. Biologia e Geologia 10º ano
 A CÉLULA Natércia Charruadas 2011 Biologia e Geologia 10º ano O entendimento dos processos biológicos depende do conhecimento da célula enquanto unidade fundamental da Vida. As dimensões das células, geralmente
A CÉLULA Natércia Charruadas 2011 Biologia e Geologia 10º ano O entendimento dos processos biológicos depende do conhecimento da célula enquanto unidade fundamental da Vida. As dimensões das células, geralmente
Transcrição: Síntese de RNA Tradução: Síntese Proteica
 Transcrição: Síntese de RNA Tradução: Síntese Proteica A estrutura química da molécula de RNA apresenta pequenas diferenças em relação ao DNA. http://www.nature.com/scitable/learning-path/theelaboration-of-the-central-dogma-701886#url
Transcrição: Síntese de RNA Tradução: Síntese Proteica A estrutura química da molécula de RNA apresenta pequenas diferenças em relação ao DNA. http://www.nature.com/scitable/learning-path/theelaboration-of-the-central-dogma-701886#url
QBQ 0102 Educação Física. Carlos Hotta. Código Genético e Tradução 16/06/15
 QBQ 0102 Educação Física Carlos Hotta Código Genético e Tradução 16/06/15 Previously... replicação DNA transcrição RNA tradução proteína RNAm possuem informação para gerar novas proteínas A RNA polimerase
QBQ 0102 Educação Física Carlos Hotta Código Genético e Tradução 16/06/15 Previously... replicação DNA transcrição RNA tradução proteína RNAm possuem informação para gerar novas proteínas A RNA polimerase
CÉLULAS 2/14/2017 FACULDADE EDUCACIONAL DE MEDIANEIRA COMPOSIÇÃO MOLECULAR DAS CÉLULAS COMPOSIÇÃO QUÍMICA DAS CÉLULAS COMPOSIÇÃO MOLECULAR DAS CÉLULAS
 2/14/2017 FACULDADE EDUCACIONAL DE MEDIANEIRA CÉLULAS Células são estruturas complexas e diversas; São capazes de autoreplicação; Realizam uma ampla variedade de papeis especializados em organismos multicelulares:
2/14/2017 FACULDADE EDUCACIONAL DE MEDIANEIRA CÉLULAS Células são estruturas complexas e diversas; São capazes de autoreplicação; Realizam uma ampla variedade de papeis especializados em organismos multicelulares:
Disciplina : Biologia Molecular: conceitos e Técnicas. Professora. Dra. Andrea Soares da Costa Fuentes
 Disciplina : Biologia Molecular: conceitos e Técnicas Professora. Dra. Andrea Soares da Costa Fuentes Revisão Geral Sumário História da Genética Molecular DNA e RNA Dogma Central Replicação Transcrição
Disciplina : Biologia Molecular: conceitos e Técnicas Professora. Dra. Andrea Soares da Costa Fuentes Revisão Geral Sumário História da Genética Molecular DNA e RNA Dogma Central Replicação Transcrição
Biologia molecular é uma disciplina híbrida
 Biologia molecular é uma disciplina híbrida Os termos: biologia molecular e genética molecular = sinônimos As técnicas da biologia molecular são aplicadas em quase todos os campos da biologia neurofisiologia
Biologia molecular é uma disciplina híbrida Os termos: biologia molecular e genética molecular = sinônimos As técnicas da biologia molecular são aplicadas em quase todos os campos da biologia neurofisiologia
ÁCIDOS NUCLÉICOS 15/6/2010. Universidade Federal de Mato Grosso Disciplina de Bioquímica. - Desoxirribose, presente no DNA; - Ribose, presente no RNA.
 Universidade Federal de Mato Grosso Disciplina de Bioquímica ÁCIDOS NUCLÉICOS Prof. Msc. Reginaldo Vicente Ribeiro Cuiabá Maio de 2010 São as biomoléculas com a função de armazenamento e expressão da informação
Universidade Federal de Mato Grosso Disciplina de Bioquímica ÁCIDOS NUCLÉICOS Prof. Msc. Reginaldo Vicente Ribeiro Cuiabá Maio de 2010 São as biomoléculas com a função de armazenamento e expressão da informação
Unidade 5 Crescimento e Renovação Celular
 Unidade 5 Crescimento e Renovação Celular 5.1 Crescimento e Renovação Celular Aula nº2-17/set/08 Prof. Ana Reis ( ) uma dupla totalmente ignorante da química dos nucleótidos, desejosa de encaixar o DNA
Unidade 5 Crescimento e Renovação Celular 5.1 Crescimento e Renovação Celular Aula nº2-17/set/08 Prof. Ana Reis ( ) uma dupla totalmente ignorante da química dos nucleótidos, desejosa de encaixar o DNA
INTRODUÇÃO A BIOLOGIA MOLECULAR DNA
 INTRODUÇÃO A BIOLOGIA MOLECULAR DNA NOS SERES HUMANOS NÚCLEO DNA CÉLULA CROMOSSOMAS (DNA+PROTEÍNAS) - Informação das proteínas e RNAs que serão sintetizadas pelas células do organismo ao longo da sua vida.
INTRODUÇÃO A BIOLOGIA MOLECULAR DNA NOS SERES HUMANOS NÚCLEO DNA CÉLULA CROMOSSOMAS (DNA+PROTEÍNAS) - Informação das proteínas e RNAs que serão sintetizadas pelas células do organismo ao longo da sua vida.
DNA. Dados relevantes para a compreensão da sua estrutura.
 DN Dados relevantes para a compreensão da sua estrutura. DN: dados relevantes para a compreensão da sua estrutura. Em 1950, Erwin hargaff, ao estudar amostras de DN de diversas espécies, constatou que
DN Dados relevantes para a compreensão da sua estrutura. DN: dados relevantes para a compreensão da sua estrutura. Em 1950, Erwin hargaff, ao estudar amostras de DN de diversas espécies, constatou que
Replicação do DNA. Prof. Edimar
 Replicação do DNA Prof. Edimar PRINCIPAIS ENZIMAS ENVOLVIDAS (SISTEMA DE REPLICAÇÃO DO DNA) 1. DNA Polimerases 2. Endonucleases 3. Helicases 4. Topoisomerases 5. Primases 6. Telomerases ENDONUCLEASES HELICASE
Replicação do DNA Prof. Edimar PRINCIPAIS ENZIMAS ENVOLVIDAS (SISTEMA DE REPLICAÇÃO DO DNA) 1. DNA Polimerases 2. Endonucleases 3. Helicases 4. Topoisomerases 5. Primases 6. Telomerases ENDONUCLEASES HELICASE
Aula 2 - Revisão DNA RNA - PROTEÍNAS
 Aula 2 - Revisão DNA RNA - PROTEÍNAS Estudo Dirigido Aula 2 - Revisão 1. Características comuns a todos os organismos vivos; 2. Domínios da Vida e tipos celulares, principais diferenças dos tipos celulares;
Aula 2 - Revisão DNA RNA - PROTEÍNAS Estudo Dirigido Aula 2 - Revisão 1. Características comuns a todos os organismos vivos; 2. Domínios da Vida e tipos celulares, principais diferenças dos tipos celulares;
Duplicação do DNA & Síntese de proteínas
 Duplicação do DNA & Síntese de proteínas Aula de Biologia Tema: Duplicação do DNA & Síntese Protéica Daniel Biólogo Planetabiologia.com ÁCIDOS NUCLÉICOS 1) Conceito: Os Ácidos Nucléicos são macromoléculas,
Duplicação do DNA & Síntese de proteínas Aula de Biologia Tema: Duplicação do DNA & Síntese Protéica Daniel Biólogo Planetabiologia.com ÁCIDOS NUCLÉICOS 1) Conceito: Os Ácidos Nucléicos são macromoléculas,
Ácidos Nucleicos: Nucleotídeos, DNA e RNA. Bianca Lobão - nº USP: Caio Lourenço - nº USP: Giulia Santos - nº USP:
 Ácidos Nucleicos: Nucleotídeos, DNA e RNA Bianca Lobão - nº USP: 9370841 Caio Lourenço - nº USP: Giulia Santos - nº USP: 9370726 Nucleotídeos Compõem a estrutura das moléculas de DNA e RNA; São compostos
Ácidos Nucleicos: Nucleotídeos, DNA e RNA Bianca Lobão - nº USP: 9370841 Caio Lourenço - nº USP: Giulia Santos - nº USP: 9370726 Nucleotídeos Compõem a estrutura das moléculas de DNA e RNA; São compostos
Membranas Biológicas. Profª Eleonora Slide de aula
 Membranas Biológicas Profª Eleonora Slide de aula Membranas biológicas Todas as células possuem na sua periferia uma membrana, a qual fornece a barreira necessária que separa o lado interno do lado externo.
Membranas Biológicas Profª Eleonora Slide de aula Membranas biológicas Todas as células possuem na sua periferia uma membrana, a qual fornece a barreira necessária que separa o lado interno do lado externo.
Capítulo 13 Acidos Nucleicos
 Capítulo 13 Acidos Nucleicos O modelo da dupla hélice O modelo da estrutura molecular do DNA foi proposto em 1953 por Watson e Crick e é aceito até hoje. Eles ganharam o prêmio Nobel de Medicina e Fisiologia
Capítulo 13 Acidos Nucleicos O modelo da dupla hélice O modelo da estrutura molecular do DNA foi proposto em 1953 por Watson e Crick e é aceito até hoje. Eles ganharam o prêmio Nobel de Medicina e Fisiologia
Qual o nome das bases pirimídicas?. R: Timina e Citosina. Quais os constituintes dos nucleótidos?
 O que significam as siglas? R: Ácido desoxirribonucleico. A molécula de tem mensagens codificadas em sequências de que contêm bases púricas e pirimídicas. R: nucleótidos Qual o nome das bases pirimídicas?.
O que significam as siglas? R: Ácido desoxirribonucleico. A molécula de tem mensagens codificadas em sequências de que contêm bases púricas e pirimídicas. R: nucleótidos Qual o nome das bases pirimídicas?.
Biologia e Bioquímica II 2009/2010
 Biologia e Bioquímica II 009/010 Módulo II - Mitocôndria Sessão 6 Ficha de trabalho 1. Qual a importância energética da glicólise se no balanço final só há produção de ATP e, por cada molécula de glicose
Biologia e Bioquímica II 009/010 Módulo II - Mitocôndria Sessão 6 Ficha de trabalho 1. Qual a importância energética da glicólise se no balanço final só há produção de ATP e, por cada molécula de glicose
Figura 1. Exemplo da estrutura de um nucleotídeo
 2 - ÁCIDOS NUCLÉICOS Na natureza há dois tipos de ácidos nucléicos: DNA ou ácido desoxirribonucléico e RNA ou ácido ribonucléico. Analogamente a um sistema de comunicação, essas informações são mantidas
2 - ÁCIDOS NUCLÉICOS Na natureza há dois tipos de ácidos nucléicos: DNA ou ácido desoxirribonucléico e RNA ou ácido ribonucléico. Analogamente a um sistema de comunicação, essas informações são mantidas
Obtenção de matéria pelos seres heterotróficos transportes transmembranares
 Obtenção de matéria pelos seres heterotróficos transportes transmembranares 1 Processos de transporte A membrana plasmática pode facilitar, dificultar ou impedir a passagem de substâncias, uma propriedade
Obtenção de matéria pelos seres heterotróficos transportes transmembranares 1 Processos de transporte A membrana plasmática pode facilitar, dificultar ou impedir a passagem de substâncias, uma propriedade
Livro Interactivo 3D Permite Fazer Anotações e Imprimir. Dúvidas Mais Comuns BIO 11. Flipping Book.
 Livro Interactivo 3D Permite Fazer Anotações e Imprimir Dúvidas Mais Comuns BIO 11 Flipping Book http://netxplica.com DÚVIDAS MAIS COMUNS :: BIOLOGIA E GEOLOGIA 11 http://netxplica.com 1. Crescimento e
Livro Interactivo 3D Permite Fazer Anotações e Imprimir Dúvidas Mais Comuns BIO 11 Flipping Book http://netxplica.com DÚVIDAS MAIS COMUNS :: BIOLOGIA E GEOLOGIA 11 http://netxplica.com 1. Crescimento e
BIOLOGIA MOLECULAR. João Carlos Bespalhok Filho Professor Associado DFF/UFPR
 BIOLOGIA MOLECULAR João Carlos Bespalhok Filho Professor Associado DFF/UFPR Definição Biologia Molecular é o campo da biologia que estuda a composição, estrutura e interações de moléculas celulares -tais
BIOLOGIA MOLECULAR João Carlos Bespalhok Filho Professor Associado DFF/UFPR Definição Biologia Molecular é o campo da biologia que estuda a composição, estrutura e interações de moléculas celulares -tais
DNA: Replicação e Transcrição. Professora: MSc Monyke Lucena
 EXTRA, EXTRA Se a mãe for (DD) e o pai (D), nenhum dos descendentes será daltónico nem portador. Se a mãe (DD) e o pai for (d), nenhum dos descendentes será daltônico, porém as filhas serão portadoras
EXTRA, EXTRA Se a mãe for (DD) e o pai (D), nenhum dos descendentes será daltónico nem portador. Se a mãe (DD) e o pai for (d), nenhum dos descendentes será daltônico, porém as filhas serão portadoras
Constituintes básicos de uma célula
 Constituintes básicos de uma célula Biomoléculas As moléculas da vida São as moléculas constituintes dos seres vivos. São formadas principalmente por C e H, ainda que também possam estar presentes na sua
Constituintes básicos de uma célula Biomoléculas As moléculas da vida São as moléculas constituintes dos seres vivos. São formadas principalmente por C e H, ainda que também possam estar presentes na sua
IMPORTÂNCIA DA GENÉTICA PARA ÁREA DA SAÚDE: Diagnóstico clínico: alteração no número ou estrutura dos cromossomos (síndrome de Down)
 Aplicações: IMPORTÂNCIA DA GENÉTICA PARA ÁREA DA SAÚDE: Diagnóstico clínico: alteração no número ou estrutura dos cromossomos (síndrome de Down) Mapeamento genético e identificação: mapeamento de genes
Aplicações: IMPORTÂNCIA DA GENÉTICA PARA ÁREA DA SAÚDE: Diagnóstico clínico: alteração no número ou estrutura dos cromossomos (síndrome de Down) Mapeamento genético e identificação: mapeamento de genes
ÁCIDOS NUCLÉICOS ESTRUTURA E FUNÇÕES
 DNA ÁCIDOS NUCLÉICOS ESTRUTURA E FUNÇÕES Prof. Edimar Campos Antes de 1950 sabia-se apenas que qualquer que fosse a natureza do material genético, ele deveria possuir 3 características importantes: O MATERIAL
DNA ÁCIDOS NUCLÉICOS ESTRUTURA E FUNÇÕES Prof. Edimar Campos Antes de 1950 sabia-se apenas que qualquer que fosse a natureza do material genético, ele deveria possuir 3 características importantes: O MATERIAL
Ácidos nucleicos (DNA e RNA) e os genes
 Disciplina: Biologia Humana Profa. Me. Vivian C. Langiano Ácidos nucleicos (DNA e RNA) e os genes De Robertis, E. Bases da Biologia Celular e Molecular. Rio de Janeiro, Guanabara Koogan, 4 ed. 2006. cap
Disciplina: Biologia Humana Profa. Me. Vivian C. Langiano Ácidos nucleicos (DNA e RNA) e os genes De Robertis, E. Bases da Biologia Celular e Molecular. Rio de Janeiro, Guanabara Koogan, 4 ed. 2006. cap
BIOQUÍMICA GERAL. Prof. Dr. Franciscleudo B. Costa UATA/CCTA/UFCG. Aula 7 Ácidos Nucleicos. Definição NUCLEOTÍDEO (RNA)
 Universidade Federal de Campina Grande Centro de Ciências e Tecnologia Agroalimentar Unidade Acadêmica de Tecnologia de Alimentos BIOQUÍMICA GERAL Definição Importância e aplicações Estrutura Geral Função
Universidade Federal de Campina Grande Centro de Ciências e Tecnologia Agroalimentar Unidade Acadêmica de Tecnologia de Alimentos BIOQUÍMICA GERAL Definição Importância e aplicações Estrutura Geral Função
Biologia. Código Genético. Professor Enrico Blota.
 Biologia Código Genético Professor Enrico Blota www.acasadoconcurseiro.com.br Biologia CÓDIGO GENÉTICO NÚCLEO E SÍNTESE PROTEICA O núcleo é de fundamental importância para grande parte dos processos que
Biologia Código Genético Professor Enrico Blota www.acasadoconcurseiro.com.br Biologia CÓDIGO GENÉTICO NÚCLEO E SÍNTESE PROTEICA O núcleo é de fundamental importância para grande parte dos processos que
DOGMA CENTRAL DA BIOLOGIA MOLECULAR
 Transcrição do DNA DOGMA CENTRAL DA BIOLOGIA MOLECULAR Replicação DNA Transcrição RNA Tradução PROTEÍNA Transcrição Processo pelo qual o DNA é copiado numa molécula de RNA (mrna, rrna e trna). Todos os
Transcrição do DNA DOGMA CENTRAL DA BIOLOGIA MOLECULAR Replicação DNA Transcrição RNA Tradução PROTEÍNA Transcrição Processo pelo qual o DNA é copiado numa molécula de RNA (mrna, rrna e trna). Todos os
Prof. Giovani - Biologia
 Prof. Giovani - Biologia 1.(UFRS) Tanto em uma célula eucarionte quanto em uma procarionte podemos encontrar: a) membrana plasmática e retículo endoplasmático. b) ribossomos e aparelho de Golgi. c) mitocôndrias
Prof. Giovani - Biologia 1.(UFRS) Tanto em uma célula eucarionte quanto em uma procarionte podemos encontrar: a) membrana plasmática e retículo endoplasmático. b) ribossomos e aparelho de Golgi. c) mitocôndrias
Exercícios de Autoduplicação, Transcrição e Tradução
 Exercícios de Autoduplicação, Transcrição e Tradução 1. (UniFoa/2003) Imagine a seguinte situação hipotética: Um aluno precisa decifrar o código de uma enzima G1, que possui uma cadeia formada por seis
Exercícios de Autoduplicação, Transcrição e Tradução 1. (UniFoa/2003) Imagine a seguinte situação hipotética: Um aluno precisa decifrar o código de uma enzima G1, que possui uma cadeia formada por seis
Estrutura e Função de Proteínas
 Estrutura e Função de Proteínas As Proteínas As proteínas observadas na natureza evoluíram pela pressão selectiva para efetuar funções específicas. As propriedades funcionais das proteínas dependem da
Estrutura e Função de Proteínas As Proteínas As proteínas observadas na natureza evoluíram pela pressão selectiva para efetuar funções específicas. As propriedades funcionais das proteínas dependem da
DNA, RNA e PROTEÍNAS. Carla Costa. Faculdade de Medicina da Universidade do Porto Serviço e Laboratório de Biologia Celular e Molecular
 DNA, RNA e PROTEÍNAS Carla Costa Faculdade de Medicina da Universidade do Porto Serviço e Laboratório de Biologia Celular e Molecular OBSERVAÇÃO http://www.csiro.au/helix/dna/index.shtml DNA A descoberta
DNA, RNA e PROTEÍNAS Carla Costa Faculdade de Medicina da Universidade do Porto Serviço e Laboratório de Biologia Celular e Molecular OBSERVAÇÃO http://www.csiro.au/helix/dna/index.shtml DNA A descoberta
BIOQUÍMICA E BIOLOGIA MOLECULAR 1º S_2009_2010_1º Teste 16/11/2009
 BIOQUÍMICA E BIOLOGIA MOLECULAR 1º S_2009_2010_1º Teste 16/11/2009 (Duração: 1,5 h) Nome do Aluno: Nº: Curso: Cada uma das questões de escolha múltipla (1 à 32) tem a cotação de 0,6 valores. Será descontado
BIOQUÍMICA E BIOLOGIA MOLECULAR 1º S_2009_2010_1º Teste 16/11/2009 (Duração: 1,5 h) Nome do Aluno: Nº: Curso: Cada uma das questões de escolha múltipla (1 à 32) tem a cotação de 0,6 valores. Será descontado
Modificação Covalente
 Controle Celular Sistema de Controle Celular Governa a Maquinaria do Ciclo Celular Ativação e desativação cíclica Ativação e Inativação Proteínas chaves e dos complexos protéicos Iniciam ou regulam Replicação
Controle Celular Sistema de Controle Celular Governa a Maquinaria do Ciclo Celular Ativação e desativação cíclica Ativação e Inativação Proteínas chaves e dos complexos protéicos Iniciam ou regulam Replicação
ÁCIDOS NUCLEICOS. Existem dois tipos de ácidos nucleicos: - Ácido desoxirribonucleico (DNA) - Ácido ribonucleico (RNA)
 ÁCIDOS NUCLEICOS ÁCIDOS NUCLEICOS Em 1868, F. Miesher, num importante trabalho de citoquímica, isolou e analisou o núcleo das células. As substâncias constituintes do núcleo foram designadas nucleínas.
ÁCIDOS NUCLEICOS ÁCIDOS NUCLEICOS Em 1868, F. Miesher, num importante trabalho de citoquímica, isolou e analisou o núcleo das células. As substâncias constituintes do núcleo foram designadas nucleínas.
Armazenamento da informação genética
 Universidade Federal do Pampa Curso de Nutrição Biologia celular e molecular Armazenamento da informação genética Profª Ms. Vanessa Retamoso Prof Ms. Vanessa Retamoso NÚCLEO INTERFÁSICO: é o núcleo da
Universidade Federal do Pampa Curso de Nutrição Biologia celular e molecular Armazenamento da informação genética Profª Ms. Vanessa Retamoso Prof Ms. Vanessa Retamoso NÚCLEO INTERFÁSICO: é o núcleo da
Estrutura do DNA. Macromoléculas Ácidos Nucleicos (DNA e RNA) Estrutura dos ácidos nucleicos. Estrutura dos ácidos nucleicos 09/03/2017
 Macromoléculas Ácidos Nucleicos (DNA e RNA) Estrutura do DNA James Watson e Francis Crick (1953) Esclareceram a estrutura do DNA: dupla hélice composta de 2 filamentos de nucleotídeosque se enrolam em
Macromoléculas Ácidos Nucleicos (DNA e RNA) Estrutura do DNA James Watson e Francis Crick (1953) Esclareceram a estrutura do DNA: dupla hélice composta de 2 filamentos de nucleotídeosque se enrolam em
Proteínas. Dra. Flávia Cristina Goulart. Bioquímica
 Proteínas Dra. Flávia Cristina Goulart Bioquímica Os aa. e as proteínas Nos animais, as proteínas correspondem à cerca de 80% do peso dos músculos desidratados, cerca de 70% da pele e 90% do sangue seco.
Proteínas Dra. Flávia Cristina Goulart Bioquímica Os aa. e as proteínas Nos animais, as proteínas correspondem à cerca de 80% do peso dos músculos desidratados, cerca de 70% da pele e 90% do sangue seco.
Aula de Bioquímica II. Tema: Transcrição. Prof. Dr. Júlio César Borges
 Aula de Bioquímica II Tema: Transcrição Prof. Dr. Júlio César Borges Depto. de Química e Física Molecular DQFM Instituto de Química de São Carlos IQSC Universidade de São Paulo USP E-mail: borgesjc@iqsc.usp.br
Aula de Bioquímica II Tema: Transcrição Prof. Dr. Júlio César Borges Depto. de Química e Física Molecular DQFM Instituto de Química de São Carlos IQSC Universidade de São Paulo USP E-mail: borgesjc@iqsc.usp.br
DNA e RNA Replicação do DNA
 DNA e RNA Replicação do DNA 1953 1952 1952 1944 1928 1869 À descoberta do Material Genético Watson e Crick Modelo do DNA dupla hélice Hershey-Chase experiências com bacteriófagos confirmam o DNA como suporte
DNA e RNA Replicação do DNA 1953 1952 1952 1944 1928 1869 À descoberta do Material Genético Watson e Crick Modelo do DNA dupla hélice Hershey-Chase experiências com bacteriófagos confirmam o DNA como suporte
Genética de microrganismos. Disciplina: Princípios de Microbiologia Professor: José Belasque Junior Monitora: Gislâine Vicente dos Reis
 Genética de microrganismos Disciplina: Princípios de Microbiologia Professor: José Belasque Junior Monitora: Gislâine Vicente dos Reis Piracicaba, outubro 2014 Histórico 1868- Primeiro a estudar o núcleo
Genética de microrganismos Disciplina: Princípios de Microbiologia Professor: José Belasque Junior Monitora: Gislâine Vicente dos Reis Piracicaba, outubro 2014 Histórico 1868- Primeiro a estudar o núcleo
IFSC Campus Lages. Transcrição. Biologia Molecular Prof. Silmar Primieri
 IFSC Campus Lages Transcrição Biologia Molecular Prof. Silmar Primieri RNA - estrutura Semelhante ao DNA, com ribose como glicídio e uracila como base nitrogenada, no lugar da timina do DNA. RNA é unifilamentar
IFSC Campus Lages Transcrição Biologia Molecular Prof. Silmar Primieri RNA - estrutura Semelhante ao DNA, com ribose como glicídio e uracila como base nitrogenada, no lugar da timina do DNA. RNA é unifilamentar
A Química da Vida. Anderson Dias Felipe Knak
 A Química da Vida Anderson Dias Felipe Knak A ÁGUA NAS CÉLULAS A água é imprescindível à vida dos indivíduos, independendo da espécie, idade, metabolismo e/ou grupo celular. A água exerce função de transporte
A Química da Vida Anderson Dias Felipe Knak A ÁGUA NAS CÉLULAS A água é imprescindível à vida dos indivíduos, independendo da espécie, idade, metabolismo e/ou grupo celular. A água exerce função de transporte
FICHA (IN)FORMATIVA Nº 3 Biologia e Geologia Módulo 2. Célula
 FICHA (IN)FORMATIVA Nº 3 Biologia e Geologia Módulo 2 A célula e o transporte transmembranar Célula Teoria celular a célula é a unidade estrutural e funcional de todos os organismos; as novas células formam-se
FICHA (IN)FORMATIVA Nº 3 Biologia e Geologia Módulo 2 A célula e o transporte transmembranar Célula Teoria celular a célula é a unidade estrutural e funcional de todos os organismos; as novas células formam-se
SÍNTESES NUCLEARES. O DNA éo suporte da informação genética. Parte 1 Replicação
 SÍNTESES NUCLEARES O DNA éo suporte da informação genética Parte 1 Replicação Estrutura do DNA Replicação do DNA Nucleótidos A informação genética das células é armazenada sob a forma de 2 moléculas similares:
SÍNTESES NUCLEARES O DNA éo suporte da informação genética Parte 1 Replicação Estrutura do DNA Replicação do DNA Nucleótidos A informação genética das células é armazenada sob a forma de 2 moléculas similares:
Replicação do DNA e Cromossomos
 Replicação do DNA e Cromossomos Características básicas da replicação do DNA In Vivo É semiconservativa, Inicia-se em origens únicas Geralmente é bidirecional a partir de cada origem de replicação. A replicação
Replicação do DNA e Cromossomos Características básicas da replicação do DNA In Vivo É semiconservativa, Inicia-se em origens únicas Geralmente é bidirecional a partir de cada origem de replicação. A replicação
BIOQUÍMICA E BIOLOGIA MOLECULAR 1º S_2013_2014_1º Teste
 BIOQUÍMICA E BIOLOGIA MOLECULAR 1º S_2013_2014_1º Teste 21/11/2013 (Duração: 1h,30 m) Nome do Aluno: Nº: Curso: Cada uma das questões de escolha múltipla (1 à 40) tem a cotação de 0,5 valores. Será descontado
BIOQUÍMICA E BIOLOGIA MOLECULAR 1º S_2013_2014_1º Teste 21/11/2013 (Duração: 1h,30 m) Nome do Aluno: Nº: Curso: Cada uma das questões de escolha múltipla (1 à 40) tem a cotação de 0,5 valores. Será descontado
3 Nucleotídeos e Ácidos Nucléicos
 1 3 Nucleotídeos e Ácidos Nucléicos - São compostos ricos em energia - Funcionam como sinais químicos - São reservatórios moleculares da informação genética a) Nucleotídeos - São encontrados polimerizados
1 3 Nucleotídeos e Ácidos Nucléicos - São compostos ricos em energia - Funcionam como sinais químicos - São reservatórios moleculares da informação genética a) Nucleotídeos - São encontrados polimerizados
Transcrição e Tradução
 Disciplina: Fundamentos de Genética e Biologia Molecular Turma: Fisioterapia (1 o Ano) Transcrição e Tradução Docente: Profa. Dra. Marilanda Ferreira Bellini E-mail: marilanda_bellini@yahoo.com Blog: http://marilandabellini.wordpress.com
Disciplina: Fundamentos de Genética e Biologia Molecular Turma: Fisioterapia (1 o Ano) Transcrição e Tradução Docente: Profa. Dra. Marilanda Ferreira Bellini E-mail: marilanda_bellini@yahoo.com Blog: http://marilandabellini.wordpress.com
LIGAÇÃO QUÍMICA NO CARBONO GEOMETRIA MOLECULAR HIBRIDAÇÃO. Geometria molecular
 LIGAÇÃO QUÍMICA NO CARBONO GEOMETRIA MOLECULAR IBRIDAÇÃO Geometria molecular O arranjo tri-dimensional dos átomos numa molécula geometria molecular A teoria da repulsão dos pares de electrões (ligantes
LIGAÇÃO QUÍMICA NO CARBONO GEOMETRIA MOLECULAR IBRIDAÇÃO Geometria molecular O arranjo tri-dimensional dos átomos numa molécula geometria molecular A teoria da repulsão dos pares de electrões (ligantes
Replicação do DNA e Cromossomos
 Replicação do DNA e Cromossomos Características básicas da replicação do DNA In Vivo É semiconservativa, Inicia-se em origens únicas Geralmente é bidirecional a partir de cada origem de replicação. A replicação
Replicação do DNA e Cromossomos Características básicas da replicação do DNA In Vivo É semiconservativa, Inicia-se em origens únicas Geralmente é bidirecional a partir de cada origem de replicação. A replicação
1 Elab.: Prof. : Gilmar
 1 Elab.: Prof. : Gilmar 2 Elab.: Prof. : Gilmar Introdução Os ácidos nucléicos são responsáveis pelo controle de todas as atividades e pela manutenção da estrutura das células, além de estarem relacionados
1 Elab.: Prof. : Gilmar 2 Elab.: Prof. : Gilmar Introdução Os ácidos nucléicos são responsáveis pelo controle de todas as atividades e pela manutenção da estrutura das células, além de estarem relacionados
MÓDULO 3 BIOLOGIA MOLECULAR
 MÓDULO 3 BIOLOGIA MOLECULAR Aula 1 - Estrutura e Propriedades dos Ácidos Nucleicos Evidências de que o DNA constitui o material genético Experimento de Frederick Griffith (1928) Pneumococcus pneumoniae
MÓDULO 3 BIOLOGIA MOLECULAR Aula 1 - Estrutura e Propriedades dos Ácidos Nucleicos Evidências de que o DNA constitui o material genético Experimento de Frederick Griffith (1928) Pneumococcus pneumoniae
Nutrição. Prof. João Ronaldo Tavares de Vasconcellos Neto ABR/2011
 Introdução a Biologia i Molecular: DNA Nutrição Prof. João Ronaldo Tavares de Vasconcellos Neto ABR/2011 HISTÓRICO Organização Células DNA + Proteínas Corpo Informação das proteínas e RNAs que serão sintetizadas
Introdução a Biologia i Molecular: DNA Nutrição Prof. João Ronaldo Tavares de Vasconcellos Neto ABR/2011 HISTÓRICO Organização Células DNA + Proteínas Corpo Informação das proteínas e RNAs que serão sintetizadas
Descoberta da Estrutura do DNA
 DNA Estrutura Descoberta da Estrutura do DNA James Watson (geneticista americano) Francis Crick (físico inglês) Esclareceram a estrutura do DNA em 1953 O que se sabia sobre os genes Fatores hereditários
DNA Estrutura Descoberta da Estrutura do DNA James Watson (geneticista americano) Francis Crick (físico inglês) Esclareceram a estrutura do DNA em 1953 O que se sabia sobre os genes Fatores hereditários
DNA RNA Proteínas. Organização estrutural e funcional do núcleo 04/04/2017. Processamento (Splicing) Tradução (citoplasma) Replicação.
 Organização estrutural e funcional do núcleo DNA RNA Proteínas Replicação Transcrição Processamento (Splicing) Tradução (citoplasma) Cromatina - Eucromatina - Heterocromatina Cromossomo - Mitose 1 DNA
Organização estrutural e funcional do núcleo DNA RNA Proteínas Replicação Transcrição Processamento (Splicing) Tradução (citoplasma) Cromatina - Eucromatina - Heterocromatina Cromossomo - Mitose 1 DNA
ESCOLA SECUNDÁRIA DE CASQUILHOS BARREIRO DISCIPLINA DE BIOLOGIA E GEOLOGIA
 ESCOLA SECUNDÁRIA DE CASQUILHOS BARREIRO DISCIPLINA DE BIOLOGIA E GEOLOGIA 10ºano Nome: Nº: Turma: Ficha de Trabalho Tema: A célula, biomoléculas e transporte. Data: / / 1. A descoberta do facto de todos
ESCOLA SECUNDÁRIA DE CASQUILHOS BARREIRO DISCIPLINA DE BIOLOGIA E GEOLOGIA 10ºano Nome: Nº: Turma: Ficha de Trabalho Tema: A célula, biomoléculas e transporte. Data: / / 1. A descoberta do facto de todos
Estrutura do DNA 16/05/2018 ÁCIDOS NUCLÉICOS HISTÓRICO ÁCIDOS NUCLÉICOS. Adenina. Guanina. Citosina. Uracila. Timina GREGOR MENDEL
 ÁCIDOS NUCLÉICOS ÁCIDOS NUCLÉICOS: são substâncias formadoras de genes, constituídas por um grande número de NUCLEOTÍDEOS, que são unidades formadas por três tipos de substâncias químicas: - Uma base nitrogenada:
ÁCIDOS NUCLÉICOS ÁCIDOS NUCLÉICOS: são substâncias formadoras de genes, constituídas por um grande número de NUCLEOTÍDEOS, que são unidades formadas por três tipos de substâncias químicas: - Uma base nitrogenada:
UTILIZAÇÃO E ORGANIZAÇÃO DOS LABORATÓRIOS ESCOLARES
 OFICINA DE FORMAÇÃO UTILIZAÇÃO E ORGANIZAÇÃO DOS LABORATÓRIOS ESCOLARES BIOLOGIA E GEOLOGIA 11.º ANO Escola Secundária Domingos Sequeira LEIRIA, 2010 INTRODUÇÃO A célula é a unidade básica funcional e
OFICINA DE FORMAÇÃO UTILIZAÇÃO E ORGANIZAÇÃO DOS LABORATÓRIOS ESCOLARES BIOLOGIA E GEOLOGIA 11.º ANO Escola Secundária Domingos Sequeira LEIRIA, 2010 INTRODUÇÃO A célula é a unidade básica funcional e
Biologia Molecular. O que é Biologia Molecular? Aplicabilidades da Biologia Molecular 3/12/2017. IFSC Campus Lages. Prof.
 IFSC Campus Lages Biologia Molecular Prof. Silmar Primieri O que é Biologia Molecular? Aplicabilidades da Biologia Molecular Genética do Câncer Doenças com herança complexa Preservação de espécies ameaçadas
IFSC Campus Lages Biologia Molecular Prof. Silmar Primieri O que é Biologia Molecular? Aplicabilidades da Biologia Molecular Genética do Câncer Doenças com herança complexa Preservação de espécies ameaçadas
Ácidos Nucleicos e suas propriedades
 UNIVERSIDADE FEDERAL DO PARANÁ SETOR DE CIÊNCIAS AGRÁRIAS DEPARTAMENTO DE FITOTECNIA E FITOSSANITARISMO AF 060- Biotecnologia Vegetal Ácidos Nucleicos e suas propriedades Professores: Bruno Portela Brasileiro
UNIVERSIDADE FEDERAL DO PARANÁ SETOR DE CIÊNCIAS AGRÁRIAS DEPARTAMENTO DE FITOTECNIA E FITOSSANITARISMO AF 060- Biotecnologia Vegetal Ácidos Nucleicos e suas propriedades Professores: Bruno Portela Brasileiro
Resposta: C Comentário: Resposta: D Comentário: Item A: falso Item B: falso Item C: falso Item D: verdadeiro Item E: falso Resposta: C Comentário:
 1. Comentário: A molécula de DNA é formada por uma dupla hélice de polinucleotídeos unidos por pontes de hidrogênio. Ao se replicar, as pontes de hidrogênio são quebradas e as hélices se separam, havendo
1. Comentário: A molécula de DNA é formada por uma dupla hélice de polinucleotídeos unidos por pontes de hidrogênio. Ao se replicar, as pontes de hidrogênio são quebradas e as hélices se separam, havendo
BIOQUÍMICA E BIOLOGIA MOLECULAR 1º S_2011_2012_1º Teste. Curso: 1. Qual dos conjuntos de moléculas representam a atmosfera da era prebiótica?
 Nome do Aluno: Nº: Curso: BIOQUÍMICA E BIOLOGIA MOLECULAR 1º S_2011_2012_1º Teste 16/11/2011 (Duração: 1,5 h) Cada uma das questões de escolha múltipla (1 à 40) tem a cotação de 0,5 valores. Será descontado
Nome do Aluno: Nº: Curso: BIOQUÍMICA E BIOLOGIA MOLECULAR 1º S_2011_2012_1º Teste 16/11/2011 (Duração: 1,5 h) Cada uma das questões de escolha múltipla (1 à 40) tem a cotação de 0,5 valores. Será descontado
ESTRUTURA E FUNÇÃO DOS GENES E CROMOSSOMOS
 Faculdade Ciência da Vida Disciplina: Genética Básica Aula 2 ESTRUTURA E FUNÇÃO DOS GENES E CROMOSSOMOS PROFESSORA: Fernanda Guimarães E-MAIL: guimaraes.biologia@gmail.com NÚCLEO Abriga do material genético
Faculdade Ciência da Vida Disciplina: Genética Básica Aula 2 ESTRUTURA E FUNÇÃO DOS GENES E CROMOSSOMOS PROFESSORA: Fernanda Guimarães E-MAIL: guimaraes.biologia@gmail.com NÚCLEO Abriga do material genético
Duplicação do DNA e Síntese de PROTEÍNAS. Telmo Giani Fonte: Internet
 Duplicação do DNA e Síntese de PROTEÍNAS Telmo Giani Fonte: Internet OS ÁCIDOS NUCLEICOS DNA Ácido fosfórico Desoxirribose Bases Púricas: A e G Bases Pirimídicas: C e T Dupla fita RNA Ácido fosfórico Ribose
Duplicação do DNA e Síntese de PROTEÍNAS Telmo Giani Fonte: Internet OS ÁCIDOS NUCLEICOS DNA Ácido fosfórico Desoxirribose Bases Púricas: A e G Bases Pirimídicas: C e T Dupla fita RNA Ácido fosfórico Ribose
Prof. Dr. Bruno Lazzari de Lima. Replicação do DNA
 Prof. Dr. Bruno Lazzari de Lima Replicação do DNA Introdução Sistemas vivos tem a capacidade de fazer cópias de si mesmos. Capacidade associada ao material genético hereditário. Compreensão do processo
Prof. Dr. Bruno Lazzari de Lima Replicação do DNA Introdução Sistemas vivos tem a capacidade de fazer cópias de si mesmos. Capacidade associada ao material genético hereditário. Compreensão do processo
Aula 2 biologia molecular
 Aula 2 biologia molecular Processo de cópia de uma molécula de DNA em duplas moléculas filhas Este processo ocorre a cada fase S do ciclo celular Apresenta processos gerais e particularidades entre procariotos
Aula 2 biologia molecular Processo de cópia de uma molécula de DNA em duplas moléculas filhas Este processo ocorre a cada fase S do ciclo celular Apresenta processos gerais e particularidades entre procariotos
Ribossomos e síntese protéica
 Ribossomos e síntese protéica SÍNTESE PROTÉICA DNA Transcrição RNA Tradução T A U T A U C G C G C G C G C C G C T A U G C G G C G O código genético é DEGENERADO PROTEÍNA Phe Ala Trp MAQUINARIA GENÉTICA
Ribossomos e síntese protéica SÍNTESE PROTÉICA DNA Transcrição RNA Tradução T A U T A U C G C G C G C G C C G C T A U G C G G C G O código genético é DEGENERADO PROTEÍNA Phe Ala Trp MAQUINARIA GENÉTICA
BIOLOGIA. Moléculas, Células e Tecidos Transcrição e Tradução. Prof. Daniele Duó
 BIOLOGIA Moléculas, Células e Tecidos Prof. Daniele Duó O código genético É a relação entre a sequência de bases no DNA e a sequência correspondente de aminoácidos, na proteína; Guarda toda informação
BIOLOGIA Moléculas, Células e Tecidos Prof. Daniele Duó O código genético É a relação entre a sequência de bases no DNA e a sequência correspondente de aminoácidos, na proteína; Guarda toda informação
Transcrição e Processamento de RNA
 Transcrição e Processamento de RNA Profa. Dra. Aline Maria da Silva Instituto de Química- USP Bibliografia: Genes VII - Benjamin Lewin Biologia Molecular Básica-Arnaldo Zaha Lenhinger Principles of Biochemistry
Transcrição e Processamento de RNA Profa. Dra. Aline Maria da Silva Instituto de Química- USP Bibliografia: Genes VII - Benjamin Lewin Biologia Molecular Básica-Arnaldo Zaha Lenhinger Principles of Biochemistry
Replicação de DNA QBQ 204 Aula 2 (biomol)
 Replicação de DNA QBQ 204 Aula 2 (biomol) Prof. João Carlos Setubal Site da disciplina http://www.iq.usp.br/setubal/qbq204/2016 Trabalho para entrega Grupos de 6 alunos Temas: processos bioquímicos relacionados
Replicação de DNA QBQ 204 Aula 2 (biomol) Prof. João Carlos Setubal Site da disciplina http://www.iq.usp.br/setubal/qbq204/2016 Trabalho para entrega Grupos de 6 alunos Temas: processos bioquímicos relacionados
Transcrição em Eucariotos. Prof. Doutor Júlio César Borges Disciplina: Bioquímica II Lenita P. Altoé Paula B. Perroni Rhaissa M.
 Transcrição em Eucariotos Prof. Doutor Júlio César Borges Disciplina: Bioquímica II Lenita P. Altoé Paula B. Perroni Rhaissa M. Bontempi _sumário _sumário Transcrição Dogma central Considerações iniciais
Transcrição em Eucariotos Prof. Doutor Júlio César Borges Disciplina: Bioquímica II Lenita P. Altoé Paula B. Perroni Rhaissa M. Bontempi _sumário _sumário Transcrição Dogma central Considerações iniciais
LISTA DE RECUPERAÇÃO I ÁCIDOS NUCLEICOS
 LISTA DE RECUPERAÇÃO I ÁCIDOS NUCLEICOS 1) (OSEC-SP) - Quanto à sua estrutura química, o DNA e o RNA são A) polipeptídeos. B) nucleoproteínas. C) polissacarídeos. D) fosfatídeos. E) polinucleotídeos. 2)
LISTA DE RECUPERAÇÃO I ÁCIDOS NUCLEICOS 1) (OSEC-SP) - Quanto à sua estrutura química, o DNA e o RNA são A) polipeptídeos. B) nucleoproteínas. C) polissacarídeos. D) fosfatídeos. E) polinucleotídeos. 2)
Resoluções das atividades
 Resoluções das atividades Aula 8 Ácidos nucleicos Atividades para sala 01 D 02 B No DNA, ocorrem duas fitas de polinucleotídios. As duas fitas são unidas por pontes de hidrogênio estabelecidas entre os
Resoluções das atividades Aula 8 Ácidos nucleicos Atividades para sala 01 D 02 B No DNA, ocorrem duas fitas de polinucleotídios. As duas fitas são unidas por pontes de hidrogênio estabelecidas entre os
Ácidos Nucleicos e suas propriedades
 UNIVERSIDADE FEDERAL DO PARANÁ SETOR DE CIÊNCIAS AGRÁRIAS DEPARTAMENTO DE FITOTECNIA E FITOSSANITARISMO AF 060- Biotecnologia Vegetal Ácidos Nucleicos e suas propriedades Prof a. Renata FaierCalegario
UNIVERSIDADE FEDERAL DO PARANÁ SETOR DE CIÊNCIAS AGRÁRIAS DEPARTAMENTO DE FITOTECNIA E FITOSSANITARISMO AF 060- Biotecnologia Vegetal Ácidos Nucleicos e suas propriedades Prof a. Renata FaierCalegario
Biologia e Geologia 10º ano. Natércia Charruadas 2011
 Biologia e Geologia 10º ano Natércia Charruadas 2011 Todos os seres vivos, logo todas as células, são constituídos por moléculas orgânicas de grandes dimensões macromoléculas. Estas são formadas por um
Biologia e Geologia 10º ano Natércia Charruadas 2011 Todos os seres vivos, logo todas as células, são constituídos por moléculas orgânicas de grandes dimensões macromoléculas. Estas são formadas por um
QBQ 0102 Educação Física. Carlos Hotta. Transcrição 12/06/15
 QBQ 0102 Educação Física Carlos Hotta Transcrição 12/06/15 Previously... replicação DNA transcrição RNA tradução proteína As fitas de DNA são antiparalelas A síntese de DNA é feita na direção 5 -> 3 A
QBQ 0102 Educação Física Carlos Hotta Transcrição 12/06/15 Previously... replicação DNA transcrição RNA tradução proteína As fitas de DNA são antiparalelas A síntese de DNA é feita na direção 5 -> 3 A
Características gerais
 Citoplasma Citoplasma Características gerais Um vertebrado é formado por dezenas de tipos de células. Citoplasma Características gerais Um vertebrado é formado por dezenas de tipos de células. Neurônio
Citoplasma Citoplasma Características gerais Um vertebrado é formado por dezenas de tipos de células. Citoplasma Características gerais Um vertebrado é formado por dezenas de tipos de células. Neurônio
Profº André Montillo
 Profº André Montillo www.montillo.com.br Definição: É um polímero, ou seja, uma longa cadeia de nucleotídeos. Estrutura Molecular dos Nucleotídeos: Os nucleotídeos são constituídos por 3 unidades: Bases
Profº André Montillo www.montillo.com.br Definição: É um polímero, ou seja, uma longa cadeia de nucleotídeos. Estrutura Molecular dos Nucleotídeos: Os nucleotídeos são constituídos por 3 unidades: Bases
